文章信息
- 边富宁, 李文革, 李先平, 卢金星. 2014.
- Bian Funing, Li Wenge, Li Xianping, Lu Jinxing. 2014.
- 利用GoPubMed对CRISPR相关文献的计量学分析
- Application of GoPubMed in bibliometric analysis of literature on CRISPR
- 中华流行病学杂志, 2014, 35(12): 1400-1403
- Chinese Journal of Epidemiology, 2014, 35(12): 1400-1403
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2014.12.020
-
文章历史
- 投稿日期:2014-06-20
GoPubMed是以PubMed为数据源,以语义检索技术为支撑,对检索结果进行分类分析的数据挖掘工具,可有效精简检索结果,高效检索目标文献,对于文献统计及研究热点的追踪提供了很大帮助[1]。CRISPR(clustered regularly interspaced short palindromic repeats)规律间隔成簇短回文重复序列,是细菌和古细菌在对抗噬菌体的生存斗争中进化出的获得性免疫系统,能对噬菌体或者外源性DNA进行切割和降解[2]。CRISPR结构最初(1987年)由日本科学家Ishino于大肠埃希菌中发现,当时并未引起关注,2002年由Jansen等将其命名为CRISPR[3, 4]。2007年Barrangou等[2]通过实验证实CRISPR系统能使宿主抵抗噬菌体、质粒等外来的DNA入侵的免疫能力。2013年CRISPR/Cas9作为基因编辑工具首次被Science报道后,许多科学家投入到这个充满魅力的技术上[5]。2013年在世界三大权威期刊(Science、Nature和Cell)共刊出CRISPR相关文章14篇。2013年12月Science公布了其评出的“2013年度十大科学突破”,CRISPR技术位列其中[6]。为了解CRISPR研究的文献增长规律和发展趋势,为从事CRISPR相关研究人员提供参考,笔者采用GoPubMed对CRISPR相关文献进行分析。
1. 数据来源:登录GoPubMed网站(www.GoPubMed.com),输入检索词“Clustered Regularly Interspaced Short Palindromic Repeats”[mesh]进行检索,检索时间为2014年5月27日。通过GoPubMed网站统计功能,分析被PubMed收录的所有CRISPR相关文献。
2. 结果与分析:经检索,PubMed收录CRISPR相关文献共758篇,检索结果均纳入统计分析。
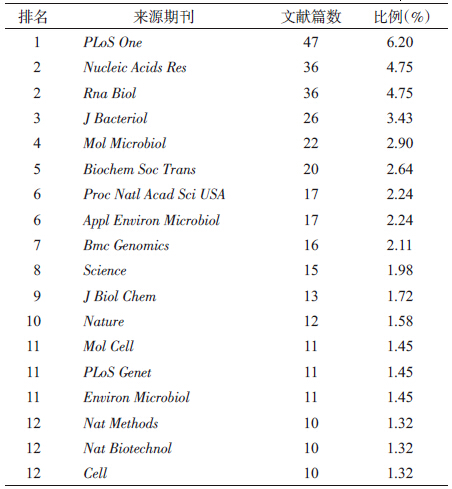
(1)文献年度分布:自2002年第一篇CRISPR相关论文发表后,2002-2012年文献数量经过缓慢增长,于2012年始呈快速增长,2012年全年PubMed收录CRISPR相关文献123篇,2013年收录文献较2012年成倍增长,共收录文章241篇,2014年至笔者检索时已收录文献193篇(图 1A)。据GoPubMed统计,CRISPR相对研究兴趣指数(relative research interest,RRI)[7]逐年提高,从2012年起增长幅度较大,2012年RRI约为0.000 240,2013年预期可达0.000 380,反映出CRISPR在所有研究领域中的关注度逐年快速增长。RRI(按当年PubMed中该研究领域加权论文数量在所有研究领域加权论文集合中所占比例计算出RRI)可较好反映研究活跃程度,比文献的绝对数量更重要(图 1B)。
 |
| 图 1 CRISPR相关文献发表的年度分布 |
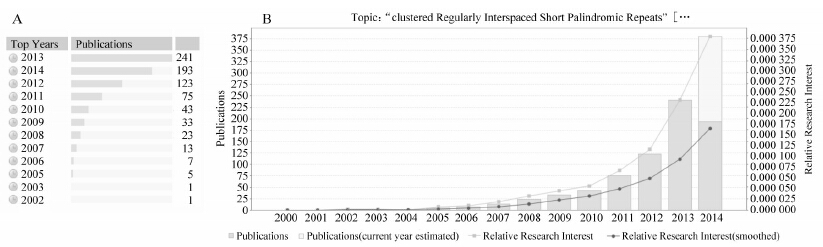
(2)国家和城市文献分布:CRISPR相关研究主要分布于北美洲、欧洲和亚洲(图 2A)。按国家排名,美国发表CRISPR相关文章288篇(27.99%),位居首位(图 2B)。中国和德国分别发表 59和57篇,排名第二、三位。如按城市分类统计文献数量,北京(中国)发表 30篇排第一位,伯克利(美国)和哥本哈根(丹麦)分列第二位和第三位(图 2C)。
 |
| 图 2 CRISPR文献数量的地域分布 |
(3)来源期刊分布:刊载CRISPR研究论文的主要来源期刊见表 1。其中 PLoS One为刊登CRISPR研究论文最多的杂志。Science、Nature等国际权威杂志也刊登CRISPR相关研究文献,其中Science刊登15篇,Nature刊登12篇,Cell刊登10篇。
(4)研究主题:目前CRISPR研究的主题集中在CRISPR结构本身的研究以及相关基因组学研究(表 2)。表明这些研究主题可代表该领域的研究趋势和热点。CRISPR作为细菌体的免疫系统,大量发表文献可出现“immunity/immunization”和“CRISPR-Cas systems”等主题词。随着CRISPR/Cas9作为基因编辑工具首次被Nature报道后,预期将会出现基因编辑、基因敲除等新的主题词[5]。
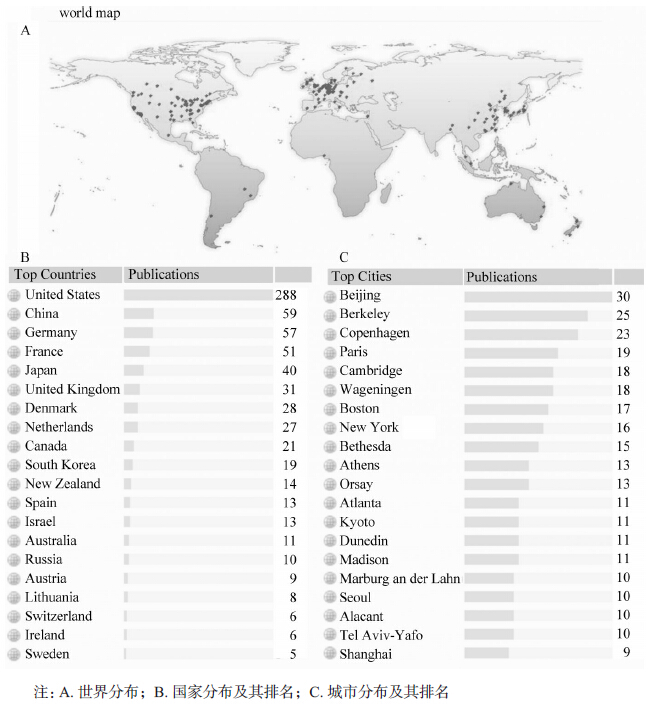
(5)高产作者及相互关系:分析表明Horvath发表CRISPR相关文章11篇,排名第一;Vergnaud发表 8篇排名第二(图 3A)。CRISPR文献的作者主要形成两大合作网络,最大的合作网络是以Horvath为核心,据图 3B中线条的复杂性和粗细,可知研究者间合作的密切程度。
 |
| 图 3 CRISPR相关文献中高产作者及其研究关系分布 |
(6)中美两国发表文献期刊比较:美国和中国发表CRISPR相关文章数量为前2名,发表论文的质量可由刊登杂志的影响因子反映出。美国在PLoS One、Proc Natl Acad Sci USA和J Bacteriol发表相关文献数量均为12篇,三者并列第一;在Science发表 10篇, 3. 评价:GoPubMed 是优秀的智能检索工具。不仅可以针对某研究课题进行分析,总结出该课题领域的最新相关情况,还可以对一个研究机构发表的文献进行分析,从而揭示其研究方向、核心作者、研究趋势等[1]。
CRISPR修饰系统为最新的基因编辑工具,较之传统的基因修饰技术,如锌指核酸酶(zinc-finger nucleases)、转录激活因子样效应物核酸酶(transcription acti vator-like effector nucleases),具有操作简单、成本较低和多基因同时操作等优势,由于其适用范围广泛,目前已应用于动物模型、植物和基础医学等多个领域[8, 9, 10, 11, 12]。
分析CRISPR相关研究文献发表的时间提示呈逐年上升趋势,且增幅较大,自2012年起开始大幅度增长。反映了当前对CRISPR的研究处于起步阶段,其论文产出不断增加,同时CRISPR正在由免疫机制研究到基因编辑技术的应用[9, 11],作为当前最有效的基因编辑技术其文献产出必然会有更大幅度的增长。分析论文分布区域,主要集中在美国、欧洲和中国,反映出我国对CRISPR研究的关注程度也在提高。北京以30篇论文名列城市排名榜首,其中不乏高影响力的国际期刊,说明北京地区在生命科学技术的研究中具有一定影响力。从文献分布可见,Science、Nature和Cell国际三大顶尖科技期刊共发表CRISPR相关研究37篇,反映出对CRISPR较高的关注度。
我国发表论文总数量排名第二,但分析中美两国发表论文的影响力,在Science、Nature和Cell上共刊登CRISPR相关研究37篇,其中美国发表 24篇,中国仅有1篇。说明在CRISPR相关研究中我国不仅在论文数量上与美国有差距,且在论文影响力方面的差距更为明显。提示我国在研究上应注重创新性,提高论文水平。

图 4 中美两国发表CRISPR相关文献期刊
| [1] Doms A,Schroeder M. GoPubMed:exploring PubMed with the Gene Ontology[J]. Nucleic Acids Res,2005,33 (Web Server Issue):W783-786. |
| [2] Barrangou R,Fremaux C,Deveau H,et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science,2007,315(5819):1709-1712. |
| [3] Ishino Y,Shinagawa H,Makino K,et al. Nucleotide sequence of the iap gene,responsible for alkaline phosphatase isozyme conversion in Escherichia coli,and identification of the gene product[J]. J Bacteriol,1987,169(12):5429-5433. |
| [4] Jansen R,Embden JD,Gaastra W,et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol,2002,43(6):1565-1575. |
| [5] Cong L,Ran FA,Cox D,et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science,2013,339(6121):819-823. |
| [6] Coontz R. Science's Top 10 Breakthroughs of 2013 [EB/OL].[2013-12-19]. http://news.sciencemag.org/2013/12/sciences- top-10-breakthroughs-2013. |
| [7] Bailey J. An assessment of the use of chimpanzees in hepatitis C research past,present and future:1. Validity of the chimpanzee model[J]. Altern Lab Anim,2010,38(5):387-418. |
| [8] Hsu PD,Lander ES,Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell,2014,157(6):1262-1278. |
| [9] Feng Z,Mao Y,Xu N,et al. Multigeneration analysis reveals the inheritance,specificity,and patterns of CRISPR/Cas-induced gene modifications in arabidopsis[J]. Proc Natl Acad Sci USA,2014,111(12):4632-4637. |
| [10] Wu Y,Liang D,Wang Y,et al. Correction of a genetic disease in mouse via use of CRISPR-Cas9[J]. Cell Stem Cell,2013,13(6):659-662. |
| [11] Bondy-Denomy J,Pawluk A,Maxwell KL,et al. Bacteriophage genes that inactivate the CRISPR/Cas bacterial immune system[J]. Nature,2013,493(7432):429-432. |
| [12] Xu R,Li H,Qin R,et al. Gene targeting using the Agrobacterium tumefaciens-mediated CRISPR-Cas system in rice[J/OL]. Rice(NY),2014,7(1):5. http://www.thericejournal.com/content/7/1/5. DOI:10.1186/s12284-014-0005-6. |
 2014, Vol. 35
2014, Vol. 35