文章信息
- 贺健梅, 邹潇白, 陈曦, 郑军. 2014.
- He Jianmei, Zou Xiaobai, Chen Xi, Zheng Jun. 2014.
- 超深度焦磷酸测序技术用于HIV-1RT基因区原发性耐药突变的研究
- The use of ultra deep sequencing technique in the screening program on HIV-1 drug resistance mutation among ART-naïve patients in Hunan province
- 中华流行病学杂志, 2014, 35(10): 1142-1145
- Chinese Journal of Epidemiology, 2014, 35(10): 1142-1145
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2014.10.015
-
文章历史
- 投稿日期:2014-4-30
虽然高效抗反转录病毒治疗(HAART)在HIV 抗病毒临床治疗上取得很好的效果,然而HIV-1基 因耐药突变也在不断增加。有研究表明即使在很高 的病毒载量水平下(≥104 copies/ml),传统HIV基因 型耐药检测技术(Sanger测序)无法检测到较低水平 的耐药突变(<20%)[1, 2]。新开发的“超深度焦磷酸 测序(UDS)”能够一次性获得更高通量的HIV基因 组测序结果,可以帮助了解病毒株中那些罕见、低频 的耐药突变位点。目前我国将UDS用于HIV耐药 检测的研究和文献少见。为掌握和灵活运用此技 术,2013年同时采用UDS和Sanger测序对90份未 接受抗病毒治疗的HIV感染者进行HIV基因型耐药 研究,并分析湖南省未治疗的HIV感染者中原发性 耐药流行趋势。 材料与方法
1. 样品来源:本实验室保存的2011年7月至 2012年6月来自衡阳市和长沙市未接受抗病毒治疗 的HIV感染者血浆90份,其中长沙20份,衡阳70 份。所有样本病毒载量均≥104 copies/ml。
2. 实验室检测:①HIV载量检测:用“COBAS TaqMan HIV-1test”全自动病毒载量检测系统(美 国罗氏公司),检测病毒载量。②UDS:cDNA合成 用罗氏公司提供的HIV试剂盒(内部研发,暂未上 市),每个样本对应HIV pol区[包括蛋白酶区和反转 录(RT)区]的4条特异引物,共1.3kb,每条特异性 引物上连接着多重标记物(MID)。扩增子使用 Agencourt AMPure XP magnetic beads试剂(美国贝 克 曼 库 尔 特 公 司)进 行 纯 化,使 用Quant-iT PicoGreen dsDNA Assay Kit(美国Invitrogen公司) 进行荧光定量分析确定核酸含量。若核酸含量低于 5ng/μl,使用Agilent2100bioanalyzer (Agilent Life Science,Santa Clara, California,US)对扩增产物长度和 质量进行分析,确定是否弃掉该扩 增产物。所有样本的4个扩增产物 等mol集合合成为1管,然后进行油 包水PCR。油包水PCR产后,将 500000个被富集的DNA beads被均 匀的铺到1个PTP芯片上,放到GS Junior上进行焦磷酸测序(正向与反 向同时测序)。数据分析采用罗氏 公 司 的Variant Analyzer(AVA)软 件。所有与HIV抗病毒治疗耐药性 相关的氨基酸位点的判断和分析均来自斯坦福大学 HIV耐药突变数据库(http://hivdb.Stanford.edu)[3]。 ③Sanger测序:用QIAamp Viral RNA Mini Kit试 剂盒(德国Qiagen公司)提取RNA为模板,采用巢 式PCR扩增HIV-1pol基因区,扩增产物长度约 为1160bp,包括蛋白酶区以及RT区,第1轮PCR使 用宝生物工程(大连)有限公司TaKaRa One Step RNA PCR Kit(AMV)试剂盒,第2轮PCR以第1轮 扩增产物为模板,使用天根生化科技(北京)有限公 司的Perfectshot TM Ex Taq Mix试剂盒,反应条件参 考相关文件[1, 2, 3, 4]。两轮PCR扩增引物见表 1。将扩 增产物送交北京诺赛基因研究中心有限公司进行 序列测定,测序引物见表 1。测得的序列应用 Contig Express和BioEdit软件拼接整理和校对,并 提交斯坦福大学的HIV序列网络数据库进行数据 分析和讨论。
3. 统计学分析:统计学分析采用SPSS13.0软 件。P<0.05为差异有统计学意义。 结 果
1. 样品亚型:检测发现75个样本为CRF01_AE 亚型(83.3%,75/90),9个为B亚型(10.0%,9/90),5个 为C亚型(5.6%,5/90),1个是G亚型(1.0%,1/90)。
2. UDS:90例样本UDS全部成功测序。72.2% (65/90)的 样 本 产 生 了 传 播 性 耐 药 突 变 位 点 (TDRs),其中突变频率>1%的RT基因区耐药突变 位点样本有38例(突变频率范围:1.02%~75.36%)。 见表 2。对核苷类反转录酶(NRTI)耐药突变者14 例(15.6%,14/90),对非核苷类反转录酶(NNRTI)耐 药突变者15例(16.7%,15/90),同时对NRTI和 NNRTI类耐药突变者9例。共发现在16个位置上发生54频次的耐药突变。其中,针对NRTI类药物 的突变位点:K65R为8例(8.89%,8/90),D67N/G为 7例(7.78%,7/90),T69N为4例(4.44%,4/90), M184I为2例(2.22%,2/90),T215I/S为2例(2.22%, 2/90),M41L、L74V、F77L以及K219Q等突变位点 均只发现了1例;针对NNRTI类药物的突变位 点:V179D/E/I15例(16.67%,15/90),Y188C/H4例 (4.44%,4/90),P225H3例(3.33%,3/90),G190E/A 2例(2.22%,2/90),而K101E和V106A均只发现了 1例(1.11%,1/90)。17例样本(18.89%,17/90)有针 对RT基因区的中度及以上的耐药突变。
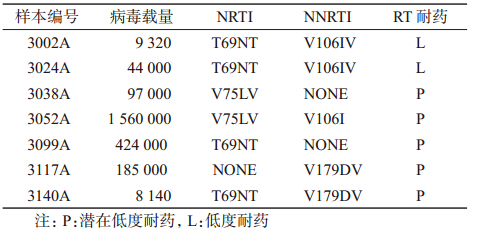
3. Sanger测序:采用Sanger测序90份样本成功 测序89例(98.9%,89/90)。RT基因区产生耐药突变 的共有7例,其中针对NRTI耐药突变的有6例, NNRTI耐药突变位点的有5例,见表 3。
4. UDS与Sanger测序的比较: UDS检测出了38例样本在16个氨基 酸位置产生了针对RT基因区的54个 频次的耐药突变。Sanger法检出7例 在4个氨基酸位置产生了11个频次的 耐药突变。Sanger法检出突变频次为 UDS检出频次的20.37%(11/54)。
本研究显示,湖南省衡阳市和长 沙 市 人 群 感 染 的HIV分 为CRF_ 01AE、B、C和G亚型,CRF_01AE在 不同感染途径的比率明显高于其他亚 型(83.33%,75/90),为当前优势毒株, 与湖南省近4年监测结果吻合[4, 5, 6, 7]。与 江西、广东、福建、广西等地类似,而不 同于我国北方省份,可能与湖南地处 华南,人群主要往来南方省份和东南 亚有关[6]。过去分离到的A、D亚型没 有再发现[8],可能这两种亚型传播人 数较少。
本研究还显示,主要原发性耐药 位点分别是:M41L,D67G/N,M184I, K65N/R,T215I/S,P225H,Y188C/H, K101E,V106A,T215I/S和G190A/E, 这些RT区耐药位点在2009年已经发 现[7],为湖南省常见耐药突变位点。 相关文献报道[9, 10, 11],M41L突变常与 215位 置 一 起 产 生,从 而 导 致 对 Zidovudine(AZT)和stavudine(d4T)产生中度以上 耐药,对tenofovir (TDF)产生低度耐药;K65N/R和 T215S/I常引起d4T低度耐药以及lamivudine (3TC) 和TDF中度耐药;M184V引起对3TC高度耐药,但 是同时也增加了针对AZT、TDF、d4T的药物敏感性。D67G/N常与70或215一起发生,导致NRTI中 3TC以外的其他药物不同程度耐药;K101E与 Y188C/H会引起nevirapine(NVP)的中度或高度耐 药以及efavirenz(EFV)低度耐药;V106A和G190A/E 导致NVP和EFV高度耐药,P225H与K103N一起 出现时会增加对EFV的耐药。湖南省初始一线抗 病毒治疗方案是由d4T,3TC,NVP,EFV,TDF,AZT 以及lopinavir/ritonovir (LPV/r)组成的三联疗法。暂 未发现引起LPV/r耐药的突变位点,这与LPV/r目前 是二线治疗方案还未普及使用有关。而在1例样本 发现了L74V,它可引发ddI和ABC耐药,ddI和ABC 并未在湖南省使用,可能是输入引起的。
本研究也显示,在UDS方法检出的54个耐药位 点中,8个为高频(≥20%)耐药突变,有6个被传统 测序同时检出,提示对高频耐药突变(≥20%)这两 种测序方法区别不大。但对于低频耐药突变(< 20%),UDS检出46个突变位点,而传统方法仅检出 5个,UDS敏感性远高于Sanger测序。UDS检测到 所有的耐药突变位点中超过85%以上为低频耐药突 变(<20%),其中绝大多数都没有被传统基因型耐 药检测方法检测到(P<0.01)。依靠传统测序方法 检测,近4年湖南省未治疗HIV感染者中原发性耐 药每年均低水平传播(<5%)。然而本研究数据显 示,采用UDS发现有18.9%(17/90)的样本产生了针 对RT中度或高度的耐药,提示通过Sanger法测序能 够鉴定出显著的耐药性突变(≥20%),但对于尚未 达到显著突变量的突变位点或稀有型抗药型突变 (<20%)不能及时发现。UDS则能更灵敏检测到低 频突变毒株[2, 12, 13, 14, 15, 16]。
当前高病毒载量伴低水平的耐药突变(1%左 右)难以被传统的检测手段所发现,有可能在药物选 择的压力下增加抗病毒治疗的失败[17, 18, 19, 20, 21]。因此选择 更高灵敏度和精确度的技术检测低水平耐药突变位 点,对于预警和研究原发性耐药毒株的流行和传播 及HIV抗病毒临床治疗有着十分重要的意义。
| [1] Roquebert B,Malet I,Wirden M,et al. Role of HIV-1 minority populations on resistance mutational pattern evolution and susceptibility to protease inhibitors[J]. AIDS,2006,20(2): 287-289. |
| [2] Wang C,MitsuyaY,Gharizadeh B,et al. Characterization of mutation spectra with ultra-deep pyrosequencing:Application to HIV-1 drug resistance[J]. Genome Res,2007,17(8):1195-1201. |
| [3] Hirsch MS,Gunthard HF,Schapiro JM,et al. Antiretroviral drug resistance testing in adult HIV-1 infection: 2008 recommendations of an international AIDS society-USA panel[J]. Clin Infect Dis,2008,47(2):266-285. |
| [4] Chen X,Xing H,He JM,et al. Study on the threshold of HIV-1 drug resistance in Hunan province[J]. Chin J Epidemiol,2008,29 (8):787-789.(in Chinese) 陈曦,邢辉,贺健梅,等. 湖南省HIV-1 耐药警戒线调查[J]. 中华流行病学杂志,2008,29(8):787-789. |
| [5] Zou XB,He JM,Zhang GQ,et al. Drug resistance analysis on AIDS patients after highly active antiretroviraltherapy in Hunan province[J]. Chin J Infect Control,2010,9(5):305-309.(in Chinese) 邹潇白,贺健梅,张国强,等. 湖南省艾滋病患者抗病毒治疗后耐药性分析[J]. 中国感染控制杂志,2010,9(5):305-309. |
| [6] Chen X,Xing H,He JM,et al. A molecular epidemiological study on HIV-1 infection in Hunan province[J]. Practical Prev Med, 2005,12(3):483-485.(in Chinese) 陈曦,邢辉,贺健梅,等. 湖南省HIV-1 分子流行病学研究[J]. 实用预防医学,2005,12(3):483-485. |
| [7] He JM,Xing H,Chen X,et al. Surveillance of transmitted HIV-1 drug resistance in Hunan province from 2009 to 2012[J]. Chin J Prev Med,2013,47(11):1065-1067.(in Chinese) 贺健梅,邢辉,陈曦,等. 2009-2012年湖南省HIV-1耐药状况分析[J]. 中华预防医学杂志,2013,47(11):1065-1067. |
| [8] Xing H,Ruan YH,Li JY,et al. HIV drug resistance and its impact on antiretroviral therapy in Chinese HIV-infected patients[J]. PLoS One,2013,8:e54917. |
| [9] Yerly S,Kaiser L,Race E,et al. Transmission of antiretroviral drug-resistant HIV-1 variants[J]. Lancet,1999,354:729-733. |
| [10] Kantor R,Katzenstein DA,Efron B,et al. Impact of HIV-1 subtype and antiretroviral therapy on protease and reverse transcriptase genotype:results of a global collaboration[J]. PLoS Med,2005, 2:e112. |
| [11] Grossman Z,Istomin V,Averbuch D,et al. Genetic variation at NNRTI resistance associated positions in patients infected with HIV-1 subtype C[J]. AIDS,2004,18:909-915. |
| [12] Avidor B,Girshengorn S,Matus N,et al. Evaluation of a Bench- Top HIV Ultra-Deep Pyrosequencing Drug-Resistance Assay in the Clinical Laboratory[J]. J Clin Microbiol,2013,51(3):880- 886. |
| [13] Kozal MJ. Drug resistant HIV[J]. Clin Microbiol Infect,2009, 15 Suppl:S169-173. |
| [14] Margulies M,Egholm M,Altman WE,et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005,437(7057):376-380. |
| [15] Hoffmann C,Minkah N,Leipzig J,et al. DNA bar coding and pyrosequencing to identify rare HIV drug resistance mutations[J]. Nucleic Acids Res,2007,35(13):e91. |
| [16] Shafer RW. Low-abundance drug-resistant HIV-1 variants:finding significance in an era of abundant diagnostic and therapeutic options[J]. J Infect Dis,2009,199:610-612. |
| [17] Johnson JA,Li JF,Wei X,et al. Minority HIV-1 drug resistance mutations are present in antiretroviral treatment-naïve populations and associate with reduced treatment efficacy[J]. PLoS Med,2008,5:1112-1122. |
| [18] Johnson JA,Li JF,Wei X,et al. Baseline detection of lowfrequency drug resistance associated mutations is strongly associated with virological failure in previously antiretroviral naïve HIV-1 infected persons[J]. Antivir Ther,2006,11:S79. |
| [19] Svarovskaia ES,Margot NA,Bae AS,et al. Low-level K65R mutation in HIV-1 reverse transcriptase of treatment-experienced patients exposed to abacavir or didanosine[J]. J Acquir Immune Defic Syndr,2007,46:174-180,700. |
| [20] Palmer S,Boltz V,Maldarelli F,et al. Selection and persistence of nonnucleoside reverse transcriptase inhibitor-resistant HIV-1 in patients starting and stopping non-nucleoside therapy[J]. AIDS,2006,20:701-710. |
| [21] Halvas EK,Wiegand A,Boltz VF,et al. Low frequency nonnucleoside reverse-transcriptase inhibitor-resistant variants contribute to failure of efavirenz-containing regimens in treatmentexperienced patients[J]. J Infect Dis,2010,201(5):672-680. |
 2014, Vol. 35
2014, Vol. 35