文章信息
- 安翠红, 孙养信, 陈宝宝, 范锁平, 霍丽霞, 佘建军, 吕文. 2014.
- An Cuihong, Sun Yangxin, Chen Baobao, Fan Suoping, Huo Lixia, She Jianjun, Lyu Wen. 2014.
- DNA条形码技术在陕西省鼠疫疫区宿主动物鉴定中的应用
- Identification on host animals for plague by DNA barcoding technology in Shaanxi province
- 中华流行病学杂志, 2014, 35(9): 1042-1045
- Chinese Journal of Epidemiology, 2014, 35(9): 1042-1045
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2014.09.016
-
文章历史
- 投稿日期:2014-2-21
陕西省鼠疫疫区位于该省西北部的定边县境 内,处于鄂尔多斯高原荒漠草原长爪沙鼠(Meriones unguiculatus)鼠疫自然疫源地的南部边缘地带,历次 监测共发现啮齿动物6科7亚科18属23种,以长爪 沙鼠为优势种,其他小型啮齿动物主要有子午沙鼠 (Meriones meridianus) 、黑 线 仓 鼠(Circetidae barabensis)、小毛足鼠(Phodopus roborovskii)、三趾跳 鼠(Dipus sagitta)、五趾跳鼠(Allactaga sibirica)、小家 鼠(Mus musculus)等。家栖鼠类有褐家鼠(Rattus norvegicus)、小家鼠等。疫源地内空间结构复杂多 样,但性质稳固。目前在疫源地鼠疫宿主动物分类 中仍存在分歧,如2010年监测中发现疑似东方田鼠 (Microtus fortis),后经分子生物学鉴定,成为陕西省 鼠疫疫源地首次发现[1]。
DNA条形码技术(DNA barcoding)[2,3]是一种通 过标准DNA片段快速、准确识别或鉴定物种的技 术,目前在动物分类中最常用的基因序列是线粒体 细胞色素C氧化酶Ⅰ亚基(COⅠ)序列。该技术的 优点在于物种鉴定无需依靠形态特征,对幼体、残体 甚至生物痕迹同样能准确鉴定,发现形态上保守的 “隐存种”(cryptic species)[3]。此外该技术还能准确 评估区域内物种遗传多样性[4],并进行有效监测[5], 通过宿主动物的识别准确评估疫情性质[6]。基于此 本研究运用DNA条形码技术对陕西省定边县鼠疫 疫区3目6科6亚科12属14种139只宿主动物及62 份残体标本进行鉴定,建立陕西省鼠疫疫源地宿主 动物DNA条形码数据库。 材料与方法
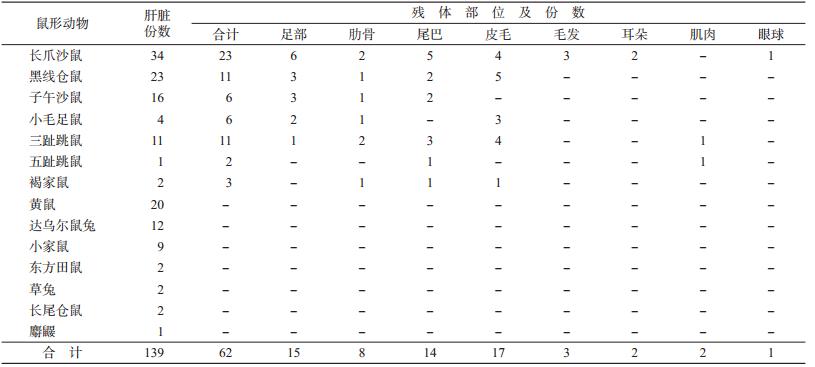
1. 材料:在定边县不同地理区域现场布点,捕鼠 疫宿主动物获取肝脏及残体组织(表 1)。
2.方法:
(1)基因组DNA提取、PCR扩增及序列测定:使 用德国Qiagen公司生产QIAamp DNA Mini Kit试剂 盒,提取肝脏及残体组织中的总DNA,应用Infinite 200酶标仪NanoQuant测定其DNA含量,通过PCR 扩增COⅠ基因,通用引物序列见表 2 [7]。反应体系: 2×Taq Mastermix12.5μl,上下游引物各1μl,模板 2 μl,补水至25 μl。PCR反应条件:94 ℃预变性 5min,94℃变性30s,55℃退火30 s,72℃延伸 1min,共35个循环。PCR产物用1.5%的琼脂糖凝 胶电泳分析,若电泳图中出现目标带即送样进行双 向测序。扩增时先选用COⅠ基因通用引物,如扩增 反应失败时,选用鸡尾酒引物[8],其中LepF1_t1、 VF1_t1、VF1d_t、VF1i_t1 的 比 例 及LepRI_t1、 VR1d_t1、VR1_t1、VR1i_t1的比例均为1∶ 1∶ 1∶ 3, 引物序列见表 2。
(2)序列分析:用Chromos软件观察评价测序峰 图质量,如果峰图质量差,不能准确判断碱基,则重 新进行扩增和测序。将测定序列在NCBI上运行 BLAST程序进行序列同源性比较。运用Mega5.0 软件比对各基因序列的碱基组成和变异,删除序列 两 端 不 能 完 全 对 齐 的 碱 基。基 于Kimura-2-parameter(K2P)[9]模型计算各物种的遗传距离;采用 邻接法构建全部COⅠ基因序列的NJ系统树,对NJ 树进行内部分支检验与1000次Bootstrap检验分 析,确定各支系的置信度[10]。 结 果
1. COⅠ基因序列分析:对陕西省定边县鼠疫疫 区宿主动物3目6科6亚科12属14种139只鼠样本 COⅠ基因进行序列分析,序列比对结果见表 3。发 现同属各种间COⅠ序列的差异度为8.9%~15.1%, 种内COⅠ序列差异度≤2%。139份样本COⅠ基因 序列的遗传距离见图 1。
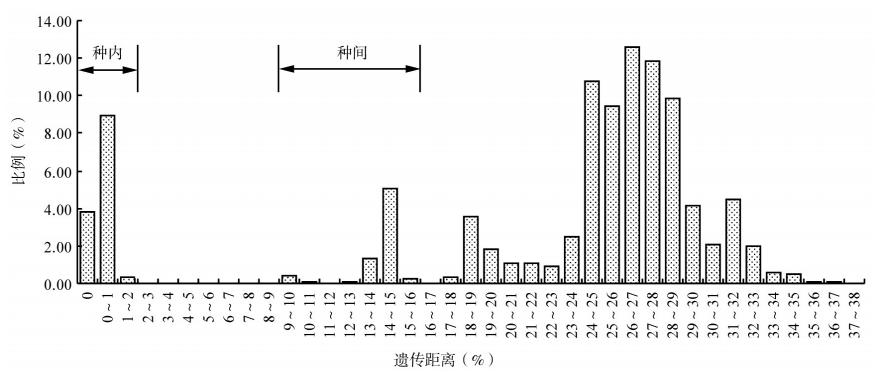 |
| 图 1 陕西省定边县鼠疫疫源地139份鼠形动物样本的遗传距离 |
2. 宿主动物NJ系统树构建:陕西省定边县鼠疫 疫源地3目6科6亚科12属14种139只宿主动物分 为14个不同的类群(图 2)。
 |
| 图 2 陕西省定边县鼠疫疫源地鼠形动物NJ系统树 |
3. 黄鼠COⅠ基因序列鉴定:在COⅠ基因序列 分析时,发现陕西省定边县黄鼠与宁夏地区黄鼠 样品的相似度>98%,而与内蒙古地区黄鼠样品的 相似度仅为92%~93%;运用Mega5.0软件基于 Kimura-2-parameter(K2P)[9]模型计算各样本的遗 传距离,显示定边黄鼠与宁夏阿拉善黄鼠遗传距 离≤1.8%,与内蒙古达乌尔黄鼠遗传距离为8%~ 9%。从Mega5.0软件构建的黄鼠NJ系统树可见 (图 3),采自宁夏、陕西定边、内蒙古地区的黄鼠样 本形成2个高支持度的单一分支,陕西定边黄鼠样 本与内蒙古达乌尔黄鼠样本分成2个独立分支,但 与宁夏阿拉善黄鼠样本聚为一类。
 |
| 图 3 陕西定边黄鼠与宁夏阿拉善黄鼠、内蒙古达乌尔黄鼠 的NJ聚类图 |
2002年Tautz等[11]将DNA序列用于生物分类。 随后,加拿大动物学家Hebert等[2]在鸟类研究中正 式提出DNA条形码的概念,并对脊椎动物和无脊椎 动物共11门13320个物种的COⅠ基因序列比较分 析,发现可以利用该基因一段长648bp的片段的 DNA碱基序列差异,进行物种的分类识别。除腔肠 动物外,98%的物种种内的差异为0~2%;种间差异 平均为11.3%。对于大部分动物群体而言,均可利 用COⅠ基因序列作为识别条形码[3]。
本研究对陕西省定边县鼠疫疫区3目6科6亚 科12属14种139只宿主动物以及7个鼠种8个不同 部位的62份残体标本COⅠ基因进行序列分析,发 现种内遗传距离为0~2%,种间遗传距离为8.9%~ 15.1%,种间遗传距离显著大于种内遗传距离;14种 宿主动物NJ系统树可区分14个不同的类群;采集 的残体标本除剪断的毛发外,均能检测到目的基 因。不仅肝脏,其他一些部位也可作为分类标本,准 确识别宿主动物。
形态学分析是鉴定啮齿动物常用的基本方法。 由于宁夏阿拉善黄鼠与内蒙古达乌尔黄鼠在形态 学方面有较多相似,采用常规形态学方法难以区分 鉴定。本研究经序列比对发现,陕西定边黄鼠与宁 夏阿拉善黄鼠样品相似度>98%,而与内蒙古达乌 尔黄鼠样品的相似度仅为92%~93%,因此将陕西 省鼠疫疫区多年命名的达乌尔黄鼠更名为阿拉善 黄鼠。
NJ系统树分析显示所有个体形成14个高分化 度的单一分支,但在黑线仓鼠物种分支内出现小支 系,分支间个体的遗传距离为8.9%~10.2%,本研究 发现种间遗传距离为8.9%~15.1%,说明黑线仓鼠 可能存在隐存种,但还有待进一步论证。
Hebert等[2,3]曾分析GenBank中同属物种的 COⅠ序列数据,发现同属不同种间COⅠ序列的平 均差异度为11.3%,种内差异度<2%,且绝大部分< 1%。马英等[12]在利用DNA条形码技术分析青海省 海东地区小型兽类鉴定时发现,种内遗传距离≤ 3%,种间遗传距离为5%~10%,说明DNA条形码数 据积累的物种判断阈值可能会对物种多样性造成一 定的误判,为此还应寻找更多的能够进行分类的基 因片段。DNA条形码技术可克服形态学鉴定的不 足,用于鼠疫疫区宿主动物及残体标本的分类鉴定, 但现阶段由于数据库中缺乏足够的序列信息,故还 需与经典分类结合使用。
| [1] An CH,Chen BB,Fan SP,et al. Identification of newly-recorded Microtus fortis species in the plaque foci of Shaanxi province[J]. Chin J Vector Biol Control,2012,23(4):306-309. (in Chinese) 安翠红,陈宝宝,范锁平,等. 陕西鼠疫自然疫源地新纪录鼠种的鉴定[J]. 中国媒介生物学及控制杂志,2012,23(4):306-309. |
| [2] Hebert PD,Stoeckle MY,Zemlak TS, et al. Identification of birds through DNA barcodes[J]. PLoS Biol,2004,2(10):312. |
| [3] Hebert PD, Ratnasingham S,de Waard JR. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Biol Sci,2003,270 Suppl 1:S96-99. |
| [4] Smith MA, Fisher BL,Hebert PD. DNA barcoding for effective biodiversity assessment of a hyperdiverse arthro-pod group:the ants of Madagascar[J]. Philos Trans R Soc Lod B Biol Sci,2005,360(1462):1825-1834. |
| [5] Hajibabaei M,Singer GA,Clare EL,et al. Design and applicability of DNA arrays and DNA barcodes in biodiversity monitoring[J]. BMC Biol,2007,5:24. |
| [6] Alcaide M, Rico C, Ruiz S,et al. Disentangling vector-borne transmission networks:a universal DNA barcoding method to identify vertebrate hosts from arthropod bloodmeals[J]. PLoS One,2009,4:e7092. |
| [7] Robins JH, Hingston M,Matisoo-Smith E, et al. Identifying Rattus species using mitochondrial DNA[J]. Mol Ecol Notes,2007,7:717-729. |
| [8] Ivanova NV, Zemlak TS, Hanner RH, et al. Universal primer cocktails for fish DNA barcoding[J]. Mol Ecol Notes,2007,7:544-548. |
| [9] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evol,1980,16(2): 111-120. |
| [10] Saitou N,Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Mol Biol Evol,1987,4(4):406-425. |
| [11] Tautz D,Aretander P,Minelli A,et al. DNA points the way ahead in taxonomy[J]. Nature,2002,418:479. |
| [12] Ma Y, Li HL, Lu L. Application for identification of small mammals by DNA barcoding in Haidong area,Qinghai province,China[J]. Biodivers Sci,2012,20(2):193-198. (in Chinese)马英,李海龙,鲁亮. DNA条形码技术在青海海东地区小型兽类鉴定中的应用[J]. 生物多样性,2012,20(2):193-198. |
 2014, Vol. 35
2014, Vol. 35