文章信息
- 黄燕惠, 柯碧霞, 孙九峰, 何冬梅, 陈清, 柯昌文, 俞守义. 2014.
- Huang Yanhui, Ke Bixia, Sun Jiufeng, He Dongmei, Chen Qing, Ke Changwen, Yu Shouyi. 2014.
- 广东省2009-2011年鼠伤寒沙门菌监测及菌株分子分型的研究
- Molecular typing and surveillance on Salmonella typhimurium strain in Guangdong province,2009-2011
- 中华流行病学杂志, 2014, 35(8): 917-924
- Chinese Journal of Epidemiology, 2014, 35(8): 917-924
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2014.08.010
-
文章历史
- 投稿日期:2014-3-14
2. 广东省疾病预防控制中心;
3. 广东省公共卫生研究院;
4. 深圳市宝安区慢性病防治院皮肤性病防治科
2 Guangdong Provincial Center for Disease Control and Prevention, Guangzhou;
3 Guangdong Provincial Institute of Public Health;
4 Department of Skin Venereal Disease Prevention and Control, Baoan District Chronic Disease Hospital
鼠伤寒沙门菌胃肠炎或食物中毒是全球食品加工及其贸易流通导致的非伤寒沙门菌食源性疾病。目前该菌引起的食物中毒事件已由先前的点源性集中暴发,转变为跨地区的“散在暴发”,而追踪污染源及传播途径依赖于菌株分子分型手段[1, 2, 3]。脉冲场凝胶电泳(PFGE)和多位点可变数目串联重复序列分析(MLVA)已成功应用于多起鼠伤寒沙门菌食源性疾病暴发的溯源[2, 3, 4]。广东省为建立实验室主动监测系统和菌株分子分型网络库,自2009年正式启动食源性疾病加强监测(ESS),以发现鼠伤寒沙门菌感染腹泻病例的优势菌型。本研究对2009-2011年ESS各监测点医院鼠伤寒沙门菌临床分离株进行PFGE、MLVA分型,以分析菌株间的分子遗传特征及其关联性,为溯源鼠伤寒沙门菌食源性疾病暴发提供依据。
材料与方法1. ESS及其病例:选择广东省珠三角(广州、中山、珠海、江门、东莞和佛山)及东(揭阳)、西(茂名、阳江)、北(韶关)部10 个城市的24 家医院为监测哨点医院。本文疑似非伤寒沙门菌病例定义为排便≥3次/日且粪便性状有改变,并符合以下任何一条标准:①发热(体温>38 ℃),或伴有头痛、寒战、疲劳;②有恶心、呕吐或腹痛等消化道症状的门诊/住院腹泻病患者。
2. 材料:
(1)实验菌株:来源于2009-2011年ESS监测系统275株鼠伤寒沙门菌;PFGE相对分子质量标准菌株为沙门菌Bmenderup血清型菌株H9812,MLVA分型的阳性对照株为鼠伤寒沙门菌LT2(ATCC29946),均由中国疾病预防控制中心分发,保存于广东省疾病预防控制中心。
(2)主要试剂和仪器:包括连四硫磺酸(TTB)增菌液、沙门菌显色培养基及双糖铁、VP、β-半乳糖苷酶(ONPG)、尿素、靛基质和赖氨酸微量生化管均购自广东环凯微生物科技有限公司,诊断血清为泰国S&A公司产品,API 20E 手工细菌生化鉴定条和Cary Blair 运送培养基为法国生物梅里埃公司产品,XLD平板、MH平板购自广州迪景微生物科技有限公司,XbaⅠ、BlnⅠ酶购自美国Promega 公司,琼脂糖为美国Cambrex 公司产品,蛋白酶K 为德国Merck公司产品。PCR反应仪为美国ABI公司产品,CHEF MAPPER琼脂糖凝胶电泳仪、CHEF MAPPER XA脉冲场凝胶电泳仪和GEL Doc EQ 凝胶成像分析系统为美国Bio-Rad 公司产品,BioMate 3S分光光度计为美国Thermo Fisher公司产品,比浊仪为美国Dade Behring公司产品,BioNumerics软件购自Applied Maths 公司。
3. 研究方法:
(1)菌分离、培养及鉴定:根据GB 4789.4-2010 中沙门菌检验方法,制定广东省ESS方案沙门菌标准检验程序[1]。
(2)PFGE分型:参照美国PulseNet沙门菌PFGE标准操作方法[5]。应用 BioNumerics(Version 5.1)软件处理 PFGE 图像,使用非加权配对算术平均法(unweightedpair group average method)进行聚类分析。
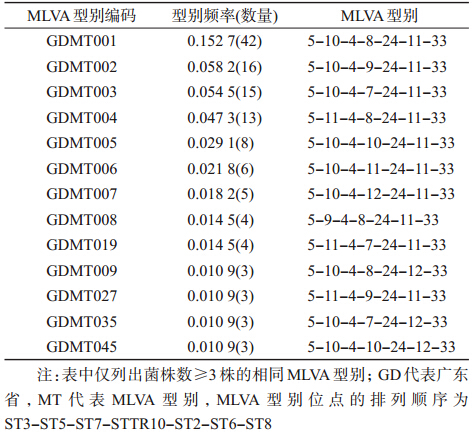
(3)MLVA分型:参考国际实验室分子分型监测网络PulseNet公布的鼠伤寒沙门菌MLVA分型标准方案[6],采用7个VNTR位点,根据位点合成相应引物,并在引物5′端标记荧光基团,根据预实验的结果调整试验退火温度和循环次数(表 1)。每株菌的扩增产物(位点按ST3-ST5、ST7-STTR10、ST2-ST6- ST8混合形式混匀)使用ABI3700测序仪进行毛细管电泳检测,计算每个VNTR位点的重复数,使用软件BioNumerics进行最小生成树(minimum spanning tree)分析,构建参数为creation of complex:1 changes。
(4)D值计算[7]:评价分型方法的分辨能力通常采用基于Simpson的分辨系数,即以D值表示,可直接比较不同分型方法及其不同组合后的效果。其公式
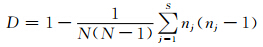
4. 统计学分析:将BioNumerics的分型结果与菌株基本资料以txt的形式导出,并整理成Excel 2003数据信息表,再导入SPSS 13.0软件并建立数据库,分析菌株主要带型的地区、时间、人群分布,组间比较采用χ2检验,α=0.05为统计学检验水准。
结果
1. 监测概况:2009-2011年广东省ESS监测系统共收集样本19 895份,沙门菌年均检出率为4.03%(802/19 895),其中鼠伤寒沙门菌年均检出率、构成比分别为1.38%(275/19 895)、34.29%(275/802),已成为广东省沙门菌优势血清型(表 2)。275株鼠伤寒沙门菌,以2010年检出的构成比最高(44.88%,136/275),每年5、9月为发病高峰(图 1),男女性别比为1.57 ∶ 1,不同年份性别间的差异无统计学意义(χ2=0.95,P>0.05),0~1 岁婴幼儿为主要发病人群(157/275,57.09%),各年份不同年龄段的差异有统计学意义(χ2=10.75,P<0.05),主要分布于广州(95/275,34.55%)、中山(59/275,21.45%)等市。
 |
| 图 1 2009-2011年广东省ESS沙门菌流行趋势 |
2. 鼠伤寒沙门菌PFGE分子分型:
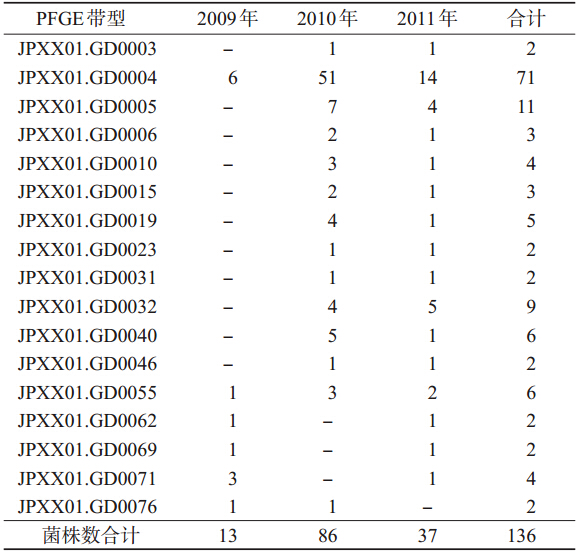
(1) PFGE-XbaⅠ带型聚类分析:275株鼠伤寒沙门菌经XbaⅠ酶切,进行PFGE分型(图 2)。结果显示菌株间相似值为56.09%~100.00%,共分为124种不同带型、7大聚类簇,A、B、E、F、G聚类簇的病例呈散在分布,聚类相似值为66.67%~73.38%,菌株数分别为2、13、5、3、6株,主要检出地区集中在广州、中山、东莞;C、D簇的聚类特点显示具有一定的地域聚集性,聚类相似值分别为77.23%、77.65%,分别由53、192株菌组成,主要来源于珠三角地区的城市,菌株在种群内、种群间的联系不仅具有时间连续性,同时亦与省内其他监测城市有空间联系。
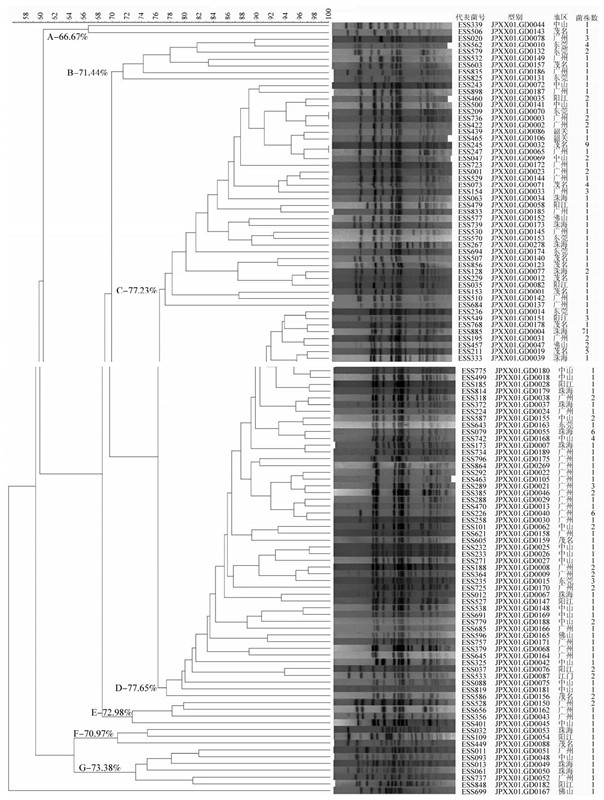 |
| 注:带型相同归为同一型别并统一编号,统计编号相同菌株,挑选有代表性的菌株条带进行聚类分析图 2 广东省275株鼠伤寒沙门菌XbaⅠ酶切PFGE分型 |
(2) PFGE-XbaⅠ带型分布:PFGE分型共获得124种带型,D=0.928 6,每种带型包含1~71株菌,平均每种带型有2.22株菌,有17 种带型在不同年份交叉出现(表 3),其余 107 种带型属于每个年份的独特带型。≥3株菌有16种带型,其带型频率分布见表 4。其中71株为优势带型JPXX01.GD0004,主要包括广州21株(29.6%)、中山17株(23.9%)、东莞14株(19.7%),均分离自不同年代的腹泻患者;时间主要集中于2010年,共51株(71.8%),其中以5月(19.72%,14/71)、9月(15.49%,11/71)居多;性别分布以男性为主(60.6%,43/71),年龄以0~1岁婴幼儿为主(53.5%,38/71),与菌株的总体流行特征一致。不同地区、年代、性别以及年龄组间分布的差异有统计学意义(P<0.05)。
3. 鼠伤寒沙门菌MLVA分子分型:
(1) MLVA型别生成树分析:所有菌株经毛细管电泳检测,计算VNTR位点的重复数,使用BioNumerics软件进行最小生成树分析(图 3)。以≤1位点的差异对菌株分型,共分为143种型别,最小生成树将所有的MLVA型分为5群(Ⅰ~Ⅴ群),其中Ⅰ群包括94个型别184株菌,主要由6个亚群组成,其中以型别002作为支点的进化群亲缘关系变化层次较多,形成Ⅰ群中最大的一簇亚群,主要源自广州、中山市腹泻病例粪便样本分离株(34株),除韶关、揭阳市,其余8个监测城市呈散在分布,而集中于珠三角地区的城市,其地域聚集分布的趋势与PFGE结果一致,其他型别亲缘关系相对较远,这与菌株来源分散有关。
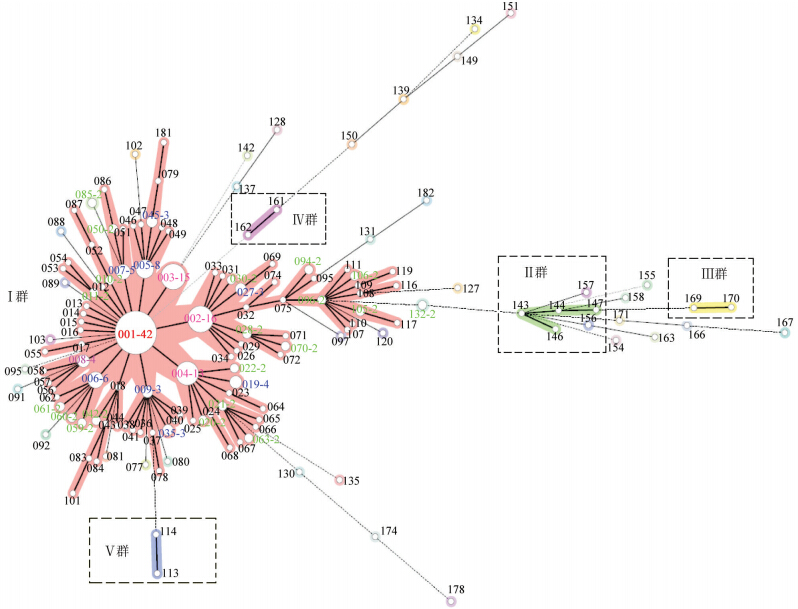 |
| 注:圆圈内(外)数字为MLVA型别编码,型别后的数字为该型别菌株数,深红色(如001-42)为菌株数>20,粉红色(如002-16)为10~20株,蓝色(如005-8)为3~9株,绿色(如010-2)为2株,型别后无数字者均表示1株菌;其他颜色表示菌株的亲缘关系,粗短线连接2个MLVA 型别意味单一VNTR基因位点不同,而细长线表示2个基因位点不同,虚线代表 2个以上基因位点的差异图 3 2009-2011年广东省275株鼠伤寒沙门菌的MLVA分型 |
(2)MLVA型别分布:275株鼠伤寒沙门菌分为143种型别,D=0.966 5,每种型别有1~42株菌,平均每种带型包含1.92株菌,≥3株型别有13种,型别频率分布见表 5。其中优势型别为001(MLVA型别:5-10-4-8-24-11-33)42株,主要分离自广州(15株,35.7%)、阳江(7株,16.7%)、中山(6株,14.3%)以及珠海(5株,11.9%),>50.0%来源于2010年的腹泻患者,每年5-9月为优势条带菌株的高峰期,占全年的81.0%(34/42),以男性为主(27/42,64.3%),并集中于0~1岁组(27/42,64.3%),<5岁儿童占92.9%。不同地区、年代、性别以及年龄组间分布的差异有统计学意义(P<0.05)。
4. MLVA和PFGE分型能力的比较:
(1)分型能力的总体比较:MLVA方法的D值(0.966 5)大于PFGE -XbaⅠ单酶切分型的D值(0.928 6),而与PFGE-XbaⅠ- BlnⅠ双酶切分辨能力(D=0.989 1)相当。主要由于ST5、STTR10、ST6及ST8的多态性较高,D值分别为0.664 8、0.754 3、0.654 6、0.419 0。
(2)分型结果的对应关系:对71株PFGE-XbaⅠ分型后的优势带型JPXX01.GD0004,进一步采用BlnⅠ酶切(PFGE-BlnⅠ),获得31种带型(表 6,其中3株BlnⅠ酶切不能比对,未计算在内),JPXA26.GD0001为优势带型。MLVA将68株菌分为41个型,其中001型6株、002型4株、003型2株、005型3株及007型2株均对应于PFGE-BlnⅠ的部分JPXA26.GD0001型及部分JPXA26. GD0006、0005、0036、0050、0017型。分析显示MLVA将BlnⅠ酶切后的条带进一步区分为不同的型别,证实MLVA分型方法的可靠性。
2009-2011 年广东省ESS中沙门菌平均检出率为4.03%,高于2008年的检出率3.69%[8],但低于2007 年的检出率6.29%[9],与国内其他城市(2%~5%)相当,但低于发达国家(5%~8%),与采样量和检测方法相关[10, 11]。广东省年均检出率较高的城市依次为佛山、珠海、东莞,这与菌株的地区分布特点、监测点医院的数目与类型(妇幼保健院)有关,上述地区工业发达,外来务工人员密集,已成为食源性疾病频发的流行地区。鼠伤寒沙门菌是非伤寒沙门菌感染性腹泻的优势菌型,其年均构成比为34.29%,且每年几乎都有“散在”暴发的报道[2, 3, 12, 13]。男性为主要感染人群,0~1 岁婴幼儿占60%,主要分布于广州和中山市,3年间菌株的流行变化趋势大致相同,呈双峰分布,高峰出现在5-6月和9-10月,表现为季节的交替变化。
PFGE已作为众多国家公共卫生实验室用以区分暴发菌株和溯源的“金标准”[14, 15]。目前早期发现沙门菌食源性疾病暴发在很大程度上依赖于菌株的分子分型技术,而MLVA作为第二代分子分型技术特别是对那些PFGE分型不理想或不适用的致病菌,显示出一定的优势。本研究对275株鼠伤寒沙门菌进行PFGE和MLVA分子分型,结果表明PFGE-XbaⅠ带型相似值较高、MLVA型别亲缘进化关系密切的菌群,其分布特点与菌株流行趋势相似,且主要集中于菌株分离较多的广州和中山地区,呈地域聚集性,感染人群则以0~1 岁男性婴幼儿为主,优势型别的流行病学趋势也有相同表现,季节流行趋势与菌株检出情况相一致。MLVA分型的D值高于PFGE-XbaⅠ单酶切,与PFGE-XbaⅠ- BlnⅠ双酶切的分辨能力相当。与PFGE-XbaⅠ条带的分析结果相比较,MLVA分型对同一血清型或同一菌群,不仅显示高的分辨力,而且基于多个VNTR位点重复数的分型,可以揭示菌群亲缘进化关系的微小变化,结合MLVA的最小生成树、MLVA型别分布以及VNTR位点重复数频率分布分析,表明高度基因多态性的同时,分型结果有一定的变化规律,即从GDMT001型为始祖的第一大菌群,常见的变化位点为STTR10、ST5、ST6,这三个位点的单独变化或者两两组合发生变化,就会表现为其他常见的MLVA型别,如STTR10的重复数为7、9、10、11、12时,由001型可变化为003、002、005、006、007,说明分型和亲缘进化关系主要与位点的多态性有关[16]。除了ST3、ST7及ST2位点的多态性较低,与张京云等[17]报道的相符合,其他VNTR位点均具有较高的多态性,D值均>0.4,主要由于位点重复数的种类多样,并且出现单菌株表现一种重复类型的情况,这与国内外相关报道相符[18, 19, 20, 21]。
MLVA分型不仅与PFGE二次酶切的结果相一致,而且可将PFGE-XbaⅠ优势带型JPXX01.GD0004二次酶切后的条带进一步区分为相同或不同的型别,通过对照流行病学资料以证实感染者在人群、地区以及时间上的联系。表明MLVA分子分型技术,不仅可从宏观上对菌株的相似性进行分析,且可从微观上揭示菌株的亲缘进化关系,用于及时监测潜在或散在的感染暴发。
| [1] Chiou CS, Hung CS, Torpdahl M, et al. Development and evaluation of multilocus variable number tandem repeat analysis for fine typing and phylogenetic analysis of Salmonella enterica serovar typhimurium[J]. Int J Food Microbiol,2010,142(1):67-73. |
| [2] Torpdahl M,Sørensen G,Lindstedt BA,et al. Tandem repeat analysis for surveillance of human Salmonella typhimurium infections[J]. Emerg Infect Dis,2007,13(3):388-395. |
| [3] Lindstedt BA,Heir E,Gjernes E, et al. DNA fingerprinting of Salmonella enterica subsp. enterica serovar typhimurium with emphasis on phage type DT104 based on variable number of tandem repeat loci[J]. J Clin Microbiol,2003,41(4):1469- 1479. |
| [4] Torpdahl M,Sorensen G,Ethelberg S,et al. A regional outbreak of S. typhimurium in Denmark and identification of the source using MLVA typing[J]. Euro Surveill,2005,11(5):134-136. |
| [5] Ribot EM,Fair MA,Gautom R,et al. Standardization of pulsed- field gel electrophoresis protocols for the subtyping of Escherichia coli O157 : H7,Salmonella,and Shigella for PulseNet[J]. Foodborne Pathog Dis,2006,3(1):59-67. |
| [6] Laboratory standard operating procedure for PulseNet MLVA of Salmonella enterica serotype typhimurium-Applied Biosystems Genetic Analyzer Platform[DB/OL]. http://www.pulsenetinternational.org/assets/PulseNet/uploads/mlva/2013%20updates/PNL24_MLVASalmTABIProtocol.pdf. |
| [7] Hunter PR,Gaston MA. Numerical index of the discriminatory ability of typing systems:an application of Simpson's index of diversity[J]. J Clin Microbiol,1988,26(11):2465-2466. |
| [8] Ke BX,Deng XL,Li BS,et al. Surveillance on Salmonella infection in Guangdong province,2008-2009[J]. Chin J Epidemiol,2011,32(8):789-792.(in Chinese)柯碧霞,邓小玲,李柏生,等. 广东省 2008-2009 年沙门菌监测[J]. 中华流行病学杂志,2011,32(8):789-792. |
| [9] Ke BX, Deng XL,Zhang LH,et al. Surveillance and pathogenic analysis on non-typhoidal Salmonella in Guangdong province,2007[J]. Chin J Epidemiol,2008,29(12):1199-1203.(in Chinese)柯碧霞,邓小玲,张丽华,等. 广东省 2007 年度非伤寒沙门菌监测及病原学特征分析[J]. 中华流行病学杂志,2008,29(12):1199-1203. |
| [10] Xu XB,Gu BK,Jin HM,et al. Development of potimum method to detected Salmonella from clinical fecal speciments and analyzed character of phenotype[J]. Chin J Epidemiol,2007,28(8) :828-829.(in Chinese)许学斌,顾宝柯,金汇明,等. 沙门菌检测在临床粪便样品的优化方法建立和表型特征分析[J]. 中华流行病学杂志,2007,28(8):828-829. |
| [11] Huang Z,Xu XB,Zhang L. Application of optimum method to detect Salmonella from clinical fecal specimens[J]. Chin J Health Lab Technol,2007,17(8):1453-1455.(in Chinese)黄峥,许学斌,张岭. 沙门菌检测在临床粪便样品的优化方法应用[J]. 中国卫生检验杂志,2007,17(8):1453-1455. |
| [12] Fuller CC,Jawahir SL,Leano FT, et al. A multi-state Salmonella typhimurium outbreak associated with frozen vacuum-packed rodents used to feed snakes[J]. Zoonoses Public Health,2008,55(8-10):481-487. |
| [13] Malorny B,Junker E,Helmuth R. Multi-locus variable-number tandem repeat analysis for outbreak studies of Salmonella enterica serotype enteritidis[J]. BMC Microbiol,2008,8(1):84. |
| [14] Thong KL,Ngeow YF,Altwegg M,et al. Molecular analysis of Salmonella enteritidis by pulsed-field gel electrophoresis and ribotyping[J]. J Clin Microbiol,1995,33(5):1070-1074. |
| [15] Landeras E, Gonzalez-Hevia MA, Alzugaray R, et al. Epidemiological differentiation of pathogenic strains of Salmonella enteritidis by ribotyping[J]. J Clin Microbiol,1996,34(9):2294-2296. |
| [16] Grissa I,Bouchon P,Pourcel C,et al. On-line resources for bacterial micro-evolution studies using MLVA or CRISPR typing[J]. Biochimie,2008,90(4):660-668. |
| [17] Zhang JY,Nie YN,Chen CX,et al. Evaluation of PFGE and MLVA in molecular typing of Salmonella typhimurium isolated in China[J]. Dis Surveill,2011,26(4):264-270.(in Chinese)张京云,聂艳妮,陈春霞,等. 脉冲场凝胶电泳和多位点串联重复序列分析应用于中国鼠伤寒沙门菌分型能力的评价[J]. 疾病监测,2011,26(4):264-270. |
| [18] Chen JC,Fan FX,Wang SJ,et al. Characteristics of molecular typing for Salmonella typhimurium isolated in China from 2006 to 2010[J]. Chin J Zoonoses,2013,28(12):1161-1166.(in Chinese)陈建才,樊粉霞,王淑京,等. 2006-2010 年中国鼠伤寒沙门菌分子分型分析[J]. 中国人兽共患病学报,2013,28(12):1161-1166. |
| [19] Wuyts V,Mattheus W,de Bex GDL,et al. MLVA as a tool for public health surveillance of human Salmonella typhimurium:prospective study in belgium and evaluation of MLVA loci stability[J]. PLoS One,2013,8(12):e84055. |
| [20] Sintchenko V,Wang Q,Howard P,et al. Improving resolution of public health surveillance for human Salmonella enterica serovar typhimurium infection:3 years of prospective multiple-locus variable-number tandem-repeat analysis (MLVA)[J]. BMC Infect Dis,2012,12(1):78. |
| [21] Broschat SL,Call DR,Davis MA,et al. Improved identification of epidemiologically related strains of Salmonella enterica by use of a fusion algorithm based on pulsed-field gel electrophoresis and multiple-locus variable-number tandem-repeat analysis[J]. J Clin Microbiol,2010,48(11):4072-4082. |
 2014, Vol. 35
2014, Vol. 35