2. 赛默飞北京环贸实验室, 北京 100013
2. Thermo Fisher Beijing Training Center, Beijing 100013, China
随着生物技术的发展,基因治疗技术逐渐走入人们的视野,溶瘤病毒作为基因治疗领域中的新热点,日益受到人们的关注。
2019年《人用基因治疗制品总论(公示稿)》为基因治疗制品的规范生产,保障制品的安全、有效和质量可控,提出了总体指导性要求。在公示稿中定义基因治疗制品为“通常由含有工程化基因构建体的载体或递送系统组成,其活性成分可为DNA、RNA、基因改造的病毒、细菌或细胞,通过将外源基因导入靶细胞或组织,替代、补偿、阻断、修正特定基因,以达到治疗疾病的目的”。
从总体上看,基因治疗制品有以下特点:由核酸序列达成治疗目的;有些核酸被包装在载体内无法直接检测;被包装核酸可不完全指导包装蛋白合成;核酸链相似度高,不易区分;载体具有一定的变异性,如病毒载体等。变异与遗传同时存在,都是生命体的固有特征。对于基因治疗制品来讲,遗传带来了批间稳定性,而变异带来了批间差异。如何在承认批间差异的前提下,保障制品的批间稳定性,即如何检测核酸序列的变异并将其控制在可接受范围内,逐渐成为热议话题。
DNA序列检测已经成为基因治疗制品质量控制的主要手段之一。目前,测序技术已经发展到第三代。第一代DNA测序技术以Fred Sanger开发的双脱氧链终止法为代表,后续发展成了荧光自动测序技术,其读长约为1 000 bp(base pair),准确性高达99.999%,但通量较低并且成本较高;第二代测序技术又称高通量测序(high-throughput sequencing)或下一代测序(next generation sequencing,NGS),以Hiseq技术、454技术和Solid技术为代表,其读长为200 bp,高通量,差错率在1%~1.5%;第三代DNA测序技术属于单分子测序技术,读长可达150 kbp,但差错率约15%[1]。
为探讨DNA测序技术在基因治疗制品中的检测特点,本文以一种改构溶瘤腺病毒制品为例,进行DNA测序,并对病毒基因组关键区段和非关键区段,以及其变异体进行了分析。
1 仪器与材料 1.1 材料溶瘤腺病毒注射液(modified oncolytic adenovirus,MOA)样品,批号201808003,为中国食品药品检定研究院重组药物室留样。病毒提取纯化试剂盒High Pure Viral Nucleic Acid Kit,批号11858874001,Roche公司。DNA文库构建试剂盒Ion XpressTM Plus Fragment Library Kit,货号4471252,Thermo Fisher公司。Ion 530TM Chip Kit-4 Reactions,货号A27763,Thermo Fisher公司。克隆载体,pClone007 Blunt Vector Kit,批号TSV-007B,Tsingke公司。
1.2 主要试剂及仪器Ion GeneStudioTM S5 Prime第二代测序仪,Thermo Fisher公司。DU-800核酸蛋白分析仪,Beckman公司。引物合成及其第一代测序均由北京博奥晶典生物技术有限公司完成,DNA测序仪型号为ABI3730XL。PureYieldTM Plasmid Miniprep System,A1222,Promega公司。Wizard SV Gel and PCR Clean-up System,A9282,Promega公司。
2 方法和结果 2.1 病毒DNA制备取样品400 μL,用病毒提取纯化试剂盒完成病毒基因组提取及纯化,纯化后DNA经水适当稀释,如50倍,用核酸蛋白分析仪检测280 nm和260 nm处的吸收值之比A260/A280(池长10 mm),结果为1.88。
将纯化所得DNA分成2份,分别用于第一代和第二代DNA测序检测。
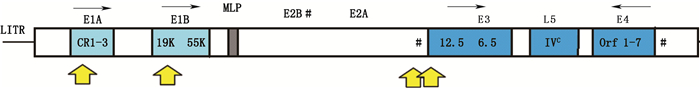
2.2 关键区段测序按照第一代测序仪的操作程序进行测序,DNA测序相关引物见表 1,对关键区段进行了8个反应,所测位置由图 1示意标出,关键区段相关解释见讨论项。
|
|
表 1 关键区段第一代DNA测序引物表 Tab.1 Primer list of first generation DNA sequencing in key regions |
|
图 1 改构溶瘤腺病毒的结构示意图(粗箭头所指为改构的关键区段) Fig.1 Structural diagram of modified oncolytic adenovirus(the key area indicated by the thick arrow is the reconstructed area) |
第一代测序结果用DNA star软件5.01版拼接分析,并与理论序列进行比较,结果与理论序列一致(相关谱图略)。
按照第二代测序文库制备试剂盒相关说明制备DNA测序文库,构建文库所用的链接DNA两端片段为测序仪特定功能性片段(序列略),测序深度控制在每个位置约300~600条文库DNA序列,制备好的DNA文库通过专属附带设备制备芯片,然后上机检测。所得数据通过仪器自带插件,PathogenNt,与预期理论序列比对。
所有测得关键区段DNA文库片段经拼接分析,结果与关键区段预期理论序列基本一致(相似度大于99.9%)。
2.3 非关键区段测序将第二代测序结果进行拼接并与预期理论DNA序列比较(包括全部DNA序列及非关键区段),相似度约99.9%;与NT数据库(Nucleotide Sequence Database)已知Human adenovirus DNA序列比较,给出序列的种属归类组成。种属归类(按相似度最高的归类计入)结果见表 2,其中合计96.4%的序列可以得到种属确认,未确认种属的序列可能为链接序列等。
|
|
表 2 与NT数据库中已知物种序列比较结果 Tab.2 Comparison results with known species sequences in NT database |
在1 900~2 050 bp区间发现变异区段进行分析。首先PCR扩增(反应体系:选择距离待测位置50~100 bp左右适当序列作为引物序列,super PCR mix 20 μL中加入模板和引物共1 μL;扩增条件:98 ℃ 2 min;98 ℃ 10 s,58 ℃ 10 s,共30循环;72 ℃ 10 s;4 ℃保存)。扩增后产物经1%琼脂糖凝胶电泳(100 V,15 min左右)挖胶后纯化(DNA纯化试剂盒),进行连接反应(反应体系:PCR产物0.5 μL,载体1 μL,Topo mix 1 μL,加水至10 μL;反应条件:室温5 min),连接产物转化感受态(42 ℃,45 s),涂布氨苄LB培养基平板,37 ℃,过夜。随机挑取9个单克隆,接种于氨苄LB液体培养基中,37 ℃,8 h。分别收集菌液并提取和纯化质粒(质粒纯化试剂盒),以此为模板,用M13F通用引物进行第一代测序分析,结果见表 3。
|
|
表 3 变异区段DNA序列分析结果 Tab.3 Results of DNA sequence analysis of variant segments |
腺病毒基因组主要包括E1区、E2区、E3区、E4区和“骨架”区[2]。其中E1区的表达产物有E1A和E1B 2种,E1A蛋白的主要功能是调节细胞代谢[3];E1B 55 kD基因产物可以下调p53基因的转录水平[4]。E2区基因表达产物可分为E2A和E2B,其中E2A为DNA结合蛋白;E2B主要产物有2种,分别是末端蛋白前体和病毒DNA聚合酶,这3种蛋白对启动腺病毒DNA复制以及病毒晚期基因的转录和翻译过程至关重要[5]。E3区基因表达产物主要功能是破坏宿主的免疫防御机制,产物之一的11.6 kD,由于可以在病毒感染的晚期裂解细胞并释放病毒颗粒而被称为腺病毒死亡蛋白[6-7];gp 19K蛋白可以在内质网上与MHC Ⅰ类分子的重链结合阻止其转运到细胞表面,并且可以延缓MHC Ⅰ的表达[8];受体内化和降解(receptor internalization and degradation,RID)α & β以及14.7 kD可以抑制由肿瘤坏死因子(TNF)诱发的细胞凋亡,促进凋亡相关因子(apoptosis related factors,Fas)降解,下调TNF受体水平[9]。E4区基因表达产物,主要与病毒信使RNA的代谢有关,还有促进病毒DNA复制以及关闭宿主蛋白合成的功能等[10]。
对腺病毒各蛋白功能的研究深入,以及作为制品的治疗目的需求,催生了对腺病毒的不同改造,包括缺失和插入等[11],改造位置包括表达蛋白基因的改造、调控序列的改造和其他功能基因的插入等。改造区段通常被定义为关键区段,根据《人用基因治疗制品总论(公示稿)》要求,关键区段范围原则上应包括删除位点两侧0.5 kb、插入位点两侧0.5 kb、插入序列全序,另还可包括生产企业认为的,对制品生产、质量控制及药学功能等有重大影响的区段,关键区段的质量标准通常制定为“应与理论序列一致”,如有同义突变或无意义突变等情况,可结合对制品的影响酌情处理,如定义为“应与理论序列一致,可有相关序列存在”(“相关序列”借鉴相关蛋白概念,可理解为序列相近,功能相同或近似[12])。
关键区段以外的其他部分为非关键区段,考虑到载体的变异性,通常采取总体控制的方式,如相似度不得低于97.5%。计算DNA序列相似性的方法较多,例如距离法[13],即在一定长度的DNA序列范围内,如100 bp,2条进行比较的DNA序列,最多有多少碱基序列相同,或反过来理解为最少变化多少碱基可将一条变为另一条。由于非关键区段的范围较大,且第一代测序的通量较小,所以通常对非关键区段测序采用第二代测序技术来完成,并且作为制品鉴别项目应用。
第一代测序技术与第二代测序技术原理略有不同,第一代测序技术对模板的要求,通常由“单分子”的扩增而来,测序后序列的判读通常采用荧光信号的丰度来判断,仪器自动选择信号丰度最高的峰作为判读结果,但当模板有突变时,尤其比例又比较高时,就会出现误判或无法判读等现象。遇到此类问题,通常的做法是将该区段定点扩增,克隆入载体,随机挑取多个单克隆后,再提取纯化DNA,然后测序,由于工作量较大,通常不能得到精度较高的比例结果。第二代测序技术由于已经实现了对模板的DNA文库建立并且较容易实现在某一点上文库DNA条数的高覆盖,虽然存在文库建立效率等一系列的定量问题,但可以通过软件分析,得到精度较高的比例结果。
由于第一代测序技术和第二代测序技术均不是单分子测序技术[14],因此在测量较长DNA分子时都采用了拼接技术[15],如果在较远的位置(大于读长)存在2个突变,判断2个突变是否在同一DNA分子上还是在2个DNA分子上,目前第一代和第二代测序技术均不能实现。因此,对变异体比例的分析,目前还是针对某一位置来定义,而非对全部基因组序列定义。
本文中提及的变异区段是一段高GC含量区,但长度只有十几个碱基,本文所用的酶为高保真DNA聚合酶(差错率低于0.1%),并且测序信号清晰,基本可以排除实验体系本身原因引起的误读。该段变异区相关分析显示并不引起制品的功能上的根本性改变,属于上文提及的“相关序列”。
本文以一种改构溶瘤腺病毒制品为例,探讨DNA测序技术在基因治疗制品中的检测特点,为基因治疗制品的质量控制和质量标准制定提供一些数据支持。
| [1] |
徐疏梅. 新一代DNA测序技术的应用与研究进展[J]. 徐州工程学院学报(自然科学版), 2018, 34(12): 60. XU SM. Application and research progress of a new generation of DNA sequencing technology[J]. J Xuzhou Inst Technol(Nat Sci Ed), 2018, 34(12): 60. |
| [2] |
ISMAIL AM, LEE JS. Adenoviromics:mining the human adenovirus species D genome[J]. Front Microbiol, 2018, 11(9): 2178. |
| [3] |
SAHA B, PARKS RJ. Human adenovirus type 5 vectors deleted of early region 1(E1) undergo limited expression of early replicative E2 proteins and DNA replication in non-permissive cells[J]. PLoS One, 2017, 12(7): e0181012. DOI:10.1371/journal.pone.0181012 |
| [4] |
SAVELYEVA I, DOBBELSTEIN M. Infection with E1B-mutant adenovirus stabilizes p53 but blocks p53 acetylation and activity through E1A[J]. Oncogene, 2011, 30(7): 865. DOI:10.1038/onc.2010.461 |
| [5] |
AMALFITANO A, HAUSER MA, HU H, et al. Production and characterization of improved adenovirus vectors with the E1, E2b, and E3 genes deleted[J]. J Virol, 1998, 72(2): 926. DOI:10.1128/JVI.72.2.926-933.1998 |
| [6] |
HAUT LH, GILL AL, KURUPATI RK, et al. A partial E3 deletion in replication-defective adenoviral vectors allows for stable expression of potentially toxic transgene products[J]. Hum Gene Ther Methods, 2016, 27(5): 187. DOI:10.1089/hgtb.2016.044 |
| [7] |
MEI YF, WU H, HULTENBY K, et al. Complete replication-competent adenovirus 11p vectors with E1 or E3 insertions show improved heat stability[J]. Virology, 2016, 497: 198. DOI:10.1016/j.virol.2016.07.026 |
| [8] |
WOLD WS, CLADARAS C, DEUTSCHER SL, et al. The 19-kDa glycoprotein coded by region E3 of adenovirus.Purification, characterization, and structural analysis[J]. J Biol Chem, 1985, 260(4): 2424. |
| [9] |
FRIEDMAN JM, HORWITZ MS. Inhibition of tumor necrosis factor alpha-induced NF-kappa B activation by the adenovirus E3-10.4/14.5K complex[J]. J Virol, 2002, 76(11): 5515. DOI:10.1128/JVI.76.11.5515-5521.2002 |
| [10] |
SORIANO AM, CRISOSTOMO L, MENDEZ M, et al. Adenovirus 5 E1A interacts with E4orf3 to regulate viral chromatin organization[J]. J Virol, 2019, 93(10): pii:e00157. |
| [11] |
CAI Z, LV H, CAO W, et al. Targeting strategies of adenovirus-mediated gene therapy and virotherapy for prostate cancer(Review)[J]. Mol Med Rep, 2017, 16(5): 6443. DOI:10.3892/mmr.2017.7487 |
| [12] |
韩春梅, 刘兰, 杨靖清, 等. 重组人粒细胞刺激因子的N端氨基酸序列异质性分析[J]. 药物分析杂志, 2018, 38(11): 1887. HAN CM, LIU L, YANG JQ, et al. Analysis of N-terminal amino acid sequence heterogeneity of recombinant human granulocyte colony-stimulating factor[J]. Chin J Pharm Anal, 2018, 38(11): 1887. |
| [13] |
JIN X, JIANG Q, CHEN Y, et al. Similarity/dissimilarity calculation methods of DNA sequences:a survey[J]. J Mol Graph Model, 2017, 76: 342. DOI:10.1016/j.jmgm.2017.07.019 |
| [14] |
VOLLGER MR, LOGSDON GA, AUDANO PA, et al. Improved assembly and variant detection of a haploid human genome using single-molecule, high-fidelity long reads[J]. Ann Hum Genet, 2019. DOI:10.1111/ahg.12364 |
| [15] |
李艳慧, 张少强. DNA测序技术及其拼接算法综述[J]. 天津师范大学学报(自然科学版), 2018, 38(5). LI Yanhui, ZHANG Shaoqiang. Overview of DNA sequencing techniques and corresponding assembly algorithms[J]. J Tianjin Norm Univ(Nat Sci Ed), 2018, 38(5). DOI:10.19638/j.issn1671-1114.20180501 |
 2020, Vol. 40
2020, Vol. 40 
