细菌DNA特征序列鉴定法在常见种属中的鉴定水平研究
冯震

,
蒋波
,
李芳
,
张宁
,
杨燕
,
秦峰
,
杨美成


上海市食品药品检验所, 国家药品监督管理局药品微生物检测技术重点实验室, 上海 201203
收稿日期:2019-06-19
基金项目:2016年上海市科委技术标准专项:《药品中微生物鉴定通用DNA条形码国家标准的建立》(16DZ0500202);2017年国家药典委员会标准提高研究项目:《标准核酸测序数据库的建立》(2017-6)、《致病菌鉴定与溯源用标准核酸序列的建立》(2017-17)
摘要:目的:开展细菌DNA特征序列鉴定法在药品质量控制中常见种属的鉴定水平研究,为药品全生命周期质量控制中的菌株鉴定与污染风险识别提供方法依据。方法:针对性地选择芽孢杆菌、库克氏菌、微球菌、柠檬酸杆菌、肠杆菌、肠球菌、假单胞菌、不动杆菌、鞘氨醇单胞菌和罗尔斯通氏菌共10个常见属中41个种72株的典型菌株,依据细菌DNA特征序列鉴定法,建立菌株鉴定的DNA特征序列,分析属内种间的菌株聚类关系,评价菌株DNA特征序列的鉴定水平。结果:10个属41个种典型菌株的DNA特征序列均具有特异性。结论:细菌DNA特征序列可满足"种"水平的鉴定,为药品质量控制中常见细菌的鉴定、分类与溯源提供研究思路和方法依据。
关键词:菌种鉴定 DNA特征序列 系统进化分析 药品质量控制 风险识别
Study on the identification level of bacterial DNA signature sequence identification in common bacterial species
FENG Zhen
,
JIANG Bo,
LI Fang,
ZHANG Ning,
YANG Yan,
QIN Feng,
YANG Mei-cheng

Shanghai Institute for Food and Drug Control, NMPA Key Laboratory for Testing Technology of Pharmaceutical Microbiology, Shanghai 201203, China
Abstract: Objective: To study on the identification level of common bacteria in the drug quality control by DNA signature sequence, and to provide reference for the identification of bacterial strains and contamination risk investigation of the whole life cycle quality control of drugs.Methods: 72 strains in 41 species in 10 common genus were selected for study, including Bacillus, Kocuria, Micrococcus, Citrobacter, Enterobacter, Enterococcus, Pseudomonas, Acinetobacter, Sphingomonas and Rolstonia. According to the bacterial DNA signature sequence identification method, the bacterial DNA signature sequences were established, the cluster relationship between the species within the genus were analyzed, and the strain identification level was evaluated.Results: The DNA signature sequences which identified by 72 strains of 14 species in 10 genus were respectively specificity in the level of species.Conclusions: The bacterial DNA signature sequence can identify bacterial in the level of species. The results may provide research ideas and methodological basis for the identification, classification and traceability of common bacteria in drug quality control.
Keywords:
bacterial identification DNA signature sequence phylogenetic analysis drug quality control risk identification
微生物污染是引发用药安全事件的重要因素,美国FDA公布的药品召回事件中,由“药品生产过程未达到无菌保障水平”、“环境微生物监控超标”、“无菌检查结果阳性”等微生物控制问题引发的事件占比将近50%。药品生产质量管理规范(GMP)2010版、美国注射剂协会技术要求指南(PDA Technical Report)等法规和规范性文件中,对于微生物的鉴定均提出了更高要求;《中华人民共和国药典》2020年版四部拟新增“细菌DNA特征序列鉴定法”(通则1021),为药品原料、辅料、制药用水、中间产品、终产品、包装材料和环境等药品质量控制环节中的细菌鉴定提供具体方法[1-3]。
根据美国FDA药品召回公告、国家药品审评核查公告等监管信息,常见细菌污染物主要有以下几个属:柠檬酸杆菌、肠杆菌、肠球菌等非无菌制剂生产工艺中的常见菌[4-5];葡萄球菌、芽孢杆菌、库克氏菌、微球菌等[6-7]生产环境中的常见菌,其中葡萄球菌已开展相关研究工作[8];假单胞菌、不动杆菌、鞘氨醇单胞菌、罗尔斯通氏菌等水系统中的常见菌[9-10]。
本文以制药行业质量控制中常见细菌污染物为研究对象,以细菌DNA特征序列鉴定法为依据,开展菌株鉴定水平的研究,为药品质量控制中常见细菌的鉴定、分类与溯源提供研究数据;为药品微生物质量控制中的细菌核酸测序鉴定标准化及药品质量标准提高等工作提供研究思路和参考依据[11]。
1 材料与方法
1.1 试验菌株
试验用标准菌株购自美国微生物菌种保藏中心(ATCC)、中国医学细菌菌种保藏管理中心(CMCC)、中国工业微生物菌种保藏管理中心(CICC)等权威菌种保藏中心;试验用分离株来源于药品质量控制的各环节,由本实验室收集、保藏。试验菌株采用革兰染色、生化反应、核糖体分型等多相分类鉴定方法进行鉴定,确证菌株遗传背景。
1.2 主要仪器与试剂
VITEK全自动微生物生化鉴定系统(Biomerieux公司);RiboPrinter全自动微生物基因指纹鉴定系统,及配套EcoR Ⅰ试剂套装(DuPont公司);ABI9700型PCR扩增仪(ABI公司);琼脂糖凝胶电泳仪及成像系统(BioRad公司);MIR-254型培养箱(SANYO公司);LABGARD型生物安全柜(NuAire公司)。
胰酪胨大豆琼脂平板(TSA,Biomerieux公司);细菌基因组DNA小量纯化试剂盒、Premix TaqTM DNA聚合酶试剂盒(TAKARA,大连宝生物工程有限公司);POP-7 Ploymer、BDT V3.1 SEQ Buffer等核酸测序试剂(Lifetech公司);其他化学试剂均为分析纯。
1.3 菌株DNA特征序列片段扩增及核酸测序鉴定
试验菌株经分离纯化获得单克隆菌落,提取菌株基因组DNA。分别以正向引物(16SV1F):5’-AGAGTTTGATCCTGGCTCAG-3’和反向引物(16SV3R):5’-GTATTACCGCGGCTGCTGGC-3’扩增细菌16S rRNA基因中V1~V3区核酸序列,获得500 bp左右单一的特异性条带。PCR扩增体系为20 μL,其中Premix 10 μL,基因组DNA 0.5 μL,引物各0.25 μL,ddH2O 9 μL。PCR反应条件:94 ℃ 3 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 45 s,32个循环;72℃ 5 min。将PCR产物纯化后进行单链扩增,扩增体系为20 μL,其中PCR产物1 μL,引物(3.2 pmol)4 μL,BigDye(2.5×)8 μL,ddH2O 7 μL;PCR反应条件:96 ℃ 1 min,96 ℃ 10 s,50 ℃ 5 s,60 ℃ 4 min,25个循环,4 ℃保温。采用试剂盒(BigDye XTerminator Purification Kit)纯化单链扩增产物后,按照核酸测序仪说明操作,上机测序。
1.4 核酸序列聚类关系分析
对获得的核酸序列进行测序质量值校核后(Q值> 30),采用Lasergene软件进行序列拼接,导入Mega 5.0序列分析软件,以邻接建树法(neighbor joining clustering method)方法进行序列比对,以Bootstrap Method(1000)、Kimura 2-Parameter Model、Uniform Rates、Pairwise Deletion的统计学计算方式,构建NJ进化树(neighbor-joining tree),聚类分析各个种属之间的进化关系。
2 实验结果
2.1 多相分类鉴定
收集整理芽孢杆菌属5个种10株、库克氏菌属4个种7株、微球菌属1个种4株、柠檬酸杆菌属5个种8株、肠杆菌属4个种8株、肠球菌属7个种12株、假单胞菌属5个种9株、不动杆菌属4个种6株、鞘氨醇单胞菌属4个种4株、罗尔斯通氏菌属2个种4株,共10个属72株细菌,用于菌株DNA特征序列鉴定,菌株信息见表 1。
表 1
Tab.1
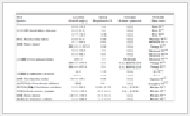
表 1 本文用于菌株DNA特性序列鉴定的菌株信息
Tab.1 Bacterial isolate information using for phylogenetic analysis in this study
序号
(number) |
属名(genus) |
种名(speices) |
试验菌株数量
(strain number) |
| 1 |
芽孢杆菌(Bacillus) |
枯草芽孢杆菌(Bacillus subtilis),苏云金芽孢杆菌(Bacillus thuringiensis),蜡样芽孢杆菌(Bacillus cereus),巨大芽孢杆菌(Bacillus megaterium),地衣芽孢杆菌(Bacillus licheniformis) |
10 |
| 2 |
库克氏菌(Kocuria) |
嗜根库克氏菌(Kocuria rhizophila),玫瑰库克氏菌(Kocuria rosea),克氏库克氏菌(Kocuria kristinae),变异库克氏菌(Kocuria varians) |
7 |
| 3 |
微球菌(Micrococcus) |
藤黄微球菌(Micrococcus luteus) |
4 |
| 4 |
柠檬酸杆菌(Citrobacter) |
弗氏柠檬酸杆菌(Citrobacter freundii),杨氏柠檬酸杆菌(Citrobacter youngae),布氏柠檬酸杆菌(Citrobacter braakii),穆氏柠檬酸杆菌(Citrobacter murliniae) |
8 |
| 5 |
肠杆菌(Enterobacter) |
产气肠杆菌(Enterobacter aerogenes),阪崎肠杆菌(Enterobacter sakazakii),阴沟肠杆菌(Enterobacter cloacae),生癌肠杆菌(Enterobacter cancerogenus) |
8 |
| 6 |
肠球菌(Enterococcus) |
粪肠球菌(Enterococcus faecalis),铅黄肠球菌(Enterococcus casseliflavus),鸡肠球菌(Enterococcus gallinarum),海氏肠球菌(Enterococcus hirae),盲肠肠球菌(Enterococcus cecorum),屎肠球菌(Enterococcus faecium),耐久肠球菌(Enterococcus durans) |
12 |
| 7 |
假单胞菌(Pseudomonas) |
施氏假单胞菌(Pseudomonas stutzeri),绿针假单胞菌(Pseudomonas chlororaphis),恶臭假单胞菌(Pseudomonas putida),铜绿假单胞菌(Pseudomonas aeruginosa),荧光假单胞菌(Pseudomonas fluorescens) |
9 |
| 8 |
不动杆菌(Acinetobacter) |
鲍曼不动杆菌(Acinetobacter baumannii),溶血不动杆菌(Acinetobacter haemolytius),印度不动杆菌(Acinetobacter indicus),拜氏不动杆菌(Acinetobacter beijerinckii) |
6 |
| 9 |
鞘氨醇单胞菌(Sphingomonas) |
血红鞘氨醇单胞菌(Sphingomonas sanguinis),少动鞘氨醇单胞菌(Sphingomonas paucimobilis),玫瑰黄鞘氨醇单胞菌(Sphingomonas roseiflava),维氏鞘氨醇单胞菌(Sphingomonas wittichii) |
4 |
| 10 |
罗尔斯通氏菌(Rolstonia) |
皮氏罗尔斯通氏菌(Rolstonia pickettii),茄科罗尔斯通氏菌(Rolstonia solanacearum) |
4 |
| 总计(total) |
|
|
72 |
|
表 1 本文用于菌株DNA特性序列鉴定的菌株信息
Tab.1 Bacterial isolate information using for phylogenetic analysis in this study |
分别采用VITEK生化鉴定和Ribotyping核糖体分型鉴定方法,对收集的菌株进行多相分类鉴定,确认菌株遗传背景(如表 2),结果显示:库克氏菌、微球菌、肠杆菌、肠球菌属中代表性菌株的多相分类鉴定结果一致;芽孢杆菌属中,枯草芽孢杆菌多相分类鉴定结果一致,苏云金芽孢杆菌、蜡样芽孢杆菌生化鉴定结果的分辨率低(lowdiscrimination,指鉴定结果无确定的菌株名称),巨大芽孢杆菌、地衣芽孢杆菌Ribotyping鉴定结果分辨率低(low probability,指仪器判读结果的菌株相似度 < 0.80或无确定的菌株名称);柠檬酸杆菌属中,弗氏柠檬酸杆菌多相分类鉴定结果一致,杨氏柠檬酸杆菌、布氏柠檬酸杆菌Ribotyping鉴定结果的分辨率低,穆氏柠檬酸杆菌无法确认菌株遗传背景;假单胞菌属中,施氏假单胞菌、铜绿假单胞菌、荧光假单胞菌多相分类鉴定结果一致,绿针假单胞菌、恶臭假单胞菌生化鉴定结果的分辨率低;不动杆菌属中,鲍曼不动杆菌、印度不动杆菌、拜氏不动杆菌生化鉴定结果的分辨率低,溶血不动杆菌Ribotyping鉴定结果的分辨率低;鞘氨醇单胞菌属中,少动鞘氨醇单胞菌Ribotyping鉴定结果的分辨率低,血红鞘氨醇单胞菌、玫瑰黄鞘氨醇单胞菌、维氏鞘氨醇单胞菌无法确认菌株遗传背景;罗尔斯通氏菌属中,皮氏罗尔斯通氏菌多相分类鉴定结果一致,茄科罗尔斯通氏菌生化鉴定结果的分辨率低。对于多相分类鉴定结果不一致或者无法确认菌株遗传背景的菌株,将测序获得的DNA特征序列与Genbank数据库进行比对,获取参比序列(RefSeq),间接佐证菌株的遗传背景。
表 2
Tab.2

表 2 多相分类鉴定结果统计
Tab.2 Bacterial multiphase identification in this study
序号
(number) |
编号
(number) |
菌株名称
(species) |
VITEK |
Ribotyping |
RefSeq |
| 1 |
ATCC 6633 |
枯草芽孢杆菌
(B. subtilis) |
枯草芽孢杆菌
(B. subtilis) |
枯草芽孢杆菌
(B. subtilis) |
/ |
| 2 |
CICC 22945 |
苏云金芽孢杆菌
(B.thuringiensis) |
低分辨率
(low discrimination) |
苏云金芽孢杆菌
(B.thuringiensis) |
NR 043403.1 |
| 3 |
ATCC 10876 |
蜡样芽孢杆菌
(B.cereus) |
低分辨率
(low discrimination) |
蜡样芽孢杆菌
(B.cereus) |
NR 074540.1 |
| 4 |
KW-05026 |
巨大芽孢杆菌
(B. megaterium) |
巨大芽孢杆菌
(B. megaterium) |
低置信度
(low probability) |
NR 116873.1 |
| 5 |
KW-05027 |
枯草芽孢杆菌
(B. subtilis) |
枯草芽孢杆菌
(B. subtilis) |
枯草芽孢杆菌
(B. subtilis) |
/ |
| 6 |
KW-05029 |
巨大芽孢杆菌
(B. megaterium) |
巨大芽孢杆菌
(B. megaterium) |
低置信度
(low probability) |
NR 116873.1 |
| 8 |
KW-05039 |
地衣芽孢杆菌
(B. licheniformis) |
地衣芽孢杆菌
(B. licheniformis) |
低置信度
(low probability) |
NR 074923.1 |
| 9 |
KW-05044 |
地衣芽孢杆菌
(B. licheniformis) |
地衣芽孢杆菌
(B. licheniformis) |
低置信度
(low probability) |
NR 074923.1 |
| 10 |
KW-05049 |
苏云金芽孢杆菌
(B.thuringiensis) |
低分辨率
(low discrimination) |
苏云金芽孢杆菌
(B.thuringiensis) |
NR 043403.1 |
| 11 |
KW-05050 |
蜡样芽孢杆菌
(B.cereus) |
低分辨率
(low discrimination) |
蜡样芽孢杆菌
(B.cereus) |
NR 074540.1 |
| 12 |
CMCC28001 |
嗜根库克氏菌
(K. rhizophila) |
嗜根库克氏菌
(K. rhizophila) |
嗜根库克氏菌
(K. rhizophila) |
/ |
| 13 |
ATCC 49321 |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
/ |
| 14 |
ATCC 27570 |
克氏库克氏菌
(K. kristinae) |
克氏库克氏菌
(K. kristinae) |
克氏库克氏菌
(K. kristinae) |
/ |
| 15 |
ATCC 15306 |
变异库克氏菌
(K. varians) |
变异库克氏菌
(K. varians) |
变异库克氏菌
(K. varians) |
/ |
| 16 |
KW-07023 |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
/ |
| 17 |
KW-07024 |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
玫瑰库克氏菌
(K. rosea) |
/ |
| 18 |
KW-07025 |
嗜根库克氏菌
(K. rhizophila) |
嗜根库克氏菌
(K. rhizophila) |
嗜根库克氏菌
(K. rhizophila) |
/ |
| 19 |
CICC 10680 |
藤黄微球菌
(M. luteus) |
藤黄/里拉微球菌
(M. luteus / lylae) |
藤黄微球菌
(M. luteus) |
/ |
| 20 |
ATCC 4698 |
藤黄微球菌
(M. luteus) |
藤黄/里拉微球菌
(M. luteus / lylae) |
藤黄微球菌
(M. luteus) |
/ |
| 21 |
KW-06031 |
藤黄微球菌
(M. luteus) |
藤黄/里拉微球菌
(M. luteus / lylae) |
藤黄微球菌
(M. luteus) |
/ |
| 22 |
KW-06032 |
藤黄微球菌
(M. luteus) |
藤黄/里拉微球菌
(M. luteus / lylae) |
藤黄微球菌
(M. luteus) |
/ |
| 23 |
ATCC43864 |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
/ |
| 24 |
CICC24146 |
杨氏柠檬酸杆菌
(C. youngae) |
杨氏柠檬酸杆菌
(C. youngae) |
柠檬酸杆菌属
(C. species) |
NR 041527.1 |
| 25 |
CICC21597 |
布氏柠檬酸杆菌
(C. braakii) |
布氏柠檬酸杆菌
(C. braakii) |
弗氏柠檬酸杆菌
(C. freundii) |
NR 028687.1 |
| 26 |
CICC24136 |
穆氏柠檬酸杆菌
(C. murliniae) |
低分辨率
(low discrimination) |
低置信度
(low probability) |
NR 028688.1 |
| 27 |
CICC21596 |
杨氏柠檬酸杆菌
(C. youngae) |
杨氏柠檬酸杆菌
(C. youngae) |
低置信度
(low probability) |
NR 041527.1 |
| 28 |
KW-12011 |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
/ |
| 29 |
KW-12012 |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
弗氏柠檬酸杆菌
(C. freundii) |
/ |
| 30 |
ATCC 13048 |
产气肠杆菌
(E. aerogenes) |
产气肠杆菌
(E. aerogenes) |
产气肠杆菌
(E. aerogenes) |
/ |
| 31 |
CICC 10293 |
产气肠杆菌
(E. aerogenes) |
产气肠杆菌
(E. aerogenes) |
产气肠杆菌
(E. aerogenes) |
/ |
| 32 |
ATCC 29544 |
阪崎肠杆菌
(E. sakazakii) |
阪崎肠杆菌
(E. sakazakii) |
阪崎肠杆菌
(E. sakazakii) |
/ |
| 33 |
CICC 21560 |
阪崎肠杆菌
(E. sakazakii) |
阪崎肠杆菌
(E. sakazakii) |
阪崎肠杆菌
(E. sakazakii) |
/ |
| 34 |
KW-11011 |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
/ |
| 35 |
KW-11012 |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
/ |
| 36 |
KW-11013 |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
阴沟肠杆菌
(E. cloacae) |
/ |
| 37 |
KW-11014 |
生癌肠杆菌
(E. cancerogenus) |
生癌肠杆菌
(E. cancerogenus) |
生癌肠杆菌
(E. cancerogenus) |
/ |
| 38 |
CICC 10396 |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
/ |
| 39 |
CICC 10433 |
铅黄肠球菌
(E. casseliflavus) |
铅黄肠球菌
(E. casseliflavus) |
铅黄肠球菌
(E. casseliflavus) |
/ |
| 40 |
CICC 24240 |
鸡肠球菌
(E. gallinarum) |
鸡肠球菌
(E. gallinarum) |
鸡肠球菌
(E. gallinarum) |
/ |
| 41 |
CICC 23228 |
海氏肠球菌
(E. hirae) |
海氏肠球菌
(E. hirae) |
海氏肠球菌
(E. hirae) |
/ |
| 42 |
CICC 21607 |
盲肠肠球菌
(E. cecorum) |
盲肠肠球菌
(E. cecorum) |
盲肠肠球菌
(E. cecorum) |
/ |
| 43 |
CICC 20089 |
屎肠球菌
(E. faecium) |
屎肠球菌
(E. faecium) |
屎肠球菌
(E. faecium) |
/ |
| 44 |
CICC 20177 |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
/ |
| 45 |
KW-09021 |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
/ |
| 46 |
KW-09022 |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
耐久肠球菌
(E. durans) |
/ |
| 47 |
KW-09023 |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
/ |
| 48 |
KW-09024 |
铅黄肠球菌
(E. casseliflavus) |
铅黄肠球菌
(E. casseliflavus) |
铅黄肠球菌
(E. casseliflavus) |
/ |
| 49 |
KW-09026 |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
粪肠球菌
(E. faecalis) |
/ |
| 50 |
CICC 10402 |
施氏假单胞菌
(P. stutzeri) |
施氏假单胞菌
(P. stutzeri) |
施氏假单胞菌
(P. stutzeri) |
/ |
| 51 |
CICC 20676 |
绿针假单胞菌
(P. chlororaphis) |
低分辨率
(Low discrimination) |
荧光假单胞菌
(P. fluorescens) |
NR 042939.1 |
| 52 |
CICC 10216 |
绿针假单胞菌
(P. chlororaphis) |
铜绿假单胞菌
(P. aeruginosa) |
绿针假单胞菌
(P. chlororaphis) |
NR 042939.1 |
| 53 |
CICC 10368 |
恶臭假单胞菌
(P. putida) |
低分辨率
(Low discrimination) |
恶臭假单胞菌
(P. putida) |
NR 114479.1 |
| 54 |
CMCC10104 |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
/ |
| 55 |
GIM1.776 |
荧光假单胞菌
(P. fluorescens) |
荧光假单胞菌
(P. fluorescens) |
荧光假单胞菌
(P. fluorescens) |
/ |
| 56 |
KW-04021 |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
/ |
| 57 |
KW-04022 |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
铜绿假单胞菌
(P. aeruginosa) |
/ |
| 58 |
KW-04040 |
荧光假单胞菌
(P. fluorescens) |
荧光假单胞菌
(P. fluorescens) |
荧光假单胞菌
(P. fluorescens) |
/ |
| 59 |
CICC 10254 |
鲍曼不动杆菌
(A. baumannii) |
低分辨率
(low discrimination) |
鲍曼不动杆菌
(A. baumannii) |
NR 117620.1 |
| 60 |
CICC 22018 |
溶血不动杆菌
(A. haemolytius) |
溶血不动杆菌
(A. haemolytius) |
低分辨率
(low discrimination) |
X 81662.1 |
| 61 |
CICC 23878 |
印度不动杆菌
(A. indicus) |
低分辨率
(low discrimination) |
低分辨率
(low discrimination) |
NR 133954.1 |
| 62 |
CICC 23877 |
拜氏不动杆菌
(A. beijerinckii) |
低分辨率
(low discrimination) |
低分辨率
(low discrimination) |
AB 859734.1 |
| 63 |
KW-14012 |
鲍曼不动杆菌
(A. baumannii) |
鲍曼不动杆菌
(A. baumannii) |
鲍曼不动杆菌
(A. baumannii) |
|
| 64 |
KW-14014 |
鲍曼不动杆菌
(A. baumannii) |
鲍曼不动杆菌
(A. baumannii) |
鲍曼不动杆菌
(A. baumannii) |
/ |
| 65 |
CICC 10892 |
血红鞘氨醇单胞菌
(S. sanguinis) |
血红鞘氨醇单胞菌
(S. sanguinis) |
鞘氨醇单胞菌属
(S. species) |
NR 043427.1 |
| 66 |
CICC 23965 |
少动鞘氨醇单胞菌
(S. paucimobilis) |
少动鞘氨醇单胞菌
(S. paucimobilis) |
鞘氨醇单胞菌属
(S. species) |
NR 115523.1 |
| 67 |
CICC23900 |
玫瑰黄鞘氨醇单胞菌
(S. roseiflava) |
少动鞘氨醇单胞菌
(S. paucimobilis) |
低置信度
(low probability) |
NR 043426.1 |
| 68 |
CICC10426 |
维氏鞘氨醇单胞菌
(S. wittichii) |
低分辨率
(low discrimination) |
低置信度
(low probability) |
NR 074268.1 |
| 69 |
CICC 10679 |
皮氏罗尔斯通氏菌
(R. pickettii) |
皮氏罗尔斯通氏菌
(R. pickettii) |
皮氏罗尔斯通氏菌
(R. pickettii) |
/ |
| 70 |
CICC 24306 |
茄科罗尔斯通氏菌
(R. solanacearum) |
低分辨率
(low discrimination) |
茄科罗尔斯通氏菌
(R. solanacearum) |
NR 044040.1 |
| 71 |
CICC 24305 |
茄科罗尔斯通氏菌
(R. solanacearum) |
低分辨率
(low discrimination) |
茄科罗尔斯通氏菌
(R. solanacearum) |
NR 044040.1 |
| 72 |
BNCC 186282 |
皮氏罗尔斯通氏菌
(R. pickettii) |
皮氏罗尔斯通氏菌
(R. pickettii) |
皮氏罗尔斯通氏菌
(R. pickettii) |
/ |
|
表 2 多相分类鉴定结果统计
Tab.2 Bacterial multiphase identification in this study |
2.2 菌株DNA特征序列鉴定与聚类分析
将遗传背景确认的菌株进行核酸测序鉴定,获得经校核的高质量核酸序列,将菌株鉴定的DNA特征序列生成fasta文件,导入Mega 5.0序列分析软件,以Bootstrap Method(1000)、Kimura 2-Parameter Model、Uniform Rates、Pairwise Deletion的统计学计算方式,构建N-J进化树(如图 1)。聚类分析结果显示:菌株鉴定的DNA特征序列在10个属41个种的水平,均具有特异性。芽孢杆菌属中的枯草芽孢杆菌、苏云金芽孢杆菌,蜡样芽孢杆菌、巨大芽孢杆菌、地衣芽孢杆菌,库克氏菌属中的嗜根库克氏菌、玫瑰库克氏菌、克氏库克氏菌、变异库克氏菌,微球菌属中的藤黄微球菌,柠檬酸杆菌属中的弗氏柠檬酸杆菌、杨氏柠檬酸杆菌、布氏柠檬酸杆菌、穆氏柠檬酸杆菌,肠杆菌属中的产气肠杆菌、阪崎肠杆菌、阴沟肠杆菌、生癌肠杆菌,肠球菌属中的粪肠球菌、铅黄肠球菌、鸡肠球菌、海氏肠球菌、屎肠球菌、盲肠肠球菌、耐久肠球菌,假单胞菌属中的铜绿假单胞菌、施氏假单胞菌、绿针假单胞菌、荧光假单胞菌、恶臭假单胞菌,不动杆菌属中的鲍曼不动杆菌、溶血不动杆菌、印度不动杆菌、拜氏不动杆菌鞘氨醇单胞菌属中的血红鞘氨醇单胞菌、少动鞘氨醇单胞菌、玫瑰黄鞘氨醇单胞菌,维氏鞘氨醇单胞菌以及罗尔斯通氏菌属中的皮氏罗尔斯通氏菌、茄科罗尔斯通氏菌的DNA特征序列均各自聚类为一个分支,可以准确鉴定到“种”的水平,并为分型溯源提供参考。
3 讨论
药品微生物质量控制中的环境监控、偏差调查与溯源分析等多个关键环节,对于菌株鉴定的要求均需达到“种”甚至“菌株”的水平,传统的形态学、显微镜检、理化分析及生化鉴定等方法已无法满足药品微生物质量控制的需求[11],采用基因型水平鉴定技术,开展微生物鉴定的标准化研究是药品质量控制发展的必然趋势,《中华人民共和国药典》2020年版首次收载“细菌DNA特征序列鉴定法”(通则1021),是该专业领域发展趋势的重要体现。
3.1 多相分类鉴定
微生物遗传背景的确认,是开展菌株DNA特征序列鉴定及建立标准核酸序列的前提。本文采用生化鉴定、核糖体分型及借助Genbank数据库参比序列相结合的方法,对于常见的10个“属”41个“种”72个菌株进行了多相分类鉴定,其中,库克氏菌属、微球菌属、肠杆菌属、肠球菌属的多相分类鉴定结果的一致性较好;芽孢杆菌属、柠檬酸杆菌属、假单胞菌属、不动杆菌属、鞘氨醇单胞菌属、罗尔斯通氏菌属均不同程度出现了生化鉴定与核糖体分型鉴定结果不一致的情况,其中穆氏柠檬酸杆菌、血红鞘氨醇单胞菌、玫瑰黄鞘氨醇单胞菌、维氏鞘氨醇单胞菌等还出现了无法确认遗传信息的情况。结果表明:(1)VITEK生化鉴定、Ribotyping核糖体分型鉴定等单一鉴定方法存在局限性,需采用表型与基因型相结合的技术手段,综合判断菌株遗传背景[12-13];(2)VITEK、RiboPrinter等自动化鉴定系统的数据库多来源于医学样本的测试验证,对于药品质量控制中常见细菌鉴定数据收载的代表性、覆盖面均不足;(3)采用DNA特征序列判定菌株的鉴定结果,客观、准确、稳定,开放性强,是菌株核酸水平鉴定的发展趋势。
3.2 细菌DNA特征序列鉴定水平
药品微生物质量控制中的常见细菌污染物主要来源于非无菌制剂生产工艺、生产环境、制药用水系统等,以革兰阳性球菌和革兰阴性杆菌最为常见,占比超过80%。本文针对性地选择了10个属41个种的细菌污染物,经核酸序列比对与聚类分析,典型细菌鉴定的DNA特征序列可以达到“种”的水平,不仅可以满足药典规定,作为药典微生物检查法的有效补充;而且也可以为种水平,甚至是菌株水平的鉴定与分型提供参考。在此基础上建立菌株鉴定信息库;针对性开展消毒、灭菌挑战性试验;抑菌剂抑菌效力验证等可以进一步提升药品微生物质量控制的整体能力和水平。
3.3 细菌DNA特征序列数据库
基于核酸序列比对分析的细菌DNA特征序列鉴定,需要建立标准核酸序列数据库[14-15]。实验室将通过扩大验证,进一步丰富细菌DNA特征序列的适用范围和数据量,为细菌核酸序列水平的鉴定结果提供准确的判定依据,为药典新增通则的实施提供有力支撑。
参考文献
| [1] |
PAUL DV, GARRITY GM, JONES D, et al. Bergey's Manual of Systematic Bacteriology.VolⅡ[M]. Berlin: Springer Berlin Heidelberg, 2011: 587.
|
| [2] |
USP 41-NF 36[S].2019: 1419
|
| [3] |
中华人民共和国药典2015年版.四部[S].2015: 393
ChP 2015.Vol Ⅳ[S].2015: 393
|
| [4] |
FERGUSON A, PATEL A, BAIRD RM. A comparison of two incubation temperatures for the isolation of gram-negative contaminants from raw materials and non-sterile pharmaceuticals[J]. J Clin Pharm Ther, 1987, 12(4): 249. DOI:10.1111/j.1365-2710.1987.tb00534.x |
| [5] |
HUGHES KA, CORNWALL J, THEIS JC, et al. Bacterial contamination of unused, disposable non-sterile gloves on a hospital orthopaedic ward[J]. Australas Med J, 2013, 30(6): 331. |
| [6] |
COBLEY M, ATKINS M, JONES PL. Environmental contamination during tracheal suction.A comparison of disposable conventional catheters with a multiple-use closed system device[J]. Anaesthesia, 1991, 46(11): 957. DOI:10.1111/j.1365-2044.1991.tb09858.x |
| [7] |
KREMER T, PATEL R. Correlation between environmental monitoring and product bioburden[J]. Biomed Instrum Technol, 2019, 53(s2): 32. DOI:10.2345/0899-8205-53.s2.32 |
| [8] |
冯震, 李芳, 宋明辉, 等. 16S核糖体RNA基因片段分析及在药品葡萄球菌污染控制应用中的探讨[J]. 药物分析杂志, 2018, 38(4): 690. FENG Z, LI F, SONG MH, et al. A detailed analysis of 16S ribosomal RNA gene segments for identification of Staphylococcus contaminants in pharmaceutical quality control[J]. Chin J Pharm Anal, 2018, 38(4): 690. |
| [9] |
TOYAMA T, KURODA M, OGATA Y, et al. Enhanced biomass production of duckweeds by inoculating a plant growth-promoting bacterium, Acinetobacter calcoaceticus P23, in sterile medium and non-sterile environmental waters[J]. Water Sci Technol, 2017, 76(6): 1418. DOI:10.2166/wst.2017.296 |
| [10] |
MORENO-FORERO SK, van der MEER JR. Genome-wide analysis of Sphingomonas wittichii RW1 behaviour during inoculation and growth in contaminated sand[J]. ISME J, 2015, 9(1): 150. DOI:10.1038/ismej.2014.101 |
| [11] |
洪小栩, 冯震, 许华玉, 等. 分子生物学技术用于药品质量控制国家标准核酸数据库的建设规划研究[J]. 中国医药工业杂志, 2016, 47(5): 651. HONG XX, FENG Z, XU HY, et al. Program of national standard nucleotide sequence database used in quality control of pharmaceutical products by molecular biology technology[J]. Chin J Pharm, 2016, 47(5): 651. |
| [12] |
KIM M, HS O, PARK SC, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. Int J Syst Evol Microbiol, 2014, 64(2): 346. |
| [13] |
PETTI CA, POLAGE CR, SCHRECKENBERGER P. The role of 16S rRNA gene sequencing in identification of microorganisms misidentified by conventional methods[J]. J Clin Microbiol, 2005, 43(12): 6123. DOI:10.1128/JCM.43.12.6123-6125.2005 |
| [14] |
NILSSON RH, RYBERG M, KRISTIANSSON E, et al. Taxonomic reliability of DNA sequences in public sequence databases:a fungal perspective[J]. PLoS One, 2006, 1(1): e59. DOI:10.1371/journal.pone.0000059 |
| [15] | |

 2019, Vol. 39
2019, Vol. 39 
