2. 中国食品药品检定研究院, 北京 100050
2. National Institutes for Food and Drug Control, Beijing 100050, China
近交系小鼠对生物学研究的准确性、敏感性和重复性有着重要的作用,使用近交系小鼠作为药物研究工具具有实验重复性好,使用数量少,试验周期短的优点,是研究新药物的重要实验材料,在药物临床前筛选和疾病发病机制等方面有着十分重要的作用。最常使用的小鼠种系有C57BL和BALB/c小鼠,C57BL小鼠细分出C57BL/6和C57BL/10 2个亚系。C57BL/6是第1个完成全基因组测序的小鼠品系,主要作为生理学与病理学实验动物模型和构建转基因动物模型[1-6]。有研究发现常用的C57Bl/6小鼠品系中发生基因重复突变,会破坏能够调节免疫细胞激活和迁移的Dock2基因的功能,导致免疫缺陷,这可能影响这些实验动物的研究结果[7]。在前期开展的试验中发现,C57BL/6和C57BL/10小鼠在接种乙肝疫苗后表现出不同的免疫应答,C57BL/10小鼠表现为免疫弱应答。因此研究C57BL/6和C57BL/10之间的基因差异对选择药物体内检测实验动物具有重要意义。
1 材料与方法 1.1 动物收集和DNA提取本实验在中国食品药品检定研究院动物伦理委员会监督下执行。由于雌鼠产生抗体效价比雄鼠高,因此本试验选择雌性小鼠。体质量约20 g的4周龄C57BL/10雌性小鼠3只由中国食品药品检定研究院实验动物资源研究所饲养。每只小鼠处死后取尾组织,立即放入液氮中保存直至使用。采用Trizol®试剂(Invitrogen,USA)并按照说明书中步骤从组织中分离小鼠DNA。1%琼脂糖电泳分析DNA的纯度和完整性。采用Qubit® DNA检测试剂盒和Qubit® 2.0荧光光度计(Life Technologies,CA,USA)测定DNA浓度。采用NanoPhotometer®分光光度计(IMPLEN,CA,USA)检测DNA纯度(A260 nm与A280 nm的比值)。
1.2 文库构建及库检每个样品提取1.5 μg DNA作为DNA样品制备的初始材料,测序文库构建采用Truseq Nano DNA HT样品制备试剂盒(Illumina USA),样品用Covaris超声波破碎仪随机打断成长度为350 bp的片段,经末端修复,加A尾,加测序接头,经AMPure XP系统纯化、PCR扩增等步骤完成整个文库制备。文库构建完成后,先使用Qubit 2.0荧光光度计进行初步定量,稀释文库至1 ng·μL-1,随后使用Agilent 2100生物分析仪对文库的insert size进行检测,insert size符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度 > 2 nmol·L-1),以保证文库质量。
1.3 上机测序库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后进行Illumina HiSeq 2500测序,通过碱基识别技术(Illumina pipeline CASAVA v1.8.2)将测得的原始图像数据转变为碱基序列数据。
1.4 信息分析的主要步骤高通量测序(如Illumina HiSeqTM2500/MiseqTM)得到的原始图像数据文件经CASAVA碱基识别(Base Calling)分析转化为原始测序序列(Sequenced Reads),即Raw Data或Raw Reads,结果以FASTQ(简称为fq)文件格式存储,其中包含测序序列(Reads)的序列信息以及其对应的测序质量信息。测序得到的原始测序序列或者Raw Reads,里面含有带接头的、低质量的Reads。为了保证信息分析质量,必须对Raw Reads过滤,得到Clean Reads,后续分析都基于Clean Reads。数据处理的步骤如下:(1)去除带接头(Adapter)的Reads Pair;(2)当单端测序Read中含有的N的含量超过该条Read长度比例的10%时,需要去除此对Paired Reads;(3)当单端测序Read中含有的低质量(Q≤5)碱基数超过该条Read长度比例的50%时,需要去除此对Paired Reads。对下机得到的Raw Data进行质控得到Clean Data;将Clean Data比对到参考基因组上,有效测序数据通过BWA软件(参数:mem-t 4-k 32-M -R)比对到参考基因组,比对结果经SAMTOOLS去除重复(参数:rmdup),参考基因组大小为3 783 309 620 bp,所有样本的比对率在99.18%~99.18%之间,对参考基因组(排除N区)的平均覆盖深度在28.51X~28.51X之间,1X覆盖度(至少有1个碱基的覆盖)在96.59%以上。根据比对结果,进行SNP和InDel的检测及注释,为了降低SNP检测的错误率,选用如下标准进行过滤:(1)SNP的Reads支持数不低于4;(2)SNP的质量值不低于20。InDel是指基因组中小片段的插入和缺失序列。利用SAMTOOLs检测长度小于50 bp的小片段插入与缺失(InDel),然后用ANNOVAR软件对检测出的InDel进行注释。为进行大范围基因表达分析,采用GOseq R package将显著表达差异基因分配到不同的功能类别。采用GO分类(http://www.geneontology.org)对差异表达基因的功能进行分类,与不同功能分类相关的不同表达聚类基因清晰地指示了分子和细胞事件[7-8]。
2 结果与讨论本次测序共产生Raw Data 80.871G,过滤后的Clean Data 79.322G,各样本的Raw Data在80 871.429M~80 871.429M之间,GC含量在42.56%~42.56%之间。测序质量合格,GC分布正常,建库测序成功。C57BL/10原始数据产量为80 871 429 000 bp,过滤之后的有效数据量为79 321 980 750 bp,过滤后获得有效数据量与原始数据量的比值为98.06 bp,碱基错误率0.04%,Phred[注:Phred=-10 log10(e)]数值大于20的碱基占总体碱基的百分比为95.38%,Phred数值大于30的碱基占总体碱基的百分比为91.48%,碱基G和C的数量总和占总的碱基数量的百分比为42.56%。4周龄雌性C57BL/6小鼠参考基因组下载地址:ftp://ftp.ensembl.org/pub/release-70/fasta/mus_musculus/dna/Mus_musculus.GRCm38.70.dna.toplevel.fa.gz.。C57BL/6参考基因组组装的序列总数为75,基因组组装结果总长度3 783 309 620,碱基G和C的含量为41.67%,组装结果中N所占的比例为29.80%,N50长度为145 439 975,N90长度为91 736 668(注:N50长度,表示组装结果中有一半的序列长度大于该值。N90长度,表示组装结果中有90%的序列长度大于该值)。
基因功能分类如图 1所示,在生物过程分类中列出了与刺激反应、催化活性调节、分子功能调节等相关差异基因;在分子功能分类中列出了与蛋白二硫化物还原酶、催化活性、磷酸化传感器激酶活性等相关差异基因。将变异检测得到的SNP位点,去掉同义突变之后,利用NCBI中BioMart进行功能注释。提取出免疫相关的转录本ID、基因ID和功能注释的信息,结果见表 1。
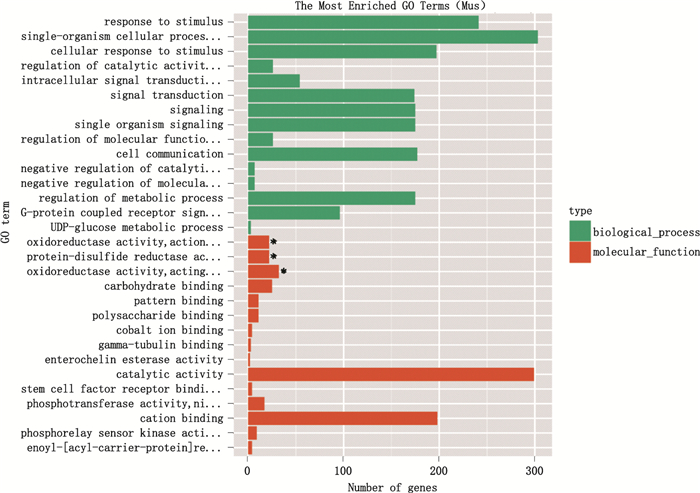
|
结果展示为2类:生物过程(BP)和分子功能(MF)[the results are summarized in two main categories:biological process(BP)and molecular function(MF)] X轴代表每类基因功能的基因数量,左边Y轴代表每类基因功能中的特定归类,“*”为富集的GO term(the X-axis indicates the number of genes in a category,the left Y-axis indicates the specific category of genes in that main category,the“*”indicates enrichment of GO term) 图 1 基因功能分类 Fig.1 Histogram of gene ontology classification |
|
|
表 1 与小鼠免疫应答相关的差异表达基因 Tab.1 Summary of differentially expressed genes involved in mice immune response |
本研究阐述了使用全基因组技术分析不同亚系小鼠的基因差异,发现了近交系小鼠C57BL/6和C57BL/10组存在显著差异基因。另外,从生物过程和分子功能两方面对差异基因进行GO分类。在与生物过程分类中发现与单组织过程类别相关的差异基因数量最多,在分子功能方面则是与催化活性相关的差异基因较多。筛选了一系列与小鼠免疫应答相关的差异基因,其中Timd 2关联蛋白可能是SEMA4A受体,后者参与调节T细胞活性。Timd 2蛋白与SEMA4A的相互作用增强了T细胞的激活反应[17]。Ighv 1-69关联蛋白与抗原结合、免疫球蛋白受体结合有关[18]。后续研究将对发现的4个差异性基因利用qPCR方法进行mRNA表达,以进一步确证这种差异。本研究为科学建立小鼠体内药物免疫应答评价模型进一步积累数据,完善了小鼠基因组信息。这些发现首次在全基因组中研究不同品系C57BL实验小鼠的基因差异,为建立生物制品体内评价实验动物模型和标准化提供了新的线索和技术手段。
| [1] |
DEACON RMJ, THOMAS CL, RAWLINS JNP, et al. A comparison of the behavior of C57BL/6 and C57BL/10 mice[J]. Behav Brain Res, 2007, 179(2): 239. DOI:10.1016/j.bbr.2007.02.009 |
| [2] |
MEKADA K, ABE K, MURAKAMI A, et al. Genetic differences among C57BL/6 strains[J]. Exp Anim, 2009, 58(2): 141. DOI:10.1538/expanim.58.141 |
| [3] |
MCCLIVE PJ, HUANG D, MORAHAN G. C57BL/6 and C57BL/10 inbred mouse strains differ at multiple loci on chromosome 4[J]. Immunogenetics, 1994, 39(4): 286. |
| [4] |
APPELBERG R, LEAL IS, PAIS TF, et al. Differences in resistance of C57BL/6 and C57BL/10 mice to infection by Mycobacterium avium are independent of gamma interferon[J]. Infect Immun, 2000, 68(1): 19. DOI:10.1128/IAI.68.1.19-23.2000 |
| [5] |
SLINGSBY JH, HOGARTH MB, SIMPSON E, et al. New microsatellite polymorphisms identified between C57BL/6, C57BL/10, and C57BL/KsJ inbred mouse strains[J]. Immunogenetics, 1995, 43(1): 72. |
| [6] |
LIPOFF DM, BHAMBRI A, FOKAS GJ, et al. Neocortical molecular layer heterotopia in substrains of C57BL/6 and C57BL/10 mice[J]. Brain Res, 2011, 1391: 36. DOI:10.1016/j.brainres.2011.03.026 |
| [7] |
MAHAJAN VS, DEMISSIE E, MATTOO H, et al. Striking immune phenotypes in gene-targeted mice are driven by a copy-number variant originating from a commercially available C57BL/6 strain[J]. Cell Rep, 2016, 15(9): 1901. DOI:10.1016/j.celrep.2016.04.080 |
| [8] |
ZHANG SB, TANG QR. Predicting protein subcellular localization based on information content of gene ontology terms[J]. Comput Biol Chem, 2016, 65: 1. DOI:10.1016/j.compbiolchem.2016.09.009 |
| [9] |
GAUDET P.The gene ontology[M]//Encyclopedia of Bioinformatics and Computational Biology.Vol 2.Amsterdam: Elsevier Inc., 2019: 1
|
| [10] |
LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754. DOI:10.1093/bioinformatics/btp324 |
| [11] |
LI H, HANDSAKER B, WYSOKER A, et al. The Sequence Alignment/Map format and SAM tools[J]. Bioinformatics, 2009, 25(16): 2078. DOI:10.1093/bioinformatics/btp352 |
| [12] |
CHEN K, WALLIS JW, MCLELLAN MD, et al. BreakDancer:an algorithm for high-resolution mapping of genomic structural variation[J]. Nat Methods, 2009, 6(9): 677. DOI:10.1038/nmeth.1363 |
| [13] |
ABYZOV A, URBAN AE, SNYDER M, et al. CNVnator:an approach to discover, genotype, and characterize typical and atypical CNVs from family and population genome sequencing[J]. Genome Res, 2011, 21(6): 974. DOI:10.1101/gr.114876.110 |
| [14] |
WANG K, LI M, HAKONARSON H. ANNOVAR:functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Res, 2010, 38(16): e164. DOI:10.1093/nar/gkq603 |
| [15] |
KRZYWINSKI M, SCHEIN J, BIROL I, et al. Circos:an information aesthetic for comparative genomics[J]. Genome Res, 2009, 19(9): 1639. DOI:10.1101/gr.092759.109 |
| [16] |
FIUME M, WILLIAMS V, BROOK A, et al. Savant:genome browser for high-throughput sequencing data[J]. Bioinformatics, 2010, 26(16): 1938. DOI:10.1093/bioinformatics/btq332 |
| [17] |
FIUME M, SMITH E J M, BROOK A, et al. Savant Genome Browser 2:visualization and analysis for population-scale genomics[J]. Nucleic Acids Res, 2012, 40(Web Server issue): W615. |
| [18] |
KUMANOGOH A, MARUKAWA S, SUZUKI K, et al. Class Ⅳ semaphorin Sema4A enhances T-cell activation and interacts with Tim-2[J]. Nature, 2002, 419(6907): 629. DOI:10.1038/nature01037 |
| [19] |
GAUDET P, LIVSTONE MS, LEWIS SE, et al. Phylogenetic-based propagation of functional annotations within the gene ontology consortium[J]. Brief Bioinform, 2011, 12(5): 449. DOI:10.1093/bib/bbr042 |
 2019, Vol. 39
2019, Vol. 39 
