洋葱伯克霍尔德菌复合体(Burkholderia cepacia complex,BCC)是一类表型近似,但在基因型上存在一定差异,环境中普遍存在的革兰氏阴性菌的总称[1]。Yabuuchi等[2]于1992年将假单胞菌属中隶属于系统发育系rRNA Ⅱ型的一些菌株重新分类为新的属,即伯克霍尔德菌属。20世纪90年代末,Vandamme等利用多相分类学,将伯克霍尔德菌复合体划分出至少5个不同的种(Burkholderia cepacia,Burkholderia multivorans,Burkholderiaceno cepacia,Burkholderia stabilis,Burkholderia vietnamiensis),并在其分类中引入基因型(Genomovar)的概念,借以指示在表型上无法区别,但基因水平存在差异的菌种[3-4]。
由于在表型上的相似性,BCC无法通过传统的生理生化实验等表型特征进行鉴别区分,对于它们的分类学研究多基于基因水平,而BCC种间98%~100%的16S rRNA同源性为使得作为细菌分类鉴别“金标准”的16S rRNA序列分析方法在BCC的分类学研究上显得捉襟见肘[5]。在BCC分类学的研究进程中,看家基因recA发挥了重要作用,recA基因编码蛋白参与DNA重组修复,在细菌基因组中普遍存在,高度保守,其序列分析多应用于遗传关系相近的物种[6-7]。BCC种间recA基因的同源性则为94%~95%,而同一基因型(Genomovar)内部的同源性则达到98%~99%[8],弥补了16S rRNA对于BCC种间区分度不足的缺陷。基于recA基因的多态性分析,结合多相分类方法,更多的BCC新种被发现(Burkholderia dolosa,Burkholderia ambifaria,Burkholderia anthina,Burkholderia pyrrocinia)[5],而随着Burkholderia latens,Burkholderia diffusa,Burkholderia arboris,Burkholderia seminalis,Burkholderia metallica以及Burkholderia ubonensis[9]的命名,recA基因的同源性分析及限制性片段长度多态性(RFLP)已成为BCC分类的主要研究手段。随着分子分型技术的进一步发展,近年来,对于BCC的分类研究拓展至其他看家基因,recA与多个保守基因联用的多位点序列分型(MLST)方法使得新的BCC成员得以被发现[10],除了较早定义的基因型Ⅰ~Ⅸ,被命名的BCC已超过20种[11-17]。
虽然16S rRNA在BCC的分类鉴别中能力有限,但20世纪初,有研究者已开始研究核糖体分型技术对BCC的分型。其中,自动核糖体分型具有重复性好,耗时短等独特优势。2000年,Risse等[18]通过对临床分离伯克霍尔德菌进行核糖体分型分析,发现其对Burkholderia gladioli,Burkholderia multivorans(GenomovarⅡ),及Burkholderia dolosa(Genomovar Ⅵ)的有效区分;2004年,该团队将法国临床分离的BCC菌株进行2种酶切(EcoRⅠ及PvuⅡ)获得的核糖体图谱进行分析,表明自动核糖体分型可对Burkholderia multivorans(GenomovarⅡ),Burkholderia cenocepacia(GenomovarⅢ),Burkholderia stabilis(Genomovar Ⅳ),Burkholderia vietnamiensis(GenomovarⅤ)及Burkholderia pyrrocinia(Genomovar Ⅸ)进行种间区分[19]。这些研究表明核糖体自动分型在BCC鉴别分析及溯源中的应用前景。
本文研究对象为某外用样品中的分离菌株,来源批次不同,以及临床样本分离菌株。考虑到待分析样本量、时间等因素,在确认待检菌株为细菌的基础上,采用自动化核糖体分型技术对大量分离株进行准确的初筛分类,进而选取不同核糖体型的代表菌株,进行生化及16S rRNA序列分析,鉴别分析结果均指向洋葱伯克霍尔德菌复合体。如上所述,生化及16S rRNA序列分析对于BCC种间的区分能力有限,因此通过补充recA基因序列分析,将8株代表菌株准确区分至种水平,结合核糖体分型结果,对此次调查中的不同来源菌株完成了准确的鉴别及溯源分析,为污染事件的原因调查提供可靠依据。
1 材料及方法 1.1 菌株产品编号1来源(某外用制剂批号1)菌株10株,分别为1-1、1-2、1-3、1-4、1-5、1-7、1-8、1-9、1-10、1-11;产品编号2来源(某外用制剂批号2)菌株8株,分别为2-1、2-2、2-3、2-4、2-5、2-6、2-7、2-8,样本来源3(临床)中分离得到菌株5株,分别为y-1、y-2、y-3、y-4、y-6。用于进化树分析的参考基因序列来自NCBI(https://www.ncbi.nlm.nih.gov/)。
1.2 核糖体分型(Ribotyping)采用核糖体自动分型系统(Riboprinter,Hygiena,USA),按照仪器操作规范,使用EcoRⅠ酶切,大肠埃希菌(Escherichia coli)核糖体基因作为探针,对上述样本中分离的所有菌株进行自动分型。条带分析软件为BioNumerics,基于皮尔森相关系数(Pearson correlation coefficient),采用非加权配对算术平均法(unweighted pair group method with arithmetic averages,UPGMA)进行聚类分析,优化系数1.2%。
1.3 生化鉴定采用Biolog Gen Ⅲ Microstation自动微生物鉴定系统,按照仪器操作规范,对上述菌株进行生化鉴定。
1.4 16S rRNA及recA基因扩增上述菌株均划线于胰酪大豆胨固体培养基(TSA)上,置于32.5 ℃培养24 h。使用细菌基因组DNA提取试剂盒(Tiangen,DP302)提取基因组,进行目标序列扩增。16S rRNA引物27f:AGA GTT TGA TCC TGG CTC AG;1492r:TAC GGC TAC CTT GTT ACG ACT T。扩增条件:95 ℃预变性4 min;95 ℃变性30 s,50 ℃引物退火45 s,72 ℃延伸1 min 30 s,35个循环;72 ℃延伸5 min。recA引物BCR1:TGA CCG CCG AGA AGA GCA A;BCR2:CTC TTC TTC GTC CAT CGC CTC[8]。扩增条件:94 ℃预变性3 min;94 ℃变性30 s,56 ℃引物退火45 s,72 ℃延伸2 min,32个循环;72 ℃延伸10 min。每25 μL反应体系中包括2×Taq Plus PCR MasterMix(Tiangen,KT205),上下游引物各0.2 μmol,模板10 ng。
1.5 序列测定及分析PCR产物委托北京诺赛基因组研究中心有限公司完成。序列比对分析通过BLAST(http://www.ncbi.nlm.nih.gov/BLAST)进行,利用MEGA 6.0进行遗传距离分析及系统发育树构建,采用邻间法,步值1 000。
2 结果分析 2.1 核糖体分型结果分析基于皮尔森相关系数,使用UPGMA算法,样本分离菌株的核糖体分型及聚类结果如图 1,不同来源的菌株的核糖体型被聚类为3个不同的分组,分别是以样本1分离菌株及样本3分离菌株(4株)为代表的group 1,以样本3分离菌株y-1构成的group 2,以样本2分离菌株为代表的group 3。核糖体分型结果表明样本3中部分分离菌株与样本1分离菌株同源,暗示样本3的污染很可能来源于样本1。3组菌株在核糖体分型中的鉴别结果的SIM值均 < 0.85,但最高比对结果均指向洋葱伯克霍尔德菌,因此在鉴别分析水平上,上述3组菌株疑似为洋葱伯克霍尔德菌。利用核糖体分型的溯源优势,选取上述3组核糖体型中的代表菌株,进行后续鉴别分析。
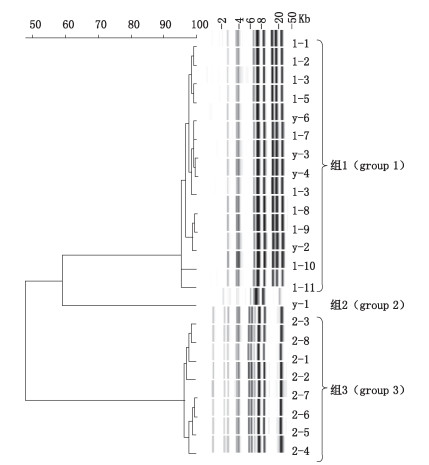
|
图 1 分离菌株核糖体分型聚类分析图 Fig.1 The diagram of cluster analysis of the ribotyping of isolated strains |
上述菌株生化鉴定结果如表 1所示。由表 1可知,待测8株菌株中5株在生化鉴定中无法确定其种水平归属,而2-1、2-2、2-3给出种水平鉴别结果,但与核糖体分型结果中,3株菌同属于group 3不符,这与文献报道中生化分析无法对BCC某些基因型进行准确区分一致。
|
|
表 1 生化鉴定结果 Tab.1 Results of automatic biochemical analysis |
将所测实验菌株的16S rRNA序列置于NCBI进行BLAST,选取BCC中12个种代表菌株的16S rRNA序列与待测菌株16S rRNA序列构建邻间树,如图 2,上述菌株被聚类为两部分,待测菌株,基因型Ⅰ、Ⅲ、Ⅳ、Ⅶ、Ⅷ、Ⅸ代表菌株及不属于上述分类的Burkholderia contaminans、Burkholderia arboris归属至第一部分,同源性为99.2%~100%;基因型Ⅱ、Ⅴ、Ⅵ的代表菌株被归属至第二部分,同源性为99.4%~99.5%,各基因型16Sr RNA同源性较高,无法明确区分。因此,除菌株y-1可明确归属至Burkholderi astabilis之外,其他菌株无法明确至种水平分类地位。显示步值> 50%结果,Burkholderia pseudomallei(ATCC23343)作为外群。
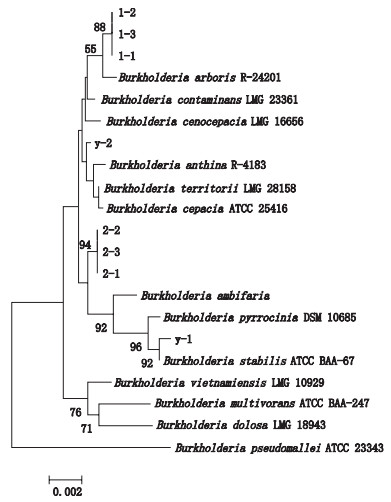
|
图 2 基于16S rRNA序列的进化树分析 Fig.2 Phylogenetic tree analysis derived from 16S rRNA sequences |
对待测菌株及BCC中不同基因型代表菌株recA基因序列进行遗传距离及系统发育分析,如图 3。BCC中recA基因的种间同源性为93%~97%,各基因型能得到明确的区分,菌株1-1、1-2、1-3、y-2同源性达到100%,与Burkholderia cepacia ATCC25416同源性达99.3%,因此,可确定1-1、1-2、1-3、y-2菌株为Burkholderia cepacia;菌株2-1、2-2、2-3与Burkholderia cenocepacia(AF143784)同源性达99%,可确定此3株菌为Burkholderia cenocepacia;y-1与Burkholderia stabilis同源性达99.6%,可确定为Burkholderia stabilis,与16S rRNA分析结果一致。显示步值> 70%结果,Burkholderia pseudomallei作为外群。
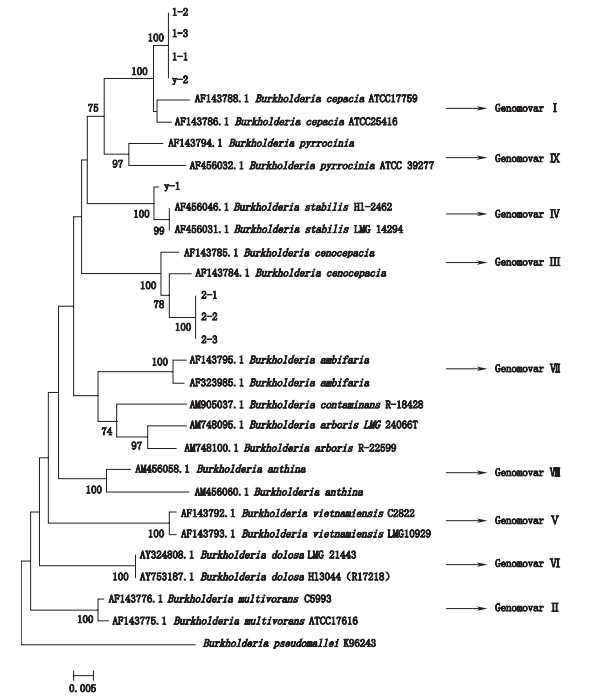
|
图 3 基于recA序列的进化树分析 Fig.3 Phylogenetic tree analysis derived from recA gene sequences |
在本实验中,涉及到的BCC种水平菌株包括Burkholderia cepacia(Genomovar Ⅰ),Burkholderia cenocepacia(Genomovar Ⅲ)以及Burkholderia stabilis(Genomovar Ⅳ),由前述研究可知,这几种基因型可通过核糖体分型进行区分,但由于数据库的限制,无法判断未知菌株的基因型,因此,无法给出可信的鉴定结果。即便如此,不同的种或者基因型并不会表现出相同的核糖体型,通过聚类将核糖体型一致或近似的菌株归属至同一分组,而后通过其他方法进一步对组内任一菌株进行鉴别分析,即可得知该组的种水平分类归属,这一优势也在本实验结果中得以体现。通过核糖体分型,临床分离的部分菌株与该外用制剂中的分离菌株核糖体型高度相似,第一时间快速地确定了污染来源,为问题调查节省了时间成本,也为后续进一步的实验结果提供了佐证。基于核糖体分型数据库的限制,后续也将尝试进行BCC本地数据库的扩增,以求使用核糖体分型方法即可完成对样品及环境中分离的疑似BCC菌株的初步鉴定和溯源分析,加快问题的分析及解决速度。
基于recA基因序列分析的研究方法经过近20年的发展,BCC特异性的recA扩增引物可以将疑似菌株定位在BCC范围内,并且根据扩增序列的同源性分析区分BCC中部分基因型,如本文所述。而作为对recA序列分析的补充,基于recA基因的RFLP方法已能够区分BCC种大多数的基因型[8, 20]。MLST的应用使得已发现的所有BCC基因型均能被准确区分,并在BCC新种的发现中起到重要作用[10]。针对BCC的MLST数据库也已建立(http://pubmlst.org/bcc/),为实验室之间的数据共享及互相验证提供平台。这一分型技术覆盖至目前已发现的所有BCC菌种,能够满足溯源鉴定中对疑似菌株更加精准的区分,以弥补某些BCC基因型无法利用单一保守序列分析方法获得准确结果的缺陷。由于本文中覆盖BCC菌种数量有限,后续将通过对BCC以及非BCC伯克霍尔德菌属微生物的MLST分型分析,进一步完善本文研究。
药品中分离微生物的鉴别和鉴定,根据目的可以选择不同的方法,常见的如生理生化、光谱、血清学、保守基因序列分析、PFGE及全基因组测序等。一般情况下,制药企业在水系统或样品中分离出疑似BCC时,应特别关注,通过风险评估采取适当措施。当中间产品或终产品出现疑似BCC污染、需要调查原因,或需要寻找污染源的时候,就需要对分离的疑似BCC进行准确鉴定至种水平甚至进行株水平的溯源分析。包括《中华人民共和国药典》在内的各国药典都规定了药品中不得检出的控制菌,例如金黄色葡萄球菌、大肠埃希菌、铜绿假单胞菌、沙门菌、梭菌等。但是,对尚未列入药典规范的不可接受微生物的鉴定方法研究较少。本研究以污染了BCC的某外用制剂为例,采用了表型和基因型的方法,对3种来源的BCC进行了鉴定分析,其中生理生化和16S rRNA序列分析将疑似菌株归属至伯克霍尔德菌属或者BCC,对后续分析结果进行佐证;核糖体分型和recA序列同源性分析则完成对疑似菌株的分型及种水平鉴别,确定了污染菌及污染来源。无论是制药企业还是是检验机构,在遇到此类问题时,应结合实际情况及目的,根据需要选择1种或几种方法进行疑似BCC的鉴别和鉴定分析,将有利于问题的快速解决。
| [1] |
SOUSA SA, RAMOS CG, LEITAO JH. Burkholderia cepacia complex:emerging multihost pathogens equipped with a wide range of virulence factors and determinants[J]. Int J Microbiol, 2010, 2011: 1. |
| [2] |
YABUUCHI E, KOSAKO Y, OYAIZU H, et al. Proposal of Burkholderia gen. nov. and transfer of seven species of the genus Pseudomonas homology group Ⅱ to the new genus, with the type species Burkholderia cepacia(Palleroni and Holmes 1981)comb. nov.[J]. Microbiol Immunol, 1992, 36(12): 1251. DOI:10.1111/j.1348-0421.1992.tb02129.x |
| [3] |
URSING JB, GARCÍA-VALDÉS E, LALUCAT J. Taxonomic note:a pragmatic approach to the nomenclature of phenotypically similar genomic groups[J]. Int J Syst Bacteriol, 1995, 45(3): 604. DOI:10.1099/00207713-45-3-604 |
| [4] |
VANDAMME P, HOLMES B, VANCANNEYT M, et al. Occurrence of multiple genomovars of Burkholderia cepacia in cystic fibrosis patients and proposal of Burkholderia multivorans sp. nov.[J]. Int J Syst Bacteriol, 1997, 47(4): 1188. DOI:10.1099/00207713-47-4-1188 |
| [5] |
COENYE T, VANDAMME P, GOVAN JR, et al. Taxonomy and identification of the Burkholderia cepacia complex[J]. J Clin Microbiol, 2001, 39(10): 3427. DOI:10.1128/JCM.39.10.3427-3436.2001 |
| [6] |
EISEN JA. The RecA protein as a model molecule for molecular systematic studies of bacteria:comparison of trees of RecAs and 16S rRNAs from the same species[J]. J Mol Evol, 1995, 41(6): 1105. |
| [7] |
KARLIN S, WEINSTOCK GM, BRENDEL V. Bacterial classifications derived from recA protein sequence comparisons[J]. J Bacteriol, 1995, 177(23): 6881. DOI:10.1128/jb.177.23.6881-6893.1995 |
| [8] |
MAHENTHIRALINGAM E, BISCHOF J, BYRNE SK, et al. DNAbased diagnostic approaches for identification of Burkholderia cepacia complex, Burkholderia vietnamiensis, Burkholderia multivorans, Burkholderia stabilis, and Burkholderia cepacia genomovars Ⅰ and Ⅲ[J]. J Clin Microbiol, 2000, 38(9): 3165. |
| [9] |
VANLAERE E, LIPUMA JJ, BALDWIN A, et al. Burkholderia latens sp. nov., Burkholderia diffusa sp. nov., Burkholderia arboris sp. nov., Burkholderia seminalis sp. nov. and Burkholderia metallica sp. nov., novel species within the Burkholderia cepacia complex[J]. Int J Syst Evol Microbiol, 2008, 58(Pt 7): 1580. |
| [10] |
BALDWIN A, MAHENTHIRALINGAM E, THICKETT KM, et al. Multilocus sequence typing scheme that provides both species and strain differentiation for the Burkholderia cepacia complex[J]. J Clin Microbiol, 2005, 43(9): 4665. DOI:10.1128/JCM.43.9.4665-4673.2005 |
| [11] |
VANLAERE E, LIPUMA JJ, BALDWIN A, et al. Burkholderia latens sp. nov., Burkholderia diffusa sp. nov., Burkholderia arboris sp. nov., Burkholderia seminalis sp. nov. and Burkholderia metallica sp. nov., novel species within the Burkholderia cepacia complex[J]. Int J Syst Evol Microbiol, 2008, 58(7): 1580. DOI:10.1099/ijs.0.65634-0 |
| [12] |
VANLAERE E, BALDWIN A, GEVERS D, et al. Taxon K, a complex within the Burkholderia cepacia complex, comprises at least two novel species, Burkholderia contaminans sp. nov. and Burkholderia lata sp. nov[J]. Int J Syst Evol Microbiol, 2009, 59(Pt 1): 102. |
| [13] |
PEETERS C, ZLOSNIK JE, SPILKER T, et al. Burkholderia pseudomultivorans sp. nov., a novel Burkholderia cepacia complex species from human respiratory samples and the rhizosphere[J]. Syst Appl Microbiol, 2013, 36(7): 483. DOI:10.1016/j.syapm.2013.06.003 |
| [14] |
de SMET B, MAYO M, PEETERS C, et al. Burkholderia stagnalis sp. nov. and Burkholderia territorii sp. nov., two novel Burkholderia cepacia complex species from environmental and human sources[J]. Int J Syst Evol Microbiol, 2015, 65(7): 2265. DOI:10.1099/ijs.0.000251 |
| [15] |
WEBER CF, KING GM. Volcanic soils as sources of novel cooxidizing paraburkholderia and Burkholderia:Paraburkholderia hiiakae sp. nov., Paraburkholderia metrosideri sp. nov., Paraburkholderia paradisi sp. nov., Paraburkholderia peleae sp. nov., and Burkholderia alpina sp. nov. a member of the Burkholderia cepacia complex[J]. Front Microbiol, 2017, 8: 207. |
| [16] |
ONG KS, AW YK, LEE LH, et al. Burkholderia paludis sp. nov., an antibiotic-siderophore producing novel Burkholderia cepacia complex species, isolated from Malaysian tropical peat swamp soil[J]. Front Microbiol, 2016, 7: 2046. |
| [17] |
MARTINA P, LEGUIZAMON M, PRIETO CI, et al. Burkholderia puraquae sp. nov., a novel species of the Burkholderia cepacia complex isolated from hospital settings and agricultural soils[J]. Int J Syst Evol Microbiol, 2018, 68(1): 14. DOI:10.1099/ijsem.0.002293 |
| [18] |
BRISSE S, VERDUIN CM, MILATOVIC D, et al. Distinguishing species of the Burkholderia cepacia complex and Burkholderia gladioli by automated ribotyping[J]. J Clin Microbiol, 2000, 38(5): 1876. |
| [19] |
BRISSE S, CORDEVANT C, VANDAMME P, et al. Species distribution and ribotype diversity of Burkholderia cepacia complex isolates from French patients with cystic fibrosis[J]. Clin Microbio, 2004, 42(10): 4824. DOI:10.1128/JCM.42.10.4824-4827.2004 |
| [20] |
COENYE T, MAHENTHIRALINGAM E, HENRY D, et al. Burkholderia ambifaria sp. nov., a novel member of the Burkholderia cepacia complex including biocontrol and cystic fibrosis-related isolates[J]. Int J Syst Evol Microbiol, 2001, 51(Pt 4): 1481. |
 2019, Vol. 39
2019, Vol. 39 
