红芪(Hedysari Radix)为豆科(Leguminosae)植物多序岩黄芪(Hedysarum polybotrys Hand.-Mazz.)的干燥根[1],在甘肃陇南地区已广泛栽培,具补气升阳、固表止汗等功效,用于治疗中气下陷等症。其野生品分布较少,栽培品是主流商品。由于野生型与栽培型中药材在化学成分含量[2-3]、质量[4-5]、遗传多样性及遗传结构[6]等方面具有差异性,导致中药材的野生品与栽培品价格波动加大,中医临床疗效参差不齐,因此野生与栽培中药材的准确鉴别一直是中药鉴定领域亟待解决的问题。
近红外光谱(near Infrared spectroscopy,NIRS)技术发展至今,以其能够反映被测物质的整体信息等特点与中医药的整体思维不谋而合,被广泛的应用于中医药的研究中,并取得了较好的成果。其在中药鉴定方面的应用潜力已被发掘[7],成功地应用到人参种植区域的鉴别[8]和天麻的产地溯源及伪品识别[9]中,为人参及天麻的产地溯源及质量研究奠定基础。同时此技术在中药及中成药的质量控制方面具有可行性与合理性[10-14],为中药的质量控制提供了研究思路,这些研究暗示出NIRS技术在中医药领域具有广泛的发展潜力。
课题组前期研究发现,不同产地野生红芪中毛蕊异黄酮和芒柄花素的含量均高于栽培红芪[15],Fe、Ca等11种无机元素的含量之间均具显著性差异[16],为本文的研究提供了研究思路。本文通过收集不同生长类型的红芪样品,采集野生与栽培红芪的近红外光谱,建立野生与栽培红芪的主成分分析-马氏距离判别模型与偏最小二乘判别模型,为野生与栽培红芪的鉴别及质量评价提供参考。
1 材料 1.1 样品收集2014年采集73份不同类型红芪样品,样品信息见表 1,样品采集地区主要有陇南市武都区(中国红芪之乡)、宕昌县等红芪的主产区及定西市的漳县、岷县、渭源县等地,样品覆盖了甘肃红芪的道地产区,具有一定的代表性。其中,野生红芪样品的直径、表面颜色、断面等性状差异性较小,且根头部均较粗,表面颜色较深(深棕色或红棕色)。同时,红芪的主产区已形成了成套的栽培技术及标准化的栽培规程,并已推广,保证了不同产地栽培红芪样品的直径、表面颜色、断面等性状具有一致性,质量方面的差异性较小。样品经甘肃中医药大学中药鉴定教研室李成义教授鉴别,为豆科植物多序岩黄芪Hedysarum polybotrys Hand.-Mazz.的干燥根。
|
|
表 1 样品信息 Table 1 The information of samples |
Nicolet-6700型傅立叶变换近红外光谱仪(配有积分球模块、RESULT 3.0光谱采集软件和TQ Analyst 8.0化学计量学软件,Thermo公司)。粉碎机(FW177型中草药粉碎机,天津市泰斯特仪器有限公司);65目不锈钢筛盘。
1.3 样品处理将“1.1”项下采集的73份红芪样品粉碎后均过4号筛(65目),装入自封袋中(红芪样品不少于30 g),贮存。
1.4 红芪样品近红外光谱采集取各红芪样品(约4 g)置测量杯中,以积分球漫反射方式采集光谱,每扫描1个样品均采集1次背景,扫描范围10 000~4 000 cm-1,扫描次数64,分辨率8 cm-1,温度20 ℃,空气相对湿度70%。73份红芪样品原始近红外光谱图见图 1。由图 1可知,红芪样品近红外原始光谱存在以下特征:(1)野生与栽培样品谱图相似度较高,依靠特征的吸收峰区别样品存在盲点,需对光谱数据做进一步处理,以便实现野生样品与栽培样品的区分;(2)红芪样品的近红外谱图受到噪声等因素的影响,其基线存在漂移现象。
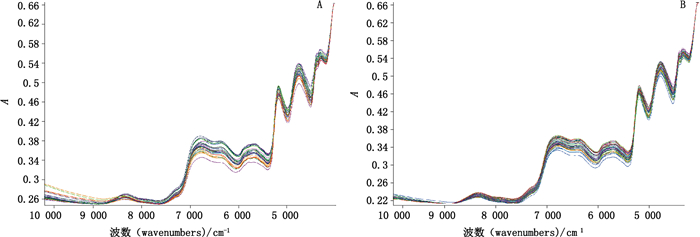
|
A.野生(wild)B.栽培(cultivated) 图 1 红芪样品近红外原始光谱 Figure 1 Original near infrared spectra of Hedysari Radix |
本文采用近红外光谱仪自带的TQ Analyst 8.0软件对光谱进行优化处理。红芪近红外光谱中除了包含样品自身的整体性化学信息外,还包括了一些无用信息、噪声等,因此本文中采用多元散射校正法(multiplication signal correction,MSC)、标准正态变换(standard normal variate,SNV)、一阶导数(first derivative,FD)、二阶导数(second derivative,SD)、SG平滑(Savitzky-Golay filter)、ND平滑(Norris derivative filter)的组合方法去除红芪光谱中存在的无用信息及噪声,以便提高判别模型的准确度,光谱预处理结果见表 2,以预测集样品判错数为指标,选取的原始光谱预处理方法为MSC、SNV、SD及ND结合的方法,最低判错数为2。
|
|
表 2 光谱预处理结果 Table 2 Results of spectral pretreatment |
在对野生与栽培红芪原始光谱预处理的基础上,采用主成分分析-马氏距离(principal component analysis-mahalanobis distance,PCA-MD)判别分析的方法,用光标法(TQ Analyst 8.0软件中可观察或选取建模光谱范围的图标)选取的光谱波段为6 071.27~4 957.55 cm-1,选取56份样品为校正集,17份样品为预测集,使用方差缩放技术(use variance scaling technique),选取的主成分数为22时,判别模型可区分野生样品与栽培样品(主成分数低于22时,野生样品与栽培样品交叉在一起,且判错数会增加,使模型的判别率下降;当主成分数大于22时模型的判错数并未减少,且判别率未增加,因此选取的主成分数为22,22个主成分对全部光谱信息贡献率达99.921 8%。),结果见图 2。对预测集样品的样品判错数均为2(表 3),说明MSC、SNV与SD及ND结合的预处理方法均能达到预期的模型效果。比较2种处理方法发现,对红芪生长类型的判别率达到88.24%,表明MSC、SNV均可以作为红芪光谱的有效预处理方法。以上分析发现优化的PCA-MD模型对野生与栽培红芪的判别率达88.24%,未达到100%,可能的原因有(1):波段选取的方法采用的是人为选取的方法,较为粗糙;(2)此波段野生与栽培样品的近红外光谱信息具有一定的特征性,但特征性不明显,同时可能是此波段的光谱信息间有较多的线性相关特点,变量间具有较强的相关性,致使野生与栽培样品光谱信息存在冗余信息,引起了PCA-MD预测能力的下降。(3)本文针对的是野生红芪与栽培红芪样品2个类别的准确区分,而无监督的PCA-MD模式识别方法要求每个类别中需包含多个典型的样本,因此PCA-MD判别模型判别率未达至100%的原因与采集的野生与栽培样品的典型性有关。尽管本文采集的样品覆盖了红芪的道地与非道地产区,但由于采样地“沟壑纵横、山大沟深”的地貌特点限制了多个典型的样本的获取,加之野生样品与栽培样品本身化学成分相似度较高,使得模型的计算较为复杂。(4)PCA-MD模型基于的原理是降维的方式,提取出特征性的主成分信息,其消除了数据的相关性并大幅减小输入数据的维数,而野生与栽培近红外原始光谱中影响建模的波数信息并没有消除掉,其依然存在与主成分中,影响了模型的预测能力。因此需要进一步建立其他判别模型以便实现野生与栽培红芪的准确区分。

|
□.野生样品(wild sample)△.栽培样品(cultivated sample) 图 2 PCA-MD模型处理结果 Figure 2 The results of PCA-MD |
|
|
表 3 PCA-MD模型预测结果 Table 3 The prediction results of PCA-MD |
由于PCA-MD判别模型并未实现对野生与栽培红芪的明显区分,因此采用SIMCA P+11.0软件,建立野生与红芪样品的偏最小二乘判别模型(partial least square discrimination analysis,PLS-DA),通过变量重要性图(variable importanceplot,VIP)选择自变量(光谱)对因变量(分类)影响大于1的光谱波段[17],4 000.000~5 418.991 cm-1、6 722.635~7 370.6 cm-1、7 467.023~7 617.44 cm-1、9 896.891~10 000 cm-1,主成分数为6。PLS-DA模型中R2Y代表累积的自变量X对Y的解释能力,Q2代表模型的预测能力。模型中R2Y值为0.652,Q2值为0.545 > 0.5,R2Y-Q2=0.107在(0~0.3之间),PLS-DA模型判别结果见图 3,由图 3可知,野生与栽培红芪的近红外光谱信息具有差异性,部分野生与栽培红芪样品存在交叉,且二者未明显区分。
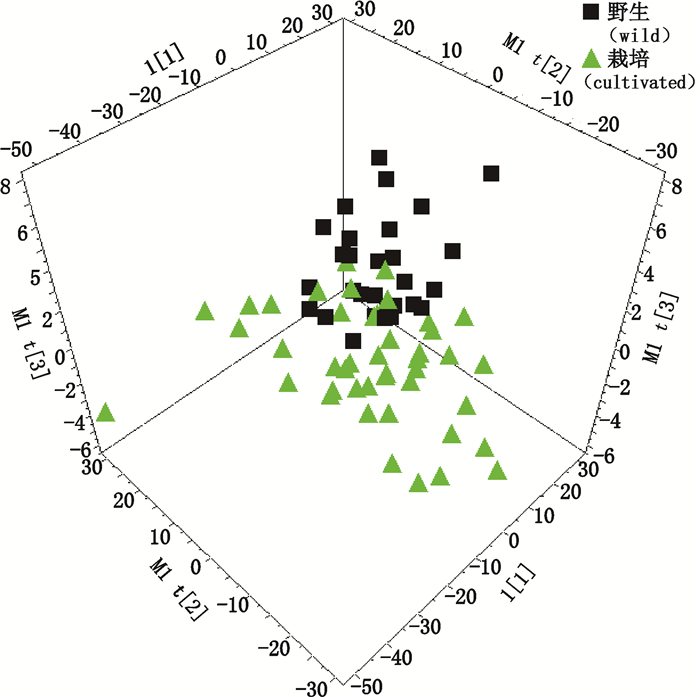
|
图 3 PLS-DA模型判别结果 Figure 3 The results of PLS-DA |
在PLS-DA模型建立的基础上,采用SIMCA-P+11软件对进行置换检验验证PLS-DA模型[18-19],排列数为200,结果见图 4,由图 4可知,R2的截距为0.085 4 < 0.4,Q2的截距值为-0.441 < 0.05,表明本文建立的模型是稳定、有效且无过度拟合,可用于判别野生与栽培红芪。

|
图 4 PLS-DA模型验证 Figure 4 Model validation of PLS-DA |
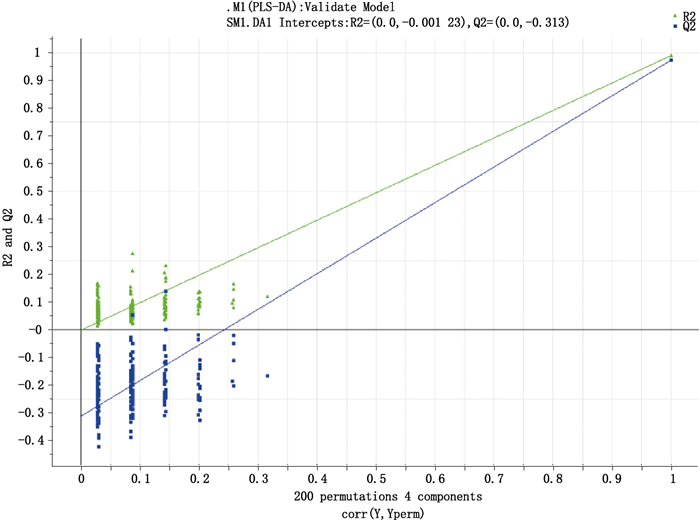
在PLS-DA模型建立及验证的基础上,进一步对选取的光谱波段进行优化,由于红芪样品二阶导数的高频波段6 722.635~7 370.6、7 467.023~7 617.44、9 896.891~10 000 cm-1在谱图上相似性较大且未发现特征性差异波段,因此在建立PLS-DA判别模型时“去除了高频波段”(预处理后光谱图)见图 5,选取4 000.000~5 418.991 cm-1的波段建立二次PLS-DA模型,主成分数为4,结果显示,模型中R2Y值为0.988,Q2值为0.972 > 0.5,R2Y-Q2=0.016在(0~0.3之间),二次PLS-DA模型判别结果见图 6,由图 6可知,野生红芪样品与栽培红芪样品被明显区分。进一步对建立的二次PLS-DA模型进行置换检验,排列数为200,结果见图 7,由图 7可知,R2的截距为-0.001 2 < 0.4,Q2的截距值为-0.313 < 0.05,表明本文建立的二次PLS-DA模型是稳定、有效且无过度拟合,可用于准确判别野生与栽培红芪。
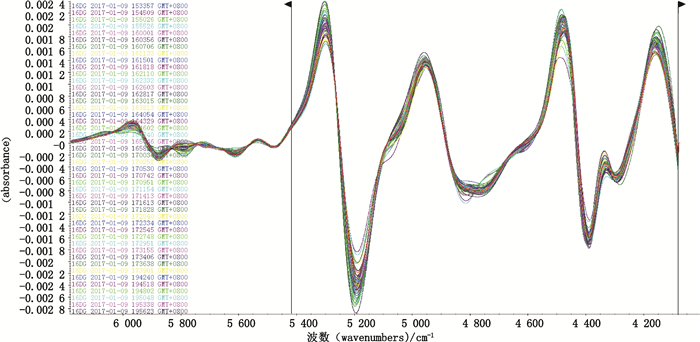
|
图 5 预处理后光谱图 Figure 5 Pretreated spectra |
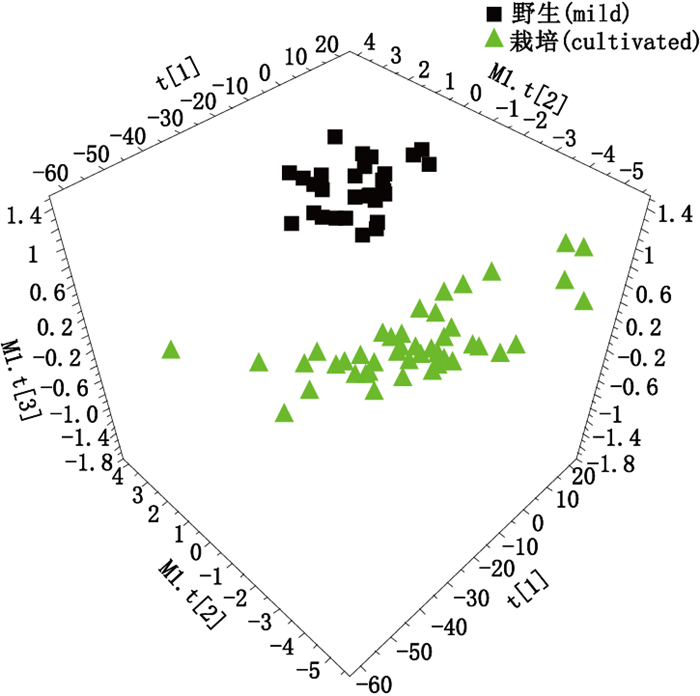
|
图 6 二次PLS-DA模型判别结果 Figure 6 The results of second PLS-DA |
|
图 7 二次PLS-DA模型验证 Figure 7 Model validation of second PLS-DA |
PCA-MD与PLS-DA模型均是模式识别的方法,PCA-MD为无监督的方法,主成分的提取是采用降维的方法,提取出有用的光谱信息,本文中建立的PCA-MD判别模型对野生与栽培红芪的鉴别率达到88.24%,未达到100%,因此建模波段与建模方法需进一步优化以便实现野生与栽培红芪的准确区分。而PLS-DA为有监督的方法,由于PLS方法同时对光谱阵和类别阵进行分解,加强了类别信息在光谱分解时的作用,以提取出与样本类别最相关的光谱信息,即最大化提取不同类别光谱之间的差异,因此PLS-DA方法通常可以得到比基于PCA的识别方法更优的分类和判别结果,基于此进一步对光谱波段进行了优化,建立了PLS-DA判别模型并对其进行验证,实现了对野生与栽培红芪的准确鉴别。
研究结果显示,野生与栽培红芪在近红外光谱上的差异性主要集中于4 000.000~5 418.991 cm-1的波段,其中4 200~4 440 cm-1为-C-H-(CH3,CH2)的组合频,5 050~5 210 cm-1为游离-OH组合频,5 130~5 230 cm-1为-C=O-的2级倍频。因此野生与栽培红芪的区别化学成分可能是含有-C-H-、-OH与-C=O-等官能团的化合物,这与课题组前期研究发现野生红芪中活性成分毛蕊异黄酮和芒柄花素(含有-C-H-、-OH与-C=O-官能团的化合物)的含量均高于栽培红芪[15],野生红芪质量优于栽培红芪的结论是相似的,因此本文建立的识别野生红芪与栽培红芪样品的方法具有实际意义。课题组前期的高效液相色谱指纹图谱实验结果显示,毛蕊异黄酮和芒柄花素是野生和栽培红芪的差异性化合物之一,但还存在其他野生和栽培红芪的差异性化合物,而其他的这些化合物是未知的,需要进一步采用UPLC-Q-TOF/MS等仪器进行识别。同时需要采集毛蕊异黄酮与芒柄花素的近红外光谱(4 000.000~5 418.991 cm-1),分析野生和栽培红芪近红外光谱的特征对应性,进一步验证毛蕊异黄酮与芒柄花素是否是造成野生与栽培样品近红外光谱差异性的主要物质。
由于野生红芪与栽培红芪受到的地理尺度、生长环境、人为干扰等因素的影响不同,可能引起了毛蕊异黄酮和芒柄花素等差异性化合物合成通路上关键酶基因的表达出现差异,导致毛蕊异黄酮和芒柄花素等差异性化合物含量呈现出野生高于栽培红芪的特征,因此在近红外光谱差异波段4 000.000~5 418.991 cm-1表现出野生红芪与栽培红芪的差异。
3 结论与讨论本文通过优化红芪近红外光谱处理方法,建立野生与栽培红芪的PCA-MD判别模型与PLS-DA判别模型,结果显示PCA-MD判别模型对野生与栽培红芪的判别率为88.24%,MSC、SNV预处理近红外光谱的方法适合于处理红芪的近红外光谱。PLS-DA判别模型显示野生与栽培红芪的近红外光谱信息具有差异性,部分野生与栽培红芪样品存在交叉,PLS-DA判别模型建立时,增大主成分数时会产生模型过拟合的现象,导致建立的模型不准确,出现野生与栽培类型的判别出现错误,2种模型均未能实现野生红芪与栽培红芪的完全区。在以上分析的基础上,去除红芪近红外光谱高频波段,建立二次PLS-DA判别模型可明显区分野生与栽培红芪,模型验证结果显示二次PLS-DA判别模型无过拟合现象存在,表明野生与栽培红芪的主要差异性光谱波段为4 000.000~5 418.991 cm-1。研究结果为其他野生型与栽培型中药材的鉴别提供了思路。
众多的证据表明,野生与栽培中药材之间存在差异性[2-6],且本文的研究也证明了此观点。这与王燕[20]研究的“不同产地野生与栽培红芪的质量存在明显差异,其中野生红芪质量优于栽培红芪结论”是一致的。而课题组前期研究发现,野生红芪居群与栽培红芪居群之间存在的遗传变异,遗传多样性野生居群高于栽培居群,野生与栽培居群总体遗传变异5.44%的变异来源于群体间,94.56%的遗传变异存在于群体内,在遗传距离及遗传一致度上具有差异性[21],因此本文中发现的野生与栽培红芪之间存在差异性光谱波段的原因可能是野生与栽培红芪的遗传多样性的差异造成的,具体的机制有待于进一步研究。
| [1] |
中华人民共和国药典2015年版.一部[S].2015: 152 ChP 2015.Vol Ⅰ[S].2015: 152 |
| [2] |
SUN H, LIU F, SUN L, et al. Proteomic analysis of amino acid metabolism differences between wild and cultivated Panax ginseng[J]. J Ginseng Res, 2016, 40(2): 113. DOI:10.1016/j.jgr.2015.06.001 |
| [3] |
华愉教, 侯娅, 王胜男, 等. 基于1H-NMR代谢组学技术的野生与栽培太子参化学成分分析[J]. 中国药学杂志, 2017, 52(4): 272. HUA YJ, HOU Y, WANG SN, et al. 1H-NMR based metabolomic analysis of chemical compositions in cultivated and wild Pseudostellariae Radix[J]. Chin Pharm J, 2017, 52(4): 272. |
| [4] |
盛萍, 安露莎, 苗莉娟, 等. 不同产地野生与栽培伊贝母药材水溶性成分指纹图谱研究[J]. 中国野生植物资源, 2014, 33(4): 19. SHENG P, AN LS, MIAO LJ, et al. HPLC fingerprints of water-soluble constituents of bulbus fritillariae pallidiflorae from different habitats and different wild growing and cultivated species[J]. Chin Wild Plant Resour, 2014, 33(4): 19. DOI:10.3969/j.issn.1006-9690.2014.04.006 |
| [5] |
皮胜玲, 张凯强, 胡玉珍, 等. 野生与栽培夏枯草5种活性成分的HPLC测定[J]. 中草药, 2017, 48(8): 1666. PI SL, ZHANG KQ, HU YZ, et al. Determination of five bioactive constituents in wild grown and cultivated Prunella vulgaris by HPLC[J]. Chin Tradit Herb Drugs, 2017, 48(8): 1666. |
| [6] |
HE J, CHEN L, SI Y, et al. Population structure and genetic diversity distribution in wild and cultivated populations of the traditional Chinese medicinal plant Magnolia officinalis subsp. biloba (Magnoliaceae)[J]. Genetica, 2009, 135(2): 233. DOI:10.1007/s10709-008-9272-8 |
| [7] |
赵中振, 梁之桃. 近红外光谱技术在中药鉴定中的应用与优势[J]. 中国中药杂志, 2012, 37(8): 1062. ZHAO ZZ, LIANG ZT. Application and advantage of near infrared spectroscopy technology in authentication of Chinese materia medica[J]. China J Chin Mater Med, 2012, 37(8): 1062. |
| [8] |
WOO YA, CHO CH, KIM HJ, et al. Classification of cultivation area of ginseng by near infrared spectroscopy and ICP-AES[J]. Microchem J, 2002, 73(3): 299. DOI:10.1016/S0026-265X(02)00094-2 |
| [9] |
LI GF, YIN QB, ZHANG L, et al. Fine classification and untargeted detection of multiple adulterants of Gastrodia elata BI.(GE) by near-infrared spectroscopy coupled with chemometrics[J]. Anal Methods, 2017, 9(12): 1897. DOI:10.1039/C7AY00153C |
| [10] |
周昭露, 李杰, 黄生权, 等. 近红外光谱技术在中药质量控制应用中的化学计量学建模:综述和展望[J]. 化工进展, 2016, 35(6): 1627. ZHOU ZL, LI J, HUANG SQ, et al. Development of chemometric modelling in the application of NIR to the quality control of Chinese herbal medicine:literature review and future perspectives[J]. Chem Ind Eng Prog, 2016, 35(6): 1627. |
| [11] |
ZHANG C, SU J. Application of near infrared spectroscopy to the analysis and fast quality assessment of traditional Chinese medicinal products[J]. Acta Pharm Sin B, 2014, 4(3): 182. DOI:10.1016/j.apsb.2014.04.001 |
| [12] |
GUO LP, HUANG LQ, ZHANG X, et al. Application of near-infrared spectroscopy (NIRS) as a tool for quality control in traditional Chinese medicine (TCM)[J]. Curr Bioact Compd, 2011, 7(2): 75. DOI:10.2174/157340711796011188 |
| [13] |
FU HY, HUAN SY, XU L, et al. Construction of an efficacious model for a nondestructive identification of traditional Chinese medicines Liuwei Dihuang pills from different manufacturers using near-infrared spectroscopy and moving window partial least-squares discriminant analysis[J]. Anal Sci Int J Japan Soc Anal Chem, 2009, 25(9): 1143. |
| [14] |
DING G, WANG Y, LIU A, et al. From chemical markers to quality markers:an integrated approach of UPLC/Q-TOF, NIRS, and chemometrics for the quality assessment of honeysuckle buds[J]. RSC Adv, 2017, 7(36): 22034. DOI:10.1039/C6RA28152D |
| [15] |
李成义, 王燕, 强正泽, 等. 甘肃不同产地野生与栽培红芪中毛蕊异黄酮和芒柄花素含量比较研究[J]. 甘肃中医学院学报, 2014, 36(6): 25. LI CY, WANG Y, QIANG ZZ, et al. Comparative study on the contents of calycosin and formononetin between wild and cultivated Hedysari Radix collected from different areas of Gansu Province[J]. J Gansu Coll Tradit Chin Med, 2014, 36(6): 25. |
| [16] |
强正泽, 李成义, 李硕, 等. 基于微量元素的野生与栽培红芪鉴别[J]. 中国实验方剂学杂志, 2016, 22(12): 47. QIANG ZZ, LI CY, LI S, et al. Identification of wild and cultivated Hedysari Radix based on trace elements[J]. Chin J Exp Tradit Med Form, 2016, 22(12): 47. |
| [17] |
赵艳丽, 张霁, 袁天军, 等. 近红外光谱快速鉴别不同产地药用植物重楼的方法研究[J]. 光谱学与光谱分析, 2014, 34(7): 1831. ZHAO YL, ZHANG J, YUAN TJ, et al. Study on rapid identification of medicinal plants of Paris polyphylla from different origin areas by NIR spectroscopy[J]. Spectrosc Spect Anal, 2014, 34(7): 1831. DOI:10.3964/j.issn.1000-0593(2014)07-1831-05 |
| [18] |
TRIBA MN, LE ML, AMATHIEU R, et al. PLS/OPLS models in metabolomics:the impact of permutation of dataset rows on the K-fold cross-validation quality parameters[J]. Mol Biosyst, 2015, 11(1): 13. DOI:10.1039/C4MB00414K |
| [19] |
柯朝甫, 武晓岩, 李康. PLS-DA模型四种诊断统计量在代谢组学应用中的比较[J]. 中国卫生统计, 2014, 31(3): 403. KE CF, WU XY, LI K. A comparative analysis of four PLS-DA diagnostic statistics in the application of metabolomics[J]. Chin J Health Stat, 2014, 31(3): 403. |
| [20] |
王燕.甘肃地产药材红芪的质量评价研究[D].兰州: 甘肃中医药大学, 2015 WANG Y.Studies on Quality Evaluation of Hedysari Radix in Gansu Region[D].Lanzhou: Gansu University of Traditional Chinese Medicine, 2015 http://cdmd.cnki.com.cn/Article/CDMD-10735-1015973433.htm |
| [21] |
强正泽.基于ISSR标记技术的甘肃地产药材红芪的遗传多样性研究[D].兰州: 甘肃中医药大学, 2016 QIANG ZZ.Studies on Genetic Diversity of Hedsari Radix from Gansu Based on ISSR[D].Lanzhou: Gansu University of Traditional Chinese Medicine, 2016 http://cdmd.cnki.com.cn/Article/CDMD-10735-1016758197.htm |
 2018, Vol. 38
2018, Vol. 38 
