2. 伦斯勒理工学院生物技术和多学科研究中心, 纽约州特洛伊 12180;
3. 浙江海洋水产研究所, 舟山 316000
2. Center for Biotechnology and Interdisciplinary Studies, Rensselaer Polytechnic Institute, Troy, NY 12180, USA;
3. Zhejiang Marine Fisheries Research Institute, Zhoushan 316000, China
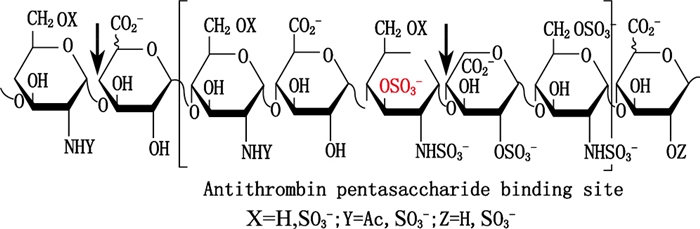
作为临床上广泛使用的抗凝血药物,肝素从发现至今已经100年了。但是肝素的结构和其在生物体中的功能仍未被完全了解[1]。肝素和其相关的糖胺聚糖-硫酸乙酰肝素是一种线性的聚阴离子多糖,由糖醛酸和氨基糖通过1,4-糖苷键交替相连而成。由图可见肝素的抗凝血活性位点肝素五糖的结构,箭头所示之处是肝素酶Ⅱ的酶切位点,经过酶切肝素水解为肝素二糖。由于肝素五糖3位硫酸化后,其右侧相邻的酶切位点被屏蔽,而得到还原端氨基糖3位硫酸化的酶抵抗肝素四糖。糖醛酸主要有β-D-葡萄糖醛酸(GlcUA),还含有少量的α-L-艾杜糖醛酸(IdoUA),氨基糖则由α-D-氨基葡萄糖胺(GlcN)组成。糖链上还存在着不同的取代基团,糖醛酸残基可以被2-O-硫酸基取代,而氨基葡萄糖的残基可以产生乙酰氨基,N-硫代,3-O-硫代和(或)6-O-硫代基团(图 1)。正是这些取代基团的随机性和多样性造就了肝素复杂的结构和丰富的生物活性[2-3]。
|
图 1 肝素的结构图 Figure 1 Structure of heparin |
肝素抗凝活性取决于它与丝氨酸蛋白酶抑制剂、抗凝血酶Ⅲ(antithrombin Ⅲ,AT Ⅲ)的相互作用。在与肝素结合的过程中,抗凝血酶(antithrombin,AT)发生了一种构象变化,增强了其不可逆转的抑制凝血酶的能力,包括凝血酶和因子Xa。肝素-AT相互识别是研究较为透彻的多糖和蛋白相互作用的实例。肝素和AT的识别依赖于肝素中特定的肝素五糖结构区域。而这特定的肝素五糖在结构上的主要特征是在五糖中间的氨基葡萄糖残基存在3-O硫酸基修饰。研究表明肝素五糖的结构也是存在变化的,特别是不同物种来源的肝素。
目前天然肝素的唯一来源是猪小肠上皮,但远远不能满足全球对肝素原料的需求,牛小肠上皮及牛肺上皮来源的肝素有望重新进入市场作为肝素的来源。所以弄清楚肝素五糖的结构有助于更清晰的了解肝素和AT或者其他蛋白作用的模式和差异,有助于进一步分析不同来源肝素活性差异的根源,提高使用的安全性。另外人工合成肝素的工作也在进行中,对肝素五糖结构和功能的研究也为人工合成肝素提供重要参考依据[4-5]。
目前对与AT结合的肝素五糖的结构的直接研究还非常困难。对其结构研究的主要方法是通过肝素酶Ⅱ对肝素进行完全酶解。一般情况肝素酶Ⅱ可以完全将肝素和低分子量肝素降解成二糖,但是肝素五糖的中心氨基葡萄糖残基存在3-O硫酸基修饰,阻碍了肝素酶Ⅱ对其非还原端相邻酶切位点的识别作用,从而形成了以3-O硫酸基修饰氨基葡萄糖为还原端的肝素四糖。作为完全酶解酶抵抗产物,肝素四糖在很大程度上能够反映肝素五糖的结构。对3位硫酸化的肝素四糖的分析将有助于还原肝素五糖的结构与功能[6-7]。
本文从肝素酶解产物中获得了3位硫酸化的肝素四糖和其相对应的同分异构体6位硫酸化的肝素四糖。通过这一对同分异构体的结构和性质的对比分析,了解3位硫酸化的肝素的结构特征,为实现3位硫酸化的肝素的整体分析及进一步研究3位硫酸化与肝素的生物活性之间的关系打下基础。
1 仪器与试药高效液相色谱仪(LC-6AD,岛津公司),电喷雾傅立叶变换离子回旋共振质谱(Apex Ultra Qh 9.4T,布鲁克公司),800 MHz液相核磁共振谱仪(AVANCE,布鲁克公司),Spherisorb S5离子交换色谱柱(4 mm×250 mm,沃特世公司),间接加热阴极电子发射器(Heat Wave公司)。
肝素酶Ⅱ来源于Flavobacterium heparinum,在大肠杆菌中异源表达并分离纯化获得,由美国伦斯勒理工学院Linhardt实验室提供。肝素(LotPH-85314,Celsus)。所用试剂均为色谱纯(Sigma)。
2 实验方法 2.1 肝素的完全酶解肝素四糖还原端的氨基葡萄糖3位硫酸化修饰后对肝素酶有抵抗作用。所以为得到3位硫酸化肝素四糖需对肝素进行完全酶解。酶解条件:500 mg肝素溶于50 mmol·L-1磷酸钠缓冲溶液(pH 7.4)100 mL中,加入肝素酶Ⅱ(30 IU,由肝素测试获得),于37 ℃水浴中酶解12 h。酶解结束后,酶通过50 mL截留相对分子质量3 000的过滤离心管来去除。过滤后溶液即为肝素完全酶解的二糖和四糖产物。
滤液冻干后采用Bio-Rad P2柱(100 mm×2.0 cm)以去离子水为流动相,对酶解产物进行分离纯化,流速0.5 mL·min-1。P2柱不仅可以将肝素二糖和肝素四糖分开,还可以进行脱盐。采用自动部分收集器收集样品,每管3 mL。肝素酶解后会在酶解位点的非还原端的糖醛酸的5位和6位形成不饱和双键,在232nm有最大紫外吸收。故在232 nm波长处检测,根据洗脱曲线按峰合并收集样品。
2.2 肝素四糖的分离纯化质谱条件:喷雾电压4.8 kV,毛细管电压-40 V,套管透镜电压-50 V,毛细管温度275 ℃。对P2柱洗脱下来的峰进行质谱分析,收集含有肝素四糖的峰。
半制备色谱条件:采用Spherisorb S5离子交换色谱柱,流动相A为去离子水(用盐酸调节pH到3.5),流动相B为pH3.5的2 mol·L-1氯化钠溶液,采用梯度洗脱的方法对样品进行洗脱(0~20 min 0%B;20~80 min 0%B→20%B;80~100 min 60%B),流速为4 mL·min-1,检测波长为232 nm。由于肝素是含有糖醛酸和硫酸基的聚阴离子结构,选择半制备强阴离子交换高效液相色谱(strong anion exchange HPLC,SAX-HPLC)进行进一步的分离纯化。由于SAX-HPLC无法确定出峰信号的结构信息,所以对分离纯化样品按峰收集,使用P2柱脱盐后冷冻干燥备用。继续采用质谱检测的方法以确定含有肝素四糖的峰。含有肝素四糖的峰还需进行一步确定是否为3位硫酸化的肝素四糖。纯化后四糖接着进行核磁共振波谱(NMR)和二级质谱(MS/MS)来确定肝素四糖的结构[8]。
2.3 NMR分析四糖结构通过质谱确定为肝素四糖后,对样品进行NMR分析。样品冷冻干燥后(4 mg)溶解在400 μL的重水中(99.96),1H NMR、HSQC NMR、1H-1HCOSY和TOCSY均在核磁共振波谱仪上使用Topspin 2.1.6处理软件分析获得。分析温度为25 ℃,氢谱扫描次数为32次,虚拟扫描为0次,谱图宽度为12×10-6,Fid大小为16 384;二维核磁扫描为32次,虚拟扫描为32次,色谱宽度为10×10-6,Fid大小为8012。
2.4 MS/MS分析四糖结构肝素四糖的二级质谱分析采用电喷雾傅立叶变换离子回旋共振高分辨质谱,配备间接加热阴极电子发射器。将待测样品溶在50:50的甲醇水溶液中(0.1 mg·mL-1)。采用负离子模式进行检测,电喷雾采用金属毛细管,喷雾电压4.8 kV,毛细管温度100 ℃,源温80 ℃,锥孔电压-7.0 V,检测激发电压120 V。谱图采集采用全扫描一级质谱,多价先驱母离子通过外部四极杆分离后在碰撞室采用碰撞诱导解离获取的二级质谱。选择窗口设定为±3.0 Da。碰撞激发电压2.0 V。碰撞气压为5.98 kPa。外部校准精度为5×10-6,内部校准采用糖苷键断裂归属清楚的产物离子,内部校准精度一般均小于1×10-6。记录信噪比(S/N)大于10的峰信号。所有的二级质谱产物采用传统的Domon and Costello命名法则(碎片离子A、B、C和X、Y、Z)。除了使用斜线区分还原端和非还原端片段以外,末端圆圈表示失去1个硫酸基团(SO3),斜线后数字表示来源的片段存在几个钠离子的交换[9-10]。
2.5 肝素四糖离子强度的对比分析与四糖的分离纯化相同,同样采用SAX-HPLC,以TriS(ΔUA2S-GlcNS6S)为对照品,根据相同条件下出峰时间的先后顺序来对比肝素四糖的离子强度。洗脱程序为流动相B的浓度:0~100 min 25%B→50%B)[11]。
2.6 肝素四糖与AT Ⅲ的结合作用采用竞争型表面等离子体共振(SPR)的方法研究肝素四糖与AT Ⅲ的结合作用。AT Ⅲ(50 nmol·L-1)与5 μg·mL-1肝素四糖溶在HBS-EP缓冲溶液[0.01 mol·L-1羟乙基哌嗪乙硫磺酸,0.15 mol·L-1氯化钠,3 mmol·L-1乙二胺四乙酸,和0.005%表面活性剂聚山梨酯20,(pH 7.4)]中。该混合体系以30 μL·min-1流速流经固定化肝素芯片,记录AT Ⅲ和固定化肝素的相互作用[12]。
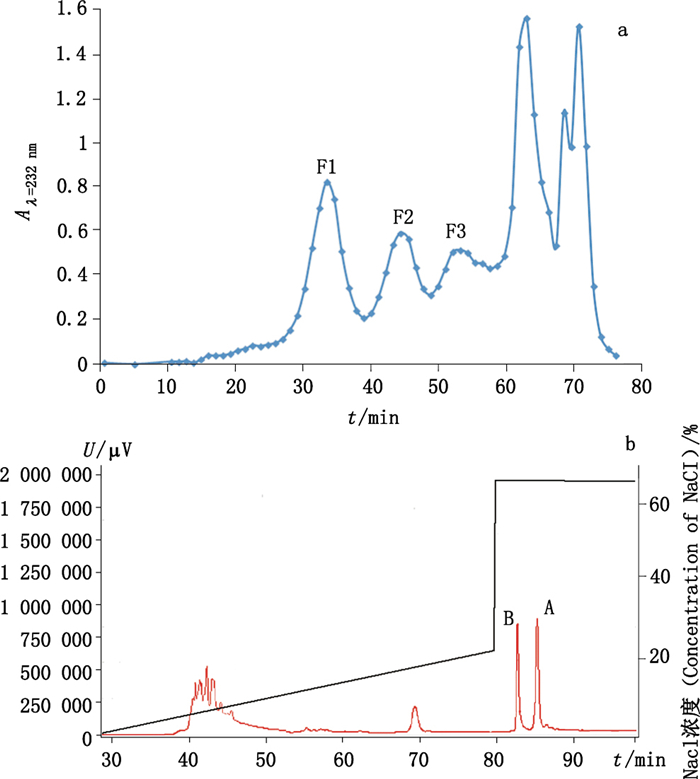
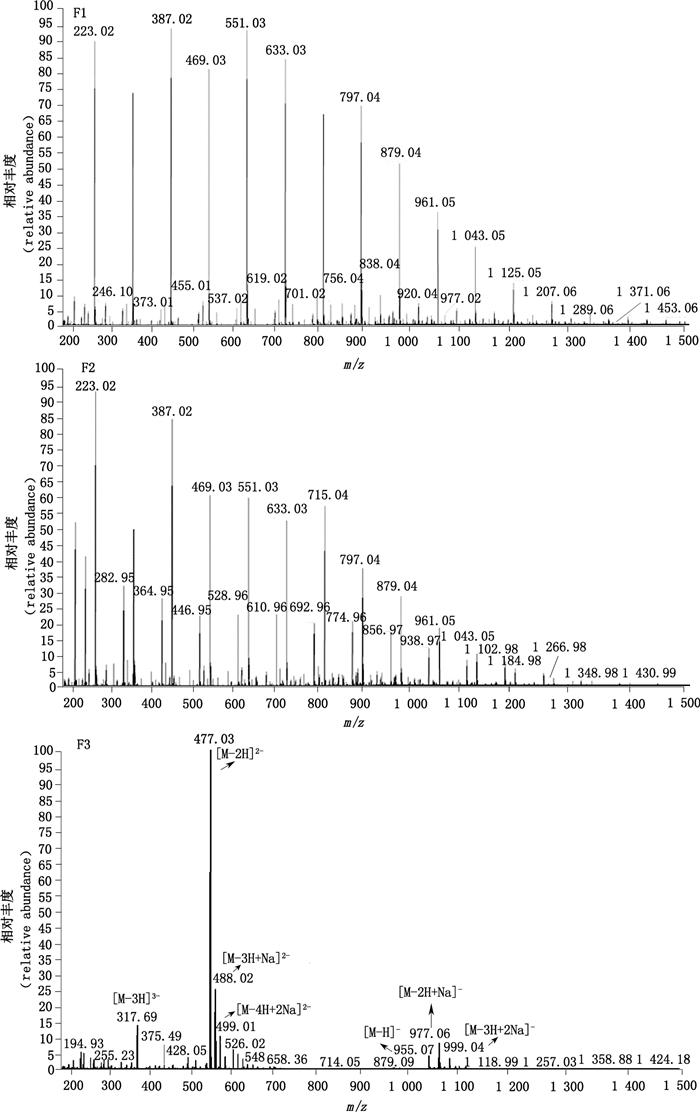
3 结果与讨论 3.1 肝素四糖的分离纯化肝素完全酶解后,通过P2柱对其进行分离纯化,分离纯化图如图 2-a所示,对各个组分通过质谱推断其组成。F1和F2组分质谱峰比较复杂,无法分析比对出肝素相关成分,推测其可能为肝素酶降解而得到的多肽成分。F3主要含有分子离子峰为m/z 955.07的片段及其加钠峰,并含有双电荷和三电荷的组峰(如图 3所示),根据肝素的结构信息判断该片段为ΔUA-GlcN-HexUA-GlcN+Ac+3SO3,也就是肝素四糖带1个乙酰基和3个硫酸基修饰。
|
图 2 肝素完全酶解产物的Bio gel P2凝胶渗透色谱图(a)和离子交换液相色谱图(b) Figure 2 The digestion products were purified by size exclusion chromatography (a)(Bio gel P2) to recover the crude tetrasaccharides.The purification (b) of tetrasaccharides by SAX-HPLC |
|
图 3 凝胶纯化含有肝素四糖组分的质谱图 Figure 3 MS of tetrasaccharides-containing fraction F3 |
将含有肝素四糖的组分进行进一步的SAX-HPLC分离纯化(图 2-b),收集各峰进行质谱分析,发现2个相近的单一组分分子离子峰(A、B)均为m/z 9955.07,说明是一对具有同分异构的肝素四糖。但无法确定是否为3位硫酸化的肝素四糖。
3.2 肝素四糖的二级质谱分析ESI-MSn由于灵敏性高,并且可以使肝素离子化成负离子,产生高电荷状态,稳定硫酸基团,避免硫酸基团的脱落而影响对硫酸基团位点的判断,已经广泛地应用于肝素寡糖的结构分析,特别是CID-MSn模式[13-14]。肝素是高硫酸化糖胺聚糖,同一个糖基上会有多个硫酸化位点,由于电荷排斥作用,同一个糖基上所有的硫酸基团很难达到全部离子化。而不全部离子化或者与金属离子配对,由于氢的重排,硫酸基团很容易脱落。所以ESI的溶剂既要保证能够进行钠离子交换而产生稳定的先驱离子,又不能降低离子化效率。所以在肝素四糖的甲醇水溶剂中按1:100(v/v)添加1% 100 μmol·L-1氢氧化钠来产生钠离子加合物。
肝素四糖A的MS/MS先驱离子质荷比m/z是332.341 6,为[M-5H+2Na]3-,表明该四糖所有可离子化质子均被离子化或取代。通过环间糖苷键断裂可以确定硫酸基团和乙酰基团在肝素四糖中的分布(图 4),如通过C1/B1和C2/B2的差值确定中间的氨基糖含有乙酰基和硫酸基团,Y1和Z1确定还原端含有2个硫酸基。最终确定四糖A应为ΔUA-GlcNAc+SO3-HexUA-GlcN+2SO3,表明硫酸基团均分布在肝素四糖的氨基糖上,糖醛酸中不含有硫酸基团的取代。还原端的氨基糖含有2个硫酸基团,中间氨基糖含有1个硫酸基团。离子片段2,4A2和0,2A2或者0,3A2和B2的差值可确定中间GlcNAc的硫酸基团在6位。还原端的2个硫酸基团位置的确定也是以此而定。还原端的氨基没有乙酰基取代就推测1个硫酸基取代发生在氨基上,质谱断裂也进一步确定。C3和2,4A4的差值可以确定另外1个硫酸基团在3位。所以肝素四糖A是3位硫酸化的肝素四糖[15]。肝素四糖A的MS/MS具体数据如表 1所示。
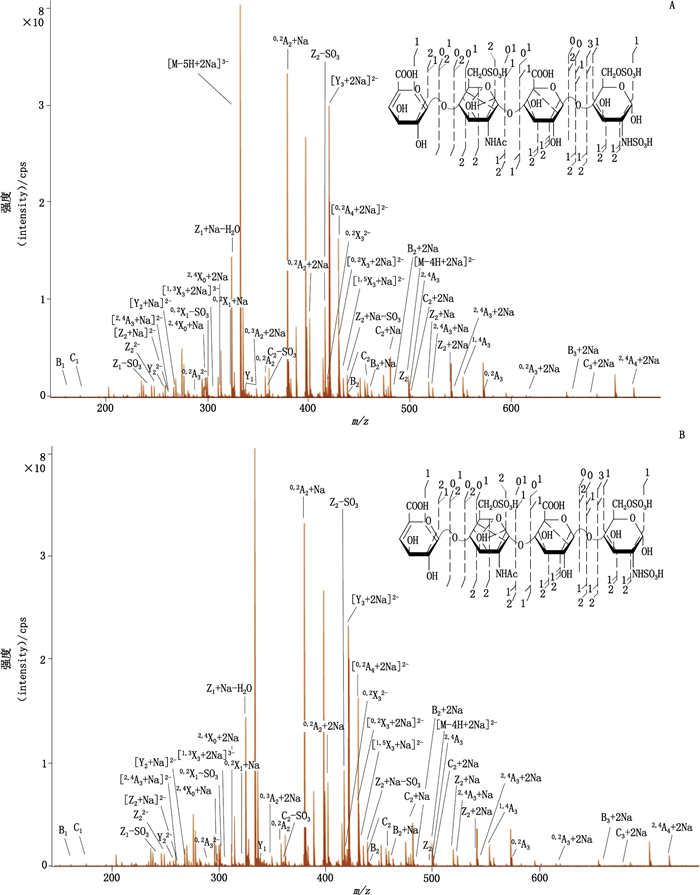
|
图 4 肝素四糖ΔUA-GlcNAc6S-GlcA-GlcNS3S(A)和ΔUA-GlcNAc6S-GlcA-GlcNS6S(B)的二级质谱图 Figure 4 MS/MS and structural annotations for heparin tetrasaccharideΔUA-GlcNAc6S-GlcA-GlcNS3S (A) and ΔUA-GlcNAc6S-GlcA-GlcNS6S (B) |
|
|
表 1 肝素四糖A和B的质谱解析 Table 1 Mass list for annotated structures of hepain tetrasaccharides A and B |
同理肝素四糖B也通过同样的方法确定为ΔUA-GlcNAc+SO3-HexUA-GlcN+2SO3,硫酸基团和乙酰基的分布与肝素四糖A完全一致。还原端的氨基糖含有2个硫酸基团,中间氨基糖含有1个硫酸基团。中间氨基糖的硫酸基团也是取代在6位。还原端氨基糖硫酸基团位置的确定通过2,4X0和0,2X0就很容易判断出来,在2位的氨基和6位。肝素四糖B的MS/MS具体数据如表 1。
肝素四糖A和B的结构非常相似,只是还原端的硫酸基修饰位点有所区别,A是NS3S取代,B是NS6S取代。所以通过肝素四糖A和B的对比分析就可以看出,MS/MS及CID模式可以提供足够的信息用于分析肝素寡糖的结构,特别是乙酰基和硫酸基的修饰位点,而且样品消耗少,灵敏准确,在区别相似结构的寡糖上有其独特的优势,见图 4。
3.3 肝素四糖的核磁共振波谱分析质谱技术在分析肝素这类具有固定连接方式但官能团修饰位点多变的化合物上优势明显,但是很难通过质谱区别肝素中的葡萄糖醛酸和艾杜糖醛酸[16]。所以还要通过NMR技术进一步验证肝素四糖A和B的结构,确定糖醛酸的组成,并研究不同取代位点对糖环化学位移的影响。
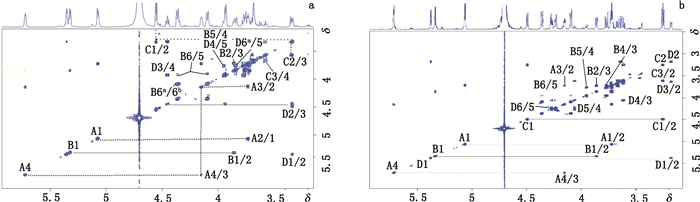
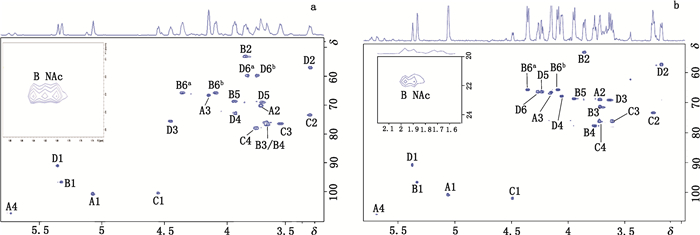
肝素四糖从非还原端到还原端4个糖基分别命名为A、B、C和D(图 5),也就是非还原端不饱和糖醛酸ΔUA(A),内部的氨基糖GlcN(B),内部的糖醛酸GlcA(C)和还原端GlcN(D)。对核磁共振波谱的解析需要首先确定各糖基的端基H的信号,再进一步实现糖环上碳氢的全归属,通过比对可确定修饰的基团及修饰的位点。首先δ 5.7低场位移的信号,不是一般端基氢可以达到,通过对四糖结构的分析确定其为非还原端不饱和糖醛酸ΔUA 4位不饱和双键上的H信号,也就是A4[17]。确定后通过H-H COSY(图 6)和TOCSY(图 7)确定A4和A1之间的相关信号,所以先确定糖环A的端基信号。通过HSQC(图 8)可以知道δ 4.5处的信号和100.3的碳信号相关,确定其为端基氢信号。此信号位于如此高场,通过和肝素的NMR数据比对可知,其为内部葡萄糖醛酸的端基信号,由于是β构型,所以位于高场。通过这一信息就确定肝素四糖中糖醛酸为葡萄糖醛酸,而非艾杜糖醛酸,确定C的信号。B和D端基氢化学位移非常接近,难以判断,但是二者相关的端基碳信号的区别非常大,1个在δ 91,1个在δ 97左右。在δ 91处可以确定是还原端GlcN的端基碳信号,所以位于高场,与之在HSQC对应的就是D的端基氢D1,而另外一个就是B的端基碳,所对应的是B1。

|
图 5 肝素四糖A(a)和B(b)的结构式与1D 1H NMR图 Figure 5 Structures and 1D 1H NMR of heparin tetrasaccharides A(a) and B(b) |
|
图 6 肝素四糖A(a)和B(b)的1H-1H COSY图 Figure 6 1H-1H COSY of heparin tetrasaccharides A(a) and B(b) |

|
图 7 肝素四糖A(a)和B(b)的1H-1H TOCSY图 Figure 7 1H-1H TOCSY of heparin tetrasaccharides A (a) and B (b) |
|
图 8 肝素四糖A(a)和B(b)的HSQC图 Figure 8 HSQC of heparin tetrasaccharides A (a) and B (b) |
确定好肝素四糖各糖基的端基氢的信号后,再根据二维核磁实现了各糖基糖环上信号的全归属。数据如表 3所示。将结果和肝素的核磁数据比对后确定肝素四糖的结构[18-20]。一般而言,对于寡糖类物质由于端基效应,还原端的糖基一般会同时出现α-和β-2种端基氢信号,而本文中发现的2种肝素四糖,还原端的GlcN均只发现了α-构型的端基信号。这就推测可能是由于还原端2位的氨基发生硫酸化修饰,由于硫酸基团的空间位阻而抑制了端基氢的自由转换,说明肝素四糖A和B还原端的GlcN均为硫酸化修饰。而通过核磁分析也进一步验证这一点。内部的GlcNAc,乙酰化后H2 δ为3.81,和肝素的数据非常接近。而还原端的GlcN H2的δ只有3.29,说明2位硫酸化修饰会使GlcN的H2的δ大大向高场位移。通过肝素四糖A的还原端GlcN和中间GlcNAc的6位核磁信号对比可以看出,还原端GlcNS的H6 δ是3.73和3.81,C6为59.8,而内部GlcNAc的H6为δ 4.32和4.05,C6为65.8,H6和C6均明显向低场位移,说明内部的GlcNAc为6位硫酸化。肝素四糖A和B还原端GlcN的H3和C3也存在很大的区别。化合物A的H3 δ为4.43,接近于GlcUA端基H的化学位移,相对于四糖B的H3(δ 3.56),有非常明显的低场位移。C3也有明显低场位移,所以判断化合物A的3位硫酸化,A为3位硫酸化的肝素四糖,结构为ΔUA-GlcNAc6S-GlcUA-GlcN3S6S。化合物B的H3和C3与内部GlcNAc差别不大,略微向高场位移,而6位则同样向低场位移至δ 4.3和4.0作用,所以肝素四糖B为6位硫酸化,其结构为ΔUA-GlcNAc6S-GlcUA-GlcNS6S。
|
|
表 3 肝素四糖的化学位移(δ)归属和耦合常数(D2O,25 ℃) Table 3 Chemical shifts (δ) and coupling constants of heparin tetrasaccharides |
结合肝素的核磁数据和二级质谱的分析,最终确定这一对肝素四糖的确切结构。通过肝素四糖A和B及同种四糖内部GlcN和还原端的GlcN核磁数据进行比较发现,当硫酸化修饰后,3位6位的碳氢的化学位移均向低场位移,而2N位被硫酸基取代后H2的化学位移向高场位移,C2向低场位移,这有可能是因为官能团的空间位阻所影响。3位和6位硫酸基团取代后对相应的4位和5位的化学位移也会产生一定的影响。
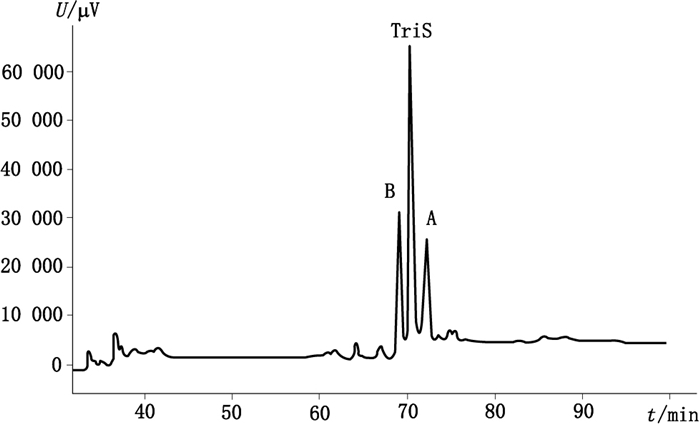
3.4 肝素四糖的离子强度的比较肝素四糖A和B除了还原端的硫酸根的取代位点有所区别以外(A为3位硫酸化。B为6位硫酸化),其他部分均一致,所以是结构非常类似的1对肝素四糖,为研究二者离子强度的区别,仍然采用SAX-HPLC的方法,采用细致缓和的盐浓度提速变化,以肝素二糖TriS为对照。在pH 3.5的流动相中肝素中的糖醛酸的羧基均为非离子状态,所以离子强度决定于硫酸基的个数和电荷密度。三者均含有3个硫酸基团。如图 9所示,三者的洗脱先后顺序是肝素B,TriS和肝素A。说明虽然硫酸基团个数相同,肝素A的硫酸基团更加集中,局部电荷密度大,而肝素四糖B电荷密度更加分散。所以从离子交换柱的洗脱顺序看,3位硫酸化的肝素四糖A比B要结合更紧密,离子强度更大,这也是这一对同分异构体能够分开的原因。
|
图 9 肝素四糖A、B和肝素二糖Tris的离子交换高效液相色谱图 Figure 9 SAX-HPLC of heparin tetrasaccharides A, B and Tris |
肝素通过肝素酶完全酶解后产生了3位硫酸化的四糖,与理论一致,但是仍然从中分离得到了非3位硫酸化的四糖,可能是由于酶解时间不够或者加酶量过低使酶解不充分所致。
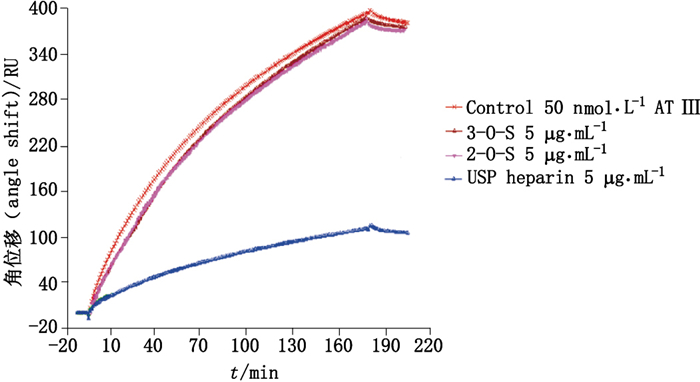
3.5 肝素四糖与AT Ⅲ的结合作用使用生物素化并固定化的肝素芯片,通过竞争性SPR技术研究肝素四糖与AT Ⅲ的相互作用。首先将肝素四糖与AT Ⅲ进行混合,再与固定化的肝素进行相互作用。一旦肝素四糖与AT Ⅲ结合,占据了肝素结合位点,AT Ⅲ与固定化肝素相互作用的强度将会下降,信号减弱。结果如图 10所示,不论是3位硫酸化肝素四糖还是其同分异构体均未与AT Ⅲ结合,产生相互作用,说明肝素四糖不存在抗凝血活性。这也说明3位硫酸化GlcN是肝素与AT Ⅲ结合产生相互作用的先决条件,但是必须有一定的长度才能发挥作用,这也验证了3位硫酸化修饰五糖是肝素活性的最小单位。
|
图 10 肝素四糖A和B与肝素的竞争型表面等离子体共振 Figure 10 Competition SPR of the two heparin tetrasaccharides A and B |
从肝素的完全酶解产物中分离纯化出1个与肝素生物活性位点处的3位硫酸化肝素四糖和它的同分异构体。MS/MS结合NMR确定二者的结构分别为ΔUA-GlcNAc6S-GlcUA-GlcNS3S和ΔUA-GlcNAc6S-GlcUA-GlcNS6S。虽然二者在结构上非常相似,但是ΔUA-GlcNAc6S-GlcUA-GlcNS3S的离子强度要略高于ΔUA-GlcNAc6S-GlcUA-GlcNS6S。二者均没有结合AT Ⅲ的能力。通过3位硫酸化肝素四糖和其同分异构体的对比分析,获得3位硫酸化肝素四糖的结构特征,为研究不同来源肝素中3位硫酸化功能域的区别,也为比较人工合成肝素与天然肝素的区别提供参考。
| [1] |
LINHARDT RJ, CLAUDE S. Hudson Award address in carbohydrate chemistry.Heparin:structure and activity[J]. J Med Chem, 2003, 46(13): 2551. DOI:10.1021/jm030176m |
| [2] |
MOON AF, EDAVETTAL SC, KRAHN JM, et al. Structural analysis of the sulfotransferase (3-o-sulfotransferase isoform 3) involved in the biosynthesis of an entry receptor for herpes simplex virus 1[J]. J Biol Chem, 2004, 279(43): 45185. DOI:10.1074/jbc.M405013200 |
| [3] |
姬胜利, 桑青, 张天民. 肝素的来源控制、结构分析以及结构与生物活性关系的研究进展[J]. 中国药学杂志, 2012, 22(9): 660. JI SL, SANG Q, ZHANG TM. Heparin source control, structural analysis, as well as the structure and biological activity relationship study progress[J]. Chin Pharm J, 2012, 22(9): 660. |
| [4] |
ONISHI A, KALIB SA, DORDICK JS, et al. Heparin and anticoagulation[J]. Front Biosci, 2016, 21(7): 1372. DOI:10.2741/4462 |
| [5] |
YAMADA S, YOSHIDA K, SUGIURA M, et al. Structural studies on the bacterial lyase-resistant tetrasaccharides derived from the antithrombin Ⅲ-binding site of porcine intestinal heparin[J]. J Biol Chem, 1993, 268(7): 4780. |
| [6] |
PETITOU M, CASU B, LINDAHL U. 1976-1983, a critical period in the history of heparin:the discovery of the antithrombin binding site[J]. Biochimie, 2003, 85(1-2): 83. DOI:10.1016/S0300-9084(03)00078-6 |
| [7] |
LI G, YANG B, LI L, et al. Analysis of 3-O-sulfo group-containing heparin tetrasaccharides in heparin by liquid chromatography-mass spectrometry[J]. Anal Biochem, 2014, 455(1): 3. |
| [8] |
MOURIER P, ANGER P, MARTINEZ C, et al. Quantitative compositional analysis of heparin using exhaustive heparinase digestion and strong anion exchange chromatography[J]. Anal Chem Res, 2015, 3(C): 46. |
| [9] |
林江慧, 张建伟, 肖晓毛, 等. 含N-乙酰化肝素寡糖的制备及序列分析[J]. 分析化学, 2015, 43(5): 689. LIN JH, ZHANG JW, XIAO XM, et al. Preparation and sequencing of N-acetylated heparin oligosaccharides[J]. Chin J Anal Chem, 2015, 43(5): 689. |
| [10] |
LEACH F, XIAO Z, LAREMORE TN, et al. Electron detachment dissociation and infrared multiphoton dissociation of heparin tetrasaccharides[J]. Int J Mass Spectrom, 2011, 308(2-3): 253. DOI:10.1016/j.ijms.2011.08.029 |
| [11] |
何传波, 李琳, 汤凤霞, 等. 巴戟天多糖在离子交换填料上吸附性能的研究[J]. 食品科学, 2009, 30(15): 109. HE CB, LI L, TANG FX, et al. Adsorption properties of poly saccharides from Morinda officinalis onto ion exchang absorbent[J]. Food Sci, 2009, 30(15): 109. DOI:10.3321/j.issn:1002-6630.2009.15.024 |
| [12] |
JING Z, LIU X, MALHOTRA A, et al. Novel method for measurement of heparin anticoagulant activity using SPR[J]. Anal Biochem, 2017, 526(1): 39. |
| [13] |
郭明全, 宋凤瑞, 陈貌连, 等. 刺五加寡糖的电喷雾多级串联质谱研究[J]. 高等学校化学学报, 2003, 24(9): 1577. GUO MQ, SONG FR, CHEN ML, et al. Studies on oligosaceharides from Acanthopanax senticosus Harms(AS) by eIectrospray ionization tandem mass spectrometry(MS)[J]. Chem J Chin Univ, 2003, 24(9): 1577. DOI:10.3321/j.issn:0251-0790.2003.09.008 |
| [14] |
陈欢欢, 赵峡, 栾晓红, 等. 电喷雾质谱在寡糖序列分析中的应用[J]. 高等学校化学学报, 2015, 36(1): 1. CHEN HH, ZHAO X, LUAN XH, et al. Application of electrospray tandem mass spectrometry in sequence analysis of oligosaccharides[J]. Chem J Chin Univ, 2015, 36(1): 1. |
| [15] |
闫存玉, 俞飚, 刘淑莹, 等. 类肝素化合物的傅立叶变换离子回旋共振-电喷雾质谱研究[J]. 分析化学, 2008, 36(7): 910. YAN CY, YU B, LIU SY, et al. Characterization of synthetic heparin-like compounds by negative electrosprary ionization fourier transform ion cycloron resonance mass spectrometry[J]. Chin J Anal Chem, 2008, 36(7): 910. DOI:10.3321/j.issn:0253-3820.2008.07.009 |
| [16] |
WOLFF JJ, CHI L, LINHARDT RJ, et al. Distinguishing glucuronic from iduronic acid in glycosaminoglycan tetrasaccharides by using electron detachment dissociation[J]. Anall Chem, 2007, 79(5): 2015. DOI:10.1021/ac061636x |
| [17] |
SHRIVER Z, SUNDARAM M, VENKATARAMAN G, et al. Cleavage of the antithrombin Ⅲ binding site in heparin by heparinases and its implication in the generation of low molecular weight heparin[J]. Proceed Nat Acad Sci USA, 2000, 97(19): 10365. DOI:10.1073/pnas.97.19.10365 |
| [18] |
XIAO Z, TAPPEN BR, LY M, et al. Heparin mapping using heparin lyases and the generation of a novel low molecular weight heparin[J]. J Med Chem, 2011, 54(2): 603. DOI:10.1021/jm101381k |
| [19] |
FU L, ZHANG FM, LI GY, et al. Structure and activity of a new low molecular weight peparin produced by enzymatic ultrafiltration[J]. J Pharml Sci, 2014, 103(5): 1375. DOI:10.1002/jps.23939 |
| [20] |
CHEN Y, LIN L, AGYEKUM I, et al. Structural analysis of heparin-derived 3-O-sulfated tetrasaccharides:antithrombin binding site variants[J]. J Pharm Sci, 2017, 106(4): 973. DOI:10.1016/j.xphs.2016.11.023 |
 2018, Vol. 38
2018, Vol. 38 
