2. 国家药典委员会, 北京 100061
2. Chinese Pharmacopoeia Commission, Beijing 100061, China
16S核糖体RNA基因(16S ribosomal RNA, 16S rRNA基因)用于细菌核酸测序鉴定已被学术界广泛认可, 《伯杰氏系统细菌学手册》(Bergey’s Manual of Systematic Bacteriology)[1], 美国药典40版通则<1113>(USP 40<1113>)[2]、日本药局方17版通则G4[3]、《中华人民共和国药典》2015年版通则(9204)[4]等国内外药品质量标准, 公开发表的研究论文[5]等均将16S rRNA基因作为细菌核酸水平鉴定的依据。16S rRNA基因全长约1 500 bp, 包含9个可变区(variable region, V区)和10个恒定区(constant region, C区), 在结构与功能上具有高度保守性, 适用于进化距离不同的各类细菌亲缘关系研究。
随着药品“全生命周期”质量控制的理念逐渐推广, 加之微生物污染的偶然性、难受控和非人为因素等, 制药行业中微生物的质量控制正逐渐从终产品控制向过程控制转变, 环境监控、偏差调查、溯源分析中微生物核酸水平鉴定的规范化和标准化备受关注。
目前, 16S rRNA基因核酸测序鉴定在药品质量控制中的应用尚需要开展以下工作:一是16S rRNA基因全长涵盖了最全面的菌株遗传进化信息, 但受到核酸测序方法操作性, 测序质量, 测序成本, 测序反应长度特别是高通量核酸测序反应读长等的限制, 多数文献报道还是采用部分核酸序列(partial sequence)进行研究, 具有鉴定和分类意义的区域尚不明确, 如: USP 40<1113>中认为16S rRNA基因5’端500 bp对于细菌“种”水平的鉴定更具有意义[2];Chan等[5]研究报道在临床分离微生物的种群鉴定时, V1~V2区更具有分类意义;Kataoka等[6]在链霉素分类鉴定中采用V1~V3区作为鉴定靶点;Becker等[7]在葡萄球菌分类鉴定中采用V1~V3区和V1~V8区的比较方法;Engelbrektson等[8]利用Roche 454核酸测序平台研究, 发现V1、V2和V8区具有更多的特异性位点;Kim等[9]借助OTUs(operational taxonomic units)分析的理论, 认为V1~V3区和V1~V4区在微生物菌群鉴定中更有分类意义;Chakravorty等[10]研究发现, 16S rRNA基因中V2、V3、V6区核酸序列串联可实现多数细菌“种”水平的鉴定和分类;Lu等[11]利用PCR和RFLP方法对于临床脑脊液分离的微生物菌群进行了分类鉴定, 认为16S rRNA基因中V4~V9区分辨率较低。二是将DNA条形码分子鉴定[12-13]与16S rRNA基因核酸测序鉴定规范法相结合, 实现细菌核酸水平鉴定的标准化, 是提升药品质量控制水平的有效手段, 但目前药品质量控制中典型细菌污染物16S rRNA基因核酸测序鉴定的规范化方法尚缺乏基础性研究数据的佐证。三是16S rRNA基因核酸测序的鉴定结果需要借助参比序列予以判定, 参比序列大多来源于NCBI、EMBL、DDBJ等开放性公用数据库, 很多非模式菌株、野生型分类株等核酸序列数据的样本信息不完整, 无法确认核酸序列所代表物种的真实性和有效性, 使得核酸测序的鉴定结果没有权威、标准化的判定依据[14]。四是目前的研究报道大多集中于临床分离致病菌和微生物菌群, 药品生产环境、制药用水系统、生物膜等微生物污染物的鉴定和分布规律的研究鲜有报道。
本文以制药行业典型致病菌和细菌污染物为研究对象, 收集整理菌株核酸水平的鉴定数据, 对于16S rRNA基因中各V区序列片段进行比较, 分析等位基因数目和单核苷酸多态性位点(SNP位点)数量;并以葡萄球菌为例进行菌株的核酸测序鉴定及序列聚类分析, 对16S rRNA基因核酸测序规范化方法及在制药行业中微生物污染控制的应用进行探讨, 为药品微生物质量控制中的细菌核酸测序鉴定标准化、标准核酸序列数据库、药品质量标准提高等方面提供研究思路和参考依据。
1 材料与方法 1.1 Genbank核酸数据库中菌株序列的筛选、片段定位登录Genbank核酸数据库网站(www.ncbi.nlm.nih.gov), 在Taxonomy项下检索制药行业常见细菌污染物的菌属名称, 如:葡萄球菌(Staphylococcus)、假单胞菌(Pseudomonas)、大肠埃希菌(Escherichia)、沙门菌(Salmonella)、肠球菌(Enterococcus)、链球菌(Streptococcus)、克雷伯菌(Klebsiella)、军团菌(Legionella)、芽孢杆菌(Bacillus)、微球菌(Micrococcus)、梭菌(Clostridium)等, 选择各类菌属16S rRNA基因核酸序列, 进一步选择序列长度在1 400~1 600 bp之间, 来源于美国微生物菌种保藏中心(ATCC)等权威机构标准菌株的参比序列(reference sequence, RefSeq), 下载核酸序列的fasta文件。
将下载后的序列导入Mega序列分析软件, 以菌株16S rRNA基因全长序列(full sequence)为参照, 将序列范围未完全覆盖并存在兼并碱基的序列, 未经确认的序列(unverified sequence)及随机打断序列(shotgun sequence)剔除, 获取可信度较高的核酸序列片段;将获取的核酸序列以C区定位, 分别截取序列长度相近(约为500 bp)、覆盖V1~V3、V2~V4、V3~V5、V4~V6、V7~V9区的核酸序列。
1.2 16S rRNA基因可变区核酸序列多样性和聚类关系分析分别将片段定位后的核酸序列导入DNASP序列分析软件, 分析各组序列中等位基因和SNP位点的数量, 评价不同V区的序列片段对细菌“种”水平的鉴定能力。以葡萄球菌为例, 分别将筛选得到的核酸序列导入Mega序列分析软件, 以“Neighbor Joining Clustering Method”方法进行序列比对, 以Bootstrap Method(1000)、Kimura 2-Parameter Model、Uniform Rates、Pairwise Deletion的统计学计算方式[15-16], 构建NJ进化树(Neighbor-Joining tree), 聚类分析各类菌属之间的进化关系。
1.3 试验菌株葡萄球菌标准菌株(共20株)分别购自美国微生物菌种保藏中心(ATCC)、中国医学细菌菌种保藏管理中心(CMCC)、中国工业微生物菌种保藏管理中心(CICC);药品生产和检验环境中的葡萄球菌分离株(共111株)为本实验室保藏。上述菌株以《中华人民共和国药典》2015年版通则(1106)方法进行分离鉴定, 以胰酪大豆胨琼脂培养基(TSA)纯化后得到单菌落, 以革兰染色和生化鉴定方法确认菌株遗传信息。
1.4 16S rRNA基因核酸测序鉴定挑取单克隆菌落, 采用细菌基因组DNA小量纯化试剂盒(TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit)提取菌株基因组DNA;分别以引物27F(5’-AGAGTTTGATCMTGGCTCAG-3’)和经设计改良的519R(5’-GTATTACCGCGGCTGCTGGC-3’)扩增葡萄球菌16S rRNA基因中V1~V3区核酸序列[9];将PCR产物进行琼脂糖凝胶电泳, 将PCR阳性扩增产物进行双向核酸测序(委托上海生工生物工程有限公司测序);以Bioedit软件校核测序峰图的质量, 以Lasergene软件拼接核酸序列;将拼接完成的核酸序列录入到Genbank数据库中进行Blast比对分析, 判定鉴定结果(同源性>98.65%的判定为同一菌种)[17], 同时将测序结果导入Mega软件, 以经过筛选的Genbank数据库中葡萄球菌参比序列为参考, 定位并截取长度相同的序列后, 参照“1.2”项下实验方法, 聚类分析葡萄球菌属内“种”之间的进化关系。
2 实验结果 2.1 16S rRNA基因可变区序列比较分析本文通过对Genbank公共核酸数据库中序列的筛选, 共得到了涵盖11个常见种属共264条序列, 菌株分类信息(见表 1)。分别以C区定位, 截取序列长度约为500 bp的V1~V3、V2~V4、V3~V5、V4~V6、V7~V9区的核酸序列, 分析不同区域序列片段中等位基因和SNP位点的数量, 如表 2中结果所示:各组V区组合的序列片段均含有不同数量的等位基因和SNP位点, 除V1~V3区的序列外, 各区域组合的序列片段长度相似, 其中V3~V5区(含有162个等位基因222个SNP位点)与V4~V6区(含有178个等位基因205个SNP位点)的序列特征相似;与前两者相比, V2~V4区含有195个等位基因251个SNP位点, 提示该区域包含较多的鉴定和分类信息;V7~V9区也含有199个等位基因, 但SNP位点的数量与等位基因相同, 提示碱基发生突变的概率较单一。
|
|
表 1 本文用于16S rRNA基因片段分析的菌株信息表 Table 1 Bacterial 16S rRNA gene sequences used in this study |
|
|
表 2 药品生产和检验环境中常见细菌污染物16S rRNA基因序列中特异性位点分析 Table 2 Specific analysis of 16S rRNA gene segments in typical bacterial contaminants of drug production and monitoring environment |
与上述区域的序列片段不同, 表 2中单独分析了V1~V3区的序列片段, 数据统计显示:不同菌属的核酸序列在V1~V3区差别较大, 除含有不同数量的等位基因和SNP位点外, 不同菌属的序列长度也不完全相同, 其中大肠埃希菌、沙门菌、克雷伯菌、假单胞菌和军团菌的序列长度相似, 分析得到的63条序列中包含了54个等位基因和168个SNP位点;葡萄球菌(50个等位基因88个SNP位点)、链球菌(22个等位基因110个SNP位点)、芽孢杆菌(20个等位基因100个SNP位点)的序列长度相似, 但各个“属”之间的序列差异较大, 与前一组的序列相比均有小片段的插入;肠球菌(16个等位基因92个SNP位点)中有较长的插入片段, 而微球菌(6个等位基因27个SNP位点)和梭菌(29个等位基因114个SNP位点)则有不同程度的序列缺失, 结果提示: V1~V3区包含更多的鉴定和分类信息, 不同菌属之间的等位基因和SNP位点也更有特异性。以上结果表明:以制药行业中常见的11个属细菌污染物为研究对象, 16S rRNA基因中的各可变区片段组合可以有效地进行菌株“属”水平的鉴定, 其中V1~V3区的鉴定效果更显著, 可以最大限度地揭示菌株核酸水平的鉴定能力。
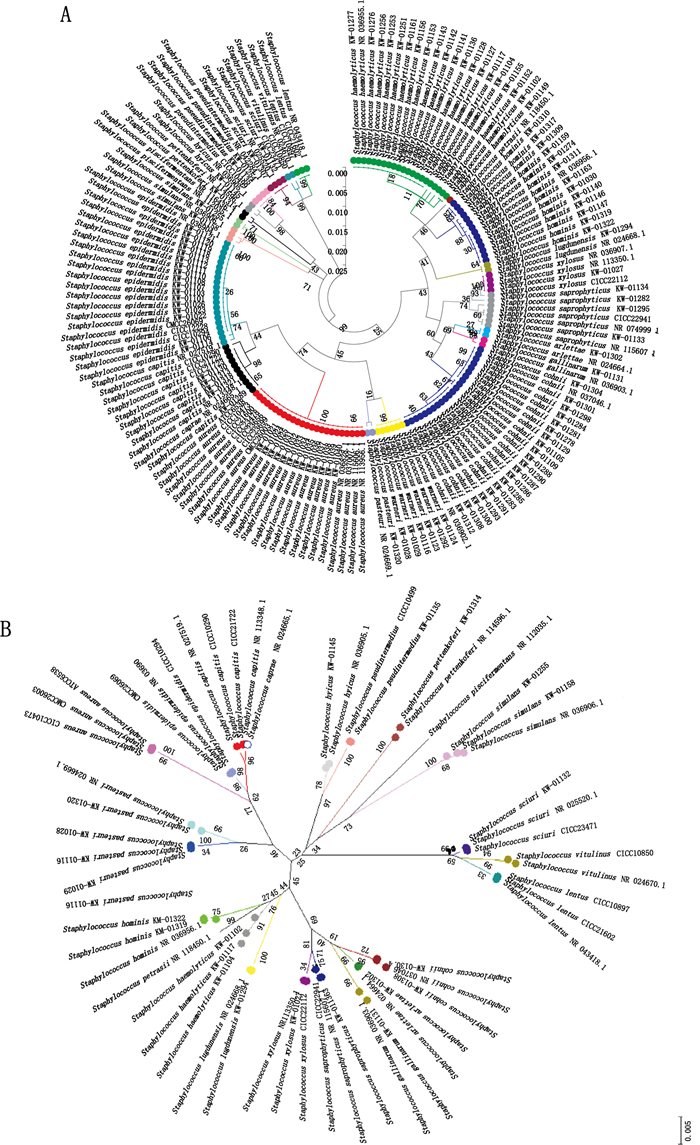
2.2 葡萄球菌16S rRNA基因核酸测序鉴定与聚类分析本文收集整理葡萄球菌标准株(20株)和实验室分离株(111株, 覆盖葡萄球菌属20个“种”), 在确证菌株遗传背景的基础上, 以V1~V3区为靶点进行核酸测序鉴定。鉴定结果经过测序峰图信号强度和特异性校核后进行拼接, 以测序引物为两端定位, 剔除引物区序列片段, 建立标准核酸序列, 进一步分析V1~V3区作为葡萄球菌核酸水平鉴定的能力。葡萄球菌核酸测序鉴定的菌株信息见表 3, 聚类分析结果见图 1, 葡萄球菌属内不同“种”水平的等位基因和SNP位点统计分析见表 4。
|
|
表 3 本文用于葡萄球菌核酸测序鉴定的菌株信息表 Table 3 Staphylococcus isolates information used for dendrogram analysis in this study |
|
图 1 131株葡萄球菌核酸测序鉴定聚类进化树状图 Figure 1 Dendrograms of 16S rRNA gene sequencing identification of 131 Staphylococcus isolates |
|
|
表 4 葡萄球菌V1~V3区核酸序列位点多样性分析 Table 4 Polymorphism analysis of V1~V3 region sequence using Staphylococcal isolates |
分别将经过测序验证的葡萄球菌标准核酸序列与Genbank数据库中筛选的核酸序列整合, 建立标准核酸序列后, 构建葡萄球菌鉴定聚类进化树状图(图 1-A), 代表性菌株的聚类结果显示:除头状葡萄球菌(S. capitis)与山羊葡萄球菌(S. capare)无法区分外, 葡萄球菌属内的20个“种”均可以特异性地聚类为1个支(图 1-B)。分别统计待测葡萄球菌属内20个“种”的种间及种内等位基因和SNP位点的数量(表 4), 结果显示: 131株葡萄球菌的16S rRNA基因V1~V3区核酸序列片段共含有37个等位基因、81个SNP位点, 不同“种”内的序列特点不同, 金黄色葡萄球菌(S. aureus)、沃式葡萄球菌(S. warneri)、松鼠葡萄球菌(S. sciuri)、假中间葡萄球菌(S. pseudintermedius)、缓慢葡萄球菌(S. lentus)和木糖葡萄球菌(S. xylosus)的种内核酸序列完全相同, 不存在突变位点;而表皮葡萄球菌(S. epidermidis)、溶血葡萄球菌(S. haemolyticus)、科氏葡萄球菌(S. cohnii)、人葡萄球菌(S. hominis)、腐生葡萄球菌(S. saprophyticus)和模仿葡萄球菌(S. simulans)均存在不同数量的等位基因和SNP位点;由于小牛肉葡萄球菌(S. vitulinus)、巴士葡萄球菌(S. pasteuri)、路邓葡萄球菌(S. lugdunensis)等菌株核酸序列数量的限制, 未作统计。上述实验结果表明:将V1~V3区作为鉴定靶点可以将常见葡萄球菌准确鉴定到“种”的水平。
3 讨论随着《药品生产质量管理规范》(GMP 2010)及《中华人民共和国药典》2015年版的实施, 微生物监测数据偏差调查及溯源性分析中对于菌株的鉴定需要达到“种”甚至“菌株”的水平, 传统的形态学、显微镜检、理化分析及生化鉴定等方法已无法满足药品微生物质量控制的需求[18], 采用分子生物学检测技术从物种的遗传物质层面, 开展微生物鉴定标准化研究是药品质量控制发展的必然趋势。《中华人民共和国药典》2020年版编制大纲中拟建设分子生物学检测技术药典标准体系[19], 技术要求通则、标准核酸信息数据库及应用平台建设, 将会为微生物污染物核酸水平的鉴定结果提供权威、准确的判定依据。本研究以细菌污染物为例, 筛选16S rRNA基因中具有鉴定意义的核酸序列片段, 认为V1~V3区核酸序列具有最显著的鉴定效果;并以葡萄球菌为例进行了应用探讨, 为细菌核酸测序鉴定标准化、高通量核酸测序、菌群分布研究等提供研究思路和参考依据。
3.1 公共数据库中核酸序列的筛选与高等动、植物相比, 细菌的基因组DNA较小, 但突变概率较大, 且无法通过肉眼识别种属遗传信息, 虽然在Genbank数据库中可参考的序列较多, 但由于菌株来源信息不明确, 序列长度和方向不一致及未经验证等原因, 实际可以利用的数据资源并不丰富。本文建立统一的筛选规则, 在获取可信度较高的核酸序列的基础上, 进行序列分段及聚类分析, 如Genbank数据库中的参比序列(RefSeq)即注释为“This record has been curated by NCBI staff.”, 提示其可信度较高。
3.2 16S rRNA基因片段分析综合考虑核酸测序反应的读长、高通量核酸测序的应用趋势、标准核酸序列长度、参考文献的报道等, 本文以500 bp的长度为单位, 对16S rRNA基因进行切段, 分为V1~V3、V2~V4、V3~V5、V4~V6、V7~V9区分别进行标准核酸序列片段的筛选和验证。
3.3 16S rRNA基因中V1~V3区具有最佳的鉴定效果根据本实验室洁净区域环境监控的结果, 结合前期的基础性研究[18], 葡萄球菌是药品生产和检验环境中最常见的细菌污染物, 占比超过40%, 通常是人为引入, 易发生交叉污染。本文以葡萄球菌为例, 进行菌株核酸测序鉴定, 建立标准核酸序列, 研究结果表明, 16S rRNA基因中V1~V3区具有最佳的鉴定效果;由于头状葡萄球菌(S. capitis)和山羊葡萄球菌(S. carpae)的16S rRNA基因全长序列未有显著性差异, 本文建立的方法未能有效区分这2个“种”, 随着菌株类型和数量的扩大验证, 需要寻找辅助序列进行进一步鉴定。
3.4 标准核酸序列数据库建设基于核酸序列比对分析的细菌鉴定方法需要建立标准核酸序列数据库。实验室将通过扩大验证, 进一步丰富核酸测序规范化方法和标准核酸序列的适用范围, 为核酸序列水平的鉴定结果提供准确的判定依据。
| [1] |
PAUL DV, GARRITY GM, JONES D, et al. Bergey's Manual of Systematic Bacteriology. Vol Ⅲ[M]. Berlin: Springer Berlin Heidelberg, 2011, 1.
|
| [2] |
USP 40-NF 35[S]. 2017: 1419
|
| [3] |
JP 17[S]. 2017: 2220
|
| [4] |
中华人民共和国药典2015年版. 四部[S]. 2015: 393 ChP 2015. Vol Ⅳ[S]. 2015: 393 |
| [5] |
CHAN ER, HESTER J, KALADY M, et al. A novel method for determining microflora composition using dynamic phylogenetic analysis of 16S ribosomal RNA deep sequencing data[J]. Genomics, 2011, 98(4): 253. DOI:10.1016/j.ygeno.2011.04.002 |
| [6] |
KATAOKA M, UEDA K, KUDO T, et al. Application of the variable region in 16S rDNA to create an index for rapid species identification in the genus Streptomyces[J]. FEMS Microbiol Lett, 1997, 151(2): 249. DOI:10.1111/j.1574-6968.1997.tb12578.x |
| [7] |
BECKER K, HARMSEN D, MELLMANN A, et al. Development and evaluation of a quality-controlled ribosomal sequence database for 16S ribosomal DNA-based identification of Staphylococcus species[J]. J Clin Microbiol, 2004, 42(11): 4988. DOI:10.1128/JCM.42.11.4988-4995.2004 |
| [8] |
ENGELBREKTSON A, KUNIN V, WRIGHTON KC, et al. Experimental factors affecting PCR-based estimates of microbial species richness and evenness[J]. ISME J, 2010, 4(5): 642. DOI:10.1038/ismej.2009.153 |
| [9] |
KIM M, MORRISON M, YU ZT. Evaluation of different partial 16S rRNA gene sequence regions for phylogenetic analysis of microbiomes[J]. J Microbiol Methods, 2011, 84(1): 81. DOI:10.1016/j.mimet.2010.10.020 |
| [10] |
CHAKRAVORTY S, HELB D, BURDAY M, et al. A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria[J]. J Microbiol Methods, 2007, 69(2): 330. DOI:10.1016/j.mimet.2007.02.005 |
| [11] |
LU JJ, PERNG CL, LEE SY, et al. Use of PCR with universal primers and restriction endonuclease digestions for detection and identification of common bacterial pathogens in cerebrospinal fluid[J]. J Clin Microbiol, 2000, 38(6): 2076. |
| [12] |
HEBERT PD, CYWINSKA A, BALL SL, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270(1512): 313. DOI:10.1098/rspb.2002.2218 |
| [13] |
HEBERT PD, PENTON EH, BURNS JM, et al. Ten species in one: DNA barcoding reveals cryptic species in the neotropical skipper butterfly Astraptes fulgerator[J]. Proc Natl Acad Sci USA, 2004, 101(41): 14812. DOI:10.1073/pnas.0406166101 |
| [14] |
NILSSON RH, RYBERG M, KRISTIANSSON E, et al. Taxonomic reliability of DNA sequences in public sequence databases: a fungal perspective[J]. PLoS One, 2006, 1(1): e59. DOI:10.1371/journal.pone.0000059 |
| [15] |
MILLER SE. DNA barcoding and the renaissance of taxonomy[J]. Proc Natl Acad Sci USA, 2007, 104(12): 4775. DOI:10.1073/pnas.0700466104 |
| [16] |
CHEN SL, YAO H, HAN JP, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PloS One, 2010, 5(1): e8613. DOI:10.1371/journal.pone.0008613 |
| [17] |
KIM M, HS O, PARK SC, et al. Towards a taxonomic coherence between average nucleotide identity and 16S rRNA gene sequence similarity for species demarcation of prokaryotes[J]. Int J Syst Evol Microbiol, 2014, 64(2): 346. |
| [18] |
PETTI CA, POLAGE CR, SCHRECKENBERGER P. The role of 16S rRNA gene sequencing in identification of microorganisms misidentified by conventional methods[J]. J Clin Microbiol, 2005, 43(12): 6123. DOI:10.1128/JCM.43.12.6123-6125.2005 |
| [19] |
洪小栩, 冯震, 许华玉, 等. 分子生物学技术用于药品质量控制国家标准核酸数据库的建设规划研究[J]. 中国医药工业杂志, 2016, 47(5): 651. HONG XX, FENG Z, XU HY, et al. Program of national standard nucleotide sequence database used in quality control of pharmaceutical products by molecular biology technology[J]. Chin J Pharm, 2016, 47(5): 651. |
 2018, Vol. 38
2018, Vol. 38 
