2. 武汉科技大学医学院公共卫生学院, 武汉 430065
2. School of Public Health, Medical College, Wuhan University of Science and Technology, Wuhan 430065, China
随着肿瘤疾病发病率的不断增长,肿瘤疾病相关的基因突变越来越引起人们的广泛关注。在人类所有致病基因突变中,近三分之一的突变属于无义突变[1]。无义突变的发生是因为某个碱基的改变,在mRNA内提前形成终止密码(premature stop codon,PTC),从而导致mRNA翻译在突变位置提前终止,形成不完整、无功能的肽链,造成编码蛋白缺失,从而导致疾病发生。研究表明,少数化合物能够诱导无义突变的通读,在类同功tRNA参与下,核糖体跨过无义突变位点,诱导突变基因的mRNA在突变位点继续翻译,进而恢复有功能的全长蛋白的表达[2-6]。对于众多无义突变所致的疾病,无义突变通读剂是一种潜在的治疗药物。目前已知的该类化合物大多数都属于氨基糖苷类抗生素类(如庆大霉素和G 418)及其衍生物(NB 74、NB 84),此外还有若干小分子化合物(PTC 124,RTC 13和RTC 14)。
作为氨基糖类抗生素的代表,庆大霉素在不同细胞和动物模型中显示了明显的通读活性,如Duchenne型肌营养不良(duchenne muscular dystrophy,DMD)、共济失调毛细血管扩张病(ataxia-telangiectasia,A-T)、囊性纤维化(cystic fibrosis,CF)等。然而,这类抗生素系统毒性大,用于长期治疗的可能很小。氨基糖苷类抗生素衍生物NB 74和NB 84具有更高的通读效率,且毒性小,体外试验已证实其对CF、Usher综合征等遗传病有通读活性[7-8],但体内研究报道甚少。PTC 124是一种研究最多的小分子通读剂,在DMD、CF、Usher综合征等疾病模型中表现出了良好的通读活性[9-14],且具有毒性低,可口服等优点,临床Ⅲ期试验已在CF及DMD患者中开展,欧洲药品管理局2014年准许其有条件上市,但美国FDA认为临床Ⅲ期试验未达到预定目标,仍拒绝在美国上市。RTC 13和RTC 14是Du等[5, 15-16]从34 000种化合物中筛选出的2个小分子化合物,其通读活性在DMD、A-T在内的多种基因中被证实,但相关研究还处于前期细胞和动物试验阶段。
由于目前临床上还没有理想的通读药物,并且可测试的活性通读剂数量极少,因此筛选和研发更多的新型有效又安全的无义突变通读剂,对于最终成功研发此类新药至关重要。为此,本研究通过利用带有无义突变型虫荧光素酶基因(本文采用虫荧光素酶)的慢病毒载体感染细胞,进而建立稳定表达无义突变型荧光素酶的细胞株,用以高效筛选无义突变通读诱导剂。
1 材料与方法 1.1 材料野生型荧光素酶质粒pGL4-WT、无义突变型荧光素酶质粒pGL4-MUT(英国邓迪大学医学研究所生命科学院分子医学系Irwin McLean教授惠赠),其中pGL4-MUT的荧光素酶编码序列第223位精氨酸遗传密码被终止密码TGA替换;限制性内切酶BamHⅠ和BglⅡ(NEB公司),CIAP酶(NEB公司),凝胶回收试剂盒(Qiagen公司),Amp固体培养基、小量质粒提取试剂盒(Promega公司),无内毒素质粒大提试剂盒(Promega公司),慢病毒浓缩纯化试剂盒、慢病毒滴度检测试剂盒、DMEM培养基、G 418、Trizol试剂盒、96孔细胞板、RT-PCR试剂盒(Qiagen公司),Western-Blot试剂盒(Pierce公司),荧光素酶报告基因检测试剂盒(Promega公司)。
1.2 RT-PCR引物用软件primer premier 5.0设计引物。luciferase-sense(5’-ACC GTC GTA TTC GTG AGC AA-3’)和luciferase-antisense(5’-GGT GGC AAA TGG GAA GTC A-3’),内参β-actin-sense(5’-TAT GGA ATC CTG TGG CAT C-3’)和β-actin-antisense(5’-GTG TTG GCA TAG AGG TCT T-3’)。
1.3 重组慢病毒载体构建利用BamHⅠ和BglⅡ同尾酶特点,参照流程图(图 1)构建重组慢病毒载体,其中插入野生型荧光素酶cDNA片段的慢病毒命名为pLVX-WT,插入无义突变型荧光素酶cDNA片段的慢病毒命名为pLVX-MUT。并对重组慢病毒载体进行EcoRⅠ单酶切,验证是否插入外源片段。为确定插入方向是否正确,对重组慢病毒载体进行定向克隆的酶切鉴定。同时设计荧光素酶引物,进一步确认荧光素酶插入到慢病毒载体中。PCR反应条件设置为95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,并用1.5%琼脂糖凝胶电泳检测。
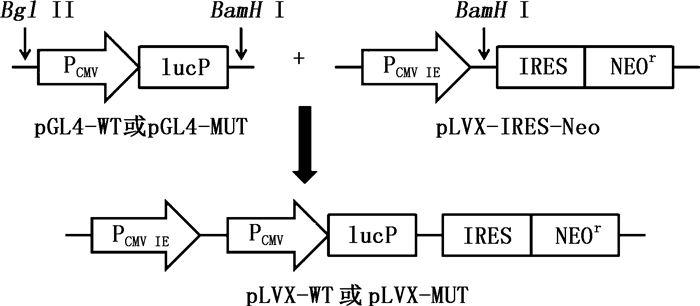
|
图 1 重组慢病毒构建流程 Figure 1 Schematic representation of construction of recombinant lentiviral vectors |
用慢病毒二质粒包装系统质粒psPAX2及pCMV-VSV-G包装病毒。慢病毒重组表达载体、psPAX2和PCMV-VSV-G按3:2:1的质量比混匀,加入无血清无双抗的高糖DMEM液体培养基中,室温孵育15 min,加入293T细胞中进行转染,培养4 d后收取上清液,并用无菌0.45 μm滤器过滤,最后利用慢病毒浓缩纯化试剂盒进行浓缩,并利用慢病毒滴度检测试剂盒进行效价测定,分装后-80 ℃冻存待用。
1.5 慢病毒稳定感染细胞筛选HEK 293和293 T接种于含10%胎牛血清的DMEM高糖培养基中,并加入终浓度为100 U·mL-1青霉素和100 U·mL-1链霉素,然后于37 ℃和5%CO2的细胞培养箱中过夜培养。当细胞密度达到80%融合率后,加1 mL病毒工作液(将“1.4”项中病毒包装后浓缩的病毒浓缩液用DMEM培养液按体积比1:10 000稀释),同时设置空白转染对照组。感染3 d之后,加入G 418(终浓度为1 mg·mL-1)进行细胞筛选。每2 d更换新鲜的加药培养基1次。当对照组中细胞完全死光后,从实验组留下的细胞中用无菌细胞挑种针挑选单克隆细胞,分别接种于96孔细胞板中培养扩增,最后常规传代培养稳定感染细胞。其中感染pLVX-WT的细胞命名为HEK293WT,感染pLVX-WT的细胞命名为HEK293MUT。
1.6 逆转录PCR方法检测细胞内荧光素酶mRNA表达在6孔板的培养细胞中按每孔1 mL加入Trizol提取总RNA,按照逆转录PCR试剂盒说明进行操作。PCR反应条件设置为95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,并用1.5%琼脂糖凝胶电泳检测。
1.7 Western blot方法检测细胞内荧光素酶蛋白表达常规培养HEK293WT和HEK293MUT细胞,用终浓度为10 μmol·L-1的G 418处理,48 h后提取总蛋白,用Western blot检测野生型和突变型荧光素酶蛋白表达,以GAPDH作为内参对照。
1.8 细胞内荧光素酶活性检测将HEK293、HEK293WT和HEK293MUT细胞接种于24孔细胞培养板上,分为无药物处理组和通读剂G 418(终浓度10 μmol·L-1)处理组,培养48 h后,破碎细胞,按照荧光素酶报告基因检测试剂盒说明操作,检测各细胞样中荧光素酶活性,以HEK293细胞发光值作为本底,每个实验样本均独立操作,实验重复3次以上,数据经统计学分析。
2 结果 2.1 重组慢病毒载体构建对质粒pGL4-WT和pGL4-MUT进行酶切并电泳分离后可得2条片段,一条3.4 kb,一条2.7 kb,利用胶回收试剂盒纯化回收2.7 kb大小的片段。如图 2所示,根据Marker指示,所切得的野生型片段和突变型片段与预期相符,说明成功切得所需片段。然后将目的片段插入到慢病毒载体中,即可构建出含野生型荧光素酶编码序列的重组慢病毒载体pLVX-WT和含无义突变荧光素酶编码序列的重组慢病毒载体pLVX-MUT。
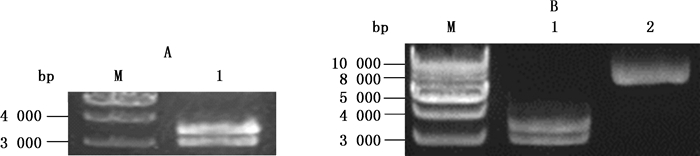
|
A. BamHⅠ和BglⅡ双酶切质粒pGL4-WT的结果(BamHⅠ and BglⅡ digestion of pGL4-WT):M. 1 kb Marker 1. pGL4-WT双酶切结果(digestion of pGL4-WT)B. BamHⅠ和BglⅡ双酶切pGL4-MUT质粒及BamHⅠ单酶切pLVX-IRES-Neo质粒的结果(BamHⅠ and BglⅡ digestion of pGL4-MUT and BamHⅠ digestion of pLVX-IRES-Neo):M. 1 kb Marker 1. pGL4-MUT双酶切结果(digestion of pGL4-WUT)2. pLVX-IRES-Neo单酶切结果(digestion of pLVX-IRES-Neo) 图 2 带目的片段的pGL4-WT和pGL4-MUT质粒及慢病毒表达质粒pLVX-IRES-Neo的酶切结果 Figure 2 Endonuclease digestion results of pGL4-WT, pGL4-MUT and pLVX-IRES-Neo |
首先对pLVX-WT、pLVX-MUT进行EcoR1单酶切,获得3.1 kb片段,与预期一致,说明荧光素酶在慢病毒载体中插入成功(图 3-A);由于不能确定插入方向是否正确,于是对其进行定向克隆的双酶切鉴定,获得2.7 kb片段,与预期一致,说明荧光素酶cDNA片段以正确方向插入慢病毒载体中(图 3-B)。同时根据虫荧光素酶特征序列设计了相应引物,用以进一步确定所得产物为插入荧光素酶编码序列的重组慢病毒载体中,结果显示所得产物均成功扩增出目的片段,即表明所得为目的重组载体(图 3-C)。
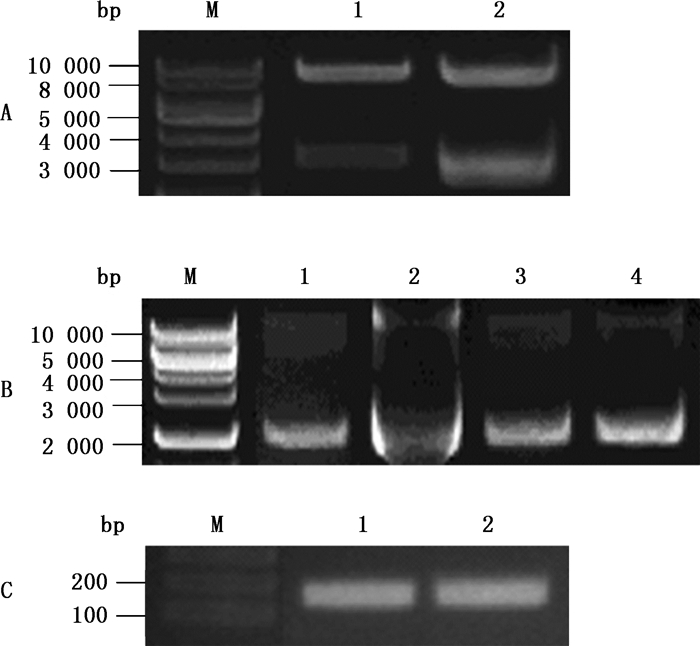
|
A.重组质粒pLVX-WT(1)与pLVX-MUT(2)的EcoRⅠ酶切鉴定[EcoRⅠ digestion of pLVX-WT(1)and pLVX-MUT(2)]:M. 1 kb Marker B.重组质粒pLVX-WT(1,2)与pLVX-MUT(3,4)的NheⅠ、BamHⅠ酶切鉴定[(NheⅠ and BamHⅠ digestion of pLVX-WT(1,2)and pLVX-MUT(3,4)]:M. 1 kb Marker C.荧光素酶2编码序列PCR鉴定(PCR analysis of luciferase 2 coding sequence):M. 100 bp Marker 1. pLVX-WT 2. pLVX-MUT 图 3 重组慢病毒表达质粒的酶切和PCR鉴定 Figure 3 Identification of recombinant lentiviral vectors by restriction analysis and PCR |
接种HEK 293细胞,分别感染pLVX-WT、pLVX-MUT获得稳定感染细胞HEK293WT和HEK293MUT。提取总RNA后,利用逆转录PCR鉴定细胞是否稳定整合并表达荧光素酶mRNA。结果显示,所建立细胞株稳定表达荧光素酶mRNA(图 4)。

|
M. 100 bp Marker 1. HEK293细胞2. HEK293WT细胞3. HEK293MUT细胞 图 4 稳定转染细胞株荧光素酶mRNA表达逆转录PCR鉴定 Figure 4 Identification of the expression of luciferase mRNA of stable transfected cells by reverse transcription PCR |
用已知无义突变阳性通读剂G 418处理HEK293WT和HEK293MUT细胞,再用Western blot杂交技术检测荧光素酶蛋白表达变化情况,结果发现,野生型HEK293WT细胞在G418处理前后均稳定表达荧光素酶蛋白,且表达水平基本相同;而突变型HEK293MUT细胞未经药物处理时,不表达荧光素酶蛋白,而G 418处理可以恢复荧光素酶表达,但表达水平低于HEK293WT细胞;作为阴性对照的HEK293细胞,药物处理前后均无荧光素酶蛋白的表达(图 5)。
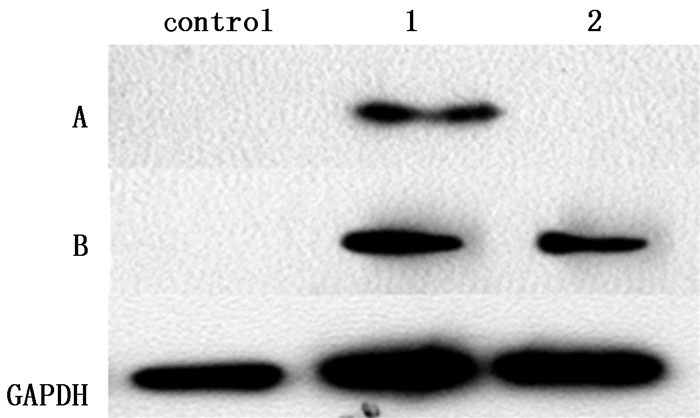
|
control. HEK293细胞(HEK293 cell)1. HEK293WT细胞(HEK293WT cell)2. HEK293MUT细胞(HEK293MUT cell) 图 5 稳定转染细胞株在通读剂G 418处理前(A)、后(B)荧光素酶蛋白表达Western检测 Figure 5 Western blot analysis of luciferase in stable transfected cells treated without(A) or with(B) G 418 |
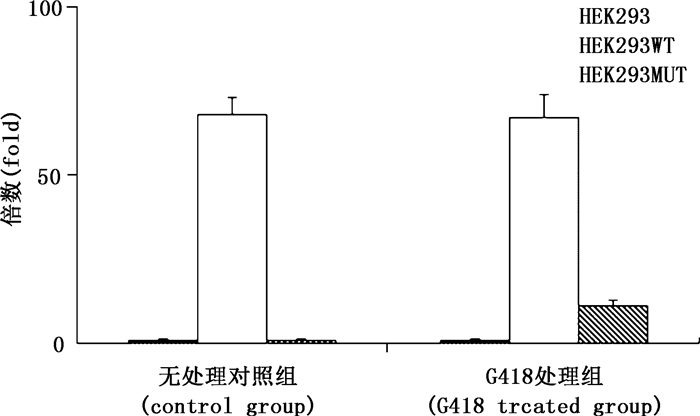
最后,利用荧光素酶活性检测技术来观察无义突变阳性通读剂G 418对HEK293WT和HEK293MUT细胞内源荧光素酶活性的影响。结果发现,HEK293WT细胞在G 418处理前后,均有较强的荧光素酶活性;未经G 418处理时,HEK293MUT细胞中未检测到荧光素酶活性,而G 418的处理可使之酶活性增强,不过,相比于HEK293WT细胞,仅为其20%左右(图 6)。
|
图 6 稳定转染细胞株在通读剂G 418处理前后荧光素酶活性检测 Figure 6 Activity of luciferase in stable transfected cells treated with or without G 418 |
目前,用于筛选新型无义突变通读剂的方法还锁定在蛋白质截短实验(protein truncation test,PTT)及双荧光素酶报告系统。PTT是一种细胞外蛋白合成实验,此方法存在非特异翻译产物的干扰,对于结果的解释并不准确,而且还具有放射性污染,因此限制了此方法的广泛应用。后期,研究人员又在PTT基础上建立了PTT-Western方法和PTT-ELISA方法[17-20]。PTT-Western方法降低了假阳性率,且避免了放射性污染,但是检测时间较长,效率并未提高。PTT-ELISA方法中的蛋白质可能由于无生物素标记而不被捕获。此后,又出现了一种全新的高通量PTT-ELISA试验(HTS-PTT-ELISA),通过巢式PCR,在蛋白质N末端和C末端分别引入c-myc和V5抗体捕获并检测。与全长产物相比,阶段蛋白由于缺乏V5标签,因此更容易被区分,从而灵敏地检测通读活性。然而,PTT是细胞外检测方法,不能真实反映细胞内的情况。
荧光素酶报告系统[21-25]是以荧光素为底物来检测萤火虫荧光素酶活性的一种报告系统。双荧光素酶即萤火虫荧光素酶与海肾荧光素酶的结合,可以更好地体现检测结果的准确性。但是,目前双荧光素酶系统一般是用瞬时转染细胞而不是稳定的细胞系,这样不仅使实验操作步骤繁杂,增加了许多不确定因素,同时导致不同批次实验间可比性较差,为了解决这个问题,增加检测的稳定性和可比性,因此需要建立一个新的稳定的细胞筛选系统。
本研究构建了含无义突变荧光素酶编码序列的重组慢病毒载体,再感染HEK 293细胞获得了可稳定表达无义突变荧光素酶的HEK293MUT细胞。用阳性通读剂G 418测试,发现该细胞可以恢复部分荧光素酶表达,并且活性相当于对照细胞的20%。这一结果说明,该细胞模型可以用于高通量筛选和开发更多新型的具有临床潜力的无义突变通读剂及其同系物,从中发现具有临床药物潜力的候选药物,用来治疗无义突变所致的众多遗传病和肿瘤。
虽然本研究所建立的细胞只含有无义突变TGA,但是研究发现,能够通读TGA的无义突变通读剂对其他2种类型无义突变(TAG、TAA)都具有较好的通读效率,TGA无义突变常用于通读剂初筛[5, 20]。当然,通过此细胞模型的成功建立,还可以构建含有其他无义突变类型的细胞模型,这将为无义突变通读诱导剂的筛选提供更广阔、更有效的、更准确的研究平台。
| [1] |
MENDELL JT, DIETZ HC. When the message goes awry:diseaseproducing mutations that influence mRNA content and performance[J]. Cell, 2001, 107(4): 411. DOI:10.1016/S0092-8674(01)00583-9 |
| [2] |
HOWARD MT, SHIRTS BH, PETROS LM, et al. Sequence specificity of aminoglycoside-induced stop condon readthrough:potential implications for treatment of Duchenne muscular dystrophy[J]. Ann Neurol, 2000, 48(2): 164. DOI:10.1002/(ISSN)1531-8249 |
| [3] |
ZINGMAN LV, PARK S, OlSON TM, et al. Aminoglycosideinduced translational read-through in disease:overcoming nonsense mutations by pharmacogenetic therapy[J]. Clin Pharmacol Ther, 2007, 81(1): 99. DOI:10.1038/sj.clpt.6100012 |
| [4] |
WELCH EM, BARTON ER, ZHUO J, et al. PTC124 targets genetic disorders caused by nonsense mutations[J]. Nature, 2007, 447(7140): 87. DOI:10.1038/nature05756 |
| [5] |
DU L, DAMOISEAUX R, NAHAS S, et al. Nonaminoglycoside compounds induce readthrough of nonsense mutations[J]. J Exp Med, 2009, 206(10): 2285. DOI:10.1084/jem.20081940 |
| [6] |
ZILBERBERG A, LAHAV L, ROSIN-ARBESFELD R. Restoration of APC gene function in colorectal cancer cells by aminoglycosideand macrolide-induced read-through of premature termination codons[J]. Gut, 2010, 59(4): 496. DOI:10.1136/gut.2008.169805 |
| [7] |
VECSLER M, ZEEV B, NUDELMAN L, et al. Ex vivo treatment with a novel synthetic aminoglycoside NB54 in primary fibroblasts from Rett syndrome patients suppresses MECP2 nonsense mutations[J]. PLoS One, 2011, 6(6): e20733. DOI:10.1371/journal.pone.0020733 |
| [8] |
BRENDEL C, BELAKHOV V, WERNER H, et al. Readthrough of nonsense mutations in Rett syndrome:evaluation of novel aminoglycosides and generation of a new mouse model[J]. J Mol Med (Berl), 2011, 89(4): 389. DOI:10.1007/s00109-010-0704-4 |
| [9] |
WELCH EM, BARTON ER, ZHUO J, et al. PTC124 targets genetic disorders caused by nonsense mutations[J]. Nature, 2007, 447(7140): 87. DOI:10.1038/nature05756 |
| [10] |
BIDOU L, ALLAMAND V, ROUSSET JP, et al. Sense from nonsense:therapies for premature stop codon diseases[J]. Trends Mol Med, 2012, 18(11): 679. DOI:10.1016/j.molmed.2012.09.008 |
| [11] |
PIBIRI I, LENTINI L, MELFI R, et al. Enhancement of premature stop codon readthrough in the CFTR gene by Ataluren (PTC124) derivatives[J]. Eur J Med Chem, 2015, 101(4): 236. |
| [12] |
KUSCHAL C, KHAN SG, ENK B, et al. Readthrough of stop codons by use of aminoglycosides in cells from xeroderma pigmentosum group C patients[J]. Exp Dermatol, 2015, 24(4): 296. DOI:10.1111/exd.2015.24.issue-4 |
| [13] |
den DUNNEN JT, van OMMEN GJ. The protein truncation test:a review[J]. Hum Mutat, 1999, 14(2): 95. DOI:10.1002/(ISSN)1098-1004 |
| [14] |
GÓMEZ-GRAU M, GARRIDO E, COZAR M, et al. Evaluation of aminoglycoside and non-aminoglycoside compounds for stop-codon readthrough therapy in four lysosomal storage diseases[J]. PLoS One, 2015, 10(8): e0135873. DOI:10.1371/journal.pone.0135873 |
| [15] |
KAYALI R, KU JM, KHITROW G, et al. Read-through compound 13 restores dystrophin expression and improves muscle function in the mdx mouse model for Duchenne muscular dystrophy[J]. Hum Mol Genet, 2012, 21(18): 4007. DOI:10.1093/hmg/dds223 |
| [16] |
DU L, JUNG ME, DAMOISEAUX R, et al. A new series of small molecular weight compounds induce read through of all three types of nonsense mutations in the ATM gene[J]. Mol Ther, 2013, 21(9): 1653. DOI:10.1038/mt.2013.150 |
| [17] |
LAI CH, CHUN HH, NAHAS SA, et al. Correction of ATM gene function by aminoglycoside-induced read-through of premature termination codons[J]. Proc Natl Acad Sci USA, 2004, 101(44): 15676. DOI:10.1073/pnas.0405155101 |
| [18] |
GITE S, LIM M, CARLSON R, et al. A high-throughput nonisotopic protein truncation test[J]. Nat Biotechnol, 2003, 21(2): 194. DOI:10.1038/nbt779 |
| [19] |
LIM MJ, FOSTER GJ, GITE S, et al. An ELISA-based high throughput protein truncation test for inherited breast cancer[J]. Breasr Cancer Res, 2010, 12(5): R78. DOI:10.1186/bcr2722 |
| [20] |
DU L, LAI CH, CONCANNON P, et al. Rapid screen for truncating ATM mutations by PTT-ELISA[J]. Mutat Res, 2008, 640(1-2): 139. DOI:10.1016/j.mrfmmm.2008.01.002 |
| [21] |
GRENTZMANN G, INGRAM JA, KELLY PJ, et al. A dualluciferase reporter system for stydying redoding signals[J]. RNA, 1998, 4(4): 479. |
| [22] |
HARGER JW, DINMAN JD. An in vivo dual-luciferase assay system for studying translational recoding in the yeast Saccharomyces cerevisiae[J]. RNA, 2003, 9(8): 1019. DOI:10.1261/rna.5930803 |
| [23] |
LOUGHRAN G, CHOU MY, IVANOV IP, et al. Evidence of efficient stop codon readthrough in four mammalian genes[J]. Nucleic Acids Res, 2014, 42(14): 8928. DOI:10.1093/nar/gku608 |
| [24] |
HALVEY PJ, LIEBLER DC, SLEBOS RJ. A reporter system for translational readthrough of stop codons in human cells[J]. FEBS Open Bio, 2012, 2(1): 56. DOI:10.1016/j.fob.2012.04.004 |
| [25] |
MCELROY SP, NOMURA T, TORRIE LS, et al. A lack of premature termination codon read-through efficacy of PTC124(Ataluren)in a diverse array of reporter assays[J]. PLoS Biol, 2013, 11(6): e1001593. DOI:10.1371/journal.pbio.1001593 |
 2017, Vol. 37
2017, Vol. 37 
