2. 安徽中医药大学, 合肥 230038
2. Anhui University of Chinese Medicine, Hefei 230038, China
人参属植物是我国传统名贵中药材,多具补气、安神、益智、提高免疫力的作用,广泛用于传统中医药及民间医学。2015年版中国药典收载了人参(Panax ginseng)、西洋参(P. quinquefolium)、三七(P. notoginseng)、竹节参(P. japonicus)、珠子参(P. japonicas var. major)以及羽叶三七(P. pseudoginseng var. bipinnatifidus)6种人参属植物。然而,由于人参属植物亲缘关系很近,其形态学、化学成分均很相似,难以进行区分,尤其是人参和西洋参[1-2]。DNA分子鉴定因其不受环境、药材形态、取样部位的影响,并且鉴别能力强、结果准确,在近年来快速发展[3-4]。基于DNA扩增图谱多态性的RAPD(随机扩增多态DNA)[5]、AFLP(扩增片段长度多态性)[6]、PCR-RFLP(PCR-限制酶切长度多态性)[7-8]、SCAR(序列特异性扩增区域)[9]、AS-PCR(位点特异性PCR)[7, 10]、MARMS(多重位点特异性PCR)[2, 11]以及基于分子杂交信号的芯片技术[12-13]等分子鉴定技术均已应用于人参属药用植物鉴定,然而这些方法往往稳定性差(如RAPD),只能鉴定单个物种(如PCR-RFLP),要求严谨的实验条件(如MARMS)或操作复杂(如DNA芯片)。DNA条形码技术(DNA barcoding)是利用标准的,具有足够变异的,易扩增且相对较短的DNA片段,并基于种内特异性和种间多样性创建的一种新的生物身份识别系统[14-21],可用于人参属中药材的鉴定[1, 22],但DNA条形码需要进行PCR产物纯化和DNA测序,其操作复杂,检测时间较长,无法满足中药分子鉴定快速、简单、可视化方面的要求。
高分辨率熔解曲线(high resolution melting,HRM)是一种基于双链DNA熔解温度不同而形成不同形态熔解曲线的基因分型技术。DNA序列长度不同,排列差异均会造成熔解曲线峰形(melting curve shapes)及熔解温度(melting temperature,Tm)的差异[23]。由于HRM技术具有很高的灵敏性,已成功用于物种鉴别[24]、SSR检测[25]、甲基化分析[26]乃至SNP分型[27]。由于其操作简单,结果容易判读,并能同时检测正伪品及其混杂品,在中药分子鉴定中具有很好的应用前景。
2011年前后,Jaakola等[28]及Ganopoulos等[29]尝试将HRM分析引入鉴定领域,已用于浆果[28]、果汁[30]、豆科植物[31]、植物油[32]等鉴定。该方法不但具有HRM分析的优点,且通用性好,可同时检测正伪品,扩增与检测可在同一PCR管内进行,避免了开盖污染。本研究把HRM分析引入人参属多来源药材物种鉴别,为中国药典收载的人参属中药真实性鉴别提供一种简单快捷的方法。在此基础上,对该分子鉴别方法的灵敏性与特异性、重复性、稳定性、检出限进行了系统考察,提出了中药分子鉴定方法学研究标准程序,为进一步规范中药分子鉴定方法学研究提供依据。
1 材料与方法 1.1 仪器LightCycler 480型实时荧光定量PCR仪(Roche公司),5810R型低温冷冻离心机(Eppendorf公司),MM 400型混合型球磨仪(Retsch公司),ND-1000型核酸定量仪(Gene公司),微量移液器(Eppendorf公司)。
1.2 试剂CTAB(Merck公司)、SpeedStar HS Taq DNA聚合酶(Takara公司)、LC Green Plus(Idaho公司)。三氯甲烷﹑无水乙醇﹑异丙醇等均为国产分析纯。
1.3 样品15份西洋参样品分别采自河北、吉林,经安徽中医药大学周建理教授鉴定;20份人参样品分别采自辽宁、吉林,经中国农业科学院许世友研究员鉴定;10份三七样品分别采自云南文山州文山县,经文山三七研究院崔秀明研究员鉴定;竹节参和珠子参样品各10份采自湖北,均由湖北轻工大学陈平研究员提供并鉴定;10份羽叶三七样品采自云南,经湖北轻工大学陈平研究员鉴定。取人参属样品的根部,硅胶干燥。凭证标本均保存于中国中医科学院中药资源中心。实验植物材料详见表 1。
|
|
表 1 植物材料 Table 1 Information of plant materials |
用75%乙醇擦拭药材表面后,刮去外表皮,取内部的药材样品约20 mg,依据CTAB法[33]提取总DNA,使用ND-1000核酸定量仪测定DNA浓度及A260/A280,并用双蒸水(ddH2O)调整终浓度约50ng·μL-1,用于PCR扩增。
1.5 PCR扩增以所提DNA为模板,在LightCycler 480型实时荧光定量PCR仪上进行HRM分析。25 μL反应体系:2.5 μL 10×buffer,1 μL 10 mmol·L-1 dNTPs,0.25 μmol·L-1上游及下游引物,0.25 U Speed Star HS Taq DNA聚合酶,1 μL(约50 ng)DNA模板,2 μL 100×LC Green Plus,加ddH2O补齐至25 μL。PCR反应条件见表 2。PCR扩增反应结束后,添加HRM分析程序:95 ℃变性1 min,40 ℃ 1 min形成异源双链DNA,而后升温至70 ℃,最后从70 ℃至97 ℃,期间每1 ℃采集15个荧光值。实验结束后使用Light Cycler 480 1.5.1软件进行HRM分析。因DNA模板或加入试剂过程中可能会存在污染或抑制而影响HRM分析准确性[34],本研究在进行HRM分析之前,通过扩增曲线排除扩增强度低于最大扩增60%的样品。
|
|
表 2 引物序列和PCR程序 Table 2 Sequence of primers and PCR program |
考察了引物psbA-F/ trnH-R、rbcL 1F/724R、ITS2/ITS3、matK 3F/1R、trnL/trnF、trnL(Intron)[1]对人参属各物种熔解曲线峰形和熔解温度(Tm)值差异的影响,筛选出合适的鉴别引物,扩增片段,引物序列及PCR反应条件如表 2所示。
1.7 HRM分析HRM分析分别采用2种方式:(1)熔解温度峰值法,对熔解曲线进行求导,获得其解链温度和求导曲线,建立标准模型,如图 1-A;(2)高分辨率熔解曲线分型(HRM genotyping),直接对熔解曲线进行比较,通过Light Cycler 480 1.5.1软件归一化后,加以对比分析,并建立标准模型,如图 1-B。
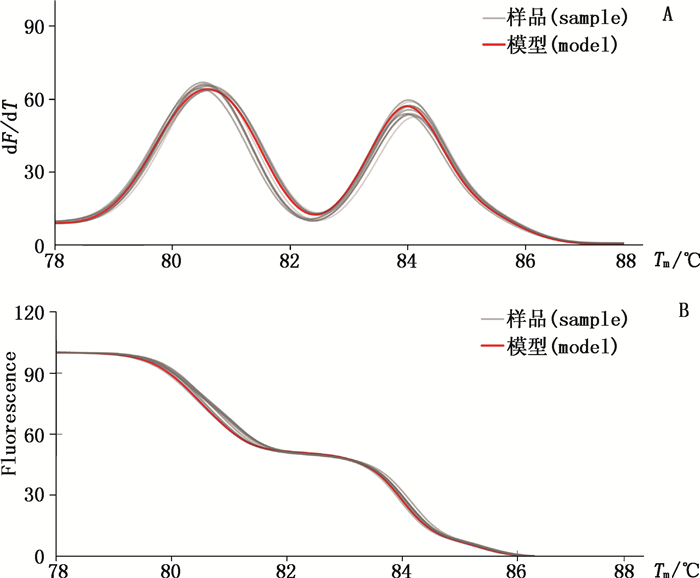
|
图 1 人参属HRM分析模型的建立 Figure 1 The establishment of standard model for HRM analysis of Panax species |
测试方法灵敏性,随机选取人参、西洋参、三七、竹节参、珠子参、羽叶三七材料各3份,提取DNA并用ddH2O调整适宜浓度,进行HRM分析,参考已建立的模型,计算比对阳性与实际阳性结果之间的百分比;测试方法特异性,计算比对阴性与实际阴性结果之间的百分比。
1.8.2 重复性测试操作重复性,取1个人参属材料DNA,分成7份,使用psbA-F/trnH-R引物进行扩增,同时进样,分析其高分辨率熔解曲线分型结果,计算其Tm值标准差及RSD。为测试仪器稳定性,取1个人参属材料DNA,分成7份,使用psbA-F/trnH-R引物进行扩增,分7 d连续进样,计算其Tm值标准差及RSD。
1.8.3 稳健性测试HRM鉴定的DNA浓度稳健性,以人参为例,取人参DNA各1个,用ddH2O调整DNA质量浓度至200 ng·μL-1,用双蒸水进行梯度稀释,形成质量浓度为200、40、8、1.6、0.32 ng·μL-1的DNA浓度梯度,使用psbA-F/trnH-R引物进行扩增,每个反应重复3次,检测其分型结果准确性,以3个重复中至少有2个重复结果与实际结果一致判定为结果准确。为测试HRM分析的退火温度稳健性,取人参属材料DNA各1个,用ddH2O调整DNA质量浓度至50 ng·μL-1,使用psbA-F/trnH-R引物,依次调整退火温度为56、58、60、62、64、66 ℃进行扩增温度,每个反应重复3次,以3个重复中至少有2个重复结果与实际结果一致判定为结果准确。为测试HRM分析的引物浓度温度稳健性,取人参属材料DNA各1个,用ddH2O调整DNA质量浓度至50 ng·μL-1,使用psbA-F/trnH-R引物,依次用ddH2O调整引物浓度为0.1、0.2、0.3、0.4 μmol·L-1进行扩增,每个反应重复3次,以3个重复中至少有2个重复结果与实际结果一致判定为结果准确。
1.8.4 线性关系与检出限测试人参属HRM分析方法的检出限,以人参为例,取人参DNA 7个,用ddH2O调整DNA质量浓度至200 ng·μL-1,用ddH2O梯度稀释,形成质量浓度为200、40、8、1.6、0.32 ng·μL-1的DNA浓度梯度,并以ddH2O为空白对照,以psbA-F/trnH-R为引物扩增,分析其HRM分析结果,记录人参属各物种对应Tm值时的荧光强度值,将DNA浓度与对应荧光强度值进行线性回归,得到线性关系,并以空白样品荧光强度值与所得标准曲线斜率的比值的3倍作为该方法检出限。
1.9 混伪品的检出针对人参和西洋参的混伪品,取两者DNA样品进行不同比例混合,再以psbA-F/trnH-R为引物扩增后进行HRM分析,分析其HRM结果。
2 结果 2.1 引物筛选为了获得适用于人参属药材HRM鉴别的引物,分别以5个人参、西洋参、三七、竹节参、珠子参、羽叶三七DNA作为模板,选择psbA-F/ trnH-R,rbcL 1F/724R,ITS2/ ITS3,matK 3F/1R,trnL/trnF,trnL(Intron)片段,分别在其适宜的退火温度及35个循环的条件下进行熔解曲线分析,对其熔解曲线Tm值及HRM分型结果进行比较,见表 3,筛选出差异最显著的引物用于HRM鉴别。结果表明,引物psbA-F/trnH-R与trnL(Intron)2个片段均可以鉴别人参属中人参、西洋参、三七、珠子参,羽叶三七、竹节参,其中在HRM分型方法中,人参属在psbA-F/trnH-R片段中差别最为明显,见图 2。
|
|
表 3 人参属不同片段熔解温度(Tm) Table 3 Melting temperature(Tm) of different fragments of Panax species |
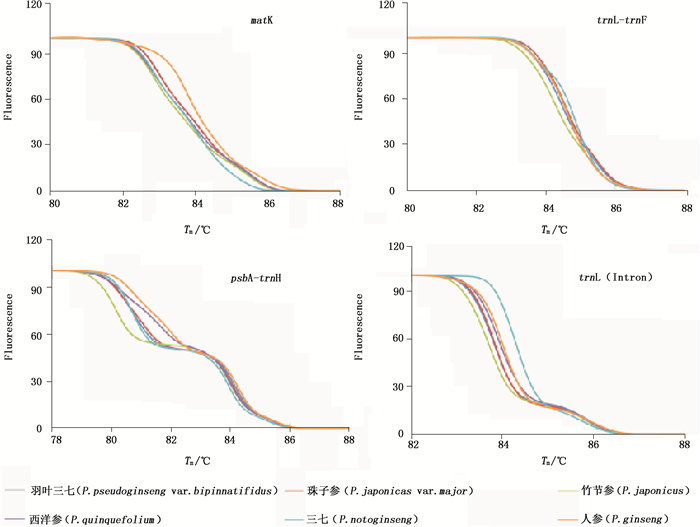
|
图 2 人参属HRM分型结果 Figure 2 The results of HRM genotyping for Panax species |
用psbA-F/trnH-R引物对所有人参属共75份样品进行扩增,利用LightCycler 480 1.5.1软件分别选择2种方法进行数据分析:(1)熔解温度峰值法,人参属6个物种均为双峰,依其Tm值,人参、西洋参、三七、竹节参均能相互鉴别,而羽叶三七与珠子参由于两者的Tm值接近,Tm熔解过程相近,无法区分,见图 3-A;(2)HRM分型,人参、西洋参、三七、羽叶三七、竹节参、珠子参样品均获得准确的HRM分型结果和HRM归一化图(the normalized graph of HRM),见图 3-B;同时也可以将珠子参熔解曲线作为基线,得到衍生出来具有放大差异作用的HRM差异化图(the difference graph of HRM),见图 3-C。
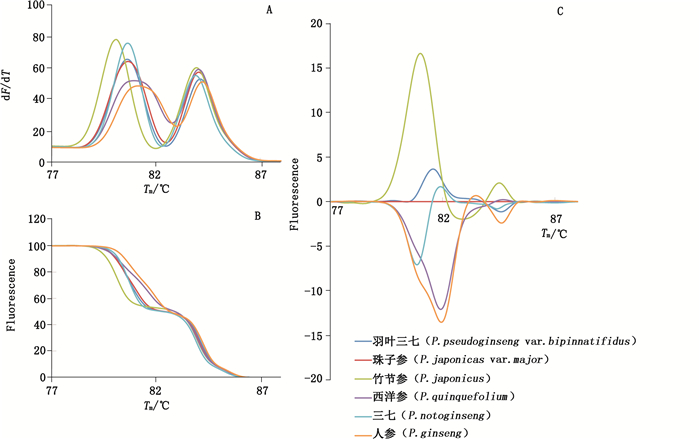
|
A.人参属熔解峰型图(the melting peaks for Panax species)B.人参属HRM归一化图(the normalized graph of HRM for Panax species)C.人参属HRM差异化图(the difference graph of HRM for Panax species) 图 3 人参属HRM高分辨率熔解曲线分析结果(psbA-trnH片段上) Figure 3 The results of HRM analysis for Panax species in psbA-trnH region |
人参、西洋参、三七、竹节参、珠子参、羽叶三七人参属HRM分型结果均与实际结果一致,故其灵敏性与特异性均为100%。
2.3.2 重复性为测试操作重复性,取1个人参属材料DNA,分成7份,使用psbA-F/trnH-R引物同时进行扩增,分析其HRM分型结果,计算其峰Tm值±标准差为(81.28±0.06)℃、(84.30±0.08)℃,RSD为0.08%和0.09%。为测试仪器稳定性,取1个人参属材料DNA,分成7份,使用psbA-F/trnH-R引物进行扩增,分7次连续进样,计算其峰Tm值±标准差为(81.30±0.06)℃、(84.33±0.09)℃,RSD为0.06%和0.09%。结果表明该方法重复性较高。
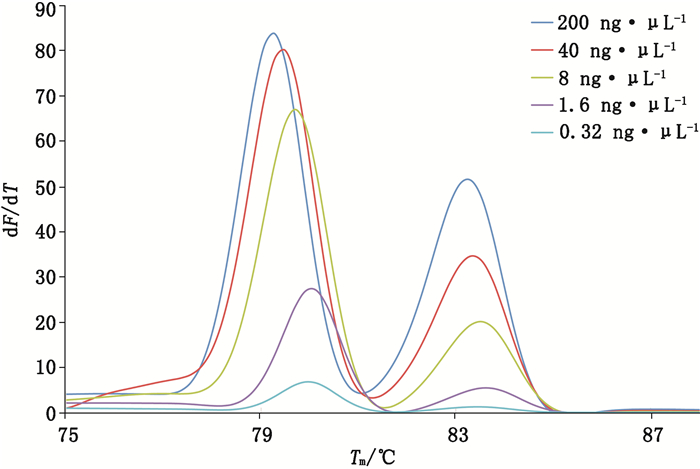
2.3.3 稳健性为测试HRM鉴定的DNA浓度稳健性,以人参为例,取1个人参DNA,以ddH2O调整DNA质量浓度至200 ng·μL-1,以ddH2O梯度稀释,形成质量浓度为200、40、8、1.6、0.32 ng·μL-1的DNA浓度梯度,使用psbA-F/trnH-R引物进行扩增,每个反应重复3次,检测其分型结果准确性,以3个重复中至少有2个重复结果与实际结果一致判定为结果准确。结果表明,其DNA质量浓度稳健范围为1.6~200 ng·μL-1,如图 4。测试HRM分析方法,结果表明其退火温度稳健范围为54~60 ℃,其引物浓度稳健范围为0.1~0.3 μmol·L-1。
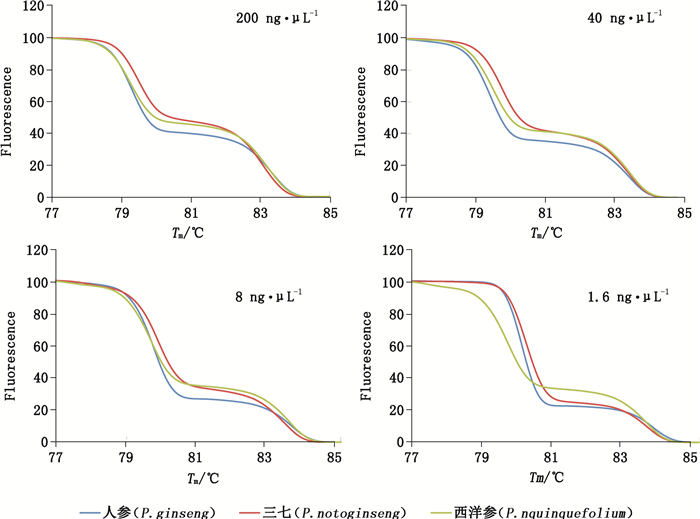
|
图 4 不同模板浓度下的HRM分型 Figure 4 HRM genotyping for different concentrations of Panax species |
DNA浓度与对应的熔解峰荧光强度值进行线性回归,得到回归方程:
Y=12.893ln X+26.354 R2=0.914 6
线性范围为200~1.6 ng·μL-1。参考检出限定义(即空白样品荧光强度值与所得标准曲线斜率的比值的3倍),人参属HRM方法检出限为0.39 ng·μL-1。
2.4 混伪品的检出以人参和三七的混伪品为例,取人参和三七DNA样品各1份,以ddH2O调整质量浓度至50 ng·μL-1,将两者混合,使三七中人参DNA所占比例,形成100%、50%、25%、12.5%、0%的浓度比例,以psbA-F/trnH-R为引物扩增后进行HRM分型分析,记录对应各浓度比例样品的Tm值,分析其HRM分型结果。结果表明,混伪品与正品信号形成明显区别,从而可以对混伪品进行检测,如图 5-A、C。针对人参和西洋参的混伪品,采用相同扩增方法,分析其HRM分型结果,如图 5-B、D。
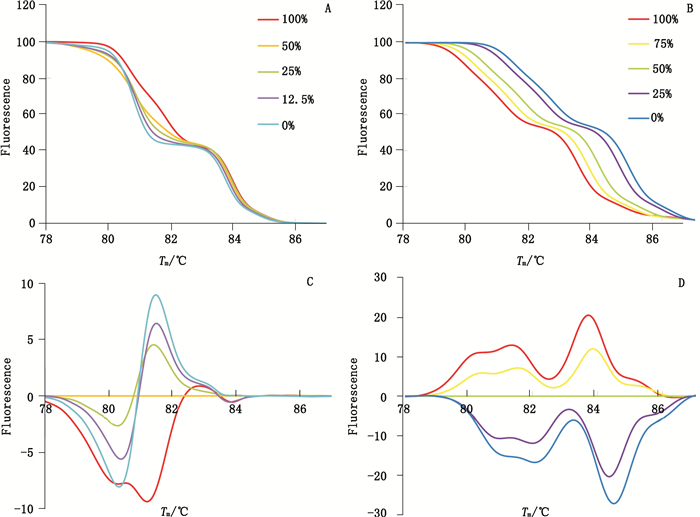
|
图 5 不同人参属中药材不同比例混合的HRM分型 Figure 5 Panax species detected by HRM genotyping |
通过对熔解温度峰值法和HRM分型2个方法进行了分析和对比,可以看出熔解温度峰值法Tm值,虽然在操作重复性与仪器重复性具有较为稳定的表现,但是熔解曲线峰位置仍然是相对的,该位置在试剂、染料或者是模板DNA浓度变化后,位置也会发生相对变化,如图 6所示,但是HRM分型分析却不存在这样的问题,故而使用归一化分析进行HRM分型时具有更好的稳健性。另一方面,目的片段越短,HRM分型结果越准确[35],为提高HRM分析精确性,寻找较短,合适的DNA条形码序列,采用精密度更高的HRM分析仪器以及使用稳定性更好的饱和染料是一种可行的途径。
|
图 6 不同模板浓度对熔解峰的影响 Figure 6 Effect of DNA concentration on melting peaks |
在分子鉴定领域内新方法的提出,往往需要方法学的验证。方法学验证包括:(1)灵敏性与特异性。灵敏性是指待测样品中测试阳性与实际阳性结果之间的百分比,即阳性结果/(阳性+假阴性),特异性是所测样品中测试阴性与实际阴性结果之间的百分比,即阴性结果/(阴性+假阳性)。灵敏性的考察主要是避免假阴性结果的出现,特异性的考察主要是避免假阳性结果的出现;(2)重复性。如操作重复性,即同一的操作者在同一天内操作的结果的稳定性,与仪器重复性,即同一操作者在不同时期内的结果的稳定性;(3)稳健性,即条件发生微小改变时结果保持稳定的能力,包括分子鉴定关键条件的稳健性;(4)检出限,是方法和仪器灵敏度体现的重要指标之一。1998年,IUPAC发表的《分析术语纲要》(IUPAC Compendium of Analysis Nomenclature)中规定:检出限以浓度(或质量)表示,是指由特定的分析步骤能够合理地检测出的最小分析信号从而求得的最低浓度(或质量),并规定应通过实验以足够多(测定次数n≥20)的测定次数求出。参考分析方法验证的指导原则,检出限的测定可分为仪器的检出限(instrument detection limit,IDL)和方法检出限(method detection limit,MDL)2种。IDL是指分析仪器能够检测的被分析物的最低量或浓度,这个浓度与特定的仪器能够从背景噪音中辨别的最小响应信号相对,即是通过已知低浓度的被测物的检测信号与基线噪音比较,确定可被检测出的最小量或浓度(一般将信噪比为3:1或2:1的量或浓度作为检出限)。该方法主要用于仪器检出限的测量和判断。MDL是指在通过某一种分析方法的全部处理和测定过程后,被测定物质产生的信号能以99%置信度区别于空白样品而被测定出来的最低浓度。方法检出限与仪器检出限相似,但考虑了样品分析前所有制备过程的影响。分析方法的检出限不仅与仪器噪音有关,还取决于整个分析过程的误差总和,与样品性质、基质及预处理过程等都有关系[36];(5)混伪品的检出,即在正品样品中混入不同比例伪品样品时伪品的检出能力。对新建立的分子鉴定方法进行此类方法学验证才能保证方法的准确与可靠性,本文建立了人参属HRM分子鉴别方法,并对其灵敏性、特异性、重复性、稳健性、检出限和混伪品检出限进行了考察[37-39]。
HRM分析方法不受样品DNA双链突变碱基位点与类型的局限,无需序列特异性探针,且闭管操作避免污染,同时实验操作与后期数据分析简单、易懂。本文以HRM分析方法建立人参属物种为例,引入了方法学验证进入中药分子鉴定方法,为分子鉴定新方法的建立,提供了一套可行性评价系统。
| [1] |
ZUO Y, CHEN Z, KONDO K, et al. DNA barcoding of Panax species[J]. Planta Med, 2011, 77(2): 182. DOI:10.1055/s-0030-1250166 |
| [2] |
ZHU S, FUSHIMI H, CAI S, et al. Species identification from ginseng drugs by multiplex amplification refractory mutation system(MARMS)[J]. Planta Med, 2004, 70(2): 189. DOI:10.1055/s-2004-815502 |
| [3] |
王川易, 郭宝林, 肖培根. 中药分子鉴定方法评述[J]. 中国中药杂志, 2011, 36(3): 237. WANG CY, GUO BL, XIAO PG. Molecuar methods for authentication of Chinese medicinal materials[J]. China J Chin Mater Med, 2011, 36(3): 237. |
| [4] |
HEUBL G. New aspects of DNA-based authentication of Chinese medicinal plants by molecular biological techniques[J]. Planta Med, 2010, 76(17): 1963. DOI:10.1055/s-0030-1250519 |
| [5] |
SHAW PC, BUT PP. Authentication of Panax species and their adulterants by random-primed polymerase chain reaction[J]. Planta Med, 1995, 61(5): 466. DOI:10.1055/s-2006-958138 |
| [6] |
HA WY, SHAW PC, LIU J, et al. Authentication of Panax ginseng and Panax quinquefolius using amplified fragment length polymorphism(AFLP) and directed amplification of minisatellite region DNA(DAMD)[J]. J Agric Food Chem, 2002, 50(7): 1871. DOI:10.1021/jf011365l |
| [7] |
DIAO Y, LIN XM, LIAO CL, et al. Authentication of Panax ginseng from its adulterants by PCR-RFLP and ARMS[J]. Planta Med, 2009, 75(5): 557. DOI:10.1055/s-0029-1185321 |
| [8] |
UM JY, CHUNG HS, KIM MS, et al. Molecular authentication of Panax ginseng species by RAPD analysis and PCR-RFLP[J]. Biol Pharm Bull, 2001, 24(8): 872. DOI:10.1248/bpb.24.872 |
| [9] |
WANG J, HA WY, NGAN FN, et al. Application of sequence characterized amplified region(SCAR) analysis to authenticate Panax species and their adulterants[J]. Planta Med, 2001, 67(8): 781. DOI:10.1055/s-2001-18340 |
| [10] |
WANG H, KIM MK, KWON WS, et al. Molecular authentication of Panax ginseng and ginseng products using robust SNP markers in ribosomal external transcribed spacer region[J]. J Pharm Biomed Anal, 2011, 55(5): 972. DOI:10.1016/j.jpba.2011.03.037 |
| [11] |
崔光红, 黄璐琦, 唐晓晶, 等. 获取人参、西洋参特定序列位点(STS)标记的新方法[J]. 中国中药杂志, 2007, 32(11): 1012. CUI GH, HUANG LQ, TANG XJ, et al. Application of multiplex allele-specific PCR for authentication of Panax ginseng and P. quinquefolius[J]. China J Chin Mater Med, 2007, 32(11): 1012. DOI:10.3321/j.issn:1001-5302.2007.11.003 |
| [12] |
BANG KH, JO IH, KIM YC, et al. Molecular identification of Korean ginseng cultivars(Panax ginseng CA Mey.) using peptide nucleic acid(PNA) microarray[J]. Korean J Med Crop Sci, 2012, 20(5): 387. DOI:10.7783/KJMCS.2012.20.5.387 |
| [13] |
LEE JW, BANG KH, CHOI JJ, et al. Development of peptide nucleic acid(PNA) microarray for identification of Panax species based on the nuclear ribosomal internal transcribed spacer(ITS) and 5.8 S rDNA regions[J]. Genes Genom, 2010, 32(5): 463. DOI:10.1007/s13258-010-0040-7 |
| [14] |
任保青, 陈之端. 植物DNA条形码技术[J]. 植物学报, 2010, 45(1): 1. REN BQ, CHEN ZD. DNA barcoding plant life[J]. Chin Bull Bot, 2010, 45(1): 1. |
| [15] |
KRESS WJ, WURDACK KJ, ZIMMER EA, et al. Use of DNA barcodes to identify flowering plants[J]. Proc Natl Acad Sci USA, 2005, 102(23): 8369. DOI:10.1073/pnas.0503123102 |
| [16] |
LI D, GAO L, LI H, et al. Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS) should be incorporated into the core barcode for seed plants[J]. P Natl Acad Sci USA, 2011, 108(49): 19641. DOI:10.1073/pnas.1104551108 |
| [17] |
CHEN S, YAO H, HAN J, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species[J]. PloS One, 2010, 5(1): e8613. DOI:10.1371/journal.pone.0008613 |
| [18] |
HOLLINGSWORTH PM, FORREST LL, SPOUGE JL, et al. A DNA barcode for land plants[J]. P Natl Acad Sci USA, 2009, 106(31): 12794. DOI:10.1073/pnas.0905845106 |
| [19] |
LI M, CAO H, BUT PP, et al. Identification of herbal medicinal materials using DNA barcodes[J]. J Syst Evol, 2011, 49(3): 271. DOI:10.1111/jse.2011.49.issue-3 |
| [20] |
LUO K, CHEN S, CHEN K, et al. Assessment of candidate plant DNA barcodes using the Rutaceae family[J]. Sci China Life Sci, 2010, 53(6): 701. DOI:10.1007/s11427-010-4009-1 |
| [21] |
TECHEN N, PARVEEN I, PAN Z, et al. DNA barcoding of medicinal plant material for identification[J]. Curr Opin Biotech, 2014, 25(2): 103. |
| [22] |
CHEN X, LIAO B, SONG J, et al. A fast SNP identification and analysis of intraspecific variation in the medicinal Panax species based on DNA barcoding[J]. Gene, 2013, 530(1): 39. DOI:10.1016/j.gene.2013.07.097 |
| [23] |
RIRIE KM, RASMUSSEN RP, WITTWER CT. Product differentiation by analysis of DNA melting curves during the polymerase chain reaction[J]. Anal Biochem, 1997, 245(2): 154. DOI:10.1006/abio.1996.9916 |
| [24] |
DRUML B, CICHN MM. High resolution melting(HRM) analysis of DNA-its role and potential in food analysis[J]. Food Chem, 2014, 158(9): 245. |
| [25] |
ARTHOFER W, STEINER FM, SCHLICK SBC. Rapid and cost-effective screening of newly identified microsatellite loci by high-resolution melting analysis[J]. Mol Genet Genom, 2011, 286(3-4): 225. DOI:10.1007/s00438-011-0641-0 |
| [26] |
WOJDACZ TK, DOBROVIC A. Methylation-sensitive high resolution melting(MS-HRM):a new approach for sensitive and high-throughput assessment of methylation[J]. Nucl Acids Res, 2007, 35(12): e41. |
| [27] |
MARTINO A, MANCUSO T, ROSSI AM. Application of high-resolution melting to large-scale, high-throughput SNP genotyping:a comparison with the TaqMan method[J]. J Biomol Screen, 2010, 15(6): 623. DOI:10.1177/1087057110365900 |
| [28] |
JAAKOLA L, SUOKAS M, HAGGMAN H. Novel approaches based on DNA barcoding and high-resolution melting of amplicons for authenticity analyses of berry species[J]. Food Chem, 2010, 123(2): 494. DOI:10.1016/j.foodchem.2010.04.069 |
| [29] |
GANOPOULOS I, MADESIS P, DARZENTAS N, et al. Barcode high pesolution melting(Bar-HRM) analysis for detection and quantification of PDO "Fava Santorinis"(Lathyrus clymenum) adulterants[J]. Food Chem, 2012, 133(2): 505. DOI:10.1016/j.foodchem.2012.01.015 |
| [30] |
FARIA MA, MAGALHAES A, NUNES ME, et al. High resolution melting of trnL amplicons in fruit juices authentication[J]. Food Control, 2013, 33(1): 136. DOI:10.1016/j.foodcont.2013.02.020 |
| [31] |
MADESIS P, GANOPOULOS I, ANAGNOSTIS A, et al. The application of Bar-HRM(Barcode DNA-high resolution melting) analysis for authenticity testing and quantitative detection of bean crops(Leguminosae) without prior DNA purification[J]. Food Control, 2012, 25(2): 576. DOI:10.1016/j.foodcont.2011.11.034 |
| [32] |
GANOPOULOS I, BAZAKOS C, MADESIS P, et al. Barcode DNA high-resolution melting(Bar-HRM) analysis as a novel close-tubed and accurate tool for olive oil forensic use[J]. J Sci Food Agric, 2013, 93(9): 2281. DOI:10.1002/jsfa.2013.93.issue-9 |
| [33] |
崔光红, 唐晓晶, 黄璐琦. 含淀粉及多糖类中药材DNA的提取方法研究[J]. 中国中药杂志, 2006, 31(16): 1365. CUI GH, TANG XJ, HUANG LQ. The studies for DNA extraction from Chinese herbal medicines which contain polysaccharides and starch[J]. China J Chin Mater Med, 2006, 31(16): 1365. DOI:10.3321/j.issn:1001-5302.2006.16.018 |
| [34] |
NORAMBUENA PA, COPELAND JA, KRENKOVA P, et al. Diagnostic method validation:high resolution melting(HRM) of small amplicons genotyping for the most common variants in the MTHFR gene[J]. Clin Biochem, 2009, 42(12): 1308. DOI:10.1016/j.clinbiochem.2009.04.015 |
| [35] |
陈康, 蒋超, 袁媛, 等. 高分辨率熔解曲线技术及其在中药DNA分子鉴定中的应用[J]. 药学学报, 2015, 50(12): 1581. CHEN K, JIANG C, YUAN Y, et al. High resolution melting and its application in identity study of traditional Chinese medicine[J]. Acta Pharm Sin, 2015, 50(12): 1581. |
| [36] |
鲁静, 付凌燕, 王旭. 质量分析方法验证中检出限和定量限测定方法探讨[J]. 中国药品标准, 2012, 13(1): 33. LU J, FU LY, WANG X. Discussion on the detecting methods for limit of detection and limit of quantification in validation of analytical method[J]. Drug Stand China, 2012, 13(1): 33. |
| [37] |
PRENCE EM. A practical guide for the validation of genetic tests[J]. Genet Test, 1999, 3(2): 201. DOI:10.1089/gte.1999.3.201 |
| [38] |
EP6-A, Evaluation of the Linearity of Quantitative Measurement Procedures:A Statistical Approach; Approved Guideline[S].2003
|
| [39] |
EP15-A, User Demonstration of Performance for Precision and Accuracy; Approved Guideline[S].2001
|
 2017, Vol. 37
2017, Vol. 37 
