治疗性单克隆抗体药物以其靶向性强,疗效明确等优势已经成为生物技术药物中增长最快的领域,由武汉生物制品研究所有限责任公司研制的抗人TNF-α单克隆抗体是一种特异性中和人肿瘤坏死因子α(TNF-α)的重组全人源IgG1κ型治疗性单克隆抗体,可用于风湿关节炎(RA)等自身免疫性疾病的治疗。与小分子药物相比,单克隆抗体类药物结构复杂且翻译后修饰众多,而CDR肽段是鉴别不同单克隆抗体的特异性序列,美国药典药物处方集中明确规定在系统适用性研究时需要采用适当的方法鉴别出抗体的CDR肽段[1],而如何在常规的质控中实现CDR肽段的识别仍然具有挑战性。肽图分析能提供丰富的结构信息,并且在一级结构的确认上具有很多优点,逐渐成为单克隆抗体类药物的常规检测指标,目前2015年版中国药典四部中肽图检查方法[2]250与2010年版相比无较大变化,均采用液相肽图的方法。2015年版中国药典三部中“人用重组DNA蛋白制品总论”及新增的“人用重组单克隆抗体产品总论”中均要求理化特性分析应尽可能采用综合的方法测定目标产品的氨基酸序列,并与基因推断的理论氨基酸序列进行比较[3],其中肽图因其高度特异性并且针对分子结构,被推荐作为专属性鉴别的一种方法,而基于液相色谱质谱联用技术的肽图分析方法因为能与理论氨基酸序列进行匹配而在现在的单克隆抗体药物质量控制中得到逐渐广泛的应用[4-7]。质谱技术现已成为治疗性单克隆抗体结构表征的关键技术手段[8],对结果的解析具有高度的精确性,可在分子水平上对单抗药物的分子量信息、N末端氨基酸序列、肽图分析[9]、二硫键定位[10-11]、糖基化修饰[12-13]以及天冬酰胺脱酰胺、天冬氨酸异构化、甲硫氨酸/色氨酸氧化、非酶作用赖氨酸糖化等化学修饰进行鉴定[14-18]。特别是在肽图分析中,常规的紫外检测方法无法阐明每个特征峰的信息,对于共流出的色谱峰也无法做出组分分析,而质谱检测的信息可为上述问题提供重要的参考信息,并且可以准确鉴别出CDR所在肽段对应的色谱峰,将逐渐成为治疗性单克隆抗体产品的整个生命周期,包括早期的质量研究到日常的产品质量控制中的关键技术。本研究在质谱技术的基础上,建立了液质联用肽图法,对武汉生物制品研究所有限责任公司研制的单克隆抗体抗人TNF-α单抗进行了肽图分析,并对各个抗体识别抗原的关键区域即互补决定区(CDR)相关的特征峰进行了质谱鉴别;对该方法按照中国药典四部(2015年版)通则9101—药品质量标准分析方法验证指导原则进行了专属性、重复性、中间精密度、耐用性等验证,并对样品的储存稳定性做了考察。
1 供试品与仪器 1.1 样品及试剂利妥昔单抗来源于上海生物制品研究所,TNF-α单抗由武汉生物制品研究所有限责任公司研发生产制备。盐酸胍、二硫苏糖醇(DTT)、碘乙酰胺(IAA)、碳酸氢铵、甲酸购自Sigma公司,Tris、尿素购自Amresco公司,色谱级乙腈及甲醇购自Tedia公司,亮氨酸脑啡肽(LE)购自Waters公司;测序用修饰级胰蛋白酶购自Promega公司,超滤浓缩管购自Merck Millipore公司。
1.2 溶液的配制精确称取0.077 g DTT溶于1 mL去离子水中,即为0.5 mol·L-1 DTT溶液;精确称取0.092 5 g IAA溶于1 mL去离子水中,即为0.5 mol·L-1 IAA溶液;称取28.66 g盐酸胍溶于50 mL 0.25 mol·L-1 pH 7.5的Tris溶液中,即为6 mol·L-1盐酸胍溶液;称取0.79 g碳酸氢铵溶于少量水,稀盐酸调pH至7.8,定容至200 mL,即为50 mmol·L-1碳酸氢铵溶液;称取12.012 g尿素溶于100 mL 50 mmol·L-1碳酸氢铵溶液中,即为含50 mmol·L-1碳酸氢铵的2 mol·L-1尿素溶液。
1.3 仪器Waters ACQUITY UPLC超高效液相色谱系统,配TUV可调波长紫外检测器、Xevo G2 Q Tof串联四极杆飞行时间质谱仪;Waters UPLC BEH300 C18(2.1 mm×150 mm,1.7 μm)色谱柱。Masslynx 4.1软件控制LC/MS仪器和采集数据,Biopharmlynx1.3.2软件用于处理采集后的数据。Nanodrop ND1000微量紫外分光光度计。
2 方法 2.1 样品处理取单抗100 μL,加入6 mol·L-1盐酸胍400 μL及0.5 mol·L-1的DTT溶液10 μL,37 ℃变性及还原1 h,再加入烷基化试剂0.5 mol·L-1的IAA溶液24 μL,室温避光放置30 min;加入0.5 mol·L-1的DTT溶液10 μL,混合均匀。用10 K超滤离心管超滤多次置换其缓冲液为50 mmol·L-1碳酸氢铵的2 mol·L-1尿素溶液,并对置换后样品采用紫外分光光度法对浓度进行测定。取适量置换后样品按样品与胰蛋白酶质量比25:1加入胰蛋白酶,置于37 ℃水浴孵育17 h,之后取出加入1 μL甲酸终止酶切反应。
2.2 色谱条件采用Waters UPLC BEH 300 C18(2.1 mm×150 mm,1.7 μm)色谱柱,以0.1%甲酸水溶液(A)-0.1%甲酸乙腈溶液(B)为流动相,梯度洗脱(5~120 min,2%B→45%B),流速为0.2 mL·min-1,检测波长为214 nm,柱温60 ℃,样品室温度设置为8 ℃。
2.3 质谱条件采用电喷雾离子源(ESI),正离子模式,脱溶剂气温度350 ℃,源温120 ℃,脱溶剂气流速600 L·h-1,毛细管电压3 kV,锥孔电压40 V,数据采集范围m/z为100~1 990。采用MSE模式获得各个肽段母离子一级质谱数据及二级质谱数据,利用质谱校正通道每间隔20 s注射亮氨酸脑啡肽(LE)溶液(200 pg·μL-1,溶剂为50%乙腈/水溶液),流速10 μL·min-1,获得Lock mass参比质量信号,对测得的质量数进行实时校准。
2.4 软件设置使用MassLynx 4.1软件控制仪器并采集数据,经Biopharmlynx 1.3.2软件处理。软件参数设置如下:选择胰蛋白酶作为酶切试剂,母离子及碎片离子的质量偏差设置为30 ppm,漏切位点设置为2,烷基化被选为固定修饰,可变修饰选择为末端赖氨酸截除,N末端焦谷氨酸环化、甲硫氨酸氧化、脱酰胺、多种N-糖基化(G0F,G1F,G2F,G0-GlcNAc,G0F-GlcNAc,G1F+SA,G2F+SA,Man5等),确证一段肽段与理论序列相符的依据为二级质谱的碎片离子b/y值大于3。
2.5 验证用样品处理 2.5.1 专属性验证TNF-α单抗、利妥昔单抗、基质对照(成品辅料溶液)均按“2.1”项方法进行变性处理并酶切获得消化溶液,胰酶对照以50 mmol·L-1碳酸氢铵的2 mol·L-1尿素溶液取代样品进行酶切获得消化溶液,采用初始流动相即2%乙腈水溶液作为空白对照,所有样品按上述色谱及质谱条件进行液质肽图分析。
2.5.2 精密度-重复性验证由1名实验人员按“2.1”项方法处理TNF-α单抗即得TNF-α单抗消化溶液,对该溶液按“2.2”项色谱条件及“2.3”项质谱条件进样分析,重复进样5次。
2.5.3 中间精密度验证由2名不同人员按“2.1”项方法分别制备2份TNF-α单抗消化溶液以及同一人员在不同时间按“2.1”项方法制备TNF-α单抗消化溶液,对不同人员同一时间制备所得的TNF-α单抗消化溶液各进样1次分析;同一人员于不同时间制备所得的TNF-α单抗消化溶液各进样1次分析。
2.5.4 耐用性验证对TNF-α单抗按“2.1”项方法处理至酶切步骤,分别按不同的抗体与胰蛋白酶质量比(20:1、25:1、30:1)加入胰蛋白酶,置于37 ℃水浴孵育17 h后终止,即得不同酶切比例下TNF-α单抗消化溶液;对TNF-α单抗按“2.1”项方法处理,按抗体与胰酶质量比25:1加入胰蛋白酶,水浴孵育不同时间(17、20、23 h)后终止,既得不同孵育时间TNF-α单抗消化溶液。对不同酶切比例下及不同孵育时间的TNF-α单抗消化溶液各进样1次分析。
2.5.5 稳定性考察制备的TNF-α单抗消化溶液在UPLC样品管理器中的样品室(8 ℃)中储存0、12、24 h时各进样1次;制备的TNF-α单抗消化溶液分装并储存于-20 ℃,于0、1、5 d时溶解各进样1次。
3 结果 3.1 TNF-α单抗的CDR鉴别TNF-α单抗为全人源IgG1单抗,按“2.1”项方法处理后获得的消化溶液,按“2.2”项色谱条件及“2.3”项质谱条件进行液质肽图分析,液质肽图结果见图 1,分析其序列覆盖率达100%(如图 2),在已知氨基酸序列内找出酶切后CDR所属理论肽段,找出b/y碎片离子可信度最高的该肽段,并根据其质量数在去卷积化处理后的质谱图内找出对应肽段,根据该肽段峰标示色谱保留时间找出对应色谱峰,即鉴定出CDR对应的特征峰。CDR所在胰酶酶切肽段、序列及在源内电离后主要荷质比形式结果见表 1,含下划线的序列为CDR序列,各CDR质谱鉴定结果见图 3,通过MSE高能量碎裂获得的CDR肽段的二级碎片b、y离子信息(图 4,以重链及轻链CDR1为例)确证了该肽段的序列。由于在该单抗的轻链部分可变区序列内存在胰酶酶切位点,2:T4及2:T9肽段只包含可变区部分氨基酸,并作为LC CDR1及LC CDR3的鉴别肽段。HC CDR2及HC CDR3经质谱解析分析两者在色谱峰图中共流出。肽段在色谱中的保留时间是由肽段的理化性质决定的,如氨基酸疏水性、肽段长度、肽段分子质量等。CDR2及CDR3相关肽段虽然质荷比不同(m/z分别为1 332.759 5和1 405.861 3),但在色谱图中体现为共流出峰,可能是由于2个肽段疏水性质相近,在反相色谱中的保留行为基本一致,两者仅通过一维的反相色谱无法区分,而质谱检测为2个共流出的CDR鉴定提供了重要的依据。
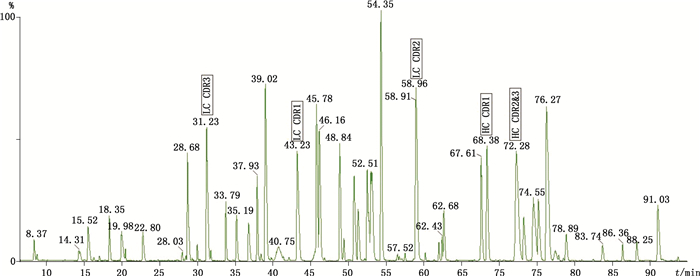
|
图 1 TNF-α单抗提取离子流色谱 Figure 1 BPI chromatogram of TNF-α monoclonal antibody(mAb) |
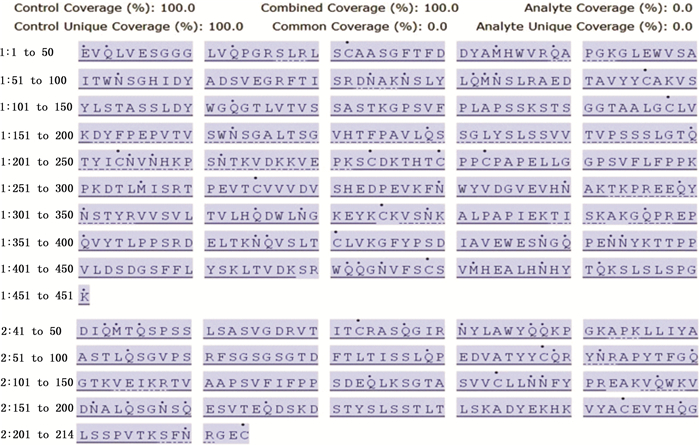
|
图 2 TNF-α单抗序列覆盖率 Figure 2 Coverage of TNF-α mAb sequence |
|
|
表 1 TNF-α单抗CDR对应肽段及离子化信息 Table 1 Related peptides of CDR and ionization information |
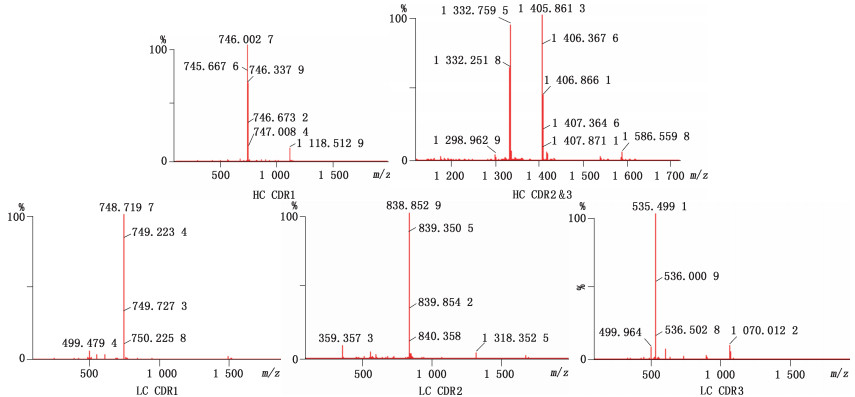
|
图 3 TNF-α单抗CDR质谱鉴别 Figure 3 Mass spectra identification of CDR of TNF-α mAb |
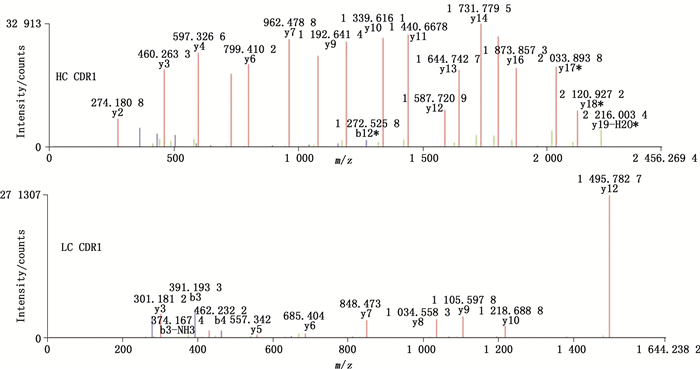
|
图 4 TNF-α单抗CDR肽段MSE谱图(HC CDR1在1:T3*肽段;LC CDR1在2:T4肽段) Figure 4 MSE spectra of CDR peptides of TNF-α mAb(HC CDR1 was at 1:T3* peptide; LC CDR1 was at 2:T4 peptide) |
专属性考察的结果表明空白对照及基质对照在样品出峰时间内无干扰峰出现,胰酶对照无自消化干扰峰出现(如图 5-a、b、c),TNF-α单抗的肽图结果如图 5-d所示,主要峰形与图 1一致;利妥昔单抗的液质肽图见图 5-e,序列覆盖率达到98.2%(结果未显示),对相应的CDR氨基酸序列所属的肽段进行搜寻,鉴定出该单抗的所有可变区,结果见表 2,各CDR质谱图见图 6。其中轻链CDR1及CDR2位于同一胰酶酶切肽段内,重链的CDR3在色谱图内无对应色谱峰,这一结果与文献报道[19]一致。利妥昔单抗消化溶液对照与TNF-α单抗消化溶液的CDR对应肽段出峰时间不一致,达到专属性鉴别的目的,可定性鉴定出TNF-α单抗。
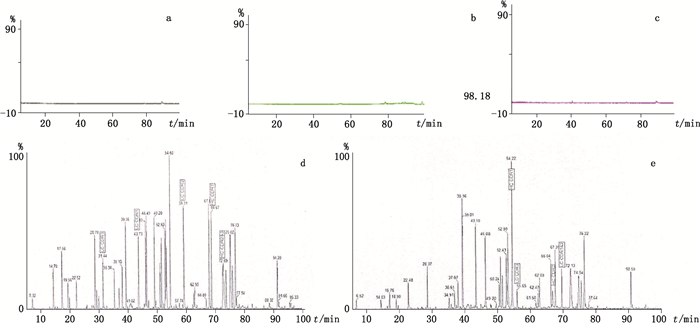
|
a.空白对照(blank control)b.基质对照(matrix control)c.胰蛋白酶对照(trypsin control)d.TNF-α单抗(TNF-α mAb)e.利妥昔单抗(rituximab) 图 5 专属性色谱图 Figure 5 Chromatograms of specificity |
|
|
表 2 利妥昔单抗CDR区对应肽段及离子化信息 Table 2 Related peptides of CDR of rituximab and ionization information |
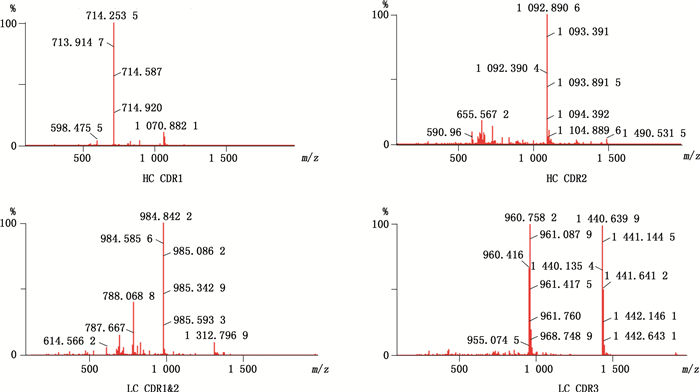
|
图 6 利妥昔单抗CDR质谱图 Figure 6 Mass spectra maps of CDR of rituximab |
反相色谱的色谱行为重现性除了肽段自身的理化性质外,色谱柱填料、流动相组成、流速、外界温度等对保留时间也会产生影响[20]。为了消除其他因素对保留时间变化的影响,选定了质荷比为1 344(M+5)的峰作为参考峰(出峰时间约为91 min),在本实验中仅考虑特征峰与此参考峰之间的相对保留变化。该色谱峰为胰蛋白酶酶切后序列最长的肽段1:T13,疏水性较强,在反相色谱中较晚出峰。在重复性试验中,以CDR的相对保留时间(特征峰的保留时间与参考峰保留时间之比)的相对标准偏差(RSD)评估重复性。重复性针对同一TNF-α单抗样品消化溶液进样5次,结果见图 7所示,CDR相对保留时间的RSD在0.57%~1.19%之间(见表 3)。
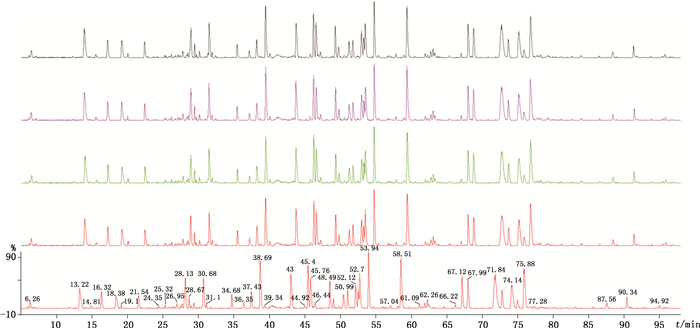
|
图 7 重复性色谱图 Figure 7 Chromatograms of repeatability |
|
|
表 3 重复性检测CDR相对保留时间变异 Table 3 RRT variation of CDR in repeatability detection |
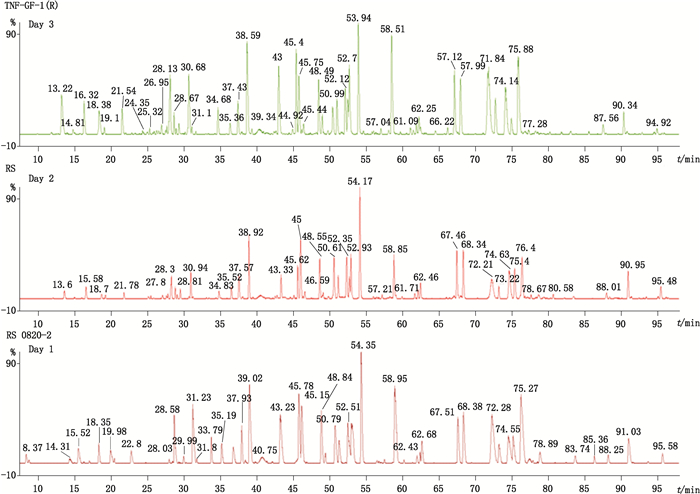
中间精密度考察不同人员及不同时间下样品制备时的变异程度,结果如表 4所示,不同人员(见图 8-a、b)及不同时间(见图 9)处理的TNF-α单抗消化溶液CDR相对保留时间的RSD在0.00%~1.08%之间(见表 4)。
|
|
表 4 不同人员及时间CDR相对保留时间变异 Table 4 RRT variation of CDR in precision detection |
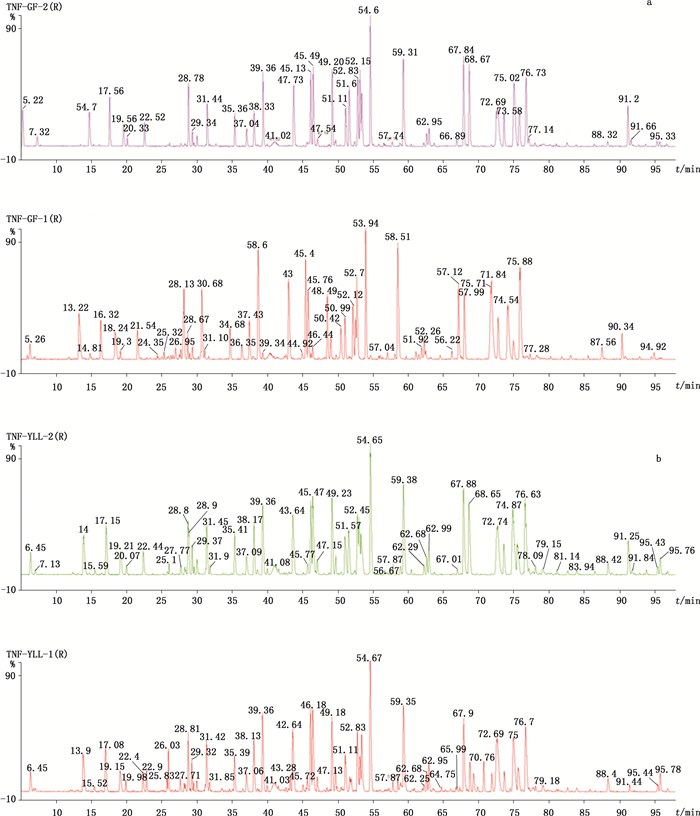
|
图 8 人员1(a)及人员2(b)制备的TNF-α单抗消化溶液图谱 Figure 8 Chromatograms of TNF-α mAb digested solution prepared by analyst 1(a) and analyst 2(b) |
|
图 9 不同时间制备的样品的图谱 Figure 9 Chromatograms of sample prepared in different time |
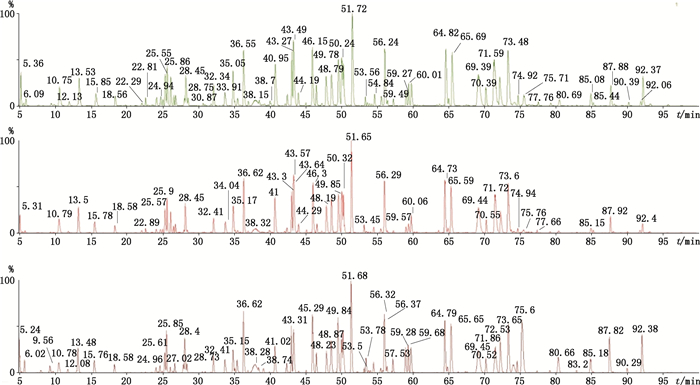
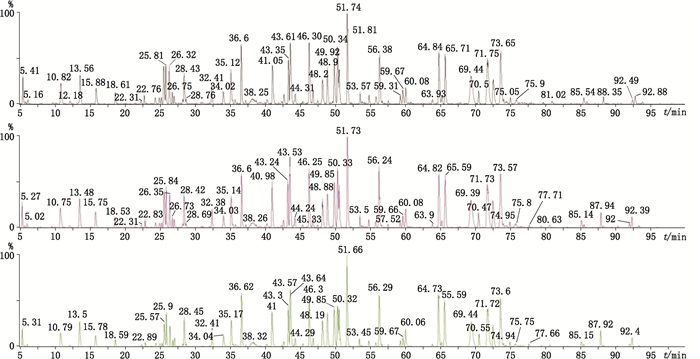
耐用性对胰蛋白酶酶切条件进行了考察,在测定条件有小的变动时,测定结果受影响的程度,为所建立的方法日后用于日常检验提供依据,以确保方法的可靠性。选择前述实验条件酶切比例(25:1)和酶切时间(17 h)等主要影响因素做均匀设计[2]374,确定主要影响因素变动范围酶切比例20:1~30:1,酶切时间17~23 h。实验结果显示在酶切比例在20:1~30:1,酶切时间17~23 h检测出的CDR相对保留时间的RSD均较小(见图 10、11及表 5)。
|
图 10 不同酶切比例色谱图(从上之下依次为30:1,25:1,20:1) Figure 10 Chromatograms of different trypsin digestion ratios(from top to bottom for 30:1, 25:1, 20:1) |
|
图 11 不同酶切时间色谱图(从上至下依次为23 h、20 h、17 h) Figure 11 Chromatograms of different trypsin digestion time(from top to bottom for 23, 20, 17 h) |
|
|
表 5 不同条件下耐用性相对保留时间变异考察 Table 5 Ruggedness of RRT variation at different conditions |
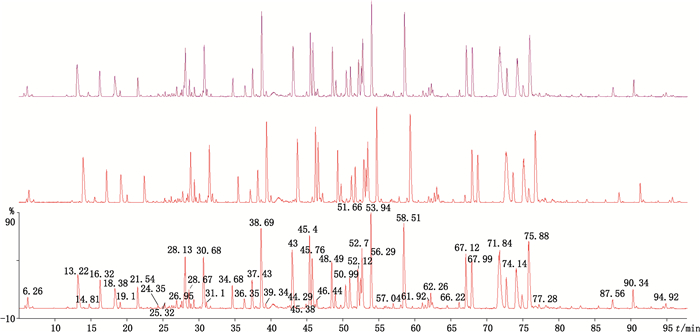
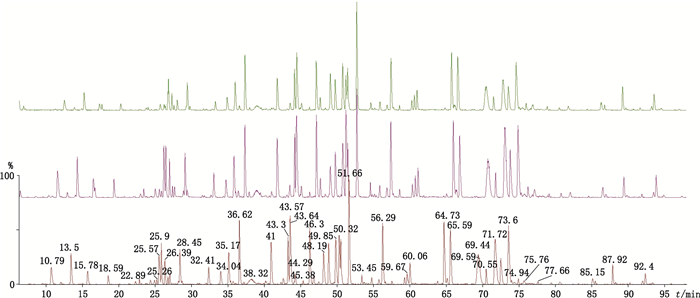
分别考察了TNF-α单抗消化溶液在样品室内(8 ℃)的储存稳定性及-20 ℃的冻存稳定性。结果表明在8 ℃下储存24 h(见图 12及表 6)及在-20℃下储存5 d(见图 13及表 7)重复性良好。
|
图 12 8 ℃储存稳定性色谱图(从上之下依次为24、12、0 h) Figure 12 Chromatograms of stock stability at 8 degrees centigrade(from top to bottom for 24, 12, 0 h) |
|
|
表 6 8 ℃下相对保留时间变异 Table 6 Variation of RRT at 8 degrees centigrade |
|
图 13 -20 ℃冻存稳定性色谱图(从上之下依次为0、1、5 d) Figure 13 Chromatograms of stock stability at 20 degrees centigrade below zero(from top to bottom for 0, 1, 5 d) |
|
|
表 7 -20 ℃下相对保留时间变异 Table 7 Variation of RRT at 20 degrees centigrade below zero |
本研究采用集高分离能力的液相色谱和高灵敏度及高选择性的质谱于一体的液质联用技术对TNF-α单抗进行肽图分析。具有不同极性的肽段经液相分离后进入电喷雾离子源离子化,经串联四极杆和飞行时间质谱对分离出来的肽段进行一级质谱数据采集及二级碎片离子MSE模式的数据采集。从一级质谱的荷质比信息数据可以大致确定肽段,根据二级质谱可以确定一段肽段内的每个氨基酸及序列信息。
对于单克隆抗体类蛋白,可变区内的互补决定区直接决定了抗体的靶点特异性,因此可以根据互补决定区的肽段序列鉴别序列差异小的单克隆抗体,在质量控制工作中,用于专属性鉴别试验[21]。
肽图样品处理过程中涉及到蛋白的胍盐变性、二硫键还原、烷基化封闭、缓冲液置换、特异性酶切等步骤,在样品处理过程中容易发生在高温和高pH条件下发生的天冬酰胺脱酰胺、N末端环化、甲硫氨酸氧化等人为造成的修饰[22],RP-HPLC方法无法对新出的色谱峰进行定性分析,故而在结果判定上往往带有主观性。而液相色谱质谱串联技术不但可通过紫外对肽段进行定量,还可以对每个色谱峰的具体信息进行质荷比分析,经软件分析得出序列覆盖率,可作为判断一致性的客观判断指标,增加方法的适用性,尤其在原研药及其类似药的质量可比性研究、工艺一致性研究、样品的稳定性研究上优势显著[23]。在对多种抗体进行序列解析时,在已知序列的基础上,结合序列覆盖率和CDR的鉴别,更有利于对单抗进行专属性的检测且意义显著。
本研究建立的液质联用肽图法用于TNF-α单抗的鉴别实验,专属性好,辅料成分及样品前处理中涉及的试剂、消化用胰酶对谱图的检测均无干扰。重复性测定CDR相对保留时间的RSD在0.57%~1.19%之间,精密度实验测得CDR相对保留时间的RSD在0.00%~1.08%之间,耐用性考察峰图基本一致,相对保留时间的RSD在0.22%~0.26%之间。不同温度下的储存稳定性考察结果也显示在8 ℃下储存24 h,-20 ℃下储存5 d,CDR相对保留时间变异较小。以上数据显示了该方法具有良好的专属性、重复性、精密度、耐用性,消化后样品能保证一定的稳定性,可用于抗人TNF-α单抗的日常质量控制及批检验放行中。
| [1] |
ZHANG JJ, QIN T, XU L, et al. Development and validation of a peptide mapping method for the characterization of adalimumab with QDa detector[J]. Chromatographia, 2016, 79(7): 395. |
| [2] |
中国药典2015年版. 四部[S]. 2015: 250, 374 ChP 2015.Vol Ⅳ[S].2015:250, 374 |
| [3] |
中国药典2015年版. 三部[S]. 2015: 37 ChP 2015.Vol Ⅲ[S].2015:37 |
| [4] |
AEBERSOLD R, MANN M. Mass spectrometry-based proteomics[J]. Nature, 2003, 422(6928): 198. DOI:10.1038/nature01511 |
| [5] |
BONGERS J, CUMMINGS JJ, EBERT MB, et al. Validation of a peptide mapping method for a therapeutic monoclonal antibody:what could we possibly learn about a method we have run 100 times[J]. J Pharm Biomed Anal, 2000, 21(6): 1099. DOI:10.1016/S0731-7085(99)00181-8 |
| [6] |
VISSER J, FEUERSTEIN I, STANGLER T, et al. Physicochemical and functional comparability between the proposed biosimilar rituximab GP2013 and originator rituximab[J]. BioDrugs, 2013, 27(5): 495. DOI:10.1007/s40259-013-0036-3 |
| [7] |
CHIRINO AJ, ANTHONY MS. Characterizing biological products and assessing comparability following manufacturing changes[J]. Nat Biotechnol, 2004, 22(11): 1383. DOI:10.1038/nbt1030 |
| [8] |
AYOUB D, JABS W, RESEMANN A, et al. Correct primary structure assessment and extensive glycoprofiling of cetuximab by a combination of intact, middle-up, middle-down and bottom-up ESI and MALDI mass spectrometry techniques[J]. MAbs, 2013, 5(5): 699. DOI:10.4161/mabs.25423 |
| [9] |
陶磊, 饶春明, 高凯, 等. 重组嵌合抗CD20 IgG1型单克隆抗体的结构验证[J]. 药学学报, 2010, 45(6): 752. TAO L, RAO CM, GAO K, et al. Structure verification of a recombinant chimeric anti-CD20 IgG1 monoclonal antibody[J]. Acta Pharm Sin, 2010, 45(6): 752. |
| [10] |
LIU HC, MAY K. Disulfide bond structures of IgG molecules:structural variations, chemical modifications and possible impacts to stability and biological function[J]. MAbs, 2012, 4(1): 17. DOI:10.4161/mabs.4.1.18347 |
| [11] |
LI XJ, XU W, PAPORELLO B, et al. Liquid chromatography and mass spectrometry with post-column partial reduction for the analysis of native and scrambled disulfide bonds[J]. Anal Biochem, 2013, 439(2): 184. DOI:10.1016/j.ab.2013.04.021 |
| [12] |
RAJU TS, JORDAN RE. Galactosylation variations in marketed therapeutic antibodies[J]. MAbs, 2012, 4(3): 385. DOI:10.4161/mabs.19868 |
| [13] |
YANG JM, AI JW, BAO YM, et al. Investigation of the correlation between charge and glycosylation of IgG1 variants by liquid chromatography-mass spectrometry[J]. Anal Biochem, 2014, 448(1): 82. |
| [14] |
PACE AL, WONG RL, ZHANG YT, et al. Asparagine deamidation dependence on buffer type, pH, and temperature[J]. J Pharm Sci, 2013, 102(6): 1712. DOI:10.1002/jps.23529 |
| [15] |
SREEDHARA A, CORDOBA A, ZHU Q, et al. Characterization of the isomerization products of aspartate residues at two different sites in a monoclonal antibody[J]. Pharm Res, 2012, 29(1): 187. DOI:10.1007/s11095-011-0534-2 |
| [16] |
HABERGER M, BOMANS K, DIEPOLD K, et al. Assessment of chemical modifications of sites in the CDRs of recombinant antibodies:Susceptibility vs.functionality of critical quality attributes[J]. MAbs, 2014, 6(2): 327. DOI:10.4161/mabs.27876 |
| [17] |
QUAN C, ALCALA E, PETKOVSKA I, et al. A study in glycation of a therapeutic recombinant humanized monoclonal antibody:where it is, how it got there, and how it affects charge-based behavior[J]. Anal Biochem, 2008, 373(2): 179. DOI:10.1016/j.ab.2007.09.027 |
| [18] |
BECK A, WAGNER-ROUSSET E, AYOUB D, et al. Characterization of therapeutic antibodies and related products[J]. Anal Chem, 2012, 85(2): 715. |
| [19] |
Rituximab USP Medicine Compendium Final Authorized Version 1.0[R].2013
|
| [20] |
刘超, 王海鹏, 付岩, 等. 肽段反相色谱保留时间预测算法及其在蛋白质鉴定中的应用[J]. 色谱, 2010, 28(6): 529. LIU C, WANG HP, FU Y, et al. Prediction of peptide retention time in reversed-phase liquid chromatography and its application in protein identification[J]. Chin J Chromatogr, 2010, 28(6): 529. |
| [21] |
张峰. 治疗性单克隆抗体类制品质量控制标准的思考[J]. 中国执业药师, 2013, 10(1): 25. ZHANG F. The quality control of therapeutic monoclonal antibody products[J]. China Licens Pharm, 2013, 10(1): 25. |
| [22] |
DICK LW JR, MATHON D, QIU D, et al. Peptide mapping of therapeutic monoclonal antibodies:Improvements for increased speed and fewer artifacts[J]. J Chromatogr B, 2009, 877(3): 230. DOI:10.1016/j.jchromb.2008.12.009 |
| [23] |
BECK A, SANGLIER-CIANFÉRANI S, Van DORSSELAER A. Biosimilar, biobetter and next generation antibody characterization by mass spectrometry[J]. Anal Chem, 2012, 84(11): 4637. DOI:10.1021/ac3002885 |
 2017, Vol. 37
2017, Vol. 37 
