文章信息
- 陈何, 王晓, 王乐, 赖钟雄, 刘生财
- CHEN He, WANG Xiao, WANG Le, LAI Zhongxiong, LIU Shengcai
- 苋菜AtrNiR基因密码子偏好性与进化分析
- Codon preference and evolution analysis of AtrNiR gene in Amaranthus tricolor L.
- 亚热带农业研究, 2021, 17(1): 48-56
- Subtropical Agriculture Research, 2021, 17(1): 48-56.
- DOI: 10.13321/j.cnki.subtrop.agric.res.2021.01.009
-
文章历史
- 收稿日期: 2021-01-22
氮素是植物生长发育必需的基本营养元素之一。硝态氮(NO3-)是植物可利用氮素的主要形式,硝态氮通过硝酸还原酶(nitrate reductase, NR)的作用转变为亚硝酸盐,亚硝酸盐再通过亚硝酸还原酶(nitrite reductase, NiR)的作用转变成铵。NiR是氮素同化途径的关键控制酶, 与NR两者偶联完成NO3-的无机同化[1]。目前,在菠菜[2]、白菜[3]和小麦[4]等植物中均克隆出了NiR基因。
在mRNA上决定1个氨基酸的3个相邻碱基称为密码子。同义密码子指编码相同氨基酸的密码子。在进化过程中,一些物种或一些基因倾向于使用特定的一组密码子进行氨基酸的编码,形成了同义密码子的使用偏好性[5]。对密码子使用偏好性进行分析,有助于了解基因转录与翻译的分子机制、确定最优密码子、选择合适外源表达宿主及提高蛋白质产量。基因密码子偏好性对基因表达水平也会有一定的影响,一般来说,基因密码子偏好性越强则其表达量越高[6]。研究密码子使用偏好性,不仅可以预测某些未知功能基因,而且可以为最佳密码子设计转基因表达载体[7]。
苋菜(Amaranthus tricolor L.)是富含甜菜色素的1年生草本植物[8],营养价值高,是我国夏季的主要蔬菜之一,在各地均有栽培。本课题组通过基因克隆克隆出苋菜AtrNiR基因,同时利用Codon W、SPSS 22、MEGA-X等软件,对苋菜AtrNiR基因与其他物种如甜菜、菠菜、藜麦的NiR基因进行比较,并比较其与拟南芥、大肠杆菌等常见模式生物基因组密码子的使用频率,以期为后续研究苋菜AtrNiR基因结构功能及选择合适的表达受体和表达系统提供参考。
1 材料与方法 1.1 序列数据来源及密码子偏好性参数分析苋菜AtrNiR(GenBank登录号:MT374155)开放阅读框(ORF)为1 785 bp,编码594个氨基酸。参照赵春丽等[8]试验方法,从GenBank数据库(http://www.ncbi.nlm.nih.gov/genbank/)获取甜菜、菠菜和藜麦等24种植物的NiR基因序列(表 1)。从Codon Usage Database数据库(http://www.kazusa.or.jp/codon/)获取甜菜(Beta vulgaris L.)、拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)、番茄(Solanum lycopersicum)、大肠杆菌(Escherichia coli)和酵母菌(Saccharomyces cerevisiae)6个模式生物的基因组密码子偏好性数据。参照王晓等[9]的数据分析方法,采用Codon W软件和EMBOSS在线程序统计密码子偏好性参数,包括苋菜AtrNiR基因及其他参试物种NiR基因编码序列的有效密码子数(effective number of codons, ENc)、密码子不同位置G/C含量(GC1、GC2、GC3)、密码子适应指数(codon adaptation index, CAI)、同义密码子相对使用度(relative synonymous codon usage, RSCU)等。
1.2 数据统计与分析 1.2.1 聚类分析及进化树构建利用Excel 2010对密码子相关参数进行重新统计,利用SPSS 22对参试物种NiR基因密码子偏好性进行聚类分析,使用MEGA-X构建不同物种NiR基因的CDS系统进化树。
1.2.2 密码子碱基组成分析分析密码子的碱基组成规律可揭示造成密码子偏好性的原因,利用Excel 2010、SPSS 22、Origin 2017软件对密码子相关参数进行分析并绘图。
1.2.3 AtrNiR基因受体系统的选择利用Excel 2010对苋菜AtrNiR与模式物种基因组密码子的使用频率进行比较,确定最适合苋菜AtrNiR的异源表达受体。
2 结果与分析 2.1 苋菜AtrNiR基因的密码子使用偏好性分析 2.1.1 GC含量及ENcENc值被认为是评价基因整体密码子偏性用法中最具参考价值的参数之一,该值在20(每个氨基酸只使用1个密码子的极端情况)到61(各个密码子均被平均使用)之间[10]。苋菜AtrNiR基因密码子选择偏好性相关参数(表 1)显示,苋菜AtrNiR基因的ENc值为54.04。该ENc值远离20,接近61,说明AtrNiR基因的密码子偏好性较弱,但在使用过程中仍存在一些偏好性较强的密码子。苋菜AtrNiR密码子的GC、GC1、GC2和GC3含量分别为45.5%、55.9%、39.1%和39.2%,说明该基因密码子偏好以A、T结尾。苋菜AtrNiR基因的CAI值为0.19,远低于1.0,进一步说明了AtrNiR对密码子偏好性较弱。
| 物种 | 基因 | 登录号 | ENc | CAI | GC含量 | GC1含量 | GC2含量 | GC3含量 | ||||
| % | % | % | % | |||||||||
| 双子叶植物 | 苋菜(Amaranthus tricolor) | AtrNiR | MT374155 | 54.04 | 0.19 | 45.50 | 55.90 | 39.10 | 39.20 | |||
| 甜菜(Beta vulgaris) | BvNiR | XM_010677274.2 | 52.89 | 0.19 | 46.70 | 57.60 | 39.20 | 40.50 | ||||
| 山茶(Camellia sinensis) | CsNiR | JX987133.1 | 56.19 | 0.22 | 51.80 | 51.50 | 40.80 | 61.10 | ||||
| 藜麦(Chenopodium quinoa) | CqNiR | XM_021870438.1 | 53.10 | 0.19 | 45.70 | 55.80 | 39.60 | 39.10 | ||||
| 菠菜(Spinacia oleracea) | SoNiR | XM_022011102.1 | 52.98 | 0.17 | 46.40 | 54.80 | 39.80 | 42.00 | ||||
| 番木瓜(Carica papaya) | CpNiR | XM_022041026.1 | 52.79 | 0.17 | 47.40 | 55.50 | 39.40 | 45.00 | ||||
| 黄檗(Phellodendron amurense) | PaNiR | KY496993.1 | 48.15 | 0.17 | 45.40 | 55.20 | 40.10 | 38.50 | ||||
| 榴莲(Durio zibethinus) | DzNiR | XM_022871500.1 | 51.84 | 0.18 | 45.70 | 55.00 | 39.70 | 39.90 | ||||
| 蓖麻(Ricinus communis) | RcNiR | XM_002518717.3 | 47.62 | 0.17 | 43.50 | 54.80 | 38.30 | 34.60 | ||||
| 黄胡萝卜(Daucus carota subsp. sativus) | DcNiR | XM_017371255.1 | 53.36 | 0.17 | 46.10 | 56.70 | 39.60 | 39.50 | ||||
| 番茄(Solanum lycopersicum) | SlNiR | XM_015201252.2 | 51.00 | 0.18 | 43.80 | 55.20 | 38.50 | 35.00 | ||||
| 烟草(Nicotiana tabacum) | NtNiR | XM_019401643.1 | 51.31 | 0.19 | 44.50 | 56.60 | 39.50 | 34.90 | ||||
| 大豆(Glycine max) | GmNiR | NM_001287455.1 | 50.38 | 0.18 | 47.10 | 54.10 | 40.30 | 44.10 | ||||
| 葡萄(Vitis vinifera) | VvNiR | NM_001281265.1 | 55.35 | 0.18 | 50.00 | 55.60 | 42.00 | 50.40 | ||||
| 川麻(Morus notabilis) | MnNiR | XM_010109635.2 | 52.55 | 0.17 | 49.10 | 56.00 | 39.70 | 49.50 | ||||
| 大桉(Eucalyptus grandis Hill) | EgNiR | XM_010030169.2 | 52.86 | 0.16 | 49.80 | 55.30 | 40.50 | 51.40 | ||||
| 辣椒(Capsicum annuum) | CaNiR | XM_016684117.1 | 50.34 | 0.18 | 44.10 | 54.60 | 39.20 | 35.80 | ||||
| 木薯(Manihot esculenta Crantz) | MeNiR | XM_021770342.1 | 48.69 | 0.16 | 45.00 | 55.00 | 38.90 | 38.20 | ||||
| 马铃薯(Solanum tuberosum) | StNiR | XM_006349563.2 | 54.88 | 0.19 | 44.80 | 55.90 | 38.80 | 37.10 | ||||
| 水蜜桃(Prunus persica) | PpNiR | XM_007201118.2 | 51.19 | 0.16 | 48.20 | 56.30 | 39.90 | 45.90 | ||||
| 拟南芥(Arabidopsis thaliana) | AtNiR | NM_127123.3 | 50.90 | 0.18 | 46.20 | 55.20 | 40.20 | 41.00 | ||||
| 单子叶植物 | 水稻(Oryza sativa) | OsNiR | XM_015786216.2 | 31.64 | 0.26 | 67.30 | 63.80 | 42.20 | 95.40 | |||
| 玉米(Zea mays) | ZmNiR | NM_001305856.1 | 31.27 | 0.30 | 67.30 | 63.20 | 44.00 | 94.30 | ||||
| 小麦(Triticum aestivum) | TaNiR | KY214455.1 | 30.29 | 0.26 | 68.90 | 64.30 | 43.30 | 98.80 | ||||
| 高粱(Sorghum) | SoNiR | XM_002454557.2 | 31.90 | 0.24 | 68.40 | 65.80 | 42.80 | 96.10 | ||||
| 平均值 | 48.70 | 0.19 | 49.95 | 56.79 | 40.22 | 50.69 | ||||||
同义密码子RSCU值能够反映密码子使用的偏性程度。当RSCU值=1,表明该密码子的使用没有偏好性;当RSCU值<l或RSCU值>1,表明该密码子的使用频率相对较低或者较高[11]。从图 1可见,苋菜AtrNiR有26个密码子的RSCU值>l,26个密码子中以A/T碱基结尾有19个,以G/C碱基结尾有7个。从图 1还可见,编码Ala的GCU的RSCU值最大(2.38),说明该密码子使用偏好性最强。编码Pro的CCU其次(2.00),表明该密码子也具有较强的使用偏好性;RSCU值=1的密码子有UUU、CAU、UGG、AUG、UUC、CAC,这些密码子不具有使用偏好性;GCG、CGG密码子RSCU值为0,说明苋菜AtrNiR基因不使用这些密码子。各氨基酸不同的密码子偏好性最终造成苋菜AtrNiR基因的ENc值偏离61。
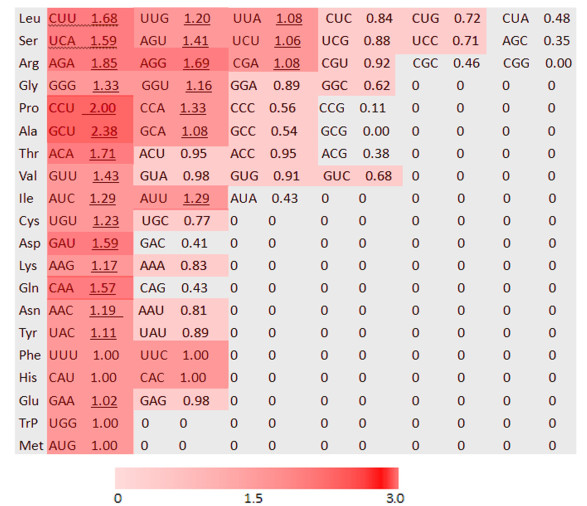
|
下划线表示苋菜AtrNiR基因对该密码子使用频率较高。 图 1 苋菜AtrNiR基因同义密码子相对使用度分析 Figure 1 Analysis of relative use frequency of synonymous codons of the amaranth AtrNiR gene |
ENc值越接近20说明密码子偏好性越强,越靠近61则偏好性越弱。从表 1可见,甜菜、藜麦、菠菜、黄胡萝卜和马铃薯与苋菜中的AtrNiR基因密码子偏好性参数相似。双子叶植物的NiR基因ENc值在47.62~56.19之间,其中最高的是山茶(56.19)、最低的是蓖麻(47.62),靠近61说明双子叶植物密码子偏好性较弱;而单子叶植物的NiR基因ENc值在30.29~31.90之间,靠近20说明单子叶植物的NiR基因密码子偏好性更强。
对GC含量分析发现,除了山茶NiR基因的GC3值为61.10%,其他双子叶植物NiR基因的GC、GC1、GC2、GC3值均低于58%。而单子叶植物中的GC2值全部低于50%,但GC、GC1、GC3值均高于62%,其中小麦NiR基因的GC3值最高,达98.8%。说明双子叶植物NiR相关基因对A或T的偏好性更强,而在单子叶植物中NiR相关基因对G或C具有更强的偏好性。综上可见,苋菜AtrNiR基因的相关参数属性符合双子叶植物密码子所具有的特性。
2.2.2 基因CDS序列及RSCU值的相关聚类分析通过邻接法对25个物种NiR基因的CDS编码序列进行进化树构建(图 2)。结果表明,绝大多数双子叶植物聚为一类,其中苋菜AtrNiR基因与藜麦、甜菜、菠菜的NiR基因聚为一支,它们的ENc值和GC3含量也较为接近(52.89≤ENc值≤54.04, 39.10%≤GC3含量≤42.00%),苋菜、藜麦、甜菜和菠菜都属于石竹目,亲缘关系相近。此外,茄科植物马铃薯、烟草、番茄、辣椒聚为一支,它们的ENc值和GC3含量也较为接近(50.34≤ENc值≤54.88, 34.90%≤GC3含量≤37.10%)。而玉米、水稻、小麦和高粱等单子叶植物的ENc值和GC3含量同样接近(30.29≤ENc值≤31.90,94.30%≤GC3含量≤98.80%)。综上表明,物种亲缘关系较近,其密码子使用偏好性也较接近。

|
分支上的数值表示1000次重复抽样符合聚类的百分数。 图 2 25个物种NiR基因系统进化树 Figure 2 An evolutionary tree of the NiR genes from 25 species |
通过SPSS 22对25个NiR相关基因的RSCU值进行聚类分析。与CDS进化树构建结果相比,两者聚类结果相似但又不相同(图 3)。在RSCU值聚类分析中,绝大多数双子叶植物依旧聚为一类,茄科植物马铃薯、烟草、番茄、辣椒和单子叶植物玉米、水稻、小麦、高粱依旧分别聚为一支。但是在RSCU值聚类分析中,苋菜AtrNiR基因与藜麦NiR基因聚为一小支,而甜菜、菠菜、黄胡萝卜和蓖麻NiR基因聚成一支。RSCU值聚类分析结果说明,即使进化关系较近的物种密码子使用偏好性有一定的相似性,但是经过漫长的进化,不同物种还是有属于自己的独特的密码子偏好性。相比之下,采用基因CDS聚类结果更接近该基因传统物种分类规律。
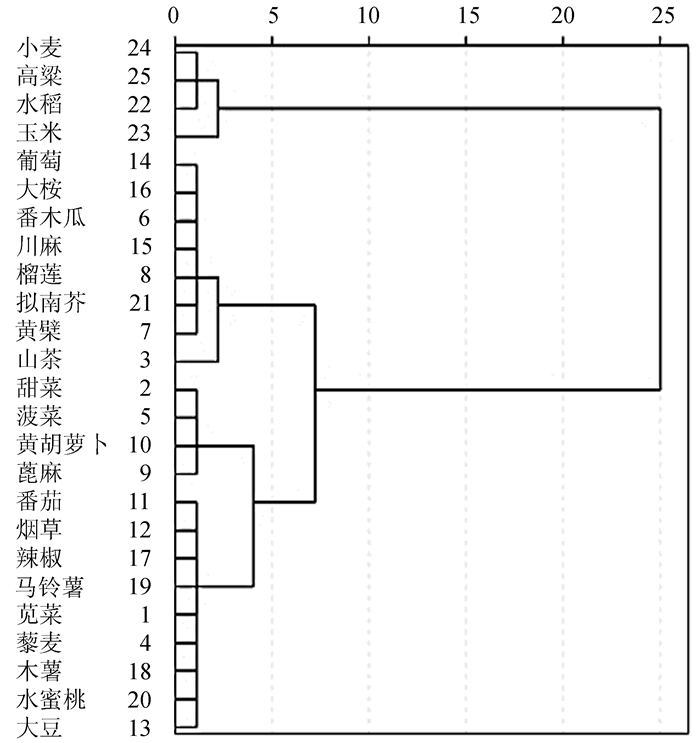
|
图 3 基于RSCU值进行NiR聚类分析 Figure 3 RSCU-based cluster analysis of the NiR genes among 25 species |
通过分析物种基因密码子的GC3与GC12(GC1与GC2的平均数)之间的关系[12-13],可以了解影响密码子偏好性的因素。利用SPSS 22中的Pearson关联分析对参试物种NiR基因密码子成分进行相关性分析(表 2)表明,不同物种NiR基因密码子的GC含量与GC1、GC2、GC3含量呈极显著正相关,说明密码子3个碱基组成相似。利用Origin 2017绘制不同物种NiR基因的中性图(图 4)。从图 4可以看出,苋菜AtrNiR基因所在的位置更靠近双子叶植物分布的位置。NiR基因GC12的范围为46.15%~54.30%,GC3的范围为34.60%~98.80%,GC3比GC12的范围更大,因此大部分物种NiR基因分布在回归线左右,和对角线成交叉分布(图 4)。说明NiR基因密码子差异可能受自然选择的压力较小,而受突变的影响比较大。
| 参数 | GC1 | GC2 | GC3 | GC | ENc |
| GC1 | 1 | ||||
| GC2 | 0.721** | 1 | |||
| GC3 | 0.863** | 0.888** | 1 | ||
| GC | 0.892** | 0.889** | 0.997** | 1 | |
| ENc | -0.902** | -0.719** | -0.886** | -0.899** | 1 |
| 1)**表示差异达0.01显著水平。 | |||||

|
图 4 NiR相关基因密码子使用偏好性中性绘图 Figure 4 A neutrality plot on the codon usage preference of NiR-related genes |
ENc与GC3的期望曲线能够说明两者之间在突变压力下的对应关系[14]。基因所对应的点越接近期望曲线,说明基因密码子偏好性受碱基突变的影响越大。从图 5可以看出,不同物种NiR基因位点都分布于期望曲线的下方,与图 4中NiR相关基因密码子偏好性更多受碱基突变的影响相一致。从图 5可看出,部分物种(如大桉、番木瓜、川麻等)的基因位点距离期望曲线相对较远,说明密码子的偏好性不仅受碱基突变的影响,还受到自然选择、基因长度和基因表达量等因素的影响[15]。

|
图 5 ENc-plot分布 Figure 5 Distribution of ENc-plot |
利用Origin 2017对不同物种NiR基因进行奇偶偏好偏差分析(图 6),结果显示,所有NiR基因的坐标值都偏离了0.5。从横坐标看,NiR基因密码子第3位碱基C的使用频率高于G;从纵坐标看,NiR基因密码子第3位碱基A的使用频率高于T。说明在进化过程中,不同物种NiR家族密码子偏好性不仅会受到突变压力影响,还会受到自然选择等其他因素的影响。
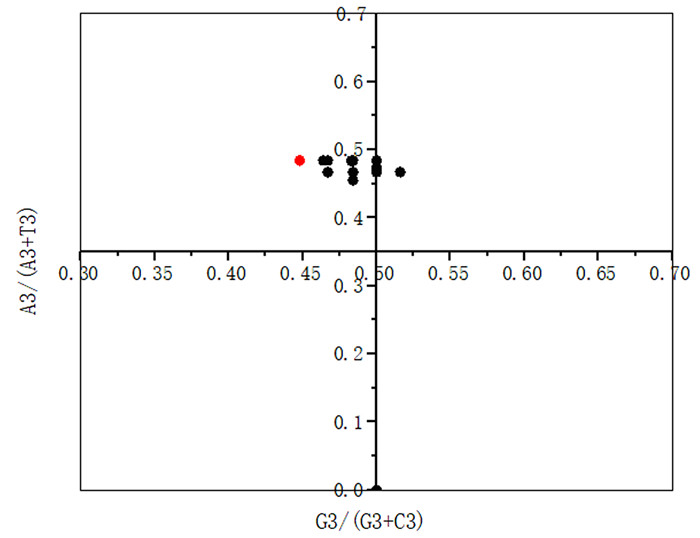
|
A3、T3、G3、C3,分别表示密码子第3位上各个碱基的含量。 图 6 参试物种NiR基因奇偶偏好偏差分析 Figure 6 Parity analysis of the NiR genes of tested plant species |
基因的原核表达和真核表达系统的受体常常是大肠杆菌和酵母菌,拟南芥、烟草、番茄等模式植物常常被用来探究相关基因的功能特征[16]。由于苋菜利用自身进行遗传转化比较困难,所以需要借助别的受体进行异源表达。本文通过计算苋菜AtrNiR基因与拟南芥、烟草、番茄、甜菜、大肠杆菌、酵母菌共6个模式植物基因组密码子使用频率的比值,筛选密码子使用差异较大的个数,确定苋菜异源表达受体。当比值≤0.5或≥2.0,则说明密码子使用差异较大[17-18]。从表 3可见,AtrNiR基因与大肠杆菌相比较,比值≤0.5或≥2.0的个数为21;与酵母菌相比较,比值≤0.5或≥2.0的个数为15。因此,酵母菌表达系统适合苋菜AtrNiR基因在真核生物中异源表达。AtrNiR基因与番茄、烟草、甜菜和拟南芥基因组密码子使用频率差异较大的个数分别为6、6、7、8,说明这4个物种都可以作为苋菜AtrNiR基因遗传转化受体,其中烟草和番茄表达系统优于拟南芥和甜菜。同为石竹目植物的甜菜也可以作为AtrNiR基因瞬时表达受体。
| 密码子 | 氨基酸 | AtrNiR | AtrNiR | AtrNiR | AtrNiR | AtrNiR | AtrNiR | AtrNiR | |||||
| 甜菜 | 拟南芥 | 烟草 | 番茄 | 大肠杆菌 | 酵母菌 | ||||||||
| GCA | Ala(A) | 16.78 | 0.76 | 0.96 | 0.73 | 0.76 | 0.67 | 1.04 | |||||
| GCC | 8.39 | 0.73 | 0.81 | 0.67 | 0.83 | 0.66 | 0.67 | ||||||
| GCU | 36.91 | 1.23 | 1.30 | 1.18 | 1.20 | 1.92 | 1.75 | ||||||
| UGC | Cys(C) | 8.39 | 1.22 | 1.17 | 1.17 | 1.25 | 3.00 | 1.78 | |||||
| UGU | 13.42 | 1.30 | 1.28 | 1.37 | 1.24 | 2.35 | 1.68 | ||||||
| GAC | Asp(D) | 10.07 | 0.58 | 0.59 | 0.60 | 0.67 | 0.54 | 0.50 | |||||
| GAU | 38.59 | 1.02 | 1.05 | 1.05 | 0.98 | 1.55 | 1.02 | ||||||
| CAA | Gln(Q) | 30.20 | 1.46 | 1.56 | 1.46 | 1.44 | 1.94 | 1.10 | |||||
| CAG | 8.39 | 0.65 | 0.55 | 0.56 | 0.60 | 0.30 | 0.69 | ||||||
| UUC | Phe(F) | 18.46 | 1.02 | 0.89 | 1.03 | 1.05 | 2.01 | 1.01 | |||||
| UUU | 18.46 | 0.76 | 0.85 | 0.74 | 0.71 | 0.79 | 0.71 | ||||||
| GGA | Gly(G) | 16.78 | 0.71 | 0.69 | 0.72 | 0.66 | 0.62 | 1.54 | |||||
| GGC | 11.74 | 1.16 | 1.28 | 1.05 | 1.21 | 0.72 | 1.21 | ||||||
| GGG | 25.17 | 2.23 | 2.47 | 2.40 | 2.33 | 0.77 | 4.19 | ||||||
| GGU | 21.81 | 0.93 | 0.98 | 0.98 | 0.91 | 0.88 | 0.91 | ||||||
| CAC | His(H) | 10.07 | 1.16 | 1.16 | 1.16 | 1.29 | 2.01 | 1.31 | |||||
| CAU | 10.07 | 0.68 | 0.73 | 0.75 | 0.65 | 1.09 | 0.73 | ||||||
| AUA | Ile(I) | 8.39 | 0.62 | 0.67 | 0.60 | 0.60 | 0.45 | 0.47 | |||||
| AUC | 25.17 | 1.55 | 1.36 | 1.81 | 1.80 | 2.21 | 1.48 | ||||||
| AUU | 25.17 | 0.95 | 1.17 | 0.91 | 0.89 | 1.14 | 0.83 | ||||||
| AAA | Lys(K) | 25.17 | 0.79 | 0.82 | 0.77 | 0.81 | 1.36 | 0.60 | |||||
| AAG | 35.23 | 1.00 | 1.08 | 1.05 | 1.14 | 2.36 | 1.15 | ||||||
| CUA | Leu(L) | 6.71 | 0.62 | 0.68 | 0.71 | 0.67 | 0.59 | 0.50 | |||||
| CUC | 11.74 | 1.00 | 0.73 | 0.95 | 1.05 | 8.39 | 2.17 | ||||||
| CUG | 10.07 | 1.16 | 1.03 | 0.99 | 0.96 | 0.23 | 0.97 | ||||||
| CUU | 23.49 | 0.94 | 0.97 | 0.98 | 0.94 | 2.55 | 1.94 | ||||||
| UUA | 15.10 | 1.13 | 1.19 | 1.13 | 1.05 | 1.25 | 0.57 | ||||||
| UUG | 16.78 | 0.78 | 0.80 | 0.75 | 0.69 | 1.97 | 0.62 | ||||||
| AUG | Met(M) | 25.17 | 1.03 | 1.03 | 1.01 | 1.02 | 1.04 | 1.20 | |||||
| AAC | Asn(N) | 26.85 | 1.50 | 1.28 | 1.50 | 1.55 | 1.26 | 1.08 | |||||
| AAU | 18.46 | 0.71 | 0.83 | 0.66 | 0.61 | 0.74 | 0.51 | ||||||
| CCU | Pro(P) | 30.20 | 1.50 | 1.86 | 1.53 | 1.57 | 3.55 | 1.66 | |||||
| CCC | 8.39 | 1.23 | 1.58 | 1.27 | 1.47 | 2.33 | 1.23 | ||||||
| CCG | 1.68 | 0.36 | 0.20 | 0.34 | 0.36 | 0.10 | 0.32 | ||||||
| CCA | 20.13 | 1.18 | 1.08 | 1.08 | 1.05 | 1.57 | 1.48 | ||||||
| GAA | Glu(E) | 43.62 | 1.24 | 1.27 | 1.21 | 1.25 | 1.57 | 0.90 | |||||
| GAG | 41.95 | 1.35 | 1.30 | 1.43 | 1.58 | 1.68 | 2.18 | ||||||
| AGA | Arg(R) | 20.13 | 1.18 | 1.06 | 1.26 | 1.23 | 2.01 | 0.95 | |||||
| AGG | 18.46 | 1.37 | 1.68 | 1.51 | 1.55 | 1.53 | 2.01 | ||||||
| CGU | 10.07 | 1.23 | 1.60 | 1.90 | 1.86 | 0.54 | 3.36 | ||||||
| CGC | 5.03 | 1.23 | 1.32 | 1.29 | 1.62 | 0.50 | 1.94 | ||||||
| CGA | 11.74 | 2.06 | 1.30 | 1.57 | 1.70 | 1.51 | 1.81 | ||||||
| AGC | Ser(S) | 3.36 | 0.35 | 0.30 | 0.34 | 0.36 | 0.26 | 0.35 | |||||
| AGU | 13.42 | 0.84 | 0.96 | 1.01 | 0.88 | 0.54 | 0.95 | ||||||
| UCA | 15.10 | 0.94 | 0.83 | 0.86 | 0.73 | 0.79 | 0.80 | ||||||
| UCC | 6.71 | 0.61 | 0.60 | 0.66 | 0.68 | 0.67 | 0.47 | ||||||
| UCG | 8.39 | 1.53 | 0.90 | 1.58 | 1.50 | 0.91 | 0.99 | ||||||
| UCU | 10.07 | 0.47 | 0.40 | 0.50 | 0.47 | 0.83 | 0.43 | ||||||
| ACA | Thr(T) | 15.10 | 0.95 | 0.96 | 0.87 | 0.84 | 0.79 | 0.85 | |||||
| ACC | 8.39 | 0.76 | 0.81 | 0.86 | 0.98 | 0.78 | 0.67 | ||||||
| ACG | 3.36 | 0.96 | 0.44 | 0.75 | 0.73 | 0.17 | 0.42 | ||||||
| ACU | 8.39 | 0.41 | 0.48 | 0.41 | 0.42 | 0.45 | 0.41 | ||||||
| GUA | Val(V) | 21.81 | 1.90 | 2.20 | 1.91 | 1.95 | 1.18 | 1.85 | |||||
| GUC | 15.10 | 1.41 | 1.18 | 1.36 | 1.50 | 1.64 | 1.30 | ||||||
| GUG | 20.13 | 1.30 | 1.16 | 1.21 | 1.26 | 0.81 | 1.90 | ||||||
| GUU | 31.88 | 1.14 | 1.17 | 1.19 | 1.14 | 2.04 | 1.45 | ||||||
| UGG | Trp(W) | 13.42 | 1.02 | 1.07 | 1.10 | 0.99 | 0.67 | 1.30 | |||||
| UAC | Tyr(Y) | 8.39 | 0.68 | 0.61 | 0.62 | 0.68 | 0.84 | 0.57 | |||||
| UAU | 6.71 | 0.38 | 0.46 | 0.38 | 0.36 | 0.23 | 0.36 |
进化过程中,不同物种或基因对基因组环境及自然选择压力适应性选择形成了密码子偏好性[19]。通过对苋菜AtrNiR基因的密码子偏好性分析后发现,编码Ala的GCT、编码Pro的CCT、编码Leu的CTT与TTG、编码Ser的AGT与TCA等密码子的偏好性较强。AtrNiR基因偏好使用第3位碱基为A/T的密码子,说明AtrNiR基因密码子符合双子叶植物的密码子使用偏好性[20]。双子叶植物NiR基因各密码子偏好性相关参数数值大小相近, 而单子叶植物NiR基因的密码子偏好性参数GC3数值远高于双子叶植物GC3数值,这符合单子叶植物偏爱以G/C结尾的密码子使用特性[9]。
3.2 苋菜AtrNiR的同源比较影响基因密码子偏好性的因素有很多,包括内含子结构、DNA甲基化等[8]。从NiR基因CDS进化树和RSCU值两个聚类分析可知,二者既有一定的相似性,又有不同。密码子偏好性的聚类分析可以作为基因CDS聚类分析的补充[12]。将二者聚类结果相结合,能更准确反映物种真实系统分类和亲缘关系[21]。因为密码子偏好性受很多因素影响,使其聚类结果往往不能如构建系统进化树般更准确反映不同物种的亲缘关系[22-23]。从NiR基因的CDS系统进化树分析表明,苋菜AtrNiR基因与藜麦、甜菜、菠菜这些石竹目植物NiR基因的同源关系较近,并且所对应的基因位点也集中分布在密码子中性分析及ENC-plot分析的图中,说明苋菜AtrNiR基因在功能上可能与这些物种的NiR相关氮代谢基因有一定的相似性。
3.3 苋菜AtrNiR异源表达和遗传转化受体系统的选择转基因过程经常会涉及到基因的异源表达,而异源表达中外源基因的表达水平会受到受体物种选择的影响。受体系统密码子偏性使用是制约外源基因翻译效率的因素之一[19]。对苋菜AtrNiR密码子使用频率的分析表明,模式植物拟南芥、烟草、番茄等均可作为苋菜AtrNiR异源表达的遗传转化受体。酵母菌更适合作为苋菜AtrNiR基因瞬时表达的受体。在6种模式植物中,甜菜与苋菜亲缘关系最近,已有研究通过注射法对甜菜进行瞬时表达[24]。本研究中通过计算密码子使用差异较大的个数发现,甜菜也可以作为AtrNiR异源转化的受体。因此,在研究苋菜AtrNiR基因时,甜菜可以成为其瞬时表达的受体。
4 结论通过分析苋菜AtrNiR基因密码子偏好性,发现AtrNiR密码子偏好以A/T结尾,与同源石竹目植物密码子使用偏好性相近,而且苋菜AtrNiR密码子偏好性主要受突变压力影响。苋菜AtrNiR基因兼容性较好,拟南芥、烟草、番茄等模式植物均可作为其异源表达的遗传转化受体,酵母菌和甜菜可以作为其瞬时表达载体。密码子偏好性分析结果对进一步开展AtrNiR转基因功能验证研究中受体物种的选择、基因异源表达水平提升以及植物遗传育种都有重要的指导作用,但基因能否高效表达还受转化效率、培养基及抗生素的选择等多种因素的影响[8]。因此,苋菜AtrNiR基因高效遗传转化的实现值得深入研究。
| [1] | 汪建飞. 营养液不同铵硝比对菠菜产量和品质影响的机理研究[D]. 南京: 南京农业大学, 2007. |
| [2] | SEITH B, SCHUSTER C, MOHR H. Coaction of light, nitrate and a plastidic factor in controlling nitrite-reductase gene expression in spinach[J]. Planta, 1991, 184(1): 74–80. |
| [3] | 孙菲菲, 蒋芳玲, 侯喜林, 等. 白菜亚硝酸还原酶基因BcNiR的克隆及表达分析[J]. 园艺学报, 2009, 36(10): 1511–1518. DOI: 10.3321/j.issn:0513-353X.2009.10.017 |
| [4] | 佘茂云, 陈朵朵, 冯晨, 等. 小麦亚硝酸还原酶基因及调控序列克隆、定位和表达分析[J]. 作物学报, 2011, 37(1): 28–39. DOI: 10.3969/j.issn.1000-2561.2011.01.006 |
| [5] | KUMAR N, BERA B C, GREENBAUM B D, et al. Revelation of influencing factors in overall codon usage bias of equine influenza viruses[J]. PLoS One, 2016, 11(4): e0154376. DOI: 10.1371/journal.pone.0154376 |
| [6] | JIANG Y, DENG F, WANG H L, et al. An extensive analysis on the global codon usage pattern of baculoviruses[J]. Archives of Virology, 2008, 153(12): 2273–2282. DOI: 10.1007/s00705-008-0260-1 |
| [7] | 梁菲菲. 密码子偏性的影响因素及研究意义[J]. 畜牧与饲料科学, 2010, 31(1): 118–119. DOI: 10.3969/j.issn.1672-5190.2010.01.058 |
| [8] | 赵春丽, 彭丽云, 王晓, 等. 苋菜AtGAI基因密码子偏好性与进化分析[J]. 中国农业大学学报, 2019, 24(12): 10–22. DOI: 10.11841/j.issn.1007-4333.2019.12.02 |
| [9] | 王晓, 赵春丽, 彭丽云, 等. 苋菜蓝光受体基因AmCRY1密码子使用模式及分析[J]. 应用与环境生物学报, 2020, 26(1): 135–143. |
| [10] | 王云, 彭丽云, 苏立遥, 等. 龙眼Hsf基因家族密码子使用模式分析[J]. 分子植物育种, 2019, 17(17): 5595–5603. |
| [11] | 时慧, 王玉, 杨路成, 等. 茶树抗寒调控转录因子ICE1密码子偏性分析[J]. 园艺学报, 2012, 39(7): 1341–1352. |
| [12] | 程丽, 李宜奎, 李晓丹, 等. 植物CPR基因密码子偏好性及聚类分析[J]. 分子植物育种, 2017, 15(5): 1672–1682. |
| [13] | 赖瑞联, 冯新, 陈瑾, 等. 橄榄查尔酮异构酶基因CHI的密码子偏好模式[J]. 应用与环境生物学报, 2017, 23(5): 945–951. |
| [14] | ZHAO Y C, ZHENG H, XU A Y, et al. Analysis of codon usage bias of envelope glycoprotein genes in nuclear polyhedrosis virus (NPV) and its relation to evolution[J]. BMC Genomics, 2016, 17(1): 677. DOI: 10.1186/s12864-016-3021-7 |
| [15] | 王占军, 李豹, 姜行舟, 等. 两种茶树全基因组数据的密码子偏好性比较分析[J]. 中国细胞生物学学报, 2018, 40(12): 2028–2039. |
| [16] | 彭丽云, 王云, 孙雪丽, 等. 苋菜AmMYB2基因密码子偏好性与进化分析[J]. 应用与环境生物学报, 2019, 25(3): 679–686. |
| [17] | 雷梦林, 冯瑞云, 郝雅萍, 等. 小麦抗逆相关转录因子DREB密码子偏好性特征分析[J]. 麦类作物学报, 2019, 39(1): 1–9. |
| [18] | 赖瑞联, 冯新, 陈瑾, 等. 橄榄转录组密码子使用偏好性及其影响因素[J]. 核农学报, 2019, 33(1): 31–38. |
| [19] | PRABHA R, SINGH DP, SINHA S, et al. Genome-wide comparative analysis of codon usage bias and codon context patterns among cyanobacterial genomes[J]. Marine Genomics, 2017, 32: 31–39. DOI: 10.1016/j.margen.2016.10.001 |
| [20] | MURRAY E E, LOTZER J, EBERLE M. Codon usage in plant genes[J]. Nucleic Acids Research, 1989, 17(2): 477–498. DOI: 10.1093/nar/17.2.477 |
| [21] | 李蓉, 谢析颖, 王雪晶, 等. 兰科植物FNR基因的密码子偏好性分析[J]. 热带作物学报, 2018, 39(6): 1137–1145. DOI: 10.3969/j.issn.1000-2561.2018.06.015 |
| [22] | 赖瑞联, 林玉玲, 钟春水, 等. 龙眼生长素受体基因TIR1密码子偏好性分析[J]. 园艺学报, 2016, 43(4): 771–780. |
| [23] | 晁岳恩, 吴政卿, 杨会民, 等. 11种植物psbA基因的密码子偏好性及聚类分析[J]. 核农学报, 2011, 25(5): 927–932. |
| [24] | HATLESTAD G J, AKHAVAN N A, SUNNADENIYA R M, et al. The beet Y locus encodes an anthocyanin MYB-like protein that activates the betalain red pigment pathway[J]. Nature Genetics, 2015, 47(1): 92–106. DOI: 10.1038/ng.3163 |
 2021,Vol. 17
2021,Vol. 17


