文章信息
- 曹代男, 李伟业, 滑留帅, 葛研, 魏玉峰, 段好冉, 龚世平
- CAO Dainan, LI Weiye, HUA Liushuai, GE Yan, WEI Yufeng, DUAN Haoran, GONG Shiping
- 用于快速识别红耳龟在中国野外分布的特异性引物筛选
- Specific primers screening for rapid identification of alien red-eared slider (Trachemys scripta elegans) in the wild of China
- 亚热带农业研究, 2018, 14(2): 86-90
- Subtropical Agriculture Research, 2018, 14(2): 86-90.
- DOI: 10.13321/j.cnki.subtrop.agric.res.2018.02.003
-
文章历史
- 收稿日期: 2018-04-19
红耳龟(Trachemys scripta elegans)是世界上100个最具威胁性的入侵物种之一[1]。现已入侵欧洲、非洲、亚洲及美洲等地区的许多国家[2], 严重威胁入侵地区的生态安全[3], 同时能够传播高致病性沙门氏菌, 对人类健康构成威胁[4]。红耳龟于20世纪80年代被引入中国, 现已在中国野外普遍存在, 潜在生态入侵危害严重[5-6]。快速准确地掌握红耳龟野外分布状况是开展入侵防控工作的重要基础, 而传统龟类分布调查以野外调查及访问调查为主[5-6], 耗时费力、效率较低, 不利于大规模快速掌握红耳龟的野外分布状况。近年来, 环境DNA(environmental DNA, eDNA)技术得到不断发展和应用, 已经成为动物野外调查的重要技术[7-8]。eDNA技术的核心是通过检测环境样品中某物种特异DNA片段的有无以判断该物种是否存在, 并且可以根据eDNA浓度来推测物种生物量[9]。目前eDNA技术已经应用于亚洲鲤鱼(Hypophthalmichthys molitrix)[10-11]、美国牛蛙(Rana catesbeiana)[12]、克氏原螯虾(Procambarus clarkii)[13]、缅甸蟒蛇(Python bivittatus)[14]、泥螺(Potamopyrgus antipodarum)[15]等物种的生物入侵研究, 并在海洋鱼类和淡水龟类调查研究中也得到有效应用[16-17]。
红耳龟在中国主要栖息于池塘、湖泊、水库和河流等淡水水体[5-6], 而这些区域同时也栖息着其他淡水龟类, 包括本土龟类和部分外来龟类[18-19]。因此, 开发红耳龟特异性引物用于eDNA识别技术十分必要。线粒体基因组COI基因具有特异性, 常作为物种识别的目标基因[20], 它不仅是物种鉴定的优良分子标记[21], 也是eDNA技术中常用的分子标记[22]。因此, 本研究拟基于红耳龟线粒体COI基因序列设计并筛选出特异性引物, 用于红耳龟的快速野外识别, 为其野外分布监测及防控提供技术支撑。
1 材料与方法 1.1 供试样品本研究采集了红耳龟及其他10种在中国野外相对常见的龟鳖类组织样品, 包括乌龟(Mauremys reevesii)、花龟(Mauremys sinensis)、黄喉拟水龟(Mauremys mutica)、山瑞鳖(Palea steindachneri)、中华鳖(Pelodiscus sinensis)5种本土龟类, 以及蛇鳄龟(Chelydra serpentina)、大鳄龟(Macroclemys temminckii)、安布闭壳龟(Cuora amboinensis)、亚洲鳖(Amyda cartilaginea)及佛罗里达鳖(Apalone ferox)5种外来龟类。采集的组织样品用体积分数为85%乙醇固定, 保存于-20 ℃冰箱备用。
1.2 试验试剂动物组织基因组DNA提取试剂盒、核酸染料、DL2000 DNA Marker及琼脂糖均购自生工生物工程(上海)股份有限公司。2×Taq Plus Master Mix购自南京诺唯赞生物科技有限公司。
1.3 DNA提取及其浓度测定采用动物组织基因组DNA提取试剂盒提取龟类基因组DNA, 使用NanoDrop 3000(Thermo)荧光分光光度计测定所提取的龟类基因组DNA浓度和纯度。
1.4 引物设计以红耳龟线粒体COI基因序列(GenBank号:KM216748.1)为模板, 采用Primer 5软件设计COI基因引物。特异性引物的设计原则是要求能鉴别出红耳龟与其他10种龟类, 引物设计中还需检查引物错配、二聚体和发夹结构等。通过设计和筛选, 最终获得1对红耳龟特异性引物(上游引物COI F:5′-GTTGGGATAGACGTAGACACC-3′; 下游引物COI R:5′-CTCCGATAGTAAAGAGGAAGATG-3′)。所用引物由生工生物工程(上海)股份有限公司负责合成。
1.5 红耳龟COI引物特异性检测分别以上述红耳龟等11种龟鳖类的DNA为模板, 检验所设计红耳龟COI特异性引物的有效性。PCR反应体系为25 μL, 其中模板DNA 2.0 μL(约100 ng), 2×Taq Plus Master Mix 12.5 μL, 上下游引物(100 nmol·L-1)各1.0 μL, ddH2O 8.5 μL。反应条件为:94 ℃ 4 min; 94 ℃ 30 s, 57 ℃ 30 s, 72 ℃ 30 s, 35个循环; 72 ℃延伸10 min。PCR产物于4 ℃下保存。对PCR产物进行琼脂糖凝胶电泳, 检测特异性引物扩增的片段有无及大小, 并对得到的扩增产物进行测序和序列分析, 进一步确认引物的有效性。
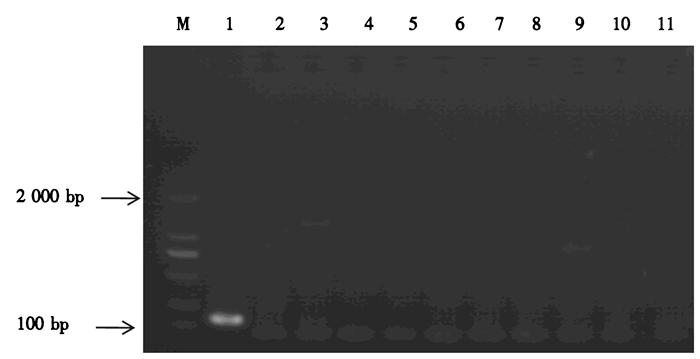
2 结果与分析 2.1 引物特异性验证以红耳龟及其他10种龟类DNA为模板, 使用本研究中设计的COI基因特异引物进行PCR扩增。电泳检测结果显示, 特异性引物能够扩增出红耳龟的目的条带, 而乌龟、花龟、黄喉拟水龟、山瑞鳖、中华鳖、蛇鳄龟、安布闭壳龟、大鳄龟、亚洲鳖及佛罗里达鳖等10种龟类均未能扩增出长度为172 bp的目的条带, 说明引物特异性较好(图 1)。
|
图 1 PCR产物电泳图
Figure 1 Electrophoretogram of PCR products
M.2 000 bp DNA Marker; 1.红耳龟; 2.花龟; 3.黄喉拟水龟; 4.蛇鳄龟; 5.安布闭壳龟; 6.山瑞鳖; 7.佛罗里达鳖; 8.亚洲鳖; 9.中华鳖; 10.大鳄龟; 11.乌龟。 |
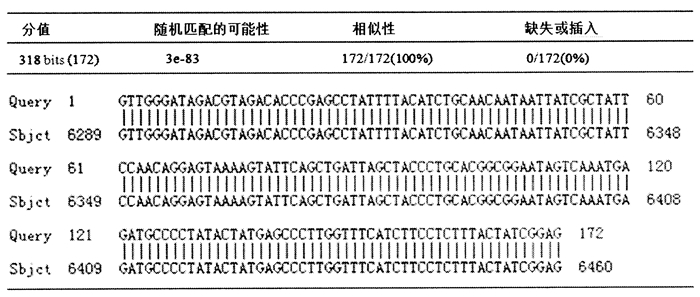
本试验所得的172 bp红耳龟COI基因PCR产物的测序结果与NCBI网站公布的红耳龟线粒体COI基因片段序列(GenBank号:KM216748.1)一致(图 2)。
|
图 2 红耳龟COI基因PCR产物与红耳龟线粒体基因组序列比对图 Figure 2 Sequence comparison between the PCR product of the mitochondrion COI gene and the complete mitochondrion genome of red-eared slider Query:线粒体COI基因PCR产物; Sbjct:红耳龟线粒体基因组序列(KM216748.1)。 |
生物入侵已成为当前威胁全球生态安全的主要因素之一, 对入侵生物生态危害的快速评估和有效防控是当前生物安全领域亟待解决的关键问题和技术难题[23]。在入侵早期发现有害物种有利于根除或遏制其扩散[10], eDNA技术可以在不采集动物标本或发现动物实体的条件下检测物种分布情况及其生物量, 是一种高效的动物野外调查监测技术[24-25], 有利于在入侵早期发现入侵物种。通过设计目标物种特异性引物, 在扩增未知模板时可以根据目标片段条带的有无把目标种类与其他种类区分开来, 此技术被称为种特异引物PCR(species-specific PCR, ssPCR)[26]。ssPCR技术是eDNA技术的基础, 其关键是筛选到目标物种的特异引物, 使用非特异性引物会影响eDNA技术对物种的检测效率。如Kelly et al[17]在研究绿海龟时发现, 使用通用引物未能检测出绿海龟eDNA, 采用特异性引物则可以检测出目标物种。
有关研究表明, 在同等条件下引物扩增的目标产物越短, 目标物种的检测率越高[27]。King et al[28]指出特异引物的目标产物片段小于300 bp能够更有效地扩增目标产物。本研究选用的红耳龟COI特异性引物扩增产物为172 bp, 在鉴定红耳龟与乌龟、花龟、黄喉拟水龟、山瑞鳖、中华鳖、蛇鳄龟、安布闭壳龟、大鳄龟、亚洲鳖及佛罗里达鳖的区别时, 具有较高的特异性。
在采用eDNA技术进行红耳龟调查时, 需要排除环境中其他龟鳖类DNA的干扰。Davy et al[16]在淡水龟eDNA标记研究中报道了可用于识别9种在加拿大分布的淡水龟类的COI基因特异性引物, 所识别的9种龟包括8种加拿大本土淡水龟类[如布氏拟龟(Emydoidea blandingii)、星点水龟(Clemmys guttata)]和外来入侵物种红耳龟。本研究所筛选的红耳龟COI特异性引物适用于中国大多数地区野外红耳龟分布识别。目前中国本土的淡水龟类普遍濒危, 在野外种群密度极低, 而且局部灭绝的现象普遍存在[29], 相反一些外来龟类却在野外比较常见[5-6, 19]。基于多年的野外调查和对相关文献的分析, 本研究选择了中国境内最有可能与红耳龟同域分布的10种龟鳖类。因此, 本文所提供的引物仅限于区分红耳龟与该10种龟类, 当野外实际的龟类物种不限于这些种类时, 需要通过基因序列对比途径来分析是否存在红耳龟, 必要时还可采用野外诱捕方法进行采集确认。
致谢: 万小娟、杨江波、蓬友红等对本研究提供了帮助, 在此谨致谢意!| [1] | LOWE S, BROWNE M, BOUDJELAS S, et al. 100 of the world's worst invasive alien species[DB/OL]. [2018-04-19]. http://www.iucngisd.org/gisd/100_worst.php. |
| [2] | RHODIN A G J, IVERSON JB, BOUR R, et al. Turtles of the world:annotated checklist and atlas of taxonomy, synonymy, distribution, and conservation status[J]. Chelonian Research Monographs, 2017, 7: 67–68. |
| [3] | 史海涛, 龚世平, 梁伟, 等. 控制外来物种红耳龟在中国野生环境蔓延的态势[J]. 生物学通报, 2009, 44(4): 1–3. |
| [4] | GONG S P, WANG F M, SHI H T, et al. Highly pathogenic Salmonella Pomona was first isolated from the exotic red-eared slider (Trachemys scripta elegans) in the wild in China:implications for public health[J]. Science of the Total Environment, 2014, 468-469: 28–30. DOI: 10.1016/j.scitotenv.2013.08.025 |
| [5] | 龚世平, 杨江波, 葛研, 等. 外来物种红耳龟在中国野外分布现状及扩散路径研究[J]. 野生动物学报, 2018, 39(2): 373–378. |
| [6] | 刘丹, 史海涛, 刘宇翔, 等. 红耳龟在我国分布现状的调查[J]. 生物学通报, 2011, 46(6): 18–21. |
| [7] | MAUVISSEAU Q, COIGNET A, DELAUNAY C, et al. Environmental DNA as an efficient tool for detecting invasive crayfishes in freshwater ponds[J]. Hydrobiologia, 2018, 805(1): 163–175. DOI: 10.1007/s10750-017-3288-y |
| [8] | THOMSEN P F, WILLERSLEV E. Environmental DNA-An emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation, 2015, 183: 4–18. DOI: 10.1016/j.biocon.2014.11.019 |
| [9] | BOHMANN K, EVANS A, GILBERT M T P, et al. Environmental DNA for wildlife biology and biodiversity monitoring[J]. Trends in Ecology & Evolution, 2014, 29(6): 358–367. |
| [10] | JERDE C L, MAHON A R, CHADDERTON W L, et al. "Sight-unseen" detection of rare aquatic species using environmental DNA[J]. Conservation Letters, 2011, 4(2): 150–157. DOI: 10.1111/conl.2011.4.issue-2 |
| [11] | MAHON A R, JERDE C L, GALASKA M, et al. Validation of eDNA surveillance sensitivity for detection of Asian carps in controlled and field experiments[J]. PLoS One, 2013, 8(3): e58316. DOI: 10.1371/journal.pone.0058316 |
| [12] | FICETOLA G F, MIAUD C, POMPANON F, et al. Species detection using environmental DNA from water samples[J]. Biology Letters, 2008, 4(4): 423–425. DOI: 10.1098/rsbl.2008.0118 |
| [13] | CAI W, MA Z X, YANG C Y, et al. Using eDNA to detect the distribution and density of invasive crayfish in the Honghe-Hani rice terrace World Heritage site[J]. PLoS One, 2017, 12(5): e0177724. DOI: 10.1371/journal.pone.0177724 |
| [14] | PIAGGIO A J, ENGEMAN R M, HOPKEN M W, et al. Detecting an elusive invasive species:a diagnostic PCR to detect Burmese python in Florida waters and an assessment of persistence of environmental DNA[J]. Molecular Ecology Resources, 2014, 14(2): 374–380. DOI: 10.1111/1755-0998.12180 |
| [15] | GOLDBERG C S, SEPULVEDA A, RAY A, et al. Environmental DNA as a new method for early detection of New Zealand mudsnails (Potamopyrgus antipodarum)[J]. Freshwater Science, 2013, 32(3): 792–800. DOI: 10.1899/13-046.1 |
| [16] | DAVY C M, KIDD A G, WILSON C C. Development and validation of environmental DNA (eDNA) markers for detection of freshwater turtles[J]. PLoS One, 2015, 10(7): e0130965. DOI: 10.1371/journal.pone.0130965 |
| [17] | KELLY R P, PORT J A, YAMAHARA K M, et al. Using environmental DNA to census marine fishes in a large mesocosm[J]. PLoS One, 2014, 9(1): e86175. DOI: 10.1371/journal.pone.0086175 |
| [18] | WANG J, SHI H T, HU S J, et al. Interspecific differences in diet between introduced redeared sliders and native turtles in China[J]. Asian Herpetological Research, 2013, 4(3): 190–196. DOI: 10.3724/SP.J.1245.2013.00190 |
| [19] | 徐婧, 周婷, 叶存奇, 等. 龟类外来种的生物入侵隐患及其防治措施[J]. 四川动物, 2006, 25(2): 420–422. |
| [20] | AGUSTI N, SHAYLER S P, HARWOOD J D, et al. Collembola as alternative prey sustaining spiders in arable ecosystems:prey detection within predators using molecular markers[J]. Molecular Ecology, 2003, 12(12): 3467–3475. DOI: 10.1046/j.1365-294X.2003.02014.x |
| [21] | SULTANA S, ALI M E, HOSSAIN M A M, et al. Universal mini COI barcode for the identification of fish species in processed products[J]. Food Research International, 2018, 105: 19–28. DOI: 10.1016/j.foodres.2017.10.065 |
| [22] | BRONNENHUBER J E, WILSON C C. Combining species-specific COI primers with environmental DNA analysis for targeted detection of rare freshwater species[J]. Conservation Genetics Resources, 2013, 5(4): 971–975. DOI: 10.1007/s12686-013-9946-0 |
| [23] | CHAFFIN B C, GARMESTANI A S, ANGELER D G, et al. Biological invasions, ecological resilience and adaptive governance[J]. Journal of Environmental Management, 2016, 183(2): 399–407. |
| [24] | THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565–2573. DOI: 10.1111/j.1365-294X.2011.05418.x |
| [25] | WELTZ K, LYLE J M, OVENDEN J, et al. Application of environmental DNA to detect an endangered marine skate species in the wild[J]. PLoS One, 2017, 12(6): e0178124. DOI: 10.1371/journal.pone.0178124 |
| [26] | SHABANI H, MEHDIZADEH M, MOUSAVI S M, et al. Halal authenticity of gelatin using species-specific PCR[J]. Food Chemistry, 2015, 184: 203–206. DOI: 10.1016/j.foodchem.2015.02.140 |
| [27] | VON BERG K, TRAUGOTT M, SYMONDSON W O C, et al. The effects of temperature on detection of prey DNA in two species of carabid beetle[J]. Bulletin of Entomological Research, 2008, 98: 263–269. |
| [28] | KING R A, READ D S, TRAUGOTT M, et al. Molecular analysis of predation:a review of best practice for DNA-based approaches[J]. Molecular Ecology, 2008, 17(4): 947–963. DOI: 10.1111/j.1365-294X.2007.03613.x |
| [29] | GONG S P, SHI H T, JIANG A W, et al. Disappearance of endangered turtles within China's nature reserves[J]. Current Biology, 2017, 27(5): 170–171. DOI: 10.1016/j.cub.2017.01.039 |
 2018,Vol. 14
2018,Vol. 14


