|
收稿日期: 2016-01-05; 修改日期: 2016-06-18; 优先数字出版日期: 2016-11-25
基金项目: 国家高技术研究发展计划(863计划)(编号:2013AA12A302);江苏省自然科学基金面上研究项目(编号:BK20151515);教育部博士点基金优先发展领域(编号:011010325);江苏高校优势学科建设工程资助项目(PAPD)
第一作者简介: 申鑫(1991—),男,硕士研究生,研究方向为遥感技术在森林资源监测中的应用。E-mail:
shenxin1903@126.com
通讯作者简介: 佘光辉(1953—),男,教授,研究方向为3S技术在森林资源监测中的应用。E-mail:
ghshe@njfu.com.cn
中图分类号: TP79
文献标识码: A
|
摘要
精确估算森林生物量对全球碳平衡以及气候变化的研究有重要意义。以亚热带天然次生林为研究对象,借助地面实测样地数据,通过对机载LiCHy(LiDAR,CCD and Hyperspectral)传感器同时获取的高光谱和高空间分辨率数据进行信息提取和数据融合,建模反演森林生物量。首先通过面向对象分割方法进行单木冠幅提取,然后融合从高光谱数据提取的光谱特征变量和从高空间分辨率数据提取的单木冠幅统计变量,构建多元回归模型估算地上、地下生物量,最后利用地面实测生物量经交叉验证评价模型精度。结果表明,综合模型的精度( R 2为0.54—0.62)高于高光谱模型( R 2为0.48—0.57);在高光谱模型中地上生物量模型精度( R 2为0.57)高于地下生物量模型( R 2为0.48);在综合模型中地上生物量模型精度( R 2为0.62)同样高于地下生物量模型( R 2为0.54)。交叉验证结果表明,与仅使用高光谱数据(单一数据源)相比,通过集成高光谱和高空间分辨率数据的生物量反演效果有所提升,可以更加有效地估算亚热带森林生物量。
关键词
亚热带森林 , 生物量估算 , 遥感反演 , 高光谱数据 , 高空间分辨率数据
Abstract
An accurate estimation of forest biomass is important for global carbon balance and climate change studies. The integration of hyperspectral and high-resolution data can provide abundant spectral and spatial detail information, and accurate biomass estimates. However, acquiring hyperspectral and high-resolution data at the same time is difficult, and related applications of integrated data are limited in the range of subtropical forests. This study used hyperspectral and high-resolution data acquired simultaneously in a subtropical natural secondary forest with field-measured data to estimate forest biomass. It provided a new approach in estimating the biomass of subtropical forests accurately. In the study, LiDAR-CCD-hyperspectral-integrated sensor was used to obtain hyperspectral and high-resolution data at the same time in a subtropical natural secondary forest. A total of 30 square plots (30×30) were established across the study site. First, tree crown features were extracted from high-resolution images by multi-scale segmentation based on edge detection. Second, five sets of spectral metrics were extracted from hyperspectral images. Third, the metrics extracted from hyperspectral and high-resolution images were integrated to build forest biomass models by using stepwise regression. Finally, cross-validations were used to assess the estimation accuracy of the models. The accuracy of integrated models ( R 2=0.54-0.62) is higher than that of hyperspectral models( R 2=0.48-0.57); the accuracy of aboveground biomass in hyperspectral model ( R 2=0.57) is higher than that of belowground biomass ( R 2=0.48);and the accuracy of aboveground biomass in the integrated model is ( R 2=0.62) higher than that of belowground biomass ( R 2=0.54). Cross-validation results indicate that the integrated model is better than the hyperspectral one, and that hyperspectral and high-resolution data obtained at the same time through integrated sensor can be used in estimating biomass in subtropical forests effectively. This study provided the effective approaches in estimating the biomass of subtropical forests by using remotely sensed data, and it proved that the integration of hyperspectral and high-resolution data can also help in estimating forest biomass accurately. This approach may provide an important basis for research on ecosystem and carbon cycle in subtropical forests.
Key words
subtropical forest , biomass estimation , remote sensing reversion , high resolution data , hyperspectral data
1 引 言
森林作为陆地生态系统的主体,在维护区域生态环境以及全球碳平衡中发挥着巨大作用( Dixon 等,1994)。森林生物量是量度森林结构和功能的重要指标,为生态系统的碳汇和碳素循环等研究提供关键数据( Luyssaert 等,2008)。精确估算森林生物量对全球碳汇、碳平衡的研究以及全球气候变化的理解有重要意义( Cramer 等,2001)。传统的生物量量测主要依靠实测法或以实测法为基础的统计分析方法,工作量大、过程复杂、周期长,需耗费大量的人力物力且对生态系统的破坏性较大( 方精云 等,1996)。遥感以其快速、准确、大尺度宏观观测的独特优势,可以弥补常规生物量量测方法的不足。
高光谱遥感技术是利用几十甚至数百个较窄的连续光谱波段,对目标物体获取遥感数据的技术以及相关的数据处理方法( 郑兰芬和王晋年,1992)。高光谱遥感具有光谱分辨率高的特点,能够记录植物体内各种生化组分对不同波长电磁波的选择性吸收特征。因此,可利用高光谱遥感数据进行植被的各种生化参数的反演,如叶绿素( Elvidge和Chen,1995)、冠层水分( Gao,1996)、纤维素和木质素( Kokaly和Clark,1999)等。同时,高光谱遥感数据也可进行叶面积指数LAI( Haboudane 等,2004)、光合有效辐射( Inoue 等,2008)和森林郁闭度( Gong 等,1994)等生物物理参数的提取。然而,高光谱遥感数据的空间分辨率通常较低,易形成混合像元,且在植被覆盖度较高的区域存在森林生物量信息饱和的现象( Brantley 等,2011)。因此,对于森林生物量的估算会产生不利影响( Lu,2006; Foody 等,2001)。高空间分辨率遥感数据能够记录丰富的空间信息,地物内部的细节结构得以表征,其这一特性有利于森林结构参数的提取和森林生物量反演精度的提升( Basti 等,2014)。以往的研究表明,高空间分辨率遥感数据在森林生物物理特征提取方面具有较大的应用潜力( Lévesque和King,2003)。
高光谱遥感数据可以获得多种不同时相和空间尺度的生物物理和化学特征( Asner 等,2011; Clark和Roberts,2012),但通常空间分辨率低,难以获得森林内部细节信息,且其光谱特征易受森林覆盖度影响。而高空间分辨率遥感数据可以获得丰富的空间结构信息,但是其往往缺乏足够的光谱信息。因此,将高光谱数据和高空间分辨率数据集成,并应用于森林参数估算具有很大的应用价值。 Thenkabail等人(2004)基于Hyperion高光谱和IKONOS高空间分辨率数据对非洲热带雨林区域进行树种分类及森林生物量估算,其结果表明,结合高光谱数据和高空间分辨率数据两者的优势可以取得更高的精度。 Takayama等(2014)利用机载HyMAP传感器获取的高光谱和高空间分辨率数据进行小范围区域的森林类型划分及森林生物量分布制图,其构建的生物量估算模型均方根误差(RMSE)为54.2 t·hm -2。但是,以上研究方法选用了较少的特征变量,并未充分提取影像的光谱信息和空间信息。 Goodenough和Bannon(2014)结合星载Hyperion和AVIRIS高光谱数据以及机载AISA数据对北美温带森林地区进行氮素、叶绿素和森林生物量估算,其中森林生物量预测模型决定系数( R 2)为0.82。 Kattenborn等人(2015)通过Hyperion高光谱数据和WorldView -2高空间分辨率数据对欧洲温带森林地区地上生物量进行估算,其中融合了光谱信息和空间信息的预测模型估算精度比单独使用光谱信息或空间信息的预测模型精度提高约20%,其综合模型的决定系数( R 2)为0.68,均方根误差(RMSE)为31.4 t·hm -2。以上研究证明了高光谱数据结合高空间分辨率数据反演森林生物量的可行性与有效性,但其高光谱和高空间分辨率数据并非同时获取,且在亚热带森林地区的相关研究较少。
本研究以亚热带天然次生林为研究对象,借助地面实测样地数据,通过同期获取的高光谱和高空间分辨率影像,在单木和样地尺度上进行多源数据的特征级融合,建模并反演森林生物量。研究还将综合模型(高光谱+高空间分辨率)与高光谱模型进行精度对比,以验证集成高光谱与高空间分辨率数据反演森林生物量方法的有效性和准确性。
2 材料与方法
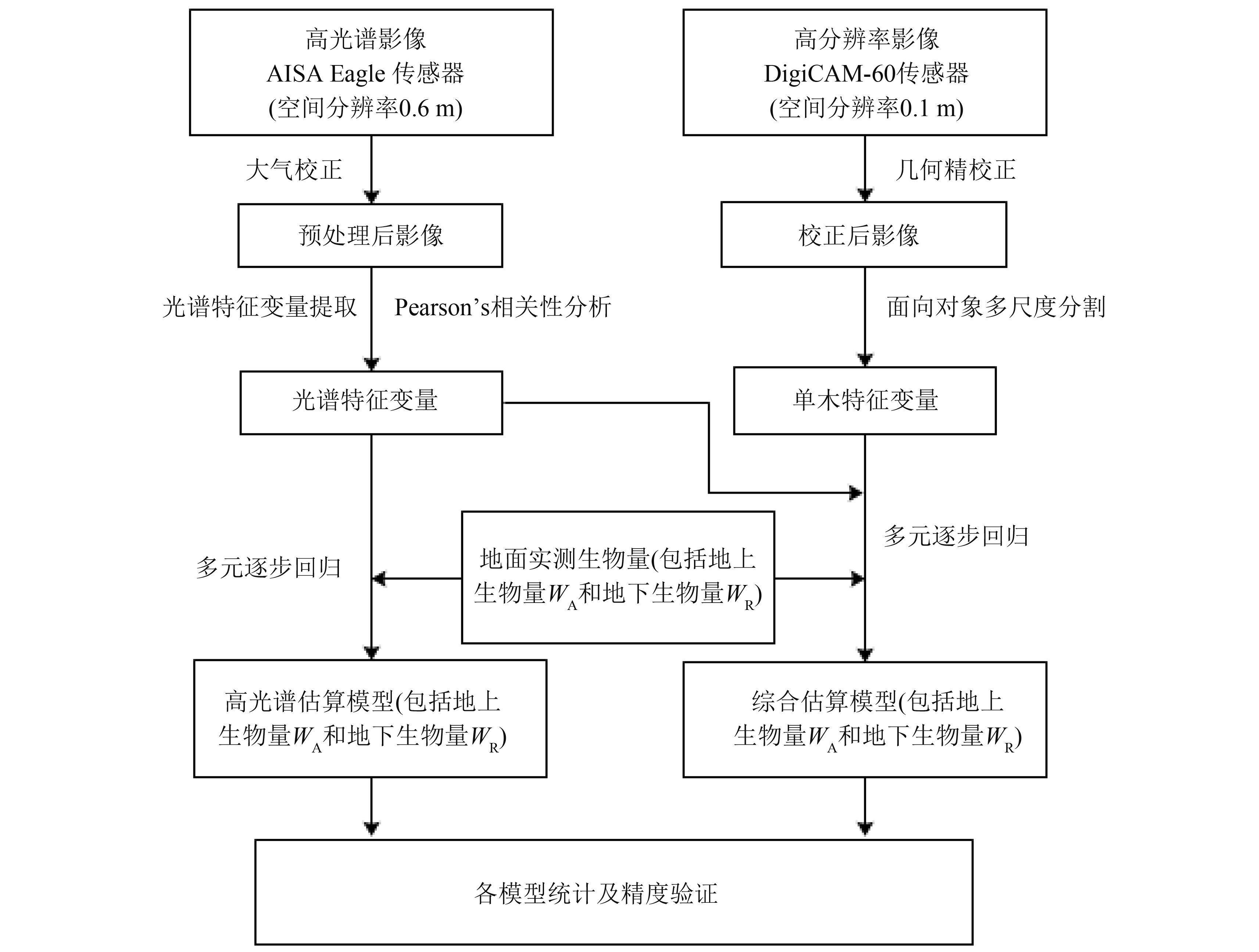
在高光谱及高空间分辨率影像预处理的基础上进行光谱特征及单木特征的提取,其中光谱特征通过特征波段选择、波段变换、植被指数计算、数理统计分析以及纹理分析进行提取,单木特征通过基于边缘检测的多尺度分割方法进行提取并汇总为单木冠幅统计变量,然后集成优化的光谱特征变量和单木冠幅统计变量并结合地面实测数据,通过构建多元回归模型分别估算森林地上、地下生物量。利用优化的光谱特征变量和地面实测数据构建多元回归模型,估算森林地上、地下生物量,最后通过地面实测数据验证模型的精度,并将两类模型进行对比以验证综合模型的准确性,研究技术路线如 图1所示。
2.1 研究区概况
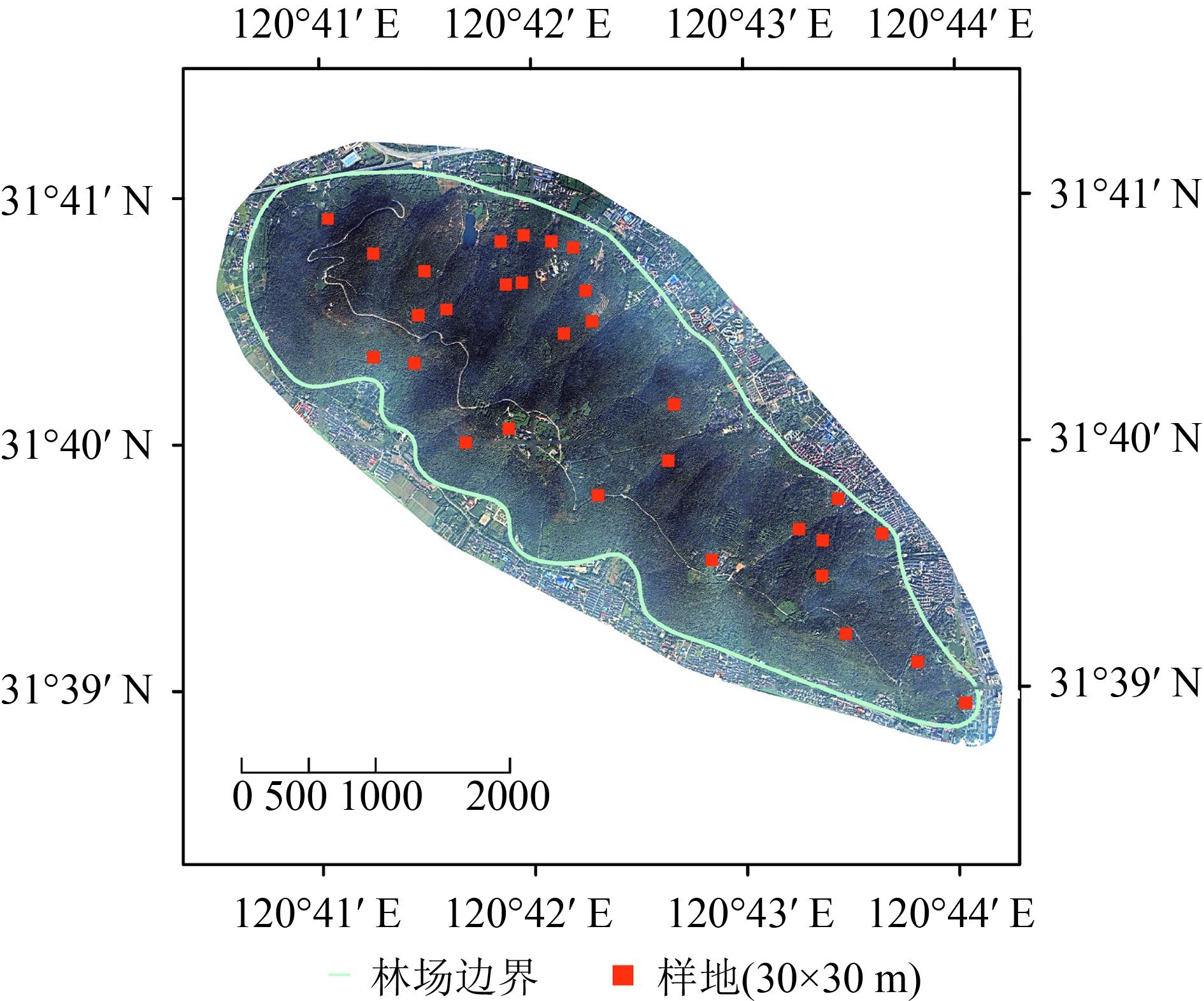
研究区选取于江苏省国营虞山林场(120°42'9.4″E—31°40'4.1″N),总面积1422 hm 2,高程变化范围为20—260 m。该研究区所处区域为典型的北亚热带季风气候区,年降水量为1062.5 mm,其中6月和7月降水量最大,分别为171.3 mm和147.0 mm,占全年降水的30%。主要土壤类型为黄棕壤,呈酸性,pH为5—6。虞山林场主要森林类型为亚热带次生混交林,可细分为针叶、阔叶和混交3个森林类型。主要针叶树种为马尾松( Pinus massoniana)、杉木( Cunninghamia lanceolata)和湿地松( Pinus elliottii)等;主要阔叶树种为麻栎( Quercus acutissima)、枫香( Liquidambar formosan)等,同时伴生冬青( Ilex chinensis)、樟树( Cinnamomum camphora)等常绿阔叶树种。研究区内林分密度较高,平均郁闭度为0.8,林木冠形为圆形或卵形,林下大多为草本类植物、低矮灌木以及乔木幼苗等。研究区及样地分布见 图2。
2.2 遥感数据获取及预处理
本研究使用的遥感数据源于机载航空平台LiCHy(LiDAR,CCD and Hyperspectral)传感器系统( Pang等,2013)。数据获取时间为2013-08-17,天气晴朗,云层较薄,太阳高度角为67.15°,飞行高度900 m。其中的高光谱数据为AISA Eagle子传感器获取,该传感器瞬时视场角(IFOV)为0.51 mrad,地面分辨率为0.6 m,波长范围为398.5—994.4 nm,光谱范围覆盖64个波段,辐射分辨率为12 bit。首先借助AISA Eagle传感器的辐射定标参数对原始影像进行辐射定标,将原始影像的DN值转化为像元辐射亮度值,然后利用经验线性模型将像元辐射亮度值转化为地表反射率值。
高空间分辨率影像为LiCHy系统中的DigiCAM-60子传感器在同一时间获取,该传感器像元分辨率为8956×6708(6000万像素),像元尺寸为6.0 μm,地面分辨率为0.1 m,辐射分辨率为8 bit。首先,对影像进行几何精校正,使其配准到高光谱影像。选取320个同名像点,采用二次多项式进行校正,并使得校正误差控制在1个像元之内,然后使用最邻近像元法进行重采样。
2.3 地面调查数据
根据虞山林场森林资源调查历史数据(2012)中的树种组成、年龄和立地分层选取30个30 m×30 m的正方形样地,样地包括针叶林(7个)、阔叶林(9个)和混交林(14个)3个森林类型。样地调查时间为2012年6—8月以及2013年8月,样地的4个角点、中心位置通过Trimble GeoXH6000 Handhelds手持GPS定位(结合JSCROS广域差分系统获得亚米级定位精度)。样地的方向和倾角通过罗盘仪测定,边界长度通过皮尺量测。在各个样地中,记录胸径≥5 cm的每棵单木的树种、胸径、树高、枝下高、冠幅、冠层透明度、树冠垂直等级CC(Crown Class)等因子。胸径采用围尺量测,树高通过Vertex IV超声波测高器获取,冠幅则在2个主方向上使用皮尺测量其投影距离。对于胸径<5 cm的树以及枯死木进行计数,但不参与生物量的计算。
借助实测的单木信息,汇总得到5个样地级森林参数,即Lorey’s树高(m)、株数(株/hm 2)、平均胸径(cm)、地上生物量(t·hm -2)和地下生物量(t·hm -2)。其中Lorey’s树高是以每株树的胸高断面积为权重,加权求和得到的平均树高;平均胸径是样地内所有树的胸径平均值。生物量的计算依据当地(或邻近区域)的异速生长方程(详见附录1),借助实测的单木胸径和树高获得单木组分生物量,最后汇总得到样地级的地上生物量 W A和地下生物量 W R( 冯宗炜等,1999; 许剑辉等,2011)。3种森林类型的5个森林参数汇总于 表1。
表 1 样地级森林参数信息汇总
Table 1 Summary of forest parameters
| 森林参数 | 针叶林(7个) | 阔叶林(9个) | 混交林(14个) | |||||
| 变化范围 | 均值 | 变化范围 | 均值 | 变化范围 | 均值 | |||
| Lorey’s树高 /m | 9.30—13.76 | 11.25 | 8.64—15.78 | 11.77 | 8.37—12.04 | 10.47 | ||
| 株数/(株·hm -2) | 633—2075 | 1443 | 510—1409 | 1076 | 965—2342 | 1554 | ||
| 平均胸径/cm | 11.45—20.90 | 14.92 | 12.63—20.62 | 14.45 | 10.42—15.69 | 13.23 | ||
| 地上生物量 W A/(t·hm -2) | 64.42—127.39 | 91.82 | 43.01—143.14 | 88.14 | 44.20—141.73 | 85.90 | ||
| 地下生物量 W R/(t·hm -2) | 20.78—39.42 | 27.55 | 20.21—37.39 | 28.23 | 15.35—43.05 | 26.29 | ||
2.4 单木特征提取及精度验证
利用基于边缘检测的多尺度分割算法对校正后的高空间分辨率影像进行面向对象分割,设置灰度阈值剔除背景信息,从而提取出单木冠幅。单木冠幅经均值统计形成样地尺度的单木统计变量,主要包括样地单木株数、冠幅面积均值、冠幅面积标准差、冠幅周长均值、冠幅周长标准差、冠幅半径均值和冠幅半径标准差共7个特征(见附录2)。另外,通过目视解译的方法分别提取出这7个单木统计特征用以验证单木冠幅自动提取的精度。
单木冠幅自动提取的精度验证采用最小二乘拟合的方式进行。将自动提取和目视解译的7个单木统计特征用最小二乘法拟合出一元线性方程,通过线性方程的系数( k)、截距( b)和决定系数( R 2)衡量冠幅自动提取的精度。
2.5 光谱特征提取
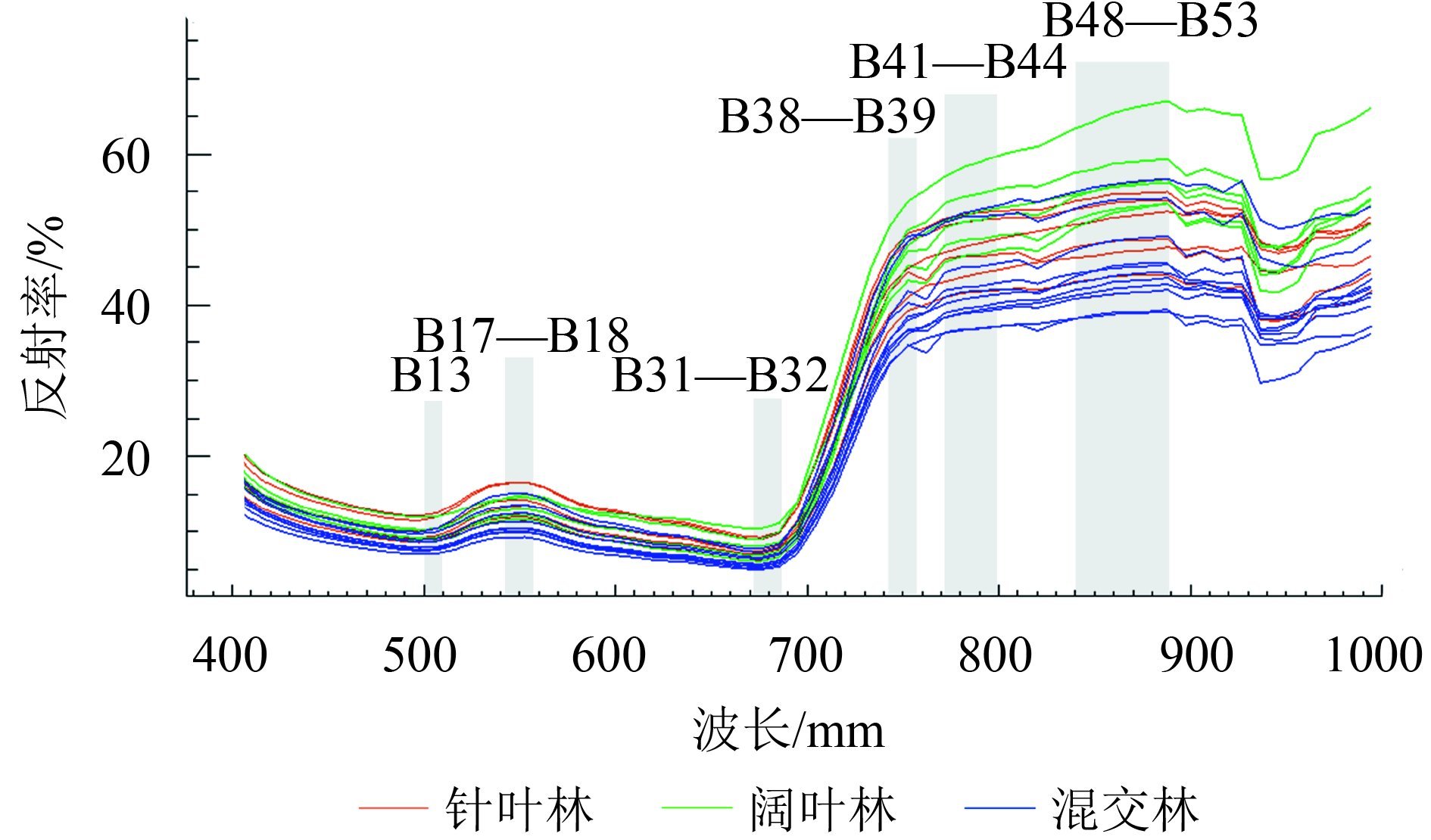
3个森林类型样地的高光谱特征曲线如 图3所示,其中同种颜色的不同光谱曲线表示同种森林类型下不同样地的光谱反射率。由图3可见,3个森林类型的光谱曲线分层较显著(尤其是近红外波段),因此选择可见光和近红外区域的部分波段作为原始单个波段特征。另外,通过对高光谱影像进行波段组合、多种植被指数变换、主成分分析、独立成分分析、最小噪声分离以及纹理分析,共提取出5组(共52个)光谱特征变量:包括17个原始单波段特征、8个波段组合特征、9个植被指数特征、9个数理统计特征以及9个纹理特征。其中,光谱特征变量已由像元尺度经均值计算转化为样地尺度。52个光谱特征变量的含义及计算公式见附录2。
2.6 特征变量优化
首先将30个正方形样地(针叶林7个、阔叶林9个、混交林14个)按照随机方式分为建模组和预测组,其中建模组21个样地,预测组9个样地。然后,在建模组中,将提取的7个单木统计变量和52个光谱特征变量与待预测变量(地上生物量 W A和地下生物量 W R)进行Pearson相关性分析,并对Pearson’s相关系数的绝对值按照从大到小的顺序排列。最后,设置阈值提取优化变量,即选取Pearson’s相关系数绝对值大于0.15的特征变量作为建模候选变量。
2.7 建模及精度评价
研究中采用多元逐步回归法,以实测地上和地下生物量作为因变量,优选的候选特征变量作为自变量建立模型。运用逐步进入法(stepwise),通过观察决定系数 R 2的情况选择进入模型的变量,经过不断进入决定系数高的变量,剔除决定系数低的变量,得到最适模型,且模型中所有变量 F检验值均达到显著水平( P值<0.05)( Næsset和Gobakken,2008)。
模型的精度采用决定系数( R 2)、均方根误差(RMSE)和相对均方根误差(rRMSE)3个指标评价。其中,均方根误差(RMSE)和相对均方根误差(rRMSE)计算公式为
| $\rm{RMSE}=\sqrt{\frac{\sum\limits_{i=1}^{n}{{{\left( {{Y}_{i}}-{{\widehat{Y}}_{i}} \right)}^{2}}}}{n-r-1}}$ | (1) |
| ${\rm{rRMSE = }}\frac{{{\rm{RMSE}}}}{{\overline {{Y_{{i}}}} }}$ | (2) |
式中,
Y
i
为生物量实测值;
通过建模组数据共建立两类模型,分别为高光谱模型和综合模型。首先,利用实测地上和地下生物量作为因变量,优选的高光谱特征变量作为自变量建立高光谱模型;然后利用实测地上和地下生物量作为因变量,优选的全部特征变量(即高光谱特征变量+高空间分辨率特征变量)作为自变量建立综合模型;最后,对高光谱模型和综合模型分别进行精度评价。
另外,在预测组中,将高光谱模型和综合模型中对应的变量分别带入,从而计算出两类模型地上、地下生物量的预测值,并与实测生物量作对比以评价两类模型的预测能力。
本研究同时采用交叉验证法评价各模型的精度。该方法是在模型参数已经确定的情况下,随机选取所有样地( n)中的一个样地作为验证样地,而其余 n—1个样地进行建模,利用拟合得到的模型对随机选取的样地进行验证,循环往复,直至所有样地都验证完毕。研究中,建模组的所有样地(针叶、阔叶、混交共21个)参与交叉验证,逐个样地进行检验。
3 研究结果
图4为利用目视解译 图4(a)和基于边缘检测的多尺度分割算法 图4(b)分别提取的单木冠幅以及利用地面实测数据对多尺度分割算法自动提取的单木冠幅进行精度评价的结果 图4(c)。 图4(b)中单木冠幅自动提取效果较好,与目视解译结果( 图4(a))较为匹配,较准确地提取出冠幅的边界,且经提取的单木冠幅半径与地面实测冠幅半径较为吻合( 图4(c))。 图5所示为面向对象方法提取的单木冠层与目视解译提取结果的比较。对比的参数包括:冠幅面积均值、冠幅面积标准差、冠幅周长均值、冠幅周长标准差、冠幅半径均值和冠幅半径标准差共6个特征。其中,除冠幅周长标准差外( R 2=0.687),其余5个特征的拟合效果都较好( R 2=0.739—0.903),且冠幅面积均值和冠幅半径均值的拟合效果最好,其直线接近于1 : 1线( k=0.984, k=1.023),截距接近于0( b=0.185, b=0.055),且决定系数接近于0.9( R 2=0.903, R 2=0.894)。因此,面向对象单木冠幅提取总体精度较高。
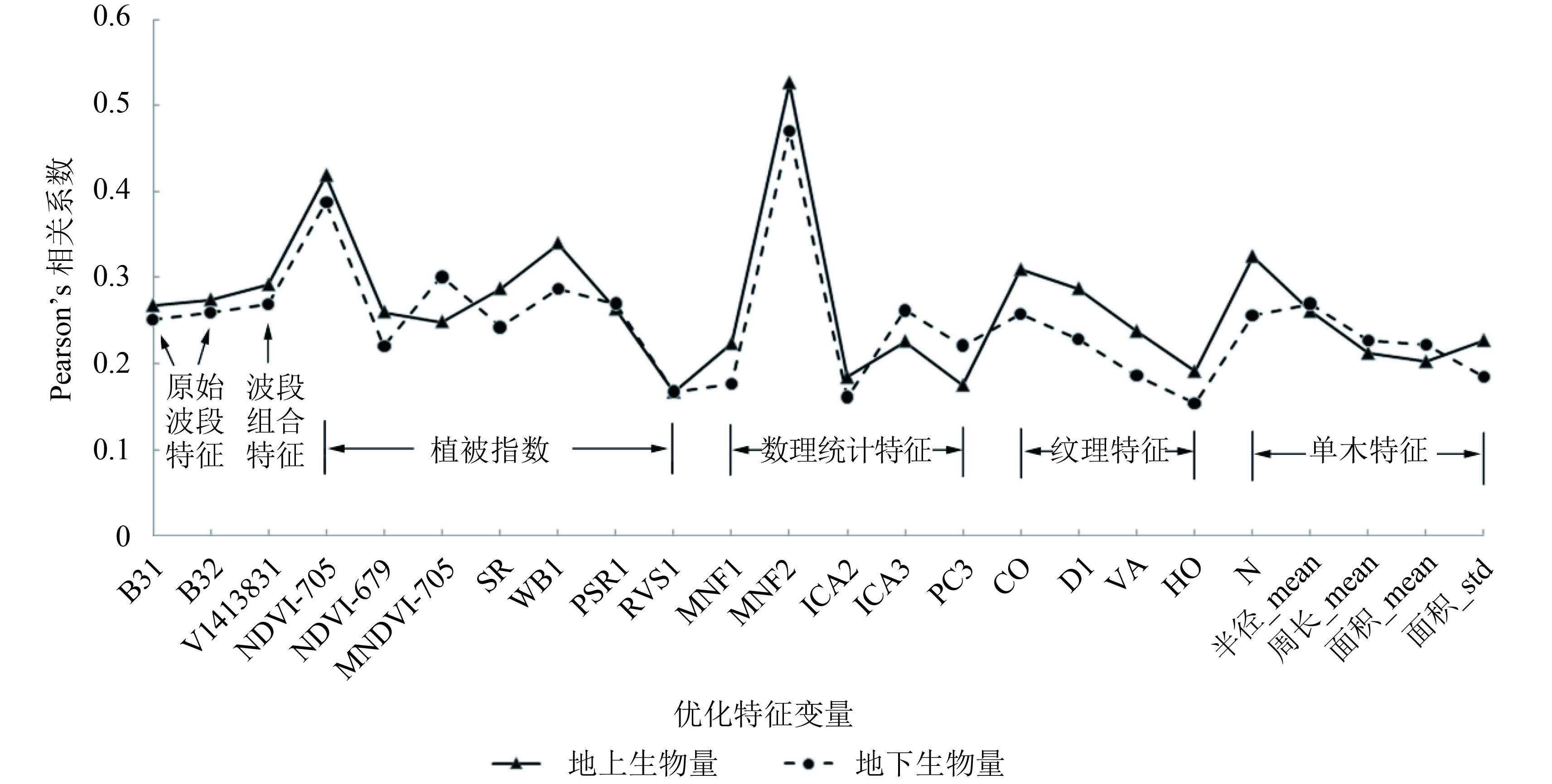
图6所示为选取的24个候选特征变量与地上和地下生物量的Pearson’s相关系数绝对值。由图中可以看出,候选高光谱特征变量为19个,候选单木统计变量为5个。在高光谱特征变量中,植被指数和数理统计特征与生物量的相关性较高,其中归一化植被指数(波长为705 nm)和提取的MNF变换第2波段与生物量的相关性最高。在单木统计变量中,株数及平均冠幅半径与生物量的相关性较高。选取的24个特征变量与生物量的相关性较高,且经独立性 T检验( Giraldo等,2008; 张彦林,2010)验证,变量之间相互独立,适合作为建模的候选变量。
表2为高光谱和综合生物量模型参数及其精度评价。由表中可见,在高光谱模型中地上生物量模型精度( R 2为0.57)高于地下生物量模型的精度( R 2为0.48);在综合模型中地上生物量模型精度( R 2为0.62)高于地下生物量模型的精度( R 2为0.54)。另外,综合模型中的地上生物量模型精度( R 2为0.62)高于高光谱模型中的地上生物量模型精度( R 2为0.57);综合模型中的地下生物量模型精度( R 2为0.54)高于高光谱模型中的地下生物量模型精度( R 2为0.48)。
表 2 高光谱和综合生物量反演模型参数及其精度评价
Table 2 Coefficients and the accuracy assessment of hyperspectral and integrated biomass models
| 高光谱模型 | 综合模型 | ||||
| 地上生物量 W A | 地下生物量 W R | 地上生物量 W A | 地下生物量 W R | ||
| 截距Intercept | 78.34 | -21.51 | -264.72 | -28.65 | |
| SR | — | 2.00 | — | — | |
| MNDVI-705 | 18.42 | 25.81 | 80.81 | 21.99 | |
| PSRI | — | -234.04 | -879.69 | -202.51 | |
| MNF1 | 0.06 | — | — | — | |
| MNF2 | 0.36 | — | — | — | |
| PC3 | 303.05 | 47.25 | — | — | |
| N | — | — | 0.87 | 0.14 | |
| 周长_mean | — | — | 10.22 | — | |
| 面积_std | — | — | — | 2.36 | |
| R 2 | 0.57 | 0.48 | 0.62 | 0.54 | |
| RMSE | 17.47 | 4.85 | 16.32 | 4.58 | |
| rRMSE /% | 19.16 | 17.59 | 17.90 | 16.61 | |
在高光谱模型和综合模型中,都含有修正型归一化植被指数705(MNDVI-705)和植被衰减指数(PSRI)这两个特征变量,由此说明植被指数与生物量的相关性较大,能够较好地反映生物量的数量状况。单木株数( N)是与生物量直接相关的特征变量,是衡量林地生物量的重要参数,因此在综合模型中都含有该特征变量。
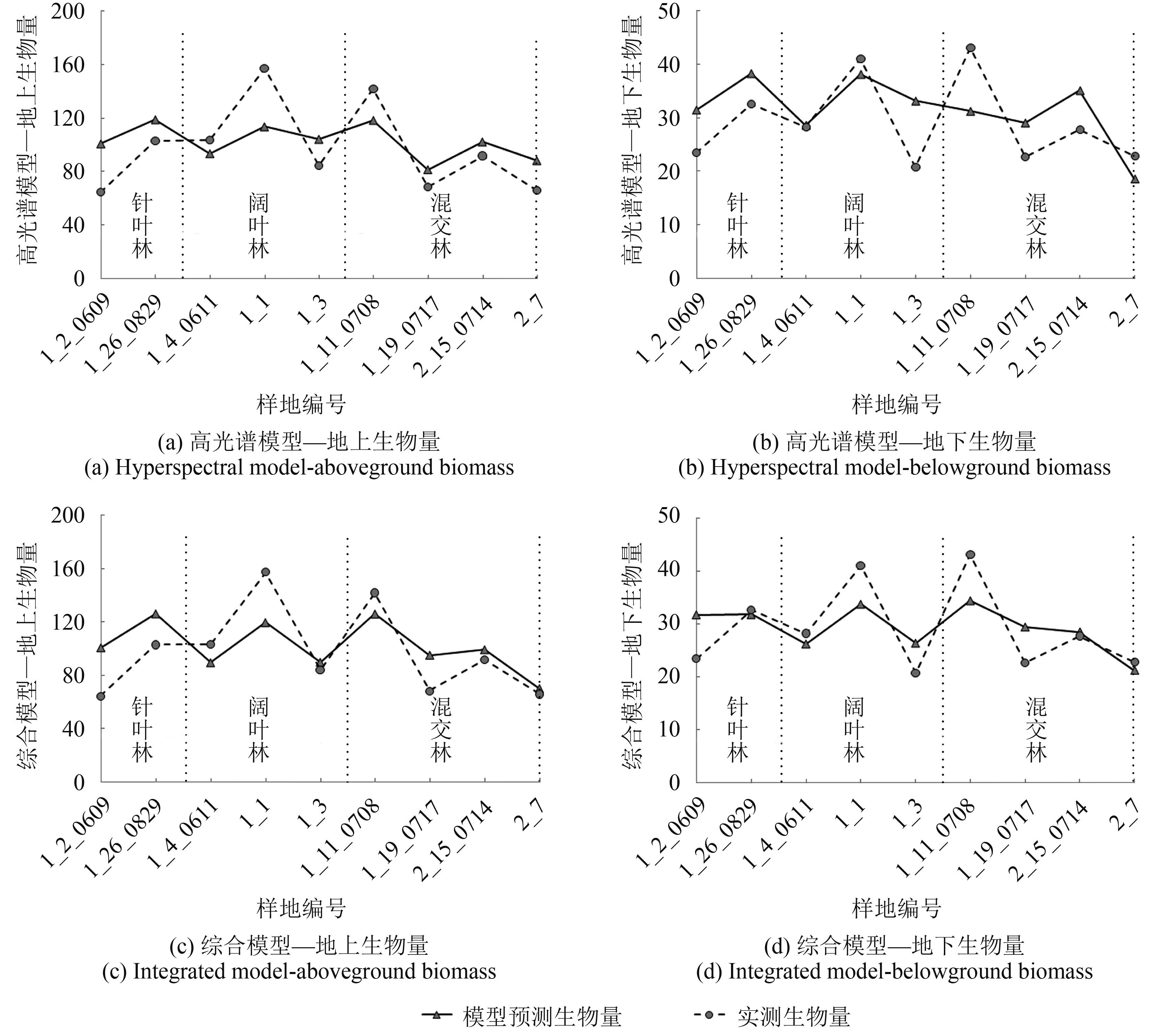
图7所示为高光谱和综合生物量模型反演地上、地下生物量与地面实测生物量对比图,其中针叶林样地2个、阔叶林样地3个、混交林样地4个,并按照从前到后的顺序排列。整体看来,高光谱模型和综合模型的预测效果都较好。其中,综合模型的预测效果比高光谱模型好,其在各个样地的模型预测值与实测生物量值差距较小;另外,在高光谱地上生物量模型中阔叶林和混交林的预测效果较好,高光谱地下生物量模型中针叶林和阔叶林的预测效果较好;在综合模型中,阔叶林和混交林的预测效果都较好。
表3为各模型交叉验证的结果。由表中可以看出,高光谱模型和综合模型预测结果与实测生物量交叉验证差异都不显著,表明两种模型预测的地上、地下生物量准确性较高,能够可靠地预测研究区的地上、地下生物量。另外,由差值范围、差值平均值和差值标准差这3个指标可以看出综合模型(0.20—4.01 t·hm -2)预测精度高于高光谱模型(0.74—4.83 t·hm -2)。
表 3 高光谱模型和综合模型交叉验证分析统计表
Table 3 The results of cross-validation of hyperspectral models and integrated models
| 模型 | 变量 | 实测平均值 | 交叉验证的差值 | ||
| 范围 | 平均值 | 标准差 | |||
| 高光谱模型 | 地上生物量 W A | 92.12 | -31.9—18.5 | -4.83 * | 15.05 |
| 地下生物量 W R | 27.41 | -19.6—14.5 | -0.74 * | 6.91 | |
| 综合模型 | 地上生物量 W A | 92.12 | -18.7—21.2 | 4.01 * | 14.06 |
| 地下生物量 W R | 27.41 | -9.04—14.5 | 0.20 * | 5.77 | |
| 注:*表示差异不显著( P>0.05)。 | |||||
4 结 论
研究中采用的高光谱和高空间分辨率数据来源于LiCHy集成传感器,属于同一时间段获取的多源高光谱和高空间分辨率影像。高光谱传感器子系统的64个波段范围覆盖整个可见光和近红外波段,可以有效捕获不同森林类型甚至不同植被在特定波段的细微光谱差异;另外其0.6 m的较高空间分辨率和12 bit的较高量化级,也增强了高光谱数据的空间识别及辐射分辨能力。高空间分辨率传感器子系统采用超高空间分辨率(0.1 m),有利于植被冠幅信息的提取。研究结果表明,高光谱和高空间分辨率数据的集成,可以丰富和完善单木冠幅和林地光谱特征信息,在充分获取细节信息的同时完善林地的光谱信息( Thenkabail等,2004)。 Kattenborn等(2015)通过集成EO-1高光谱影像和WorldView-2高空间分辨率影像对北欧温带森林进行生物量反演,其高光谱模型 R 2最高为0.56,与本研究接近;但其综合高光谱特征和高空间分辨率特征所建立的模型 R 2最高为0.68,比本研究略高。这可能是由于温带森林结构较亚热带森林简单,且其使用的样地内树种组成较为单一。本研究采用面向对象的基于边缘检测多尺度分割算法,自上而下建立不同尺度的分割等级,逐层进行分割以提取单木冠幅信息,相比传统的直接(在郁闭度较低的林分影像上)量测法( 林辉等,2004)自动化程度和精度都有所提升,同时与复杂的空间统计学理论( 冯益明等,2006)相比,更易于推广。另外,本次研究利用目视解译方法提取的单木冠幅信息来验证面向对象方法提取信息的精度,保证了多尺度分割算法提取信息的可靠性,为后续的生物量反演提供了精度保证。
与高光谱模型相比,综合生物量模型中的单木冠幅特征包括株数、周长均值和面积标准差3个变量,其中株数分别出现在地上、地下生物量模型中。另外,随着单木统计特征的加入,地上生物量模型的精度由0.57提升到0.62,地下生物量模型的精度由0.48提升到0.54,单木统计特征作为森林的重要参数,与生物量有密切联系,从而可以有效提升其反演精度( Gleason和Im,2012)。植被指数常被用来作为衡量植物生长状况的指标,经研究发现植被指数与生物量之间存在显著的统计关系( Hardisky等,1984; 张志东和臧润国,2009),这在本研究也得到验证。研究中,植被指数与地上、地下生物量的Pearson’s相关系数较高,并且经多元逐步回归后修正型归一化植被指数705(MNDVI-705)和植被衰减指数(PSRI)这2个植被指数都出现在高光谱模型和综合模型中。这充分表明,植被指数与生物量之间具有很好的相关性,可以用来建立预测和估算生物量的统计模型。
由模型预测生物量可知,在高光谱生物量模型中阔叶林预测效果较好,这是因为虞山林场处于次生演替阶段,早期人工栽培的针叶树种(如马尾松、杉木等)逐渐被本土阔叶树种(枫香、麻栎等)所代替,阔叶树种生长旺盛,占优势地位,造成了建模光谱特征变量中阔叶树种的属性较丰富所致。由 图7可以看出,模型具有一定的趋中性,且表现为当样地生物量较高时,模型预测生物量的精度偏低。究其原因,或许由于成熟林分的郁闭度高,冠层重叠较大,利用面向对象方法分割时造成了一定的信息损失;另一方面,研究区内成熟林分存在生物量饱和(saturation)现象,光谱特征对其敏感性降低,进而引起模型预测能力减弱( Song,2013)。由于高光谱和高空间分辨率数据通常只能获得森林的冠幅上表面2维(光谱)信息,缺乏空间3维结构信息,且亚热带天然次生林森林结构复杂,次生演替现象明显。故在以后的研究中,一方面可尝试从辐射传输机理层面研究生物量的反演方法,另一方面可尝试集成机载激光雷达数据、高空间分辨率数据和高光谱数据,以及分样地类型分别建立预测模型,以期进一步提高亚热带森林生物量的反演精度。
本研究以虞山林场为研究对象,利用集成传感器LiCHy获取的同时相高光谱和高空间分辨率影像,并结合外业实测数据,通过多元逐步回归法反演亚热带森林地上、地下生物量。结果表明,综合模型的精度( R 2为0.54—0.62)高于高光谱模型( R 2为0.48—0.57);在高光谱模型中地上生物量模型精度( R 2为0.57)高于地下生物量模型( R 2为0.48);在综合模型中地上生物量模型精度( R 2为0.62)同样高于地下生物量模型( R 2为0.54)。交叉检验结果表明,与仅使用高光谱数据(单一数据源)相比,通过集成高光谱和高空间分辨率数据的生物反演效果有所提升,可以更加有效地估算亚热带森林生物量。
表 1 用于计算研究区各主要树种组分生物量的异速生长模型
Table 1 Allometric equations for biomass components of dominant tree species in the study site
| 树种 | 组分 | a | b |
| 马尾松( Pinus massoniana) | 树干生物量( W s) | 0.141 | 1.092 |
| 树枝生物量( W b) | 0.065 | 0.991 | |
| 叶生物量( W f) | 0.132 | 0.745 | |
| 根生物量( W r) | 0.005 | 0.890 | |
| 杉木( Cunninghamia lanceolata) | 树干生物量( W s) | 0.124 | 0.680 |
| 树枝生物量( W b) | 0.203 | 0.385 | |
| 叶生物量( W f) | 0.850 | 0.189 | |
| 根生物量( W r) | 0.337 | 0.418 | |
| 湿地松( Pinus elliottii) | 树干生物量( W s) | 0.235 | 0.900 |
| 树枝生物量( W b) | 0.080 | 1.064 | |
| 叶生物量( W f) | 0.456 | 0.610 | |
| 根生物量( W r) | 0.007 | 1.014 | |
| 麻栎( Quercus acutissima) | 树干生物量( W s) | 0.018 | 1.034 |
| 树枝生物量( W b) | 0.00008 | 1.468 | |
| 叶生物量( W f) | 0.004 | 0.769 | |
| 根生物量( W r) | 0.020 | 0.882 | |
| 枫香( Liquidambar formosana) | 树干生物量( W s) | 0.093 | 0.801 |
| 树枝生物量( W b) | 0.083 | 0.649 | |
| 叶生物量( W f) | 1.084 | 0.217 | |
| 根生物量( W r) | 0.055 | 0.107 | |
| 注: 模型的格式为 W=a( D 2 H) b. H=树高(m), D=胸径(cm); a, b为模型参数。 | |||
表 2 特征变量汇总表
Table 2 Summary of metrics used in the research
| 特征变量 | 变量描述 | |
| 原始单个波段 | B13、 B17—18、 B31—32、 B38—39、 B41—44、 B48—53 | 高光谱原始第13、17—18、31—32、38—39、41—44、48—53波段 |
| 波段组合 | VI(39,52,53) | ( B 39+ B 52+ B 53)/3 |
| VI(42,38,53) | ( B 42+ B 38+ B 53)/3 | |
| VI(43,38,53) | ( B 43+ B 38+ B 53)/3 | |
| VI(44,38,53) | ( B 44+ B 38+ B 53)/3 | |
| VI(51,38,39) | ( B 51+ B 38+ B 39)/3 | |
| VI(41,38,31) | ( B 41- B 38)/ B 31 | |
| VI(40,15) | ( B 40- B 15)/( B 40+ B 15) | |
| VI(45,31) | B 45- B 31 | |
| 植被指数 | 简单比值植被指数(SR) | B 44/ B 31 |
| 修正型简单比值植被指数(MSR) | ( B 39- B 6)/( B 34- B 6) | |
| 归一化植被指数679(NDVI-679) | ( B 44- B 31)/( B 44+ B 31) | |
| 归一化植被指数705(NDVI-705) | ( B 39- B 34)/( B 39+ B 34) | |
| 修正型归一化植被指数705(MNDVI-705) |
|
|
| 土壤调整植被指数(SAVI) |
|
|
| 红边植被胁迫指数(RVSI) |
|
|
| 植被衰减指数(PSRI) | ( B 31- B 12)/ B 39 | |
| 植被水含量指数(WBI) | B 54/ B 62 | |
| 数理统计特征 | 第一主成分(PC1) | 提取的主成分分析第一波段 |
| 第二主成分(PC2) | 提取的主成分分析第二波段 | |
| 第三主成分(PC3) | 提取的主成分分析第三波段 | |
| 独立成分分析第一波段(IC1) | 提取的独立成分分析第一波段 | |
| 独立成分分析第二波段(IC2) | 提取的独立成分分析第二波段 | |
| 独立成分分析第三波段(IC3) | 提取的独立成分分析第三波段 | |
| 最小噪声分离变换第一波段(MNF1) | 提取的MNF变换第一波段 | |
| 最小噪声分离变换第二波段(MNF2) | 提取的MNF变换第二波段 | |
| 最小噪声分离变换第三波段(MNF3) | 提取的MNF变换第三波段 | |
| 纹理特征 | 相关度(CR) | |
| 对比度(CO) | ||
| 相异性(DI) | ||
| 信息熵(EN) | ||
| 均匀度(HO) | ||
| 均值(ME) | ||
| 二阶矩(SM) | ||
| 偏斜度(SK) | ||
| 方差(VA) | ||
| 单木统计变量 | 株数(N) | 样地尺度单木株数 |
| 单木冠幅面积平均值(面积_mean) | 样地尺度单木冠幅面积平均值 | |
| 单木冠幅面积标准差(面积_std) | 样地尺度单木冠幅面积标准差 | |
| 单木冠幅周长平均值(周长_mean) | 样地尺度单木冠幅周长平均值 | |
| 单木冠幅周长标准差(周长_std) | 样地尺度单木冠幅周长标准差 | |
| 单木冠幅半径平均值(半径_mean) | 样地尺度单木冠幅半径平均值 | |
| 单木冠幅半径标准差(半径_std) | 样地尺度单木冠幅半径标准差 | |
| 注:Pi,jki,j/ | ||
参考文献(References)
-
Asner G P, Martin R E, Knapp D E, Tupayachi R, Anderson C, Carranza L, Martinez P, Houcheime M, Sinca F, Weiss P.2011.Spectroscopy of canopy chemicals in humid tropical forests. Remote Sensing of Environment, 115 : 3587–3598. DOI: 10.1016/j.rse.2011.08.020.
-
Bastin J F, Barbier N, Couteron P, Adams B, Shapiro A, Bogaert J, De Cannière C.2014.Aboveground biomass mapping of African forest mosaics using canopy texture analysis: toward a regional approach. Ecological Applications, 24 : 1984–2001. DOI: 10.1890/13-1574.1.
-
Brantley S T, Zinnert J C, Young D R.2011.Application of hyperspectral vegetation indices to detect variations in high leaf area index temperate shrub thicket canopies. Remote Sensing of Environment, 115 : 514–523. DOI: 10.1016/j.rse.2010.09.020.
-
Clark M L, Roberts D A.2012.Species-level differences in hyperspectral metrics among tropical rainforest trees as determined by a tree-based classifier. Remote Sensing, 4 : 1820–1855. DOI: 10.3390/rs4061820.
-
Cramer W, Bondeau A, Woodward F I, Prentice I C, Betts R A, Brovkin V, Cox P M, Fisher V, Foley J A, Friend A D, Kucharik C, Lomas M R, Ramankutty N, Sitch S, Smith B, White A, Young-Molling C.2001.Global response of terrestrial ecosystem structure and function to CO 2 and climate change: results from six dynamic global vegetation models . Global Change Biology, 7 : 357–373. DOI: 10.1046/j.1365-2486.2001.00383.x.
-
Dixon R K, Solomon A M, Brown S, Houghton R A, Trexier M C, Wisniewski J.1994.Carbon pools and flux of global forest ecosystems. Science, 263 : 185–190. DOI: 10.1126/science.263.5144.185.
-
Elvidge C D, Chen Z K.1995.Comparison of broad-band and narrow-band red and near-infrared vegetation indices. Remote Sensing of Environment, 54 : 38–48. DOI: 10.1016/0034-4257(95)00132-K.
-
Fang J Y, Liu G H, Xu S L.1996.Biomass and net production of forest vegetation in China. Acta Ecologica Sinica, 16 : 497–508. ( 方精云, 刘国华, 徐嵩龄. 1996. 我国森林植被的生物量和净生产量. 生态学报, 16 : 497–508. )
-
Feng Y M, Li Z Y, Zhang X.2006.Estimating forest stand crown based on high spatial resolution image. Scientia Silvae Sinicae, 42 : 110–113. ( 冯益明, 李增元, 张旭. 2006. 基于高空间分辩率影像的林分冠幅估计. 林业科学, 42 : 110–113. )
-
Feng Z W, Wang X K, Wu G.1999.. The Forest Ecosystem Biomass and Productivity in China : 99–187. ( 冯宗炜, 王效科, 吴刚. 1999. . 中国森林生态系统的生物量和生产力 : 99–187. )
-
Foody G M, Cutler M E, McMorrow J, Pelz D, Tangki H, Boyd D S, Douglas I.2001.Mapping the biomass of Bornean tropical rain forest from remotely sensed data. Global Ecology and Biogeography, 10 : 379–387. DOI: 10.1046/j.1466-822X.2001.00248.x.
-
Gao B C.1996.NDWI-A normalized difference water index for remote sensing of vegetation liquid water from space. Remote Sensing of Environment, 58 : 257–266. DOI: 10.1016/S0034-4257(96)00067-3.
-
Giraldo M A, Bosch D, Madden M, Usery L, Kvien C.2008.Landscape complexity and soil moisture variation in South Georgia, USA, for remote sensing applications. Journal of Hydrology, 357 : 405–420. DOI: 10.1016/j.jhydrol.2008.05.029.
-
Gleason C J, Im J.2012.Forest biomass estimation from airborne LiDAR data using machine learning approaches. Remote Sensing of Environment, 125 : 80–91. DOI: 10.1016/j.rse.2012.07.006.
-
Gong P, Miller J R, Spanner M.1994.Forest canopy closure from classification and spectral unmixing of scene components-multisensor evaluation of an open canopy. IEEE Transactions on Geoscience and Remote Sensing, 32 : 1067–1080. DOI: 10.1109/36.312895.
-
(Goodenough D G and Bannon D. 2014. Hyperspectral forest monitoring and imaging implications//Proc. SPIE 9104, Spectral Imaging Sensor Technologies: Innovation Driving Advanced Application Capabilities. Baltimore, Maryland, USA: SPIE [DOI: 10.1117/12.2057637])
-
Haboudane D, Miller J R, Pattey E, Zarco-Tejada P J, Strachan I B.2004.Hyperspectral vegetation indices and novel algorithms for predicting green LAI of crop canopies: modeling and validation in the context of precision agriculture. Remote Sensing of Environment, 90 : 337–352. DOI: 10.1016/j.rse.2003.12.013.
-
Hardisky M A, Daiber F C, Roman C T, Klemas V.1984.Remote sensing of biomass and annual net aerial primary productivity of a salt marsh. Remote Sensing of Environment, 16 : 91–106. DOI: 10.1016/0034-4257(84)90055-5.
-
Inoue Y, Peñuelas J, Miyata A, Mano M.2008.Normalized difference spectral indices for estimating photosynthetic efficiency and capacity at a canopy scale derived from hyperspectral and CO 2 flux measurements in rice . Remote Sens Environ, 112 : 156–172. DOI: 10.1016/j.rse.2007.04.011.
-
Kattenborn T, Maack J, Faßnacht F, Enßle F, Ermert J, Koch B.2015.Mapping forest biomass from space-Fusion of hyperspectral EO1-hyperion data and Tandem-X and WorldView-2 canopy height models. International Journal of Applied Earth Observation and Geoinformation, 35 : 359–367. DOI: 10.1016/j.jag.2014.10.008.
-
Kokaly R F, Clark R N.1999.Spectroscopic determination of leaf biochemistry using band-depth analysis of absorption features and stepwise multiple linear regression. Remote Sensing of Environment, 67 : 267–287. DOI: 10.1016/S0034-4257(98)00084-4.
-
Lévesque J, King D J.2003.Spatial analysis of radiometric fractions from high-resolution multispectral imagery for modelling individual tree crown and forest canopy structure and health. Remote Sensing of Environment, 84 : 589–602. DOI: 10.1016/S0034-4257(02)00182-7.
-
Lin H, Ning X B, Lü Y.2004.Compiling the standing volume table of Chinese fir based on the high-resolution satellite image. Scientia Silvae Sinicae, 40 : 33–39. ( 林辉, 宁晓波, 吕勇. 2004. 基于高分辨率卫星图像的立木材积表的编制. 林业科学, 40 : 33–39. )
-
Lu D S.2006.The potential and challenge of remote sensing-based biomass estimation. International Journal of Remote Sensing, 27 : 1297–1328. DOI: 10.1080/01431160500486732.
-
Luyssaert S, Schulze E D, Börner A, Knohl A, Hessenmöller D, Law B E, Ciais P, Grace J.2008.Old-growth forests as global carbon sinks. Nature, 455 : 213–215. DOI: 10.1038/nature07276.
-
Næsset E, Gobakken T.2008.Estimation of above-and below-ground biomass across regions of the boreal forest zone using airborne laser. Remote Sensing of Environment, 112 : 3079–3090. DOI: 10.1016/j.rse.2008.03.004.
-
(Pang Y, Li Z Y, Ju H B, Liu Q W, Si L, Li S M, Tan B X, Zhong K T, Lu H, Dian Y Y, Liu L X and Jia W. 2013. LiCHy: CAF’s LiDAR, CCD and hyperspectral airborne observation system//Proceedings of 13th International Conference on LiDAR Applications for Assessing Forest Ecosystems. Beijing: Chinese Academy of Forestry: 45-54)
-
Song C H.2013.Optical remote sensing of forest leaf area index and biomass. Progress in Physical Geography, 37 : 98–113. DOI: 10.1177/0309133312471367.
-
(Takayama T, Iwasaki A and Kashimura O. 2014. Optimal segmentation of classification and prediction maps for monitoring forest condition with spectral and spatial information from hyperspectral data//Proceedings of the 2014 IEEE Geoscience and Remote Sensing Symposium. Quebec City, QC: IEEE: 3498-3501)
-
Thenkabail P S, Enclona E A, Ashton M S, Legg C, De Dieu M J.2004.Hyperion, IKONOS, ALI, and ETM+ sensors in the study of African rainforests. Remote Sensing of Environment, 90 : 23–43. DOI: 10.1016/j.rse.2003.11.018.
-
Xu J H, Wang M L, Huang Q F, Gong S F.2011.Study on aboveground biomass model of natural individual trees of Quercus acutissima . Anhui Forestry Science and Technology, 37 : 3–6. ( 许剑辉, 汪茂林, 黄庆丰, 宫守飞. 2011. 天然麻栎单木地上生物量模型研究. 安徽林业科技, 37 : 3–6. )
-
Zhang Y L.2010.Application of t Test in modeling of remote sensing data. Journal of Anhui Agricultural Science, 38 : 2730–2733. ( 张彦林. 2010. t检验在遥感数据建模中的应用. 安徽农业科学, 38 : 2730–2733. )
-
Zhang Z D, Zang R G.2009.Modelling the spatial distribution of aboveground biomass based on vegetation index in a tropical forest in Bawangling, Hainan Island, South China. Chinese Journal of Plant Ecology, 33 : 833–841. ( 张志东, 臧润国. 2009. 基于植被指数的海南岛霸王岭热带森林地上生物量空间分布模拟. 植物生态学报, 33 : 833–841. )
-
Zheng L F, Wang J N.1992.A study on imaging spectrometry and its extraction of image spectral information. Remote Sensing of Environment China, 7 : 49–58. ( 郑兰芬, 王晋年. 1992. 成像光谱遥感技术及其图像光谱信息提取的分析研究. 环境遥感, 7 : 49–58. )