| 机载激光雷达及高光谱的森林乔木物种多样性遥感监测 |
收稿日期: 2017-08-30
2. College of Resources and Environmental, University of Chinese Academy of Sciences, Beijing 100049, China
3. State Key Laboratory of Vegetation and Environmental Change, Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China
森林的物种多样性作为生物多样性的关键组成部分,在维持生态平衡、生态过程以及生态服务等方面起着不可替代的作用(张煜星和王祝熊,2007)。乔木层是森林生态系统的主要部分,决定着林下灌木层和草本层的组成和结构(赵淑清 等,2004)。森林乔木物种多样性是指森林生态系统中乔木物种的数量和物种分布的均匀程度,体现了一定区域内森林生物资源的丰富性。近年来,随着人类活动的加剧和气候变化的影响,森林乔木物种多样性正面临着严重的威胁。为了制定有效的森林生物多样性保护措施,需要对森林乔木物种多样性的现状和变化趋势进行动态监测。
传统的森林乔木物种多样性监测方法主要有编目法和指示种法(徐文婷和吴炳方,2005)。编目法主要通过大规模的标本采集进行分类鉴定,建立物种、生态系统以及遗传资源的数据库并对这些数据进行分类、排序、数量化和制图的过程(Heywood,1995)。指示种法是根据经验和分析研究,选择某个或某些具有指示性意义的品种、物种或生态系统,用来表示研究地区内物种多样性丰富、贫乏、受到保护或损害的程度。传统方法虽然监测精度高,但是耗时耗力,受到研究区环境因素的限制。
遥感技术具有探测范围广、高时空分辨率等优势,从20世纪90年代初开始应用于森林生物多样性的监测研究(Davis 等,1990;Warner和Shank,1997),主要基于以下4个理论:光谱变异假说、生产力假说、种—面积关系和栖息地空间异质性(魏彦昌 等,2008)。Rocchini等人(2004)利用Quickbird影像结合主成分分析方法证实了光谱异质性与物种多样性存在一定的联系;Kooistra等人(2008)结合植被动态模型与遥感估算的NPP模拟了莱茵河下游平原地区的生物多样性;Westman等人(1989)基于种—面积关系利用从遥感影像提取的栖息地破碎度等景观指数预测了随栖息地面积减小受威胁的物种的数量;Oindo等人(2003)利用遥感获取的土地数据与环境异质性参数间接进行了物种多样性的估算。传统遥感监测森林物种多样性的方法由于低空间分辨率和光谱分辨率,难以获取丰富的光谱分辨率与森林结构信息,制约着遥感监测森林物种多样性的精度。随着遥感技术的快速发展,CASI、PHI等高光谱成像光谱仪和ALS、LMS等激光雷达(LiDAR)机载传感器的出现,使得获取高分辨率区域尺度的森林物种光谱与结构特征成为可能,为开展森林乔木物种多样性监测提供了更加有效的手段。
机载激光雷达LiDAR通过主动发射高频脉冲能够直接穿透植被冠层,获取高精度的植被3维结构信息和生境结构信息(Heinzel和Koch,2011;Coops 等,2016)。通过提取冠层高度、叶面积指数与郁闭度等结构参数,建立结构参数与实测多样性指数的关系,可以监测区域尺度的森林物种多样性。如Lopatin等人(2016)利用机载LiDAR离散点云数据分别从乔木、灌木等4层植被中提取了12个森林结构参数与地形参数,结合广义线性模型和随机森林模型预测了研究区的物种多样性(R2=0.5—0.66,P<0.001)。另一方面,机载平台上的高光谱数据可以获得连续的地物光谱信息,现已广泛应用于森林物种多样性的监测(Kapos,2017;Asner 等,2017;Fassnacht 等,2016;Möckel 等,2016),其监测方法主要分为两类:一是利用光谱角度匹配SAM(Cho 等,2010)、支持向量机SVM(Feret,2013)、线性判别分析LDA(Clark 等,2005)等分类方法通过森林树种识别监测森林物种多样性;二是利用高光谱数据从特定波段计算出的植被指数来探测植被的生化组分以识别不同树种,如Asner(2008)在夏威夷热带森林利用机载高光谱数据和实测生化组分含量,证实了几乎每一个物种都拥有唯一的化学脚印,建立了物种光谱特征与生化组分之间的相关关系。
将LiDAR和高光谱数据相结合监测森林冠层物种多样性,主要基于将LiDAR提取的结构信息与高光谱提取的光谱信息协同构建估算模型,其中模型构建的方式可分为两种:一是直接与实测多样性数据建立关系模型(Naidoo 等,2012;Ceballos 等,2015);二是利用树种的特征参数进行聚类分析。前者算法成熟,较为简便,但精度受训练样本约束;后者阐明机理,具有一定的区域适用性。由于不同区域的树种具有不同的生化特性与结构差异,通过实测多样性指数来挑选适用于研究区的最优植被指数与最优结构参数,能够提高特定区域内乔木物种多样性的估算能力。如Asner和Martin(2009)利用机载高光谱数据和实测生化组分含量数据,选择了叶绿素、纤维素、木质素等7个叶片生化组分,实现了热带森林物种多样性成图;Simonson等人(2012)利用机载LiDAR离散点云数据提取了平均植被高度等9个森林结构参数,通过与实测数据的相关分析表明95%分位数高度和植被穿透率指数与Shannon-Wiener指数拟合关系最好(R2=0.57,P<0.001)。
综上,森林物种多样性监测可以通过树种的结构特征和光谱特征来予以区分,根据其相似程度通过聚类的方法,利用聚类数量确定物种丰富度。其中,自适应模糊C均值聚类算法克服了传统聚类方法需要预先指示初始类的缺点,能够自动地确定最佳聚类数(Li和Yu,2009)。为了获取不同树种的结构与光谱特征,需要在对树种进行单木分离的基础上进行聚类,以监测区域尺度森林乔木物种多样性。本文以神农架国家自然保护区为研究区,同步获取机载激光雷达与高光谱数据,基于光谱变异理论,提出适用于森林乔木物种多样性监测的最优结构参数和最优植被指数,并在单木尺度上,利用自适应模糊C均值聚类方法结合最优结构参数与最优植被指数,探讨区域尺度森林乔木物种多样性的遥感监测方法。
2、研究区与数据 (2.1) 研究区概况研究区位于湖北省神农架国家级自然保护区,地理坐标为东经110°2′56″—110°31′47″,北纬31°16′34″—31°22′30″,研究区总面积为154 km2(图1)。研究区受北亚热带季风气候影响,全年降雨量800—2500 mm,年平均气温12.5 ℃,年平均日照时数为1682.9 h。该区域处于亚热带与暖温带两种气候的过渡地区,同时还处于中部山地与东部平原丘陵间两种地貌过渡地区,土壤类型主要为黄棕壤。在研究区内,植被垂直带谱完整,植物区系异常丰富,从低海拔到高海拔依次表现为常绿阔叶林带—常绿落叶阔叶混交林带—落叶阔叶林带—针阔混交林带—亚高山针叶林带(Zeng 等,2008)。

|
| 图 1 研究区PHI-3高光谱影像及典型研究区地面样方分布 Figure 1 PHI-3 hyperspectral image of the study area and the fieldlocations of filed measured sample plots in the typical region |
另外,在研究区内将中国科学院神农架生物多样性定位研究站所在的区域作为典型研究区,用于森林乔木物种多样性监测模型的构建。该典型研究区的植被垂直带分布明显,涵盖了20多个森林优势树种,同时该区域还能够反映人类活动对物种多样性分布的影响。
(2.2) 数据及预处理 2.2.1. 机载遥感数据LiDAR数据的获取时间为2013年10月,采用运5飞机搭载的ALS70-HP机载激光扫描系统,飞行最低绝对航高大于2800 m,最低相对航高800 m,共敷设57条航线,平均点云密度大于4个/m2。基于国家测绘地理信息局发布的《机载激光雷达数据处理技术规范》与《机载激光雷达数据后处理技术规范》标准,经过分类将LiDAR点云数据分为地面点、建筑物、低矮植被、中间植被和高层植被5类,再利用点云数据中的首次回波点插值生成数字表面模型DSM(Digital Surface Model),利用地面点建立不规则三角网TIN(Triangular Irregular Network),基于TIN的构网原理,采用双线性内插法插值生成数字高程模型DEM(Digital Elevation Model)(邬伦 等,2001)。随后,将DEM与DSM做差值并结合冠层高度模型优化算法(Zhao 等,2014),得到经过无效值填充的冠层高度模型CHM(Canopy Height Model)。在开展林冠结构参数提取时,还需要将点云数据的高程值减去对应DEM的栅格像元值,得到点距离地面的相对高度,即去除地形影响后的归一化点云数据。
机载高光谱数据是由同步搭载在运5飞机上的PHI-3(Push broom Hyperspectral Image-3)高光谱传感器获得,两个探测器的光谱范围和分辨率分别为450—1000 nm(5 nm)和1000—2500 nm(10 nm),其中可见光近红外波段数为128,短波红外波段数为256,视场角为40°,航飞对应的高光谱成像空间分辨率为1 m。高光谱数据的预处理包括辐射定标、几何校正、地形校正和大气校正。其中,几何校正是利用与LiDAR点云数据同步获取的CCD影像进行几何配准,配准精度在1—2个像元;地形校正是基于LiDAR数据生成的精度优于1 m的DEM数据,采用ACTOR4软件中的起伏地形校正模型完成的;大气校正是利用ATCOR4软件基于MODTRAN5辐射传输代码计算的大气校正函数查找表LUTs(Look Up Tables)进行纠正。大气校正前,将440—2500 nm光谱范围内受水汽影响较大的波段(1326—1518 nm和1823—2015 nm)剔除,最终保留了376个波段。由于高光谱数据是由21条航带组成且不同航带获取数据的时间有所差异,为了消除不同天顶角和太阳位置对森林冠层光谱产生的差异,利用ATCOR4软件提供的经验BRDF校正模型将大气校正后的高光谱影像地表反射率统一校正到传感器天顶角为0°,太阳天顶角为45°。
2.2.2. 野外调查数据野外数据调查时间为2013年9月到10月和2016年9月,两次调查共获得30 m×30 m的样方37个(图1)。样方的四角点坐标使用差分GPS定位(定位精度+1 m),其中还有4个样方使用全站仪测量了单木树冠中心及树干根部位置(精度≤10 cm),用于单木分离结果的验证。样方内获取的单木参数包括:树种名称、胸径(DBG≥5 cm)、树高、树下高和冠幅。其中,胸径用胸径尺测量,树高采用激光测高仪,冠幅是利用东西和南北两个走向的长度获得。该研究区的优势树种主要包括化香树(Platycarya strobilacea)、米心水青冈(Fagus engleriana)、亮叶桦(Betula luminifera)、锐齿槲栎(Quercus aliena)、马尾松(Pinus massoniana)、杉木(Cunninghamia lanceolata)和华山松(Pinus armandii)等。
基于调查数据计算了每个样方的Shannon-Wiener指数。Shannon-Wiener指数是一种信息不确定性测度公式,即随机从群落中抽取个体,它属于哪个种是不确定的,森林物种数越多,其不确定性越大。该指数是目前评价森林物种多样性中使用最为广泛的指标,既能反映区域内物种数量的多少,也可以反映物种分布的多样性。其计算公式如下
| ${\rm{SW}} = \sum\limits_{i = 1}^n - {p_i}{\ln}{p_i}$ | (1) |
式中,pi表示存在S个物种的群落中第i个物种的个数占所有物种总数的比例。
3、研究方法本研究的总体技术路线如图2所示。物种丰富度表征了区域内物种的数量,Shannon-Wiener指数既能体现区域内物种数量的多少,也可以反映物种分布的均匀性(Pielou,1966)。利用遥感技术获取能够区分物种的结构和光谱特征,需要首先从常用的结构和光谱参数中筛选出与实测Shannon-Wiener指数相关性最高的结构参数与植被指数。为了消除林下灌、草对乔木树种冠层光谱的影响,在利用冠层高度模型CHM进行单木分离的基础上,计算单木的最优结构参数与最优光谱参数,再基于自适应模糊C均值聚类方法,以30 m×30 m为窗口获取最佳聚类数(物种丰富度)和Shannon-Wiener指数,以实现在典型研究区及神农架国家自然保护区的森林乔木物种多样性遥感监测成图。

|
| 图 2 基于机载LiDAR与机载高光谱数据的林冠物种多样性监测技术路线 Figure 2 Flowchart of main processing for predicting forest biodiversity by hyperspectral and LiDAR data. |
单木分离的方法是以LiDAR数据生成的并经过无效值填充的冠层高度模型CHM为数据源(Zhao 等,2013),采用基于结合形态学冠层控制的分水岭算法对单木进行分离(Zhao 等,2014)。算法首先引入形态学冠层控制来确定树冠的区域,即对CHM应用近圆形结构元素的形态学闭算子,利用目标区域树木的枝下高作为阈值确定冠层区域;再对原始的CHM灰度影像进行中值滤波得到CHM灰度影像,对当前确定的冠层区域采用局部极值法对冠层区域求极值确定树冠顶点位置,同时对冠层高度模型CHM进行两次分水岭变换来探测CHM上高凸山峰状的树冠,从而勾绘出树冠的形状,两次分水岭变换中间需要通过一次图像重建来移除第一次分水岭变换结果中的过分割树冠。最后对每个树冠进行形态学开运算,还原真实的树冠形状,结合高度归一化的点云计算每棵单木的树高,得到最终的单木分离结果。
(3.2) 基于激光雷达的森林冠层结构参数提取利用机载LiDAR获取的归一化点云数据提取了典型区37个样方的森林冠层结构参数。冠层结构参数分为3类,包括垂直、水平以及内部结构参数(表1)。其中,垂直结构参数中95%分位数高度和标准差是利用分类后的离散点云中植被回波数据直接统计获取;平均植被高度是所有植被回波的平均高度。水平结构中,冠层盖度通过首次回波中高度大于1.8 m的回波数所占点云总数的比例计算的;冠幅是基于CHM利用分水岭分割算法提取的;叶面积指数是基于分类后的点云数据根据比尔朗伯定律建立林窗空隙与LAI的关系获取的。森林冠层结构的内部参数中,植被穿透率是分类后植被首次回波点数与总点云数的比例;叶高度多样性强调的是叶密度和高度分布,类似于物种多样性中的Shannon-Wiener指数,不同的是公式中的P是指LiDAR在不同高度间隔所占的比例。基于相关分析法对37个实测样方的Shannon-Wiener指数与LiDAR结构参数的关系进行定量关系,分别从3类结构参数中选择相关性最高的结构参数用于监测森林乔木物种多样性。
|
|
表 1 LiDAR森林冠层结构参数提取 Table 1 Canopy structure index derived from LiDAR |
植被指数是多个波长范围内地物反射率的组合运算,在一定条件下可定量表明植被的长势、生化含量及光能利用效率等(赵英时,2003)。基于对前人研究的分析与总结,在本研究中选取了16个常用的高光谱植被指数(表2)。其中,改进型叶绿素吸收比值植被指数TCARI、优化土壤调节植被指数OSAVI、MCARI/OSAVI指数以及红边位置指数REIP等能够反映叶绿素含量;Gitelson等人(2002)发展了类胡萝卜素反射率指数CRI用来反演类胡萝卜素含量;Serrano等人(2002)发展了归一化氮指数NDNI和归一化木质素指数NDLI用以反演冠层氮含量和木质素含量。光化学植被指数PRI可以反映叶黄素指标。Asner(2008)证实了这些生化指标对物种的区分具有一定指示性。因此,本文首先计算了37个样方中单木树冠的高光谱植被指数,但该指数受到冠层结构变化的影响,为了排除了冠层结构对树种间光谱特征的影响,通过除以基于LiDAR提取的单木尺度LAI,简单的实现了冠层到叶片的尺度转换(Zarco-Tejada 等,2010)。随后,利用实测的Shannon-Wiener指数与样方内16个植被指数的标准差开展相关分析,选择适用于表征研究区树种光谱差异的最优植被指数。
|
|
表 2 植被指数计算公式 Table 2 Vegetation indices equations |
模糊C均值聚类基本思想为对于给定的样本数据集X={Xi,i=1,
| $J\left( {U,V} \right) = \sum\limits_{j = 1}^n {\sum\limits_{i = 1}^n {u_{ij}^md_{ij}^2} } $ | (2) |
式中,
其中,目标函数满足的约束条件为
| $\left\{ {\begin{array}{*{20}{c}}{{u_{ij}} \in \left[ {0,1} \right]}\\ {\displaystyle\sum\limits_{i = 1}^c {{u_{ij}}} = 1}\end{array}} \right.$ | (3) |
模糊C均值聚类的基本原则为通过迭代调整隶属度矩阵U和聚类中心V。使得目标函数J(U, V)取得极小值,通过拉格朗日乘法求得J(U, V)的条件极值,分别得到U和V的迭代形式,其中k表示迭代步数,ε为迭代阈值,
| $u_{ij}^{\left(k \right)} = \frac{1}{{\displaystyle\mathop \sum \limits_{r = 1}^c {{\left({\displaystyle\frac{{d_{ij}^{\left(k \right)}}}{{d_{rj}^{\left(k \right)}}}} \right)}^{\frac{2}{{m - 1}}}}}}$ | (4) |
| $v_i^{\left({k + 1} \right)} =\displaystyle \frac{{\displaystyle\mathop \sum \limits_{j = 1}^n {{\left({u_{ij}^{\left(k \right)}} \right)}^m}{x_j}}}{{\displaystyle\mathop \sum \limits_{j = 1}^n {{\left({u_{ij}^{\left(k \right)}} \right)}^m}}}$ | (5) |
自适应模糊C均值聚类算法能够自动确定最佳聚类数,改进了基本模糊C均值算法聚类结果对初始聚类数过度依赖的缺点,通过用聚类有效性函数L(c)(Li和Yu,2009)来确定最佳聚类数。
| $\left\{ {\begin{array}{*{20}{c}} {L\left({{c}} \right) = \displaystyle\frac{{\displaystyle\mathop \sum \limits_{i = 1}^c \mathop \sum \limits_{j = 1}^n u_{ij}^m{v_i} - {{\bar x}^2}/\left({c - 1} \right)}}{{\displaystyle\mathop \sum \limits_{i = 1}^c \mathop \sum \limits_{j = 1}^n u_{ij}^m{x_j} - {v_i}^2/\left({n - c} \right)}}} \\ {\bar x =\displaystyle \frac{1}{n}\mathop \sum \limits_{i = 1}^c \mathop \sum \limits_{j = 1}^n u_{ij}^m{x_j}} \end{array}} \right.$ | (6) |
式中,L(c)的分子分母分别表示类内和类间距离和。L(c)越大表示类间距离足够大,类内距离足够小的聚类原则。自适应C均值聚类的基本步骤为通过随机设定的聚类中心开始,通过计算目标函数的最小值,根据设定的迭代阈值不断迭代计算更新隶属度矩阵与新的聚类中心,当满足
将整个研究区37个样方的单木分离结果与实测单木数进行对比验证,精度如图3(a)所示(R2=0.88, RMSE=13.17(P<0.001))。在37个样方中有4个利用全站仪进行了单木定位(定位精度≤10 cm),其中样方20由全站仪测量的单木位置(树根)与激光雷达数据单木分离后的树冠位置进行匹配的结果如图3(b)所示,单木分离存在过分割和欠分割现象,过分割的原因是单木树冠较大,树冠顶点不突出导致一个树冠被误分为多个树冠;欠分割主要是由于研究区森林郁闭度较高,多个树冠重叠在一起,造成单木分离时被分割成一个树冠。因欠分割问题导致该研究区单木分离结果略低于实测单木数。

|
| 图 3 基于CHM的单木分离验证结果和样方20单木分离结果图(绿框为样方边界,白框为分离的单木树冠,红点为实测单木根部位置) Figure 3 Individual tree isolation result based on CHM and the result of plot 20 (the green polygon represents plot border, the white polygons represent canopy location, the red points represent the field-measured position of tree root) |
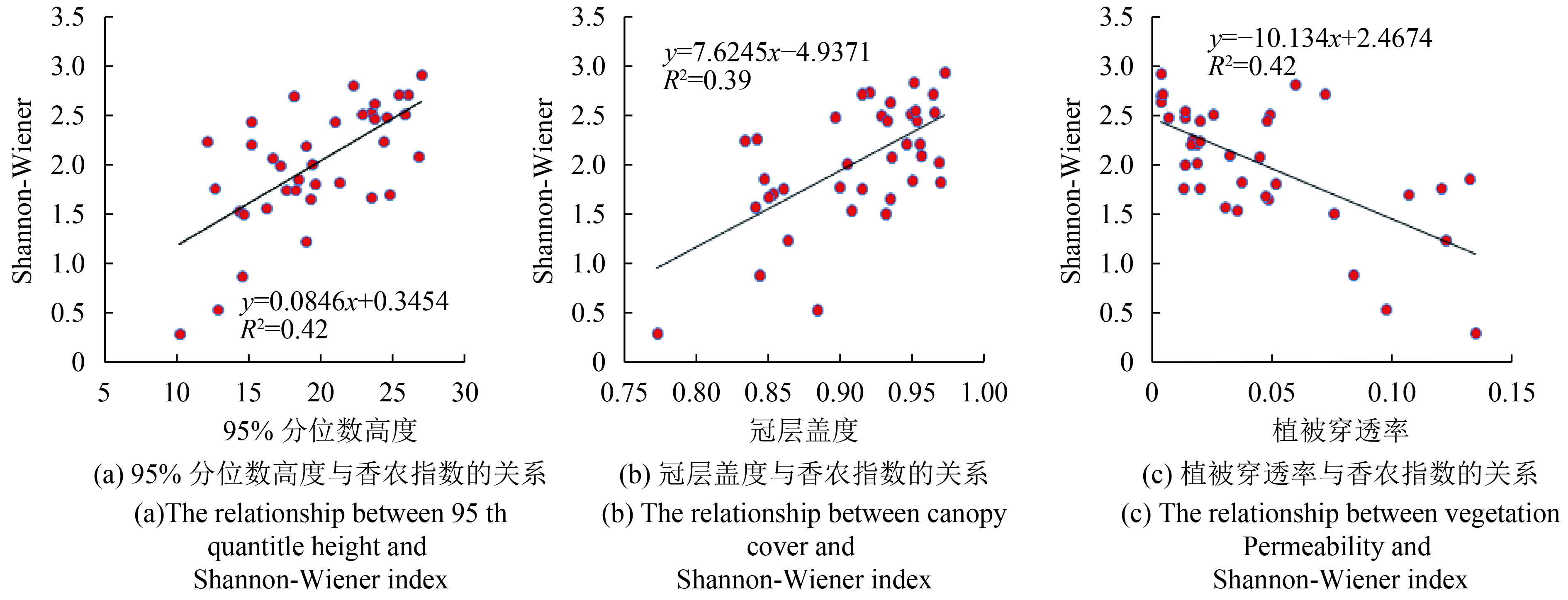
相关分析表明(图4),在垂直结构中Shannon-Wiener指数与95%分位数高度的相关性最高(R2=0.42,P<0.01),其次为标准差(R2=0.27,P<0.01),平均植被高度最低(R2=0.25,P<0.01);水平结构参数中冠层盖度参数的相关性最高(R2=0.39,P<0.01),其次为叶面积指数(R2=0.27,P<0.01),冠幅最低(R2=0.17,P<0.01);内部结构参数中植被穿透率的相关性最高(R2=0.42,P<0.01),其次为叶高度多样性(R2=0.22,P<0.01),偏度最低(R2=0.14,P<0.01)。因此,从3类结构特征参数中选择的适用于该研究区的最优结构参数分别为95%百分位高度、冠层盖度和植被穿透率。该结论与前人的研究较为一致,如Simonson等人(2012)的研究表明,95%分位数高度和植被穿透率对Shannon-Wiener指数拟合性最高(R2=0.57,P<0.001),从而证实了LiDAR结构参数对区域内森林乔木物种多样性的预测能力。
|
| 图 4 林冠结构参数与Shannon-Wiener多样性指数的关系 Figure 4 Relationship between LiDAR-derived forest canopy structure indices and Shannon-Wiener index |
在典型研究区分别提取的最优结构参数如图5所示,靠近位于中心地带的人类活动区域(生态站附近),95%分位数高度与冠层盖度较低,植被穿透率较高。但在植被生长茂盛的区域,如生态站西部,植被的冠层盖度较高,植被穿透率较低,但是95%分位数高度却不高,说明该地区虽然植被长势好但冠层高度较低。另外,在生态站东北部区域和生态站以南地区,分别有两个呈条带状的植被分布区域,该区域95%分位数高度与冠层盖度明显高于其他区域,植被穿透率也较低,表明位于阴坡的植被不但长势好且冠层高度高。

|
| 图 5 最优LiDAR林冠结构参数 Figure 5 Optimal LiDAR-derived forest canopy structure indices |
相关分析的结果表明(图6),样方内Shannon-Wiener指数与CRI、OSAVI、Narrow band NDVI、SR、Vogelmann RedEdge index1、PRI标准差的相关性最高(R2=0.37—0.45,P<0.01),其余植被指数的相关性都在0.30以下。因此,在16个植被指数中选择了以上6个植被指数,作为该区域开展森林乔木物种多样性监测的最优植被指数。其中,CRI可以反映冠层中类胡萝卜素的含量(Gitelson 等,2002);Vogelmann RedEdge Index1对冠层叶绿素浓度与水分含量十分敏感;PRI不仅可以反映叶绿素含量、还与碳吸收效率及氮胁迫的光合作用效率有关。此外,Narrow band NDVI和SR与植被的茂密程度有关,还能够与净初级生产力等间接反映物种丰富度的指数存在一定的关系(Turner,2014);OSAVI能够进一步减少土壤背景、大气和水分吸收以及宽波段植被指数的饱和问题(Tapp和Siwak,2006)。该研究与Zhao等人(2016)在同一区域开展的森林物种多样性、生化多样性及光谱多样性相关分析的结论一致,证实了与物种多样性具有较高相关性的植被指数可用来表征该区域内的树种差异。

|
| 图 6 植被指数标准差与Shannon-Wiener多样性指数的关系 Figure 6 Relationship between standard deviation of Vegetation indices and Shannon-Wiener index |
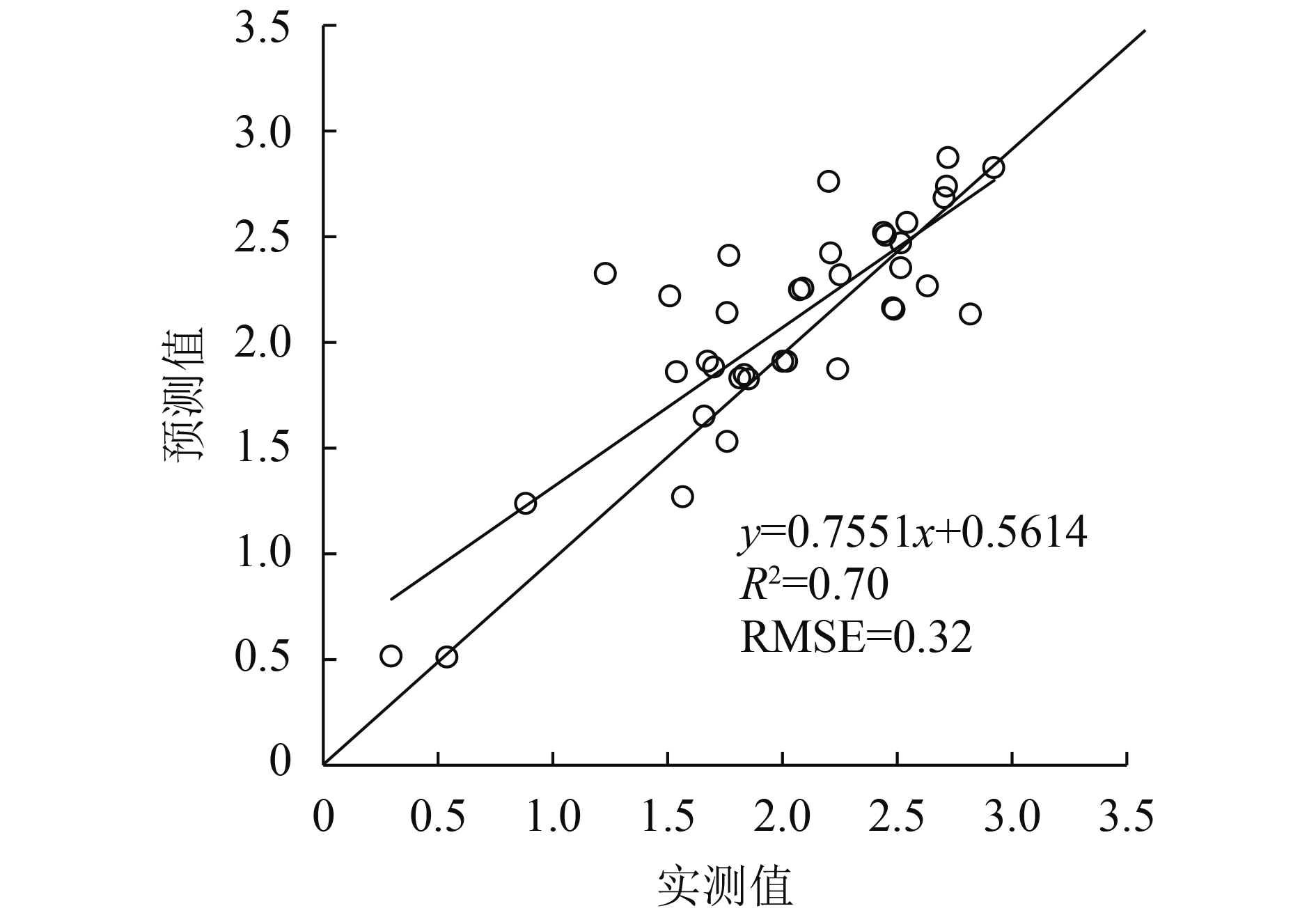
在单木尺度上提取的林冠最优结构参数和最优植被指数,利用自适应模糊C均值聚类算法以30 m×30 m窗口开展森林物种多样性监测。其中,典型研究区监测结果如图7(a)—7(b)所示,利用37个样方实测的物种丰富度和Shannon-Wiener指数对监测结果进行精度验证。物种丰富度的聚类结果(表3)表明样方内最大预测物种数为20,与实测样方最大物种数22较为接近,物种丰富度估算的精度为R2=0.69,RMSE=3.11,Shannon-Wiener指数的精度验证结果为R2=0.70,RMSE=0.32(图8)。

|
| 图 7 森林乔木物种丰富度和Shannon-Wiener指数遥感估算结果 Figure 7 Remote sensing monitoring result of species richness and Shannon-Wiener index |
|
|
表 3 物种丰富度聚类及验证结果 Table 3 The cluster and validation results of species richness |
|
| 图 8 Shannon-Wiener指数结果验证 Figure 8 The validation result of Shannon-Wiener index |
在典型研究区估算的物种多样性空间分布如图7(a)—7(b)所示,该研究区地势复杂,在坡度较缓的地方,植被容易受到人类活动的影响,如生态站附近区域的乔木物种多样性明显偏低。生态站西北与东南方向分别有两条平行的物种多样性高值带,这两个区域都位于阴坡。由于阴坡与阳坡植被受到的光照、降水条件不同,导致乔木物种的多样性有所差异。通过与最优结构参数的分布图(图5)进行对比发现,生态站东南方向物种多样性高值带区域的植被冠层高度与冠层盖度均高于阳坡,说明该研究区阴坡植被不但多样性较丰富,植被长势也好于阳坡。
将估算模型应用于整个航飞区,神农架国家自然保护区的森林乔木物种多样性遥感监测结果如图7(c)—7(d)所示。该研究区多样性变化幅度较大,主要与海拔关系密切,且呈中间膨胀型分布,即在海拔600—1400 m乔木物种多样性最高,而在低于600 m和高于1400 m的地区多样性降低,这一分布特征主要受人类活动干扰、水热条件和植被类型分布的影响。该区域植被类型从低海拔到高海拔分布规律为常绿阔叶林带—常绿落叶阔叶混交林带—落叶阔叶林带—针阔混交林带—亚高山针叶林带。航飞区西南角的海拔在1300—1400 m,主要植被类型为常绿落叶阔叶混交林与针阔混交林,由于坡度较陡、地形复杂,受人工干扰的影响较小,乔木物种多样性相对较高。航飞区西北角的平均海拔约为1600 m,该地区主要植被类型为亚高山针叶林,乔木植被种类较单一,物种多样性较低。此外,在研究区中部和东南方向植被生长茂盛区域,物种多样性也较为丰富,该区域主要位于阴坡,这与在典型研究区乔木物种多样性的分布规律一致。
聚类的基本思想是将具有相同或相似属性的个体分到同一类,聚类的核心是聚类算法与聚类尺度的有效性。自适应C均值聚类算法不用预先指定而能自动确定最佳聚类数,且该算法既能保证类间距离足够大,又能保证类内距离足够小,因此适用于森林乔木物种多样性监测模型的构建。在该研究区选择30 m×30 m窗口开展聚类,主要考虑到每一个聚类窗口与实际样方大小一致,便于开展验证。
5、结 论本文主要结果与结论如下:
(1) 单木分离有助于去除低矮灌丛、冠层结构对物种间光谱差异的影响,提高森林乔木物种多样性的监测精度。基于LiDAR数据的冠层高度模型,利用结合形态学冠层控制的分水岭算法分割了单木树冠,单木分割的精度为(R2=0.88,RMSE=13.17,P<0.001)。
(2) 机载激光雷达提取的结构参数能够反映森林乔木冠层结构分布特征,单木尺度上的3维结构参数反映了树种的结构差异,增加了对研究区森林乔木物种多样性解释能力。从冠层3维结构角度出发,在9个提取的LiDAR结构参数中,通过相关分析法得到适用于物种多样性监测的最优结构参数,分别为95%百分位高度(R2=0.42,P<0.01、冠层盖度(R2=0.42,P<0.01)和植被穿透率(R2=0.39,P<0.01)。
(3) 机载高光谱数据提取的单木尺度冠层植被指数,从光谱变异角度出发,可通过光谱特征有效反映不同树种之间的差异,通过相关分析从16个常用的植被指数中结合研究区实测多样性数据,选择了适用于物种多样性监测的最优植被指数,分别为CRI、OSAVI、SR、Narrow band NDVI、Vogelmann index1、PRI(R2=0.37—0.45,P<0.01)。
(4) 基于机载高光谱和LiDAR协同监测森林物种多样性,在单木尺度上刻画出冠层的结构特征与光谱特征,以30 m×30 m的窗口开展聚类,能够有效的监测研究区森林乔木物种多样性。结果表明,自适应模糊C均值聚类算法可预测的最大森林乔木物种数为20,物种丰富度的预测精度为R2=0.69,RMSE=3.11,Shannon-Wiener指数的预测精度为R2=0.70,RMSE=0.32。
从本研究还可以看出,单木分离的精度是影响利用单木聚类方法估算森林乔木物种多样性精度的重要因素,由于受到郁闭度较高森林单木树冠间的重叠、LiDAR点云采样密度的限制和单木分离算法的分割误差,最终都会影响单木分离的精度。在后续的研究中拟考虑利用全波形LiDAR数据,获取更精细的森林冠层3维结构,以提高单木分离的精度。本文选择了9个最优聚类参数基于自适应模糊C均值聚类方法在样方内能够识别的最大物种数为20,较适用于亚热带森林物种多样性的监测,对于乔木物种数较多的热带雨林,会受到一定的限制。今后的研究还可以从单木树冠的生物物理属性出发,挑选具有代表性的叶片光谱指数,同时加入本底环境数据,如地形、降水等,结合模式识别、机器学习等科学计算方法,将多维结构参数、光谱指数与环境数据融合建模。
| [1] | Antonarakis A S, Richards K S and Brasington J. Object-based land cover classification using airborne LiDAR[J]. Remote Sensing of Environment, 2008, 112 (6) : 2988 –2998. DOI: 10.1016/j.rse.2008.02.004 |
| [2] | Asner G P. 2008. Hyperspectral remote sensing of canopy chemistry, physiology, and biodiversity in tropical rainforests // Hyperspectral Remote Sensing of Tropical and Sub-Tropical Forests. Boca Raton: CRC Press: 261–296 [DOI: 10.1201/9781420053432.ch12] |
| [3] | Asner G P and Martin R E. Airborne spectranomics: mapping canopy chemical and taxonomic diversity in tropical forests[J]. Frontiers in Ecology and the Environment, 2009, 7 (5) : 269 –276. DOI: 10.1890/070152 |
| [4] | Asner G P, Martin R E, Knapp D E, Tupayachi R, Anderson C B, Sinca F, Vaughn N R and Llactayo W. Airborne laser-guided imaging spectroscopy to map forest trait diversity and guide conservation[J]. Science, 2017, 355 (6323) : 385 –389. DOI: 10.1126/science.aaj1987 |
| [5] | Ceballos A, Hernández J, Corvalán P and Galleguillos M. Comparison of airborne LiDAR and satellite hyperspectral remote sensing to estimate vascular plant richness in deciduous mediterranean forests of central chile[J]. Remote Sensing, 2015, 7 (3) : 2692 –2714. DOI: 10.3390/rs70302692 |
| [6] | Cho M A, Debba P, Mathieu R, Naidoo L, Aardt J V and Asner G. Improving discrimination of savanna tree species through a multiple-endmember spectral angle mapper approach: Canopy-Level analysis[J]. IEEE Transactions on Geosciences & Remote sensing, 2010, 48 : 4133 –4142. DOI: 10.1109/TGRS.2010.2058.579 |
| [7] | Clark M L, Roberts D A and Clark D B. Hyperspectral discrimination of tropical rain forest tree species at leaf to crown scales[J]. Remote Sensing of Environment, 2005, 96 (3/4) : 375 –398. DOI: 10.1016/j.rse.2005.03.009 |
| [8] | Clawges R, Vierling K, Vierling L and Rowell E. The use of airborne lidar to assess avian species diversity, density, and occurrence in a pine/aspen forest[J]. Remote Sensing of Environment, 2008, 112 (5) : 2064 –2073. DOI: 10.1016/j.rse.2007.08.023 |
| [9] | Coops N C, Tompaski P, Nijland W, Rickbeil G J M, Nielsen S E, Bater C W and Stadt J J. A forest structure habitat index based on airborne laser scanning data[J]. Ecological Indicators, 2016, 67 : 346 –357. DOI: 10.1016/j.ecolind.2016.02.057 |
| [10] | Daughtry C S T, Walthall C L, Kim M S, de Colstoun E B and EMcMurtrey J E III. Estimating corn leaf chlorophyll concentration from leaf and canopy reflectance[J]. Remote Sensing of Environment, 2000, 74 (2) : 229 –239. DOI: 10.1016/S0034-4257(00)00113-9 |
| [11] | Davis F W, Stoms D M, Estes J E, Scepan J and Michael S J. An information systems approach to the preservation of biological diversity[J]. International Journal of Geographical Information Systems, 1990, 4 (1) : 55 –78. DOI: 10.1080/02693799008941529 |
| [12] | Fassnacht F E, Latifi H, Stereńczak K, Modzelewska A, Lefsky M, Waser L T, Straub C and Ghosh A. Review of studies on tree species classification from remotely sensed data[J]. Remote Sensing of Environment, 2016, 186 : 64 –87. DOI: 10.1016/j.rse.2016.08.013 |
| [13] | Feret J B and Asner G P. Tree species discrimination in tropical forests using airborne imaging spectroscopy[J]. IEEE Transactions on Geoscience and Remote Sensing, 2013, 51 (1) : 73 –84. DOI: 10.1109/TGRS.2012.2199323 |
| [14] | Gamon J A, Serrano L and Surfus J S. The photochemical reflectance index: an optical indicator of photosynthetic radiation use efficiency across species, functional types, and nutrient levels[J]. Oecologia, 1997, 112 (4) : 492 –501. DOI: 10.1007/s004420050337 |
| [15] | Gitelson A and Merzlyak M N. Spectral reflectance changes associated with autumn senescence of Aesculus hippocastanum L. and Acer platanoides L. leaves. Spectral features and relation to chlorophyll estimation [J]. Journal of Plant Physiology, 1994, 143 (3) : 286 –292. DOI: 10.1016/S0176-1617(11)81633-0 |
| [16] | Gitelson A A, Zur Y, Chivkunova O B and Merzlyak M N. Assessing carotenoid content in plant leaves with reflectance spectroscopy[J]. Photochemistry and Photobiology, 2002, 75 (3) : 272 –281. DOI: 10.1562/0031-8655(2002)0750272ACCIPL2.0.CO2 |
| [17] | Guyot G, Baret F and Major D J. High spectral resolution: determination of spectral shifts between the red and the near infrared[J]. International Archives of Photogrammetry and Remote Sensing, 1988, 11 : VII-750 –VII-760. |
| [18] | Heinzel J and Koch B. Exploring full-waveform LiDAR parameters for tree species classification[J]. International Journal of Applied Earth Observation and Geoinformation, 2011, 13 (1) : 152 –160. DOI: 10.1016/j.jag.2010.09.010 |
| [19] | Hernández-Stefanoni J L and Dupuy J M. Mapping species density of trees, shrubs and vines in a tropical forest, using field measurements, satellite multiespectral imagery and spatial interpolation[J]. Biodiversity and Conservation, 2007, 16 (13) : 3817 –3833. DOI: 10.1007/s10531-007-9182-6 |
| [20] | Heywood V H. 1995. Global Biodiversity Assessment. Cambridge: Cambridge University Press |
| [21] | Jordan C F. Derivation of leaf-area index from quality of light on the forest floor[J]. Ecology, 1969, 50 (4) : 663 –666. DOI: 10.2307/1936256 |
| [22] | Kapos V. Seeing the forest through the trees[J]. Science, 2017, 355 (6323) : 347 –349. DOI: 10.1126/science.aal5120 |
| [23] | Kooistra L, Wamelink W, Schaepman-Strub G, Schaepman M, van Dobben H, Aduaka U and Batelaan O. Assessing and predicting biodiversity in a floodplain ecosystem: assimilation of net primary production derived from imaging spectrometer data into a dynamic vegetation model[J]. Remote Sensing of Environment, 2008, 112 (5) : 2118 –2130. DOI: 10.1016/j.rse.2007.10.010 |
| [24] | Lefsky M A, Cohen W B, Harding D J, Parker G G, Acker S A and Gower S T. Lidar remote sensing of above-ground biomass in three biomes[J]. Global Ecology and Biogeography, 2002, 11 (5) : 393 –399. DOI: 10.1046/j.1466-822x.2002.00303.x |
| [25] | Li Y and Yu F S. 2009. A new validity function for fuzzy clustering // International Conference on Computational Intelligence and Natural Computing. Wuhan, China: IEEE: 462–465 [DOI: 10.1109/CINC.2009.100] |
| [26] | Lopatin J, Dolos K, Hernández H J, Galleguillos M and Fassnacht F E. Comparing Generalized Linear Models and random forest to model vascular plant species richness using LiDAR data in a natural forest in central Chile[J]. Remote Sensing of Environment, 2016, 173 : 200 –210. DOI: 10.1016/j.rse.2015.11.029 |
| [27] | Möckel T, Dalmayne J, Schmid B C, Prentice H C and Hall K. Airborne hyperspectral data predict fine-scale plant species diversity in grazed dry grasslands[J]. Remote Sensing, 2016, 8 (2) : 133 . DOI: 10.13310.3390/rs8020133 |
| [28] | Moffiet T, Mengersen K, Witte C, King R and Denham R. Airborne laser scanning: Exploratory data analysis indicates potential variables for classification of individual trees or forest stands according to species[J]. Isprs Journal of Photogrammetry and Remote Sensing, 2005, 59 (5) : 289 –309. DOI: 10.1016/j.isprsjprs.2005.05.002 |
| [29] | Mutanga O and Skidmore A K. Narrow band vegetation indices overcome the saturation problem in biomass estimation[J]. International Journal of Remote Sensing, 2004, 25 (19) : 3999 –4014. DOI: 10.1080/01431160310001654923 |
| [30] | Naidoo L, Cho M A, Mathieu R and Asner G. Classification of savanna tree species, in the Greater Kruger National Park region, by integrating hyperspectral and LiDAR data in a Random Forest data mining environment[J]. Isprs Journal of Photogrammetry and Remote Sensing, 2012, 69 : 167 –179. DOI: 10.1016/j.isprsjprs.2012.03.005 |
| [31] | 国家测绘地理信息局. 2012. 机载激光雷达数据处理技术规范. 北京: 测绘出版社 National Administration of Surveying, Mapping and Geoinformation. 2012. Technical specification for data processing of airborne laser radar. Beijing: Surveying and Mapping Press |
| [32] | Nelson R, Krabill W and Tonelli J. Estimating Forest Biomass and Volume Using Airborne Laser Data[J]. Remote Sensing of Environment, 1988, 24 (2) : 247 –267. DOI: 10.1016/0034-4257(88)90028-4 |
| [33] | Oindo B O, Skidmore A K and De Salvo P. Mapping habitat and biological diversity in the Maasai Mara ecosystem[J]. International Journal of Remote Sensing, 2003, 24 (5) : 1053 –1069. DOI: 10.1080/01431160210144552 |
| [34] | Pielou E C. The measurement of diversity in different types of biological collections[J]. Journal of Theoretical Biology, 1966, 13 : 131 –144. DOI: 10.1016/0022-5193(66)90013-0 |
| [35] | Qi J, Chehbouni A, Huete A R, Kerr Y H and Sorooshian S. A Modified Soil Adjusted Vegetation Index[J]. Remote Sensing of Environment, 1994, 48 (2) : 119 –126. DOI: 10.1016/0034-4257(94)90134-1 |
| [36] | Riaño D, Valladares F, Condés S and Chuvieco E. Estimation of leaf area index and covered ground from airborne laser scanner (Lidar) in two contrasting forests (Lidar) in two contrasting forests. Agricultural and Forest Meteorology, 124(3/4): 269–275 [DOI: 10.1016/j.agrformet.2004.02.005[J]. Agricultural and Forest Meteorology, 2004, 124 (3/4) : 269 –275. DOI: 10.1016/j.agrformet.2004.02.005 |
| [37] | Richardson A J and Everitt J H. Using spectral vegetation indices to estimate rangeland productivity[J]. Geocarto International, 1992, 7 (1) : 63 –69. DOI: 10.1080/10106049209354353 |
| [38] | Richardson J J, Moskal L M and Kim S H. Modeling approaches to estimate effective leaf area index from aerial discrete-return LIDAR[J]. Agricultural and Forest Meteorology, 2009, 149 (6/7) : 1152 –1160. DOI: 10.1016/j.agrformet.2009.02.007 |
| [39] | Rocchini D, Chiarucci A and Loiselle S A. Testing the spectral variation hypothesis by using satellite multispectral images[J]. Acta Oecologica, 2004, 26 (2) : 117 –120. DOI: 10.1016/j.actao.2004.03.008 |
| [40] | Rondeaux G, Steven M and Baret F. Optimization of soil-adjusted vegetation indices[J]. Remote Sensing of Environment, 1996, 55 (2) : 95 –107. DOI: 10.1016/0034-4257(95)00186-7 |
| [41] | Serrano L, Peñuelas J and Ustin S L. Remote sensing of nitrogen and lignin in Mediterranean vegetation from AVIRIS data: decomposing biochemical from structural signals[J]. Remote Sensing of Environment, 2002, 81 (2/3) : 355 –364. DOI: 10.1016/S0034-4257(02)00011-1 |
| [42] | Simonson W D, Allen H D and Coomes D A. Use of an airborne lidar system to model plant species composition and diversity of mediterranean oak forests[J]. Conservation Biology, 2012, 26 (5) : 840 –850. DOI: 10.1111/j.1523-1739.2012.01869.x |
| [43] | Simpson E H. Measurement of diversity[J]. Nature, 1949, 163 (4148) : 688 –688. DOI: 10.1038/163688a0 |
| [44] | Sims D A and Gamon J A. Relationships between leaf pigment content and spectral reflectance across a wide range of species, leaf structures and development stages[J]. Remote Sensing of Environment, 2002, 81 (2-3) : 337 –354. |
| [45] | Tapp P D and Siwak C T. 2006. The canine model of human brain aging: cognition, behavior, and neuropathology // Handbook of Models for Human Aging. New York: Academic Press: 415–434 [DOI: 10.1016/B978-012369391-4/50036-9] |
| [46] | Turner W. Sensing biodiversity[J]. Science, 2014, 346 (6207) : 301 –302. DOI: 10.1126/science.1256014 |
| [47] | Vogelmann J E, Rock B N and Moss D M. Red edge spectral measurements from sugar maple leaves[J]. International Journal of Remote Sensing, 1993, 14 (8) : 1563 –1575. DOI: 10.1080/01431169308953986 |
| [48] | Warner T A and Shank M C. Spatial autocorrelation analysis of hyperspectral imagery for feature selection[J]. Remote Sensing of Environment, 1997, 60 (1) : 58 –70. DOI: 10.1016/S0034-4257(96)00138-1 |
| [49] | 魏彦昌, 吴炳方, 张喜旺, 杜鑫. 生物多样性遥感研究进展[J]. 地球科学进展, 2008, 23 (9) : 924 –931. Wei Y C, Wu B F, Zhang X W and Du X. Advances in remote sensing research for biodiversity monitoring[J]. Advances in Earth Science, 2008, 23 (9) : 924 –931. DOI: 10.3321/j.issn:1001-8166.2008.09.003 |
| [50] | Westman W E, Strong L L and Wilcox B A. Tropical deforestation and species endangerment: the role of remote sensing[J]. Landscape Ecology, 1989, 3 (2) : 97 –109. DOI: 10.1007/BF00131173 |
| [51] | 邬伦, 刘瑜, 张晶. 2001. 地理信息系统: 原理、方法和应用. 北京: 科学出版社 Wu L, Liu Y and Zhang J. 2001. Geographic Information System: Principles, Methods and Applications. Beijing: Science Press |
| [52] | 徐文婷, 吴炳方. 遥感用于森林生物多样性监测的进展[J]. 生态学报, 2005, 25 (5) : 1199 –1204. Xu W T and Wu B F. Progress on measuring forest biodiversity with remote sensing technique[J]. Acta Ecologica Sinica, 2005, 25 (5) : 1199 –1204. DOI: 10.3321/j.issn:1000-0933.2005.05.037 |
| [53] | Zarco-Tejada P J, Miller J R, Noland T L, Mohammed G H and Sampson P H. Scaling-up and model inversion methods with narrowband optical indices for chlorophyll content estimation in closed forest canopies with hyperspectral data[J]. IEEE Transactions on Geoscience and Remote Sensing, 2001, 39 (7) : 1491 –1507. DOI: 10.1109/36.934080 |
| [54] | Zeng Y, Schaepman M E, Wu B, Clevers J and Bregt A. Scaling-based forest structural change detection using an inverted geometric-optical model in the Three Gorges region of China[J]. Remote Sensing of Environment, 2008, 112 (12) : 4261 –4271. DOI: 10.1016/j.rse.2008.07.007 |
| [55] | 张煜星, 王祝熊. 2007. 遥感技术在森林资源清查中的应用研究. 北京: 中国林业出版社 Zhang Y X and Wang Z X. 2007. Research on the Application of Remote Sensing Technology in Inventory of Forest Resources. Beijing: China Forestry Publishing House |
| [56] | Zhao D, Pang Y, Li Z Y and Liu L J. Isolating individual trees in a closed coniferous forest using small footprint lidar data[J]. International Journal of Remote Sensing, 2014, 35 (20) : 7199 –7218. DOI: 10.1080/01431161.2014.967886 |
| [57] | Zhao D, Pang Y, Li Z Y and Sun G Q. Filling invalid values in a lidar-derived canopy height model with morphological crown control[J]. International Journal of Remote Sensing, 2013, 34 (13) : 4636 –4654. DOI: 10.1080/01431161.2013.779398 |
| [58] | Zhao Y J, Zeng Y, Zhao D, Wu B F and Zhao Q J. The optimal leaf biochemical selection for mapping species diversity based on imaging spectroscopy[J]. Remote Sensing, 2016, 8 (3) : 216 . DOI: 10.3390/rs8030216 |
| [59] | 赵英时. 2003. 遥感应用分析原理与方法. 北京: 科学出版社 Zhao Y S. 2003. Principles and Methods of Remote Sensing Application Analysis. Beijing: Science Press |
| [60] | 赵淑清, 方精云, 宗占江, 朱彪, 沈海花. 长白山北坡植物群落组成、结构及物种多样性的垂直分布[J]. 生物多样性, 2004, 12 (1) : 164 –173. Zhao S Q, Fang J Y, Zong Z J, Zhu B and Shen H H. Composition, structure and species diversity of plant communities along an altitudinal gradient on the northern slope of Mt[J]. Changbai, Northeast China. Chinese Biodiversity, 2004, 12 (1) : 164 –173. DOI: 10.3321/j.issn:1005-0094.2004.01.020 |

