|
收稿日期: 2015-05-13; 修改日期: 2016-02-29;
优先数字出版日期: 2016-07-25
基金项目: 江苏省自然科学基金(编号:BK20151515);江苏省高校自然科学研究项目(编号:14KJB220002);江苏高校优势学科建设工程资助项目(PAPD)
第一作者简介:
曹林(1983-),男,博士研究生,主要从事遥感技术在林业中的应用研究。E-mail:ginkgocao@gmail.com
通讯作者简介: 佘光辉(1953-),男,教授,主要从事森林监测与"3S"技术在林业中的应用研究
中图分类号: TP79
文献标识码: A
文章编号: 1007-4619(2016)04-0665-14
|
摘要
以亚热带天然次生林为研究对象,借助一个条带的少量LiDAR点云数据和覆盖整个研究区的免费LandsatOLI多光谱数据,并结合地面实测数据,探索森林生物量低成本高精度制图方法。首先,提取了OLI和LiDAR特征变量,并与地上和地下生物量进行相关分析以筛选变量;然后,借助LiDAR数据覆盖区的样地和条带LiDAR数据构建“LiDAR生物量模型”;再从LiDAR反演生物量的结果中进行采样,结合OLI特征变量构建“LiDAR-OLI模型”;最后,与单独使用OLI多光谱数据建立的“OLI估算模型”结果进行比较,分析精度并验证新方法的效果。结果表明,“LiDAR-OLI模型”对地上和地下生物量的模型拟合效果较好且均优于“OLI模型”,且其交叉验证的精度也较高并优于“OLI模型”,从而证明了新方法的可靠性及有效性。本研究为主、被动遥感技术在中小尺度上协同反演森林参数提供了实验基础,也为基于全覆盖免费OLI多光谱数据及条带LiDAR数据的低成本森林生物量制图探索了技术路线。
关键词
升尺度 , 生物量反演 , 亚热带森林 , 机载LiDAR数据 , Landsat 8 OLI影像 , 多元回归模型
Abstract
Accurate estimation of forest biomass is critical for modeling the carbon cycle and mitigating climate changes. Integration of multi-spectral satellite data and airborne LiDAR data can accurately estimate the biomass. However, the application of this strategy is lim-ited in subtropical forests, particularly in China. In this study, a novelapproach was assessed using one strip of LiDAR point cloud and "wallto-wall" Landsat OLI free multi-spectral data combined with field-measured plot data to generate a low-cost and high-accuracy forest biomass map in a subtropical secondary forest in southeast China. Sixty square plots (30 m×30 m) were established across the study site. First, the OLI data were processed by atmospheric and geometric correction, and LiDAR point clouds were extracted from the raw full-waveform LiDAR data. Second, fivesets of OLI and three sets of LiDAR metrics were extracted, and correlation analysis was performed with the field estimates of above-and below-ground biomass foroptimal metrics selection. Third, the "LiDAR biomass model" was fitted to LiDAR metrics extracted from the strip of LiDAR point cloud and the field plots within the strip. The "LiDAR-OLI biomass model" was fitted tothe OLI metrics and forest biomass estimated by the LiDAR data. Finally, the performance of the predictive models and the accuracy of the cross-validation results were evaluated through comparison with the accuracy assessment results of the "OLI biomass model." [Result] The "LiDAR-OLI biomass model" (R2 of above-and below-ground biomass estimation=0.69 and 0.56, respectively) exhibited improved performance than the "OLI biomass model" (R2 of above-and below-ground biomass estimation=0.69 and 0.56, respectively). The relative biases of above-and below-ground biomass estimation increased by 14% and 15%, respectively. The mean differences in the cross-validation results for the "LiDAR-OLI biomass model" (mean differences in above-and below-ground biomass estimation=-12.9 and -0.15, respectively) were more accurate than the "OLI model" (mean differences in above-and below-ground biomass estimation=-18.99 and -0.33, respectively). The ranges of above-and below-ground biomasses were 49.9-214.6 and 15.6-59.0 t·hm-2, respectively, in the entire study site. Moreover, the spatial distributions of above-and below-ground biomasses were similar to each other. Forests with high biomass were located in valleys and flat areas, whereas those with low biomass were located in the mountain ridge. This study provides an experimental basis for estimation of medium-scale forest parameters by synergizing active and passive remote sensing technologies. The study also explores the technological route of using one strip of LiDAR point cloud and "wall-to-wall" Landsat OLI free multi-spectral data for biomass mapping. These methods are relatively inexpensive and exhibit potential in supporting management and policies for addressing carbon stocks and understanding the effect of subtropical forest ecosystems under climate changes in China and elsewhere.
Key words
upscale , forest biomass inversion , subtropical forest , airborne LiDAR data , Landsat 8 OLI imagery , multiple regression model
1 引 言
森林生态系统是陆地生物圈的主体,其生物量约占整个陆地生态系统的85%,在减缓全球气候变化中具有不可替代的作用(Post等,1982)。森林在生长过程中通过同化作用吸收大气中的CO2,并以生物量形式将其长期固定。亚热带树种丰富且森林生产力高,不仅对区域生态环境有很大影响,而且对维持全球碳平衡也具有重要作用(Cao等,2014)。传统的生物量调查方法耗时费力,且只能获得有限的“点”上信息(曹林等,2014)。而遥感技术却能够准确、快速地获取各个尺度的“面”上连续分布的森林信息(董立新,2008;马利群和李爱农,2011),在森林资源动态监测及森林碳储量反演方面具有重要作用。
Landsat系列卫星自1972年起对地连续观测,获取了高质量的中等分辨率多光谱数据,并已成为全球最大的对地观测影像数据库。该数据为林业生产和研究提供了高质量的森林信息,特别是在2008年美国地质调查局(USGS)宣布其向公众免费开放之后,其贡献更为显著(Wulder和Coops,2014)。2013年启用的Landsat 8 OLI多光谱传感器在波段设置及对植被的敏感性上比ETM等传感器有所提升,体现在信噪比、光谱和辐射分辨率等(徐涵秋和唐菲,2013)。然而光学遥感难以穿透森林冠层获得其垂直结构信息,且在森林覆盖度高(植被生长旺盛)的区域获取森林生物量信息时易饱和,故对于亚热带森林的生物量估算有所影响(徐婷等,2015)。激光雷达LiDAR(Light Detection And Ranging)是近年来迅速发展的主动遥感技术,能透过植被冠层获取垂直结构信息和高精度3维数据,从而大大地提高森林生物量的估测精度,可以更好地反演森林碳储量(庞勇等,2005)。以往研究表明LiDAR在精确反演不同森林类型的生物物理特性方面具有较大潜力(Dubayah和Drake,2000)。
Landsat遥感数据可以获取连续、实时的森林生物物理特性的水平空间分布信息,且免费获取,但易“饱和”且难于获取森林的结构参数;而LiDAR发射的激光脉冲可以穿透植被冠层获得其三维结构和能量信息(Lefsky等,2002),但是数据获取的成本高,难以在较大尺度上应用。针对多光谱影像与LiDAR数据各自的特性,将两者结合并应用于森林参数获取一直是研究热点。Popescu等人(2004)基于小光斑 LiDAR 数据和多光谱数据进行小样区水平的材积和生物量的反演,结果表明两种技术相结合的森林参数反演精度更高;Guo等人(2010)利用环境卫星多光谱数据和ICESat/GLAS 波形数据,以塔河林场为研究区反演了地上生物量,结果表明 80%的验证点生物量误差在20 t·hm-2以内(Guo等,2010)。但以上方法要求LiDAR数据与多光谱数据覆盖整个研究区,成本较高,工作量较大,不适合在更大尺度上推广和应用。黄克标等人(2013)结合机载LiDAR数据对星载GLAS光斑范围内的森林地上生物量进行估测,并利用MODIS以及MERIS 土地覆盖产品进行了云南省森林地上生物量升尺度估算,其构建的生物量估算模型的决定系数(R2)为0.52,均方根误差(RMSE)为31 Mg·hm-2。庞勇等人(2011)通过非连续的机载LiDAR和星载LiDAR GLAS数据以及连续的光学遥感数据MERIS对大湄公河次区域的森林地上生物量进行连续分布制图。连续覆盖生物量估计的模型总体误差为34 t·hm-2,升尺度模型的相关系数为0.7(庞勇等,2011)。以上研究证明了LiDAR与光学遥感数据结合进行森林参数反演和连续分布制图的可行性及有效性,但其升尺度模型自变量相对单一(如MERIS数据只提供了NDVI作为反演参数)且制图分辨率较低(300 m)。
针对以上研究空缺,本文以江苏南部丘陵地区的北亚热带天然次生林为研究对象,借助地面调查样地数据,探索结合少量(一个条带)LiDAR点云数据和覆盖整个研究区的Landsat OLI数据估算森林生物量的方法;并与直接使用OLI数据估算生物量方法做精度比对,从而验证新方法的有效性。通过提取多组LiDAR和OLI体征变量并进行统计建模,为基于LiDAR和OLI数据的中等尺度(制图分辨率30 m)森林信息升尺度反演和森林生物量连续分布制图提供实验基础和切实可行的思路。
2 材料与方法
本文的技术路线见图 1。其中方法一是以全覆盖的Landsat 8 OLI多光谱数据为基础,结合少量(一个条带)的LiDAR数据和地面调查样地数据,建立LiDAR-OLI生物量反演模型(即从LiDAR反演生物量的结果中进行采样,结合OLI特征变量构建模型)。方法二是直接利用Landsat 8 OLI多光谱数据结合地面调查数据建立OLI生物量反演模型。通过比较“直接使用OLI数据估算生物量”(方法二)及“结合条带LiDAR数据和OLI数据估算生物量”(方法一),从而验证“方法一”的可行性及优越性。最后选择精度较高的模型生成研究区的生物量空间分布图(分辨率30 m×30 m)。
2.1 研究区概况
研究区位于江苏省常熟市国营虞山林场(120°42′9.4″ E,31°40′4.1″ N),属亚热带季风气候,气候温和,年平均降水量1054 mm,面积约1103 hm2,其海拔高度为20—261m。虞山林场属于北亚热带次生混交林,主要森林类型为针叶林,阔叶林和混交林,其中主要针叶树种有马尾松(Pinus massoniana)、杉木(Cunninghamia lanceolata)和湿地松(Pinus elliottii)等;主要阔叶树种有麻栎(Quercus acutissima)、枫香(Liquidambar formosana)及部分常绿阔叶树种,如壳斗科植物(Fagaceae)、樟科植物(Lauraceae)和山茶科植物(Theaceae)。研究区及样地分布如图 2。
2.2 地面调查数据
根据二类调查历史数据(2012年)中的森林类型、年龄和立地指数等指标在研究区范围内设置60个方形样地(大小:30 m×30 m,设置时间: 2013年8月)。样地调查过程中,对于胸径大于5 cm的树,逐一测定单木的树种、胸径(用围尺测量)、树高和枝下高(利用Vertex IV激光测高器测量)以及冠幅(即两个主方向上的投影距离,用皮尺测量),对于胸径小于5 cm的和枯死木进行计数,但不参与生物量的计算。样地西南角坐标使用差分GPS(Trimble GeoXH6000 Handheld GPS units,美国加利福尼亚州森尼韦尔市Trimble导航公司)测定,通过接收JSCORS广域差分信号定位,精度优于0.5 m。
根据单木调查数据汇总样地尺度的相关森林参数,包括每块样地单位面积的胸高断面积(m3·hm-2);胸高断面积加权的平均树高(Lorey’s mean height,即以单木胸高断面积为权重汇总的样地平均高);样地尺度上的单位面积地上、地下生物量(t·hm-2)。生物量信息通过异速生长方程(遵从就近原则)计算单木的生物量,并汇总得到每块样地的胸径、树高、Lorey’s高、单位面积地上生物量(WA)以及地下生物量(WB)(冯宗炜等,1999)。样地信息汇总于表 1。
表 1 地面调查样地信息汇总
Table 1 Summary of ground-truth plot data
| 森林参数 | 针叶林 | 阔叶林 | 混交林 | |||||
| 数值范围 | 均值 | 数值范围 | 均值 | 数值范围 | 均值 | |||
| Lorey’s高/m | 5.46—13.98 | 10.47 | 7.67—18.52 | 11.85 | 7.89—14.63 | 10.36 | ||
| 胸径/cm | 10.34—20.87 | 13.09 | 12.47—22.43 | 15.17 | 10.85—20.52 | 14.21 | ||
| 地上生物量/t·hm-2 | 43.02—126.29 | 72.43 | 32.12—219.67 | 95.63 | 54.63—142.71 | 89.21 | ||
| 地下生物量/t·hm-2 | 14.42—39.25 | 22.60 | 10.12—61.67 | 29.06 | 15.59—43.21 | 27.26 | ||
2.3 LiDAR条带数据获取及处理
研究采用Riegl LMS-Q680i LiDAR传感器获取的数据(2013年8月17日),该传感器搭载在LiCHy(LiDAR,CCDandHyperspectral)集成遥感装置中(Pang等,2013)。传感器记录了完整的激光脉冲返回波形信息,其时间采样间隔为1ns(即每个采样单元间隔为15 cm)。激光发射脉冲所在波段为近红外(波长为1055 nm),脉冲发射频率(PRF)为360 kHz,扫描频率(SF)为112 线·s-1,扫描角为±30°;遥感平台的飞行高度为900 m,飞行速度为55 m·s-1,旁向重叠度为60%;地面光斑直径约为25 cm,平均地面光斑距离约为0.48 m。最终获取的数据格式为LAS 1.3(美国摄影测量与遥感协会)标准格式。每个激光返回点内包含三维坐标值(x,y,z)、强度值(intensity)和返回类型(return type)等信息。本研究使用LiDAR数据其中一个条带范围的数据。
根据LiDAR点云数据计算了9个特征变量:冠层覆盖度变量(c);4个高度百分位数变量: h25、h50、h60、h75、h95;4个高度变量:最大高度(hmax)、最小高度(hmin)、高度变异系数(hcv)以及平均高度(hmean)。9个特征变量的含义及计算公式见附录1。
2.4 OLI多光谱数据获取及处理
研究采用2013年7月19日的Landsat 8 OLI影像(条带号119/38),完全覆盖整个虞山林场,本研究使用其中的2—7波段。首先借助OLI传感器的辐射定标参数对原始影像进行辐射定标。影像空间分辨率为30 m,辐射分辨率为12 bit,光谱范围覆盖11个波段,将原始影像灰度值(DN值)转化为像元辐射亮度值。再以FLAASH模型对影像进行大气校正(大气模型:Tropical;气溶胶模型:Urban;气溶胶反演法:2-Band(K-T);初始能见度:40;波谱响应函数:ldcm_oli.sli),从而将辐射亮度值转化为地表实际反射率。然后对影像进行几何精校正,选取40个同名地物点,采用二次多项式进行校正,校正误差控制在0.1个像元以内,并采用最邻近像元法进行重采样。
通过对OLI影像进行波段组合、缨帽变换、纹理信息提取、主成分分析、最小噪声分离变换以及多种植被指数变换,提取5组(共53个)特征变量,(1) 6个原始单波段变量:经过辐射校正和几何精校正的B2—B7;(2) 10个波段组合变量:Albedo、B4/Albedo、B547、B65、B74等;(3) 10个信息增强组变量:缨帽变换绿度TG、亮度TB、湿度TW;最小距离变换前4个波段MNF1、MNF2、MNF3、MNF4以及主成分分析前3个波段PC1、PC2、PC3);(4) 18个植被指数变量:增强型植被指数(EVI)、归一化植被指数(NDVI)、土壤调节植被指数(SAVI)、有效叶面积指数(SLAVI)等;(5) 9个纹理信息变量:对比度(CO)、相关性(CR)、相异性(DI)、信息熵(EN)、均匀度(HO)、均值(ME)、二阶矩(SM)、偏斜度(SK)以及方差(VA)。其中纹理分析针对主成分分析的第一主成分进行,窗口大小为3×3,滞后距离为1个像元。53个变量的含义及计算公式见附录 2。
2.5 特征变量优化提取及统计分析
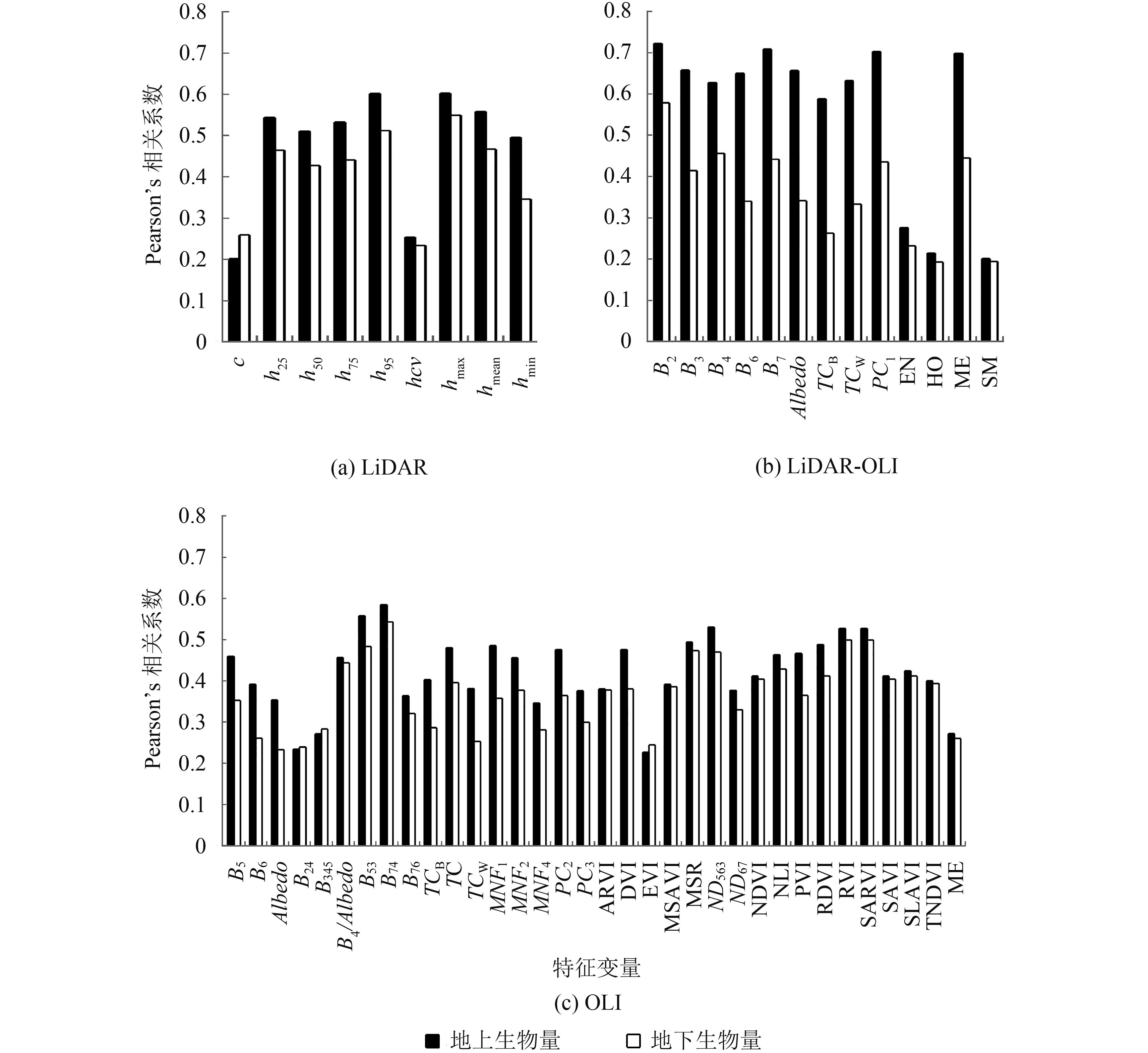
利用样地GPS坐标点位置,基于9景包含LiDAR特征变量信息的影像和53景包含OLI特征变量信息的影像,分别提取样地中心位置的像元值,并借助pearson’s相关系数矩阵分析特征变量之间的相关性,去除相关性高的特征变量。并将其与需要预测的参数进行pearson’s相关性分析,选取相关系数绝对值高于0.2 的特征变量作为建模候选变量。本研究共建立了3个生物量反演模型(LiDAR模型、LiDAR-OLI模型和OLI模型),各模型建模因子与生物量的pearson’s相关系数绝对值如图 3。
将地面实测汇总的生物量信息作为因变量,遥感方法提取的特征变量作为自变量,建立多元回归模型。运用逐步进入法(stepwise)和检验决定系数(R2)的变化情况来选择进入模型的合适变量(Næsset和Gobakken,2008)。如果有自变量使统计量F值过小并且T检验达不到显著水平(P值> 0.1),则予以剔除;F值较大且T检验达到显著水平(P值< 0.05)则得以进入(付甜等,2011)。为保证自变量之间的低信息重叠,本研究通过控制因子κ(即最大特征根的平方根除以最小特征根所得到的值)进一步筛选模型。κ通过PCA分析计算相关关系矩阵得到,其值低于30则表明自变量之间相关性较低(Næsset,2002)。采用决定系数(R2)、均方根误差(RMSE)和相对均方根误差(rRMSE)评价回归模型的精度。rRMSE作为一个相对量,与评价量本身的数量级水平无关,能够更好地体现总体值域差别较大的模型的预测精度。
本研究运用两种方法建立虞山生物量反演模型,第1种方法是以OLI数据结合LiDAR数据,进行虞山森林生物量反演,步骤如下:首先以重叠区(少量LiDAR数据与OLI影像的重叠区域)样地(31个)LiDAR特征变量为自变量,样地实测生物量为因变量构建LiDAR生物量反演模型(以下简称LiDAR模型),再利用LiDAR模型计算出的重叠区所有像元的生物量,以其作为因变量,与重叠区的OLI特征变量进行多元线性拟合,建立重叠区生物量反演模型(以下称为LiDAR-OLI模型)。第2种方法是直接利用OLI数据进行虞山森林生物量反演,步骤如下:以重叠区样地实测生物量为因变量,重叠区样地OLI特征变量为自变量构建重叠区生物量反演模型(以下称为OLI模型)。验证并比较两种方法反演生物量的精度,并将精度较高的模型应用于整个虞山林场,绘制出虞山林场地上和地下生物量信息空间分布图。
3 研究结果
3.1 OLI数据结合LiDAR数据反演森林生物量
表 2汇总了利用OLI数据结合LiDAR数据方法估算森林生物量过程中所建立的LiDAR模型和LiDAR-OLI模型的精度评价情况,图 4是根据LiDAR模型绘制的重叠区生物量信息空间分布图,图 5描述了LiDAR-OLI模型地上和地下生物量样地实测值和模型预测值的拟合散点图(95%置信区间内)。从表 2可以看出,基于重叠区样地生物量和LiDAR特征变量建立的LiDAR模型,地上和地下生物量反演精度R2分别为0.67、0.54,对应的RMSE分别为24.73 t·hm-2、7.40 t·hm-2,对应的rRMSE分别为26%、28%。由图 4可以看出,重叠区地上生物量分布范围在38.05—273.44 t·hm-2之间,地下生物量分布范围在15.53—61.83 t·hm-2之间。由表 2和图 5可以看出,利用LiDAR模型计算得出的条带区生物量值和OLI特征变量建立的LiDAR-OLI模型中,地上和地下生物量反演精度R2分别为0.69、0.56,对应的RMSE分别为17.48 t·hm-2、3.33 t·hm-2,对应的rRMSE分别为18%、13%。
表 2 OLI数据结合LiDAR数据生物量反演模型及其参数精度评价
Table 2 Regression models(based on OLI data and LiDAR data)and parameters used for estimation of biomass of plots in strip
| 特征变量 | |||||||||||
| b0 | h25 | h50 | hcv | hmax | hmean | c | R2 | RMSE | rRMSE/% | ||
| LiDAR模型 | 地上生物量 | -18.49 | -134.47 | 164.15 | 152.23 | -0.50 | 0.67 | 24.73 | 26 | ||
| 地下生物量 | -30.48 | 14.08 | -14.93 | 105.35 | 4.18 | 0.54 | 7.40 | 28 | |||
| b0 | B2 | B3 | B4 | Albedo | TCB | R2 | RMSE | rRMSE/% | |||
| LiDAR-OLI模型 | 地上生物量 | -962.28 | 0.22 | -0.11 | -0.04 | 0.02 | 0.69 | 17.48 | 18 | ||
| 地下生物量 | -180.17 | 0.05 | -0.03 | -0.003 | 0.005 | 0.56 | 3.33 | 13 | |||
3.2 直接利用OLI数据反演森林生物量
表 3汇总了利用OLI数据反演森林生物量过程中所建立的OLI模型的精度评价情况,图 6描述了OLI模型地上和地下生物量样地实测值和模型预测值的拟合散点图。从表 3和图 6可以看出,基于重叠区样地生物量和OLI特征变量建立的生物量反演模型(OLI模型),地上和地下生物量反演精度R2分别为0.49、0.55,对应的RMSE为30.80 t·hm-2、7.16 t·hm-2,对应的rRMSE分别为32%、28%。
表 3 OLI数据生物量反演模型及其参数精度评价
Table 3 Regression models(based on OLI data)and parameters used for estimation of biomass of plots in strip
| 特征变量 | |||||||||||
| b0 | NDVI | RDVI | RVI | SAVI | DVI | TNDVI | ME | R2 | RMSE | rRMSE/% | |
| 地上生物量 | 1363.81 | -6506130 | -45.58 | 84.23 | 4338117.01 | 0.49 | 30.80 | 32 | |||
| 地下生物量 | 6022.94 | 5668.26 | -0.03 | 14257.05 | 16.38 | 0.55 | 7.27 | 28 | |||
3.3 两种方法精度比较
3.3.1 比较两种模型反演生物量的精度
将重叠区外的29个样地数据作为验证数据,分别对LiDAR-OLI模型和OLI模型进行交叉验证,并对结果进行配对样本t检验,验证模型精度。由表 4可知,对于地上和地下生物量,两种模型检验结果都表现为实测值和预测值差异不显著,从交叉验证差值的平均值和范围来看,LiDAR-OLI模型(平均值为-12.92— -0.15)精度高于OLI模型(平均值为-18.98— -0.33)的精度。该结果与模型拟合的验证结果相一致。LiDAR-OLI模型拟合的效果(R2=0.56—0.69,rRMSE=13 %—18 %)同样优于OLI模型拟合的效果(R2=0.49—0.55,rRMSE=28%—32 %)。
表 4 LiDAR-OLI模型和OLI模型交叉验证均值差值分析统计结果
Table 4 Statistics for the observed mean and differences in cross-validation of the LiDAR-OLI model and OLI model
| 变量 | 模型 | 实测值平均值 | 交叉验证的差值 | ||
| 平均值 | 标准差 | 范围 | |||
| 地上生物量 | LiDAR-OLI | 85.29 | -12.92NS | 32.58 | -71.68-41.72 |
| OLI | -18.99NS | 67.59 | -218.8-73.24 | ||
| 地下生物量 | LiDAR-OLI | 26.17 | -0.15NS | 5.77 | -11.62-9.59 |
| OLI | -0.33NS | 13.33 | -26.77-25.51 | ||
| 注:NS表示差异不显著(P> 0.05) | |||||
3.3.2 虞山生物量空间分布制图
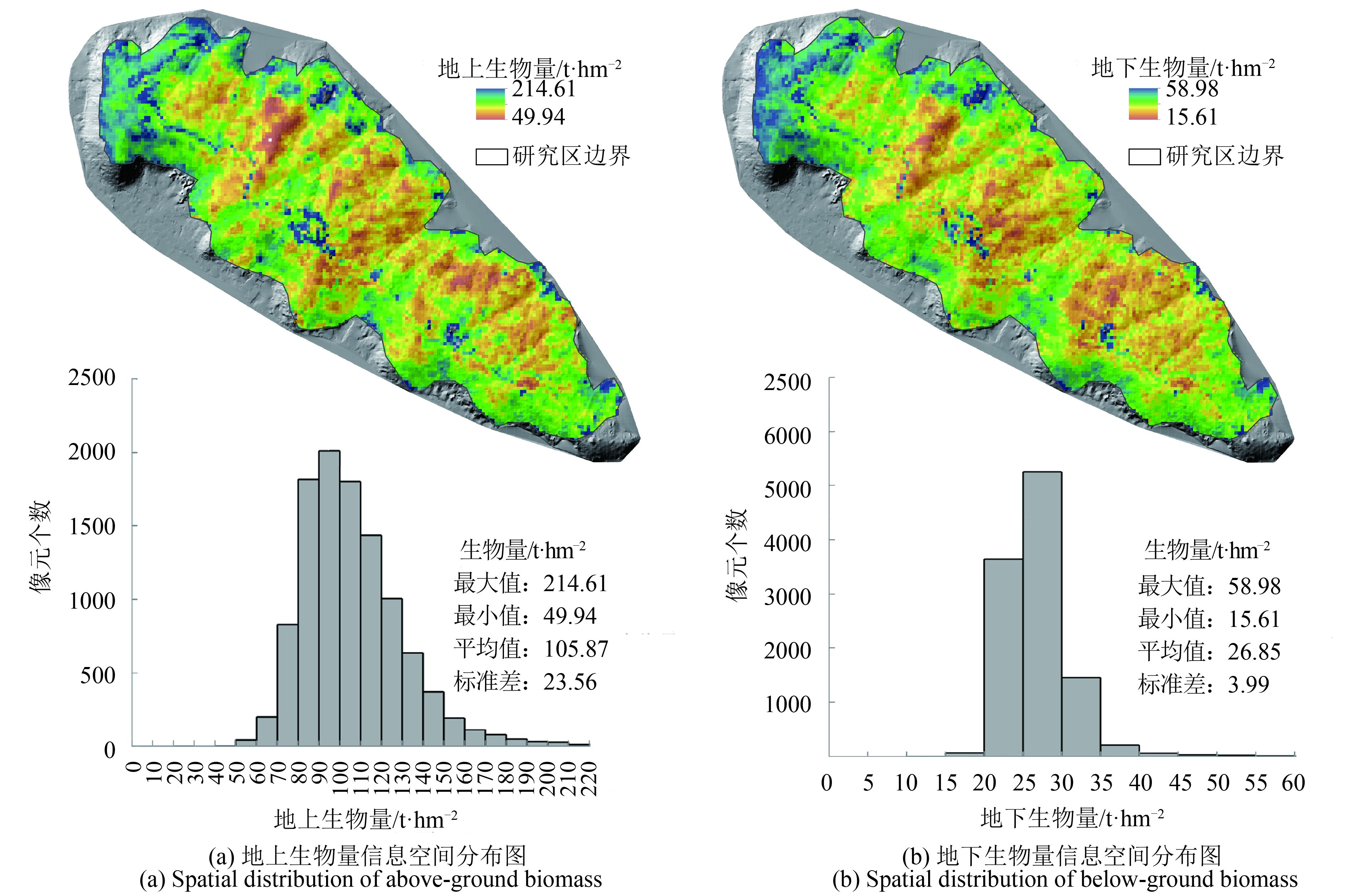
利用LiDAR-OLI生物量估算模型反演得到虞山地上和地下生物量信息空间分布图(图 7),地上生物量分布范围在49.94—214.61 t·hm-2,地下生物量分布范围在15.61—58.98 t·hm-2之间,由分布直方图可以看出,地上生物量集中分布在70—130 t·hm-2,地下生物量集中分布在20—35 t·hm-2。低值区主要分布山脊处,高值区主要分布在山谷处和林场边缘平坦的地区。地上和地下生物量分布趋势一致。
4 结 论
本研究以北亚热带天然次生林为研究对象,借助地面调查样地数据,探索结合少量(一个条带)LiDAR点云数据和覆盖整个研究区的Landsat OLI数据估算连续森林生物量分布及制图的方法。与直接使用OLI数据反演生物量的方法相比,通过两阶段(即首先构建LiDAR模型,再构建LiDAR-OLI模型)升尺度估算的方法都获得了较好的模型拟合效果及估算精度。该研究还通过提取多组LiDAR和OLI体征变量进行统计建模,并基于优化模型生成了优质的中等尺度(制图分辨率30 m)森林生物量连续分布图。由图 5和表 1可知,通过LiDAR-OLI模型反演出的生物量值的范围在野外地面调查计算得到的生物量值的范围内,这不仅说明样地的选择较合理,而且说明本研究反演的生物量模型适用于整个研究区。另外,从生物量信息空间分布图还可以看出,地上和地下生物量分布趋势基本一致,低值区主要在山脊等陡峭的地区,高值区主要分布在山谷等平坦的地区。由图 5可以看出,西北角有一块低值区,主要是因为那里地势陡峭且土壤薄不利于树木生长,虞山东北部有一个明显的高值区,是因为那里有大片的阔叶林。
本研究拟合的LiDAR-OLI模型效果(R2=0.56—0.69,rRMSE=3.33—17.48 Mg·hm-2)要优于前人在亚热带地区的类似研究。如黄克标等人(2013)结合机载LiDAR数据、星载GLAS、MODIS及MERIS 土地覆盖产品对云南省森林地上生物量进行了升尺度估算,其生物量估算模型的决定系数(R2)为0.52,均方根误差(RMSE)为31 Mg·hm-2。庞勇等通过类似方法对大湄公河次区域的森林地上生物量进行连续分布制图,其连续覆盖生物量估计模型的总体误差为34 Mg·hm-2(庞勇等,2011)。究其原因,有研究区的差异,同时也源于数据源和方法的差异。在研究区方面,由于本研究区坐落在亚热带北缘,则该森林类型没有亚热带南部和热带(如云南省及大湄公河次区)地区的森林结构复杂。另外,由于本研究结合了地面实测样地数据,提取的建模特征变量丰富,且制图分辨率为30m(高于前人研究的300 m等),故模型拟合效果更好且制图精度更高;再之,MODIS及MERIS 土地覆盖产品本身的误差会累积从而增加最终制图的误差(降低前人研究的精度);最后,由于本研究所获取的样地、LiDAR数据和OLI数据差异在一个月之内,故减少了数据获取时相上的不一致性。
从本研究所选取的数据源来看,OLI传感器对ETM+的近红外波段的设置进行了优化,波段范围由ETM+的0.775—0.900 μm缩减到0.845—0.885 μm,有效避开0.825 μm附近水汽吸收特征,提高了近红外波段的灵敏度。另外,在数据量化级别与传感器扫描方式上,采用了12 bit的数据量化级,增强了影像数据的辐射分辨能力,并避免了8 bit数据在高反射地区出现的数据饱和现象,采用线阵“推扫式”扫描方式,这种平行轨道的扫描方式能够获得更高的信噪比和几何定位精度,使影像数据产品更加准确有效(姜高珍等,2013)。Landsat 8 OLI数据的这些特性对本研究生物量估算具有积极意义。许多国内外研究表明,应用机载激光雷达数据能够精确地估计森林生物量。Means等人(1999)使用由NASA发展起来的SLICER(Scanning LiDAR Imager of Canopies by Echo Recovery)成功估测了太平洋西北方的松林结构,包括总生物量及叶生物量,且决定系数范围达到0.84—0.96。Lim等人利用机载离散回波LiDAR对加拿大东部不同年龄的成熟及过熟林生物量组分进行了估算。通过LiDAR百分位高度拟合模型,每个模型的决定系数都大于0.8(Lim等,2003)。在本研究中,利用LiDAR数据估算重叠区生物量精度达到0.54—0.67,也比较理想。
由于OLI多光谱遥感数据难以穿透森林冠层获得其垂直结构信息,且在森林覆盖度高(植被生长旺盛)的区域获取森林生物量信息时易饱和,故在该研究区直接反演生物量的估算精度低于LiDAR数据(Cao等,2014;徐婷等,2015)。通过本研究还可以看出,LiDAR-OLI模型反演生物量的结果比单独使用OLI模型的反演结果精度高。多光谱数据能够通过地物对光谱的响应实现森林水平结构参数的提取;激光雷达技术则通过主动向研究对象发射脉冲,并接收反射回波的方式,获取传统光学遥感所不能获取的森林垂直结构信息(刘丽娟,2011),主、被动遥感技术的协同,理论上可以实现数据的优势互补,一定程度上能够提高参数提取的精度。运用大范围的Landsat 8 OLI数据结合少量LiDAR数据反演森林生物量不仅可以提高反演精度还能降低成本。
研究结果表明,“LiDAR-OLI模型”对地上和地下生物量的模型拟合效果较好且均优于“OLI模型”,且其交叉验证的精度也较高并优于“OLI模型”,从而证明了新方法的可靠性及有效性。该研究为主、被动遥感技术在中小尺度上协同反演森林参数提供了实验基础,也为基于全覆盖免费OLI多光谱数据及条带LiDAR数据的低成本森林生物量制图探索了切实可行的方法。本研究所获取的地面生物量信息是基于实测的样地内单木树高和胸径,并结合前人文献提供的临近研究区异速生长模型计算得到的,即生物量本身并非实测,从而给建模引入了误差,也影响了精度验证的效果。日后可尝试采用地基LiDAR获取样地内的单木“精细”森林结构信息,并通过各组分(如树干,树枝,树叶)直接建模方法精确获取地上生物量。同时,日后可以借鉴本研究所探索的技术路线对更多森林类型(如热带、温带和寒带森林)进行建模,并借助多个森林类型中的样地及覆盖这些区域的遥感数据探寻在更大尺度上适用性更广的通用生物量反演模型。
附表 1 LiDAR特征变量汇总
Appendix 1 Summary of metrics computed from LiDAR
| 特征变量 | 变量描述 |
| 高度百分位数h25,h50,h60,h75,h95 | 激光雷达植被第一回波返回点的百分位数高度(25th,50th,60th,75th,95th) |
| 高度变量hmin | 激光雷达树高的最小值 |
| 高度变量hmean | 激光雷达树高的平均值 |
| 高度变量hmax | 激光雷达树高的最大值 |
| 高度变量hcv | 激光雷达树高的高度变异系数 |
附表 2 OLI特征变量汇总
Appendix 2 Summary of metrics computed from OLI multispectral data
| 特征变量 | 变量描述 |
| ${{原始单波段}_{{{B}_{2}}-{{B}_{7}}}}$ | OLI第2-7波段(经过大气校正和几何精校正) |
| 波段组合 ${Albedo}$ | $Albedo = {B_2} + {B_3} + {B_4} + {B_5} + {B_6} + {B_7}$ |
| 波段组合 ${B_4}/Albedo$ | ${B_4}/Albedo = {B_4}/({B_2} + {B_3} + {B_4} + {B_5} + {B_6} + {B_7})$ |
| 波段组合 ${B_{{24}}}$ | ${B_{24}} = {B_2}/{B_4}$ |
| 波段组合 ${B_{{74}}}$ | ${B_{74}} = {B_7}/{B_4}$ |
| 波段组合 ${B_{{76}}}$ | ${B_{76}} = {B_7}/{B_6}$ |
| 波段组合 ${B_{{547}}} $ | ${B_{547}} = {B_5} \cdot {B_4}/{B_7}$ |
| 波段组合 ${B_{{65}}}$ | ${B_{65}} = {B_6}/{B_5}$ |
| 波段组合 ${B_{{345}}}$ | ${B_{345}} = {B_3} \cdot {B_4}/{B_5}$ |
| 波段组合 ${B_{{53}}}$ | ${B_{53}} = {B_5}/{B_3}$ |
| 波段组合 ${VIS_{{234}}}$ | $VI{S_{234}} = {B_2} + {B_3} + {B_4}$ |
| 绿度( ${TC_{{G}}}$ ) | 提取缨帽变换绿度波段 |
| 亮度( ${TC_{{B}}}$ ) | 提取缨帽变换亮度波段 |
| 湿度( ${TC_{{W}}}$ ) | 提取缨帽变换湿度波段 |
| 第一主成分( ${PC_{{1}}}$ ) | 提取主成分分析第一波段 |
| 第二主成分( ${PC_{{2}}}$ ) | 提取主成分分析第二波段 |
| 第三主成分( ${PC_{{3}}}$ ) | 提取主成分分析第三波段 |
| 最小噪声分离变换第一波段( ${MNF_{{1}}}$ ) | 提取MNF变换第一波段 |
| 最小噪声分离变换第二波段( ${MNF_{{2}}}$ ) | 提取MNF变换第二波段 |
| 最小噪声分离变换第三波段( ${MNF_{{3}}}$ ) | 提取MNF变换第三波段 |
| 最小噪声分离变换第四波段( ${MNF_{{4}}}$ ) | 提取MNF变换第四波段 |
| 增强型植被指数(EVI) | ${\rm EVI} = 2.5\left({\displaystyle\frac{{{B_5} - {B_4}}}{{{B_5} + 6{B_4} - 7.5{B_2} + 1}}} \right)$ |
| 归一化植被指数(NDVI) | ${\rm NDVI} = \left({{B_5} - {B_4}} \right)\left({{B_5} + {B_4}} \right)$ |
| 土壤调整植被指数(SAVI) | ${\rm SAVI} = \displaystyle\frac{{\left({1 + L} \right)\left({{B_5} - {B_4}} \right)}}{{{B_5} + {B_4} + L}}\left({L = 0.5} \right)$ |
| 有效叶面积指数(SLAVI) | ${\rm SLAVI} = \displaystyle\frac{{{B_5}}}{{{B_4} + {B_7}}}$ |
| 简单比值植被指数(RVI) | ${\rm RVI} = \displaystyle\frac{{{B_5}}}{{{B_4}}}$ |
| 中红外植被指数(VI3) | ${\rm VI}3 = \displaystyle\frac{{{B_5} - {B_6}}}{{{B_5} + {B_6}}}$ |
| 垂直植被指数(PVI) | ${\rm PVI} = \sqrt {{{\left({0.355{B_5} - 0.149{B_4}} \right)}^2} + {{\left({0.355{B_4} - 0.852{B_5}} \right)}^2}} $ |
| 土壤调整比值植被指(SARVI) | ${\rm SARVI} = \displaystyle\frac{{{B_5}}}{{{B_4} + 0.084726/0.96916}}$ |
| 差值植被指数(DVI) | ${\rm DVI} = {B_5} - {B_4}$ |
| 转换型植被指数(TNDVI) | ${\rm TNDVI} = \sqrt {NDVI + 0.5}$ |
| 大气阻抗植被指数(ARVI) | ${\rm ARVI} = \displaystyle\frac{{{B_5} - 2{B_4} + {B_2}}}{{{B_5} + 2{B_4} - {B_2}}}$ |
| 修正型土壤调整植被指数(MSAVI) | ${\rm MSAVI} = {B_5} + 0.5\sqrt {{{\left({{B_5} + 0.5} \right)}^2} - 2\left({{B_5} - {B_4}} \right)} $ |
| 修正型简单比值植被指数(MSR) | ${\rm MSR} = \displaystyle\frac{{{B_5}/{B_4} - 1}}{{\sqrt {{B_5}/{B_4} + 1} }}$ |
| 非线性指数( $\rm NLI$ ) | ${\rm NLI} = \displaystyle\frac{{{B_5}^2 - {B_4}}}{{{B_5}^2 + {B_4}}}$ |
| 重归一化植被指数( $\rm RDVI$ ) | ${\rm RDVI} = \displaystyle\frac{{{B_5} - {B_4}}}{{\sqrt {{B_5} + {B_4}} }}$ |
| 归一化植被指数( ${{\rm ND}_43}$ ) | ${\rm N}{{\rm D}_{43}} = \left({{B_4} - {B_3}} \right)\left({{B_4} + {B_3}} \right)$ |
| 归一化植被指数( ${{\rm ND}_67}$ ) | ${\rm N}{{\rm D}_{67}} = \left({{B_6} - {B_7}} \right)\left({{B_6} + {B_7}} \right)$ |
| 归一化植被指数( ${{\rm ND}_563}$ ) | ${\rm N}{{\rm D}_{563}} = \left({{B_5} + {B_6} - {B_3}} \right)\left({{B_5} + {B_6} + {B_3}} \right)$ |
| 相关度( ${\rm CR}$ ) | ${\rm CR} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}} \left| {\frac{{\left({i - {\rm ME}} \right)\left({j - {\rm ME}} \right)}}{{\sqrt {{\rm V}{{\rm A}_i} \cdot {\rm V}{{\rm A}_j}} }}} \right|$ |
| 相关度( ${\rm CO}$ ) | ${\rm CO} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}} $ |
| 相异性( ${\rm DI}$ ) | ${\rm DI} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}} \left| {i - j} \right|$ |
| 信息熵( ${\rm EN}$ ) | ${\rm EN} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}} \left({ - \ln {P_{i,j}}} \right)$ |
| 均匀度( ${\rm HO}$ ) | ${\rm HO} = \sum\limits_{i,j = 0}^{N - 1} {i\frac{{{P_{i,j}}}}{{1 + {{\left({i - j} \right)}^2}}}} $ |
| 均值( ${\rm ME}$ ) | ${\rm ME} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}} $ |
| 二阶矩( ${\rm SM}$ ) | ${\rm SM} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}^2} $ |
| 偏斜度( ${\rm SK}$ ) | ${\rm SK} = \frac{{\left| {\sum {{{\left({{P_{i,j}} - {\rm ME}} \right)}^2}} } \right|}}{{\left({{N^2} - 1} \right){{\left({{\rm VA}} \right)}^{\frac{3}{2}}}}}$ |
| 方差( ${\rm VA}$ ) | ${\rm VA} = \sum\limits_{i,j = 0}^{N - 1} {i \cdot {P_{i,j}}{{\left({i - {\rm ME}} \right)}^2}} $ |
| 注:表中Bi表示高光谱第i波段数据;其中 ${{P}_{i\text{,}j}}={{D}_{i\text{,}j}}\text{ }/\sum\limits_{i\text{,}j=0}^{N-1}{{{D}_{i\text{,}j}}}\text{ }$ ,Dij表示第i行和第j列位置上的像元亮度值,N表示计算纹理特征时窗口的大小或尺寸。 | |
附表 3 中文用于计算研究区各主要树种组分生物量的异速生长模型标题
Appendix 3 Allometric equations for aboveground biomass components of dominant tree species in the study site
| 树种 | 组分 | a | b |
| 马尾松(Pinus massoniana) | 树干生物量(Ws) | 0.141 | 1.092 |
| 树枝生物量(Wb) | 0.065 | 0.991 | |
| 叶生物量(Wf) | 0.132 | 0.745 | |
| 根生物量(Wr) | 0.005 | 0.890 | |
| 杉木(Cunninghamia lanceolata) | 树干生物量(Ws) | 0.124 | 0.680 |
| 树枝生物量(Wb) | 0.203 | 0.385 | |
| 叶生物量(Wf) | 0.850 | 0.189 | |
| 根生物量(Wf) | 0.337 | 0.418 | |
| 湿地松(Quercus acutissima) | 树干生物量(Ws) | 0.235 | 0.900 |
| 树枝生物量(Wb) | 0.080 | 1.064 | |
| 叶生物量(Wf) | 0.456 | 0.610 | |
| 根生物量(Wr) | 0.007 | 1.014 | |
| 麻栎(Quercus acutissima) | 树干生物量(Ws) | 0.018 | 1.034 |
| 树枝生物量(Wb) | 0.00008 | 1.468 | |
| 叶生物量(Wf) | 0.004 | 0.769 | |
| 根生物量(Wr) | 0.020 | 0.882 | |
| 枫香(Liquidambar formosana) | 树干生物量(Ws) | 0.093 | 0.801 |
| 树枝生物量(Wb) | 0.083 | 0.649 | |
| 叶生物量(Wf) | 1.084 | 0.217 | |
| 根生物量(Wr) | 0.055 | 0.107 | |
| 注: 模型的格式为 $W = a{({D^2}H)^{\rm b}}$ . H=树高(m),D=胸径(cm); a,b 为模型参数. | |||
参考文献(References)
-
Cao L, Coops N C, Innes J, Dai J S, She G H.2014.Mapping above-and below-ground biomass components in subtropical forests using small-footprint LiDAR. Forests, 5 (6) : 1356–1373 . [DOI:10.3390/f5061356]
-
Cao L, Dai J S, Xu J X, Xu Z Q, She G H.2014.Optimized extraction of forest parameters in subtropical forests based on airborne small footprint LiDAR technology. Journal of Beijing Forestry University, 36 (5) : 13–21 .
-
( 曹林, 代劲松, 徐建新, 许子乾, 佘光辉. 2014. 基于机载小光斑LiDAR技术的亚热带森林参数信息优化提取. 北京林业大学学报, 36 (5) : 13–21. )
-
Dong L X. 2008. Estimation of Forest Canopy Height and Biomass in Three Gorges Reservoir Area Based on Multi-Source Remote Sensing Data. Beijing:Institute of Remote Sensing Applications
-
-
Dubayah R O, Drake J B.2000.Lidar remote sensing for forestry. Journal of Forestry, 98 (6) : 44–46 .
-
Feng Z W, Wang X K, Wu G. The Forest Ecosystem Biomass and Productivity in China. Beijing: Science Press 1999 : 99 -187.
-
( 冯宗炜, 王效科, 吴刚. 1999. 中国森林生态系统的生物量和生产力. 北京: 科学出版社 : 99 -187. )
-
Fu T, Pang Y, Huang Q F, Liu Q W, Xu G C.2011.Prediction of subtropical forest parameters using airborne laser scanner. Journal of Remote Sensing, 15 (5) : 1092–1104 .
-
( 付甜, 庞勇, 黄庆丰, 刘清旺, 徐光彩. 2011. 亚热带森林参数的机载激光雷达估测. 遥感学报, 15 (5) : 1092–1104. )
-
Guo Z F, Chi H, Sun G Q.2010.Estimating forest aboveground biomass using HJ-1 satellite CCD and ICESat GLAS waveform data. Science China Earth Sciences, 53 (1) : 16–25 . [DOI:10.1007/s11430-010-4128-3]
-
Huang K B, Pang Y, Shu Q T, Fu T.2013.Aboveground forest biomass estimation using ICESat GLAS in Yunnan. China, 17 (1) : 165–179 .
-
( 黄克标, 庞勇, 舒清态, 付甜. 2013. 基于ICESat GLAS的云南省森林地上生物量反演. 遥感学报, 17 (1) : 165–179. )
-
Jiang G Z, Han B, Gao Y B, Yang C J.2013.Review of 40-year earth observation with Landsat series and prospects of LDCM. Journal of Remote Sensing, 17 (5) : 1033–1048 .
-
( 姜高珍, 韩冰, 高应波, 杨崇俊. 2013. Landsat系列卫星对地观测40LDCM前瞻. 遥感学报, 17 (5) : 1033–1048. )
-
Lefsky M A, Cohen W B, Parker G G, Harding D J.2002.Lidar remote sensing for ecosystem studies. BioScience, 52 (1) : 19–30 . [DOI:10.1641/0006-3568(2002)052[0019:LRSFES]2.0.CO;2]
-
Lim K, Treitz P, Wulder M, St-Onge B, Flood M.2003.LiDAR remote sensing of forest structure. Progress in Physical Geography, 27 (1) : 88–106 . [DOI:10.1191/0309133303pp360ra]
-
Liu L J. 2011. Forest Parameters Inversion Using Airborne LiDAR and Hyperspectral Data fusion. Harbin:Northeast Forestry University
-
-
Ma L Q, Li A N.2011.Review of application of LiDAR to estimation of forest vertical structure parameters. World Forestry Research, 24 (1) : 41–45 .
-
( 马利群, 李爱农. 2011. 激光雷达在森林垂直结构参数估算中的应用. 世界林业研究, 24 (1) : 41–45. )
-
Means J E, Acker S A, Harding D J, Blair J B, Lefsky M A, Cohen W B, Harmon M E, McKee W A.1999.Use of large-footprint scanning airborne lidar to estimate forest stand characteristics in the western cascades of Oregon. Remote Sensing of Environment, 67 (3) : 298–308 . [DOI:10.1016/S0034-4257(98)00091-1]
-
Næsset E.2002.Predicting forest stand characteristics with airborne scanning laser using a practical two-stage procedure and field data. Remote Sensing of Environment, 80 (1) : 88–99 . [DOI:10.1016/S0034-4257(01)00290-5]
-
Næsset E, Gobakken T.2008.Estimation of above-and belowground biomass across regions of the boreal forest zone using airborne laser. Remote Sensing of Environment, 112 (6) : 3079–3090 . [DOI:10.1016/j.rse.2008.03.004]
-
Pang Y, Huang K B, Li Z Y, Tian X L and Chen E X. 2011. Forest aboveground biomass analysis using remote sensing in the Greater Mekong Subregion, 33(10):1863-1869
-
( 庞勇, 黄克标, 李增元, 覃先林, 陈尔学. 2011. 基于遥感的湄公河次区域森林地上生物量分析. 资源科学, 33 (10) : 1863–1869. )
-
Pang Y, Li Z Y, Chen E X, Sun G Q.2005.Lidar remote sensing technology and its application in forestry. Scientia Silvae Sinicae, 41 (3) : 129–136 .
-
( 庞勇, 李增元, 陈尔学, 孙国清. 2005. 激光雷达技术及其在林业上的应用. 林业科学, 41 (3) : 129–136. )
-
Pang Y, Li Z Y, Ju H B, Liu Q W, Si L, Li S M, Tan B X, Zhong K T, Lu H, Dian Y Y, Liu L X and Jia W. 2013. LiCHy:CAF's LiDAR, CCD and Hyperspectral airborne observation system//Proceeding of SILVILASER 2013. Beijing:Chinese Academy of Forestry:45-54
-
Popescu S C, Wynne R H, Scrivani J A.2004.Fusion of small-footprint Lidar and multispectral data to estimate plot-level volume and biomass in deciduous and pine forests in Virginia, USA. Forest Science, 50 (4) : 551–565 .
-
Post W M, Emanuel W R, Zinke P J, Stangenberger A G.1982.Soil carbon pools and world life zones. Nature, 298 (5870) : 156–159 . [DOI:10.1038/298156a0]
-
Wulder M A, Coops N C.2014.Make Earth observations open access. Nature, 513 (7516) : 30–31 . [DOI:10.1038/513030a]
-
Xu H Q, Tang F.2013.Analysis of new characteristics of the first landsat 8 image and their eco-environmental significance. Acta Ecologica Sinica, 33 (11) : 3249–3257 .
-
( 徐涵秋, 唐菲. 2013. 新一代Landsat系列卫星:Landsat 8遥感影像新增特征及其生态环境意义. 生态学报, 33 (11) : 3249–3257. [DOI:10.5846/stxb2013030912] )
-
Xu T, Cao L, She G H.2015.Feature extraction and forest biomass estimation based on Landsat 8 OLI. Remote Sensing Technology and Application, 30 (2) : 226–234 .
-
( 徐婷, 曹林, 佘光辉. 2015. 基于Landsat 8 OLI的特征变量优化提取及森林生物量反演. 遥感技术与应用, 30 (2) : 226–234. [DOI:10.11873/j.issn.1004-0323.2015.2.0226] )