A small polyp objects network integrating boundary attention features
-
摘要: 从结肠图像中分割息肉小目标病变区域对于预防结直肠癌至关重要,它可以为结直肠癌的诊断提供有价值的信息。然而目前现有的方法存在2个局限性:一是不能稳健捕获全局上下文信息,二是未能充分挖掘细粒度细节特征信息。因此,提出融合边界注意力的特征挖掘息肉小目标网络(transformer feature boundary network,TFB-Net)。该网络主要包括3个核心模块:首先,采用Transformer辅助编码器建立长程依赖关系,补充全局信息;其次,设计特征挖掘模块进一步细化特征,学习到更好的特征;最后,使用边界反转注意力模块加强对边界语义空间的关注,提高区域辨别能力。在5个息肉小目标数据集上进行广泛实验,实验结果表明TFB-Net具有优越的分割性能。Abstract: Segmentation of small target lesion areas, such as polyps in colon images, is essential for the prevention and diagnosis of colorectal cancer. However, existing methods face two main limitations: either the global context information cannot be captured robustly or the fine-grained detail information cannot be fully mined. To address these issues, this study proposes TFB-Net, a feature mining network for small target polyps that integrates boundary attention. The network consists of three core modules: First, a Transformer is used to establish long-term dependencies and supplement global information. Second, the feature mining module is designed to further optimize and enhance the learned features. Finally, the boundary inversion attention module strengthens attention to the boundary semantic space, which consequently improves regional discrimination. Extensive experiments were conducted on five small polyp target datasets, and the results show that TFB-Net achieves superior segmentation performance.
-
结直肠癌作为世界第三大常见癌症,已经严重威胁人们的健康甚至生命[1]。研究表明,结直肠癌发现于早期,死亡率仅为10%[2]。在结直肠癌病变初期,息肉通常体积较小,并且形状各异,这大大增加了诊断难度。目前,结肠镜检查是筛查和预防结直肠癌的主要技术。然而,这种方法不仅耗费人力成本,而且微小息肉很容易被漏检。因此,实现高效、准确的息肉小目标自动分割具有重要的临床意义[3]。
与传统分割方法相比,基于编码器-解码器的深度学习方法[4-5]可达到更高的分割精度。U-Net[6]作为首个编码器−解码器结构分割网络,已成功应用于生物医学图像分割领域。随后出现的UNet++[7]通过集成不同深度U-Net和一系列嵌套、密集跳跃连接,缩小收缩路径和扩展路径特征图间语义差距,从而灵活高效聚合不同语义尺度特征。而UNet3+[8]应用全尺度跳跃连接和深度监督策略,模型分割性能得到极大提高。随后相继提出ResUNet(road extraction by deep residual U-net)[9]以及ResUNet++[10]等,通过不断促进特征有效融合,这些网络进一步提高息肉分割精度,在许多医学图像分割任务中表现出良好性能。然而,这些方法捕获丰富全局上下文信息和细节信息的能力不足。
为捕获全局上下文信息,使用扩大感受野策略和Transformer模型2种优化方案。DilatedSegNet(dilated segmentation network)[11]通过将膨胀卷积模块的输出进行连接,分割息肉目标。MKDCNet(multiple kernel dilated convolution network)[12]利用多核膨胀卷积块扩展感受野,学习更稳定和异构的特征表示。HSNet(hybrid semantic network)[13]以金字塔Transformer为编码器,通过交互机制来交换2种类型的语义信息,缩小高级和低级特征间差距;PVT(pyramid vision tranformer)[14]以Transformer为编码器,不仅能够有效抑制噪声,还能显著增强特征表达能力。此外,为进一步增强特征提取能力,MIA-Net(multi-information aggregation network)[15]利用Transformer和CNNs(convolutional neural networks)双编码器架构,分别提取全局和局部特征;FAT-Net(feature adaptive transformers)[16]作为特征自适应Transformer网络,为有效捕获长程依赖关系,集成一个额外的Transformer分支;Transfuse(fusing transformers)[17]结合CNNs和Transformer,有效捕获全局信息和低级空间细节特征;为捕获丰富全局上下文信息,MNFE-Net (multiscale nonlocal feature extraction network)[18]基于Transformer和CNNs提出并行编码器结构,提高网络分割性能。以上4种模型均是以Transformer和CNNs作为双分支对输入图像进行编码。
为捕获更多细节特征信息,注意力机制在医学图像分割任务中的重要性已经凸显。ACSNet(adaptive context selection )[19]提出局部上下文注意力(local context attention, LCA)模块,进一步加强对复杂区域关注,补充局部细节信息;为捕获更多细节特征,PraNet(parallel reverse attention network)[20]采用反转注意力(reverse attention, RA)模块对目标区域进行反转,逐步挖掘息肉区域信息;但在息肉小目标分割任务中,获取的全局信息和局部信息仍不够全面。为缓解以上问题,CaraNet(context axial reverse attention network)[21]提出上下文轴向反转注意力网络来提高息肉小目标分割性能。该网络使用轻量级通道特征金字塔模块提取丰富特征信息;为获取更准确特征信息,设计轴向反转注意力模块对位置信息进行分析。为进一步提高息肉小目标分割性能,充分考虑卷积核较小的卷积运算和膨胀卷积的优势,并结合注意力机制,这能够获得息肉小目标更丰富的全局上下文信息和细粒度局部细节特征。
结合膨胀卷积和注意力机制虽然能够有效提高网络分割性能,但对于息肉小目标分割任务,仍存在漏检和误检问题,导致模型分割性能不理想。针对上述问题,本文提出融合Transformer辅助编码器和边界反转注意力的特征挖掘息肉小目标分割网络(transformer feature boundary network, TFB-Net),来促进结肠镜图像中息肉小目标分割。其中Transformer辅助编码器(Transformer auxiliary encoder, TAE)捕获输入图像全局上下文信息,利用自注意力机制建立像素间长程依赖关系,弥补CNNs中固有局限性;特征挖掘模块(feature mining module, FMM)将每层融合的特征进行深层次挖掘,进一步细化特征,学习特征表示;边界反转注意力(boundary reverse attention, BRA)模块用于加强对边缘关注,有效抑制不相关噪声,提升区域辨别能力。在5个可用息肉小目标数据集上对TFB-Net进行评估,大量的实验结果表明所提出的TFB-Net显著提高息肉小目标分割精度。综上所述,本文的贡献可归纳如下:
1)提出一种新的融合Transformer的分割网络推进结肠息肉小目标分割,称为TFB-Net。TFB-Net利用Transformer辅助CNNs编码器进行特征提取。该网络可以补充CNNs编码器底部3层缺少的全局特征,从而提高息肉小目标分割精度。
2)利用特征挖掘模块替代传统跳跃连接,恢复上采样过程中的细节信息,以不同感受野提取详细特征,针对不同层级采用不同特征挖掘模块,有效缩小编码器和解码器间语义鸿沟。
3)采用边界反转注意力模块加强对特征边界区域关注,有效抑制不相关噪声,增强边界信息处理能力,提高特征整体鲁棒性。
4)在5个息肉小目标数据集上进行广泛实验,相比于10种先进分割网络,实验结果表明TFB-Net在息肉小目标分割上具有优越的分割性能,特别在ETIS-LaribPolyPDB*数据集上,该网络在Dice和IoU指标上分别超过CaraNet网络2.35百分点和3.91百分点。
1. 相关工作
Transformer最初专注于自然语言处理(natural language processing, NLP)领域,由于自注意力机制在建模长程依赖关系的优异表现,Transformer已经广泛应用在各种计算机视觉任务中。视觉Transformer (vision Transformer, ViT)[22]首次将Transformer应用于图像分类任务,相比于卷积网络,通过应用Transformer获得令人满意的分类效果;语义分割Transformer (semantic segmentation Transformer, SETR)[23]利用Transformer作为编码器,将输入图像编码为图像序列块,提取丰富全局上下文特征,获得较好的语义分割结果;数据高效图像Transformer (data-efficient image Transformers, DeiT)[24]引入老师−学生策略进一步提高精度。
为学习更稳定的特征表示,PVT[14]将金字塔结构Transformer作为编码器,不仅减少整体计算量,而且获得较高输出分辨率。PVT分割网络继承Transformer和CNNs优点,有效抑制图像中噪声,显著提高特征表达能力。相较于PVT,Transfuse[17]在PVT基础上添加一条CNNs分支,以并行分支结构结合CNNs和Transformer,有效捕获全局上下文信息和低级空间细节特征;MGW-Net[25]采用可以捕捉多尺度信息的PVT编码器和实现不同层之间多尺度信息交互的并行多级特征融合解码器组成,进一步改善了分割性能,从而实现更高的分割精度。
为了充分利用自注意力机制和分层结构的优势,考虑在息肉小目标分割任务中使用金字塔Transformer。不同于以往单一Transformer编码器和并行双分支编码器,考虑将金字塔Transformer作为辅助编码器,将其底部3层特征分别和CNNs中同级特征进行有效融合,增强特征稳健性,实现更好分割效果。
2. 网络模型
本文提出融合Transformer和边界反转注意力的特征挖掘息肉小目标分割网络,称为TFB-Net,其网络结构整体框架如图1所示。从图1中可得出TFB-Net网络结构主要包括3个核心模块,包括Transformer辅助编码器、特征挖掘模块以及边界反转注意力模块。Transformer辅助编码器弥补CNNs编码器固有局限性,建立输入图像像素间长程依赖关系,补充全局特征信息;设计特征挖掘模块代替跳跃连接,融合不同尺度卷积和膨胀卷积操作生成更精细的特征图,进一步细化全局语义空间特征;在此基础上,利用边界反转注意力模块加强对边界区域关注,获取更多边界细节信息,减少特征噪声,使特征图更具稳健性。下面将逐一介绍各个模块。
2.1 Transformer辅助编码器
相比于卷积神经网络,Transformer通过建模长程依赖关系捕获全局上下文信息的极好表现使其频繁出现在各种计算机视觉任务中。因此,本文考虑引入一个额外的Transformer作为辅助编码器与CNNs分支进行分层连接,将局部特征信息和全局上下文信息相融合。此外,由于PVT网络在多个密集检测任务中实现最先进的性能,通过空间缩减序列有效降低计算负担,所以TFB-Net采用预训练PVT对全局特征进行分层编码,得到由粗到细的4层金字塔特征。
首先,将输入图像
$ {\boldsymbol{X}} \in {{\bf{R}}^{H \times W \times 3}} $ 平均分成$ N = \dfrac{H}{P} \times \dfrac{W}{P} $ ,其中P是每个图像块大小,N是图像块总数,为匹配Transformer层的输入,通过对这些二维图像块应用线性映射函数转化为一维嵌入图像序列$ {{\boldsymbol{X}}_e} \in {{\bf{R}}^{N \times {C_e}}} $ ,输出维度为$ {C_e} $ 。为保留图像原本的空间位置信息,将其对应的相同维度可学习位置嵌入信息添加到生成的图像序列$ {{\boldsymbol{X}}_e} \in {{\bf{R}}^{N \times {C_e}}} $ 中。最后将生成的图像嵌入$ {{\boldsymbol{X}}_l} \in {{\bf{R}}^{N \times {C_e}}} $ 输入到Transformer块中,Transformer模块结构如图2所示。1个经典Transformer块通常包括3个关键部分:多头自注意力、多层感知机和层归一化。其中高效的自注意力机制作为Transformer块的核心,通过在每个层中聚合全局信息并行更新每个图像嵌入块状态。由生成的输出图像块嵌入
$ {{\boldsymbol{X}}_l} $ 或者前一个Transformer输出$ {{\boldsymbol{X}}_m} $ 计算得到图像块的3个序列向量,包括查询向量Q、键向量K和值向量V。然后,通过对Q和K进行点积运算来计算注意力得分。此外,为确保所有值均为正数且相加为数值1,对生成的向量应用Softmax激活函数来归一化得分。最后,将每个V与归一化得分相乘获得输出向量,并对加权值V求和。自注意力的整个计算过程为$$ {\boldsymbol{Q}} = {{\boldsymbol{X}}_m} * {{\boldsymbol{W}}_{\boldsymbol{Q}}},{\boldsymbol{K}} = {{\boldsymbol{X}}_m} * {{\boldsymbol{W}}_{\boldsymbol{K}}},{\boldsymbol{V}} = {{\boldsymbol{X}}_m} * {{\boldsymbol{W}}_{\boldsymbol{V}}} $$ $$ \mathrm{SA}(\boldsymbol{X})=\mathrm{Softmax}\left(\frac{\boldsymbol{Q}\boldsymbol{K}^{\text{T}}}{\sqrt{d_k}}\right)\boldsymbol{V} $$ 式中:
$ {{\boldsymbol{W}}_{\boldsymbol{Q}}} \in {{\bf{R}}^{{C_e} \times {d_q}}} $ 、$ {{\boldsymbol{W}}_{\boldsymbol{K}}} \in {{\bf{R}}^{{C_e} \times {d_k}}} $ 和$ {{\boldsymbol{W}}_{\boldsymbol{V}}} \in {{\bf{R}}^{{C_e} \times {d_v}}} $ 是3个参数可学习的Q、K以及V。多头自注意力具有h个独立自注意层,将Q、K和V映射到h个头上,并投射到原始高维空间的不同子空间以并行方式进行自注意力计算。最后,将不同子空间中用于计算自注意得分的多个头进行拼接。
从TAE中可得到4个不同层次语义特征
$\{ {{\boldsymbol{T}}_i},i = 1,2,3,4\}$ ,并将其底部3层特征进行$ 1 \times 1 $ 和$ 3 \times 3 $ 卷积操作处理。进而与CNNs中底部3层特征$\{ {{\boldsymbol{X}}_i},i = 2,3,4\}$ 按通道进行连接分别得到$ \{ {{\boldsymbol{C}}_i},i = 1,2,3\} $ ,此时,TAE辅助CNNs编码器在底部3层跳跃连接上获得全局上下文信息。2.2 特征挖掘模块
在通道维度上直接融合TAE和CNNs编码器特征可能会导致输出特征不够细化。受跳跃连接启发,本文设计了特征挖掘模块(FMM),将前期提取的特征
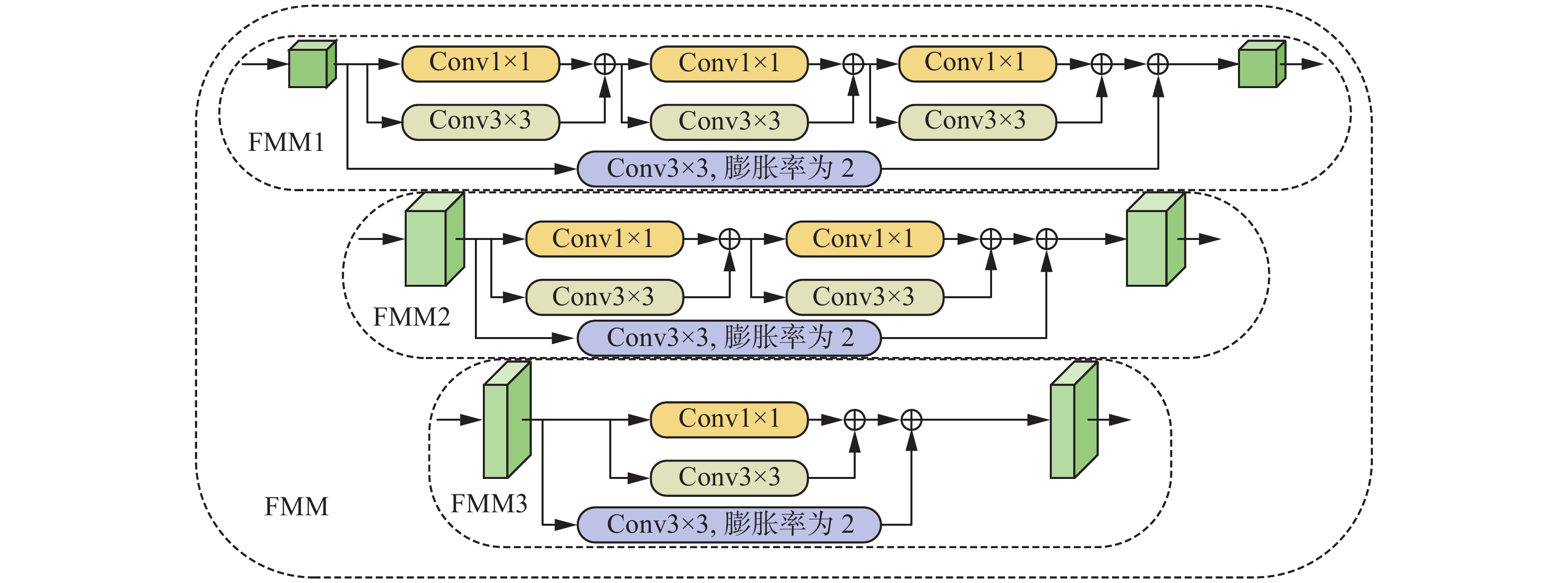
$ \{ {{\boldsymbol{C}}_i},i = 1,2,3\} $ 通过FMM进行优化并传递到对应的上采样过程中,挖掘全局语义空间特征,自适应匹配编码器和解码器之间的特征分布,促进特征融合。特征挖掘模块具体结构如图3所示。针对息肉小目标分割问题,膨胀率不宜设置过大,故设置膨胀率为2。针对不同层的特征表示具有差异性,在FMM模块中分别考虑3种方式以进一步精细化全局特征,进而学习更好特征表示,其中包括FMM1、FMM2以及FMM3。将一个核大小为
$3 \times 3$ 的卷积操作和另一个核大小为$1 \times 1$ 的卷积操作按元素进行相加,并将其设定为Sub-FMM。由于编码器第2层特征图具有相对低级的特征,在FMM3中设置1个Sub-FMM来更好匹配编码器和解码器间特征分布。而编码器第3层和第4层特征图相对比较高级,因此,在FMM2和FMM1中分别设置2个和3个Sub-FMM,缩小编码器和解码器间特征间隙。前期融合的3层全局特征作为FMM模块的输入,经不同卷积计算和融合操作,可以进一步从原始全局特征中提取更详细特征。同时,在FMM1、FMM2以及FMM3的输入特征处分别应用1个核大小为
$3 \times 3$ 且膨胀率为2的卷积操作,通过按元素相加的方式分别与上述输出特征进行融合操作。2.3 边界反转注意力模块
由于息肉小目标图像同样呈边缘模糊、成像亮度不一以及背景复杂等特点,受注意力机制启发,为提高特征鲁棒性,本文部署边界反转注意力(BRA)模块,有效增强边界细节信息,突出模型的抗噪能力。在上采样过程中,经BRA模块处理可以极大地恢复上采样过程中丢失细节,缩小编码器和解码器间语义鸿沟。边界反转注意力模块如图4所示。
通过将输入特征分别与边缘注意力特征图和反转注意力特征图按通道执行相乘操作。将获得的2种注意力特征按通道维度进行拼接,并输入到卷积层,使通道变为原来的1/2,接着与原输入特征执行按像素相加操作,这将有效提高区域辨别能力,从而实现更准确分割。从图1中可得到,通过将
$ \{ {{\boldsymbol{F}}_i},i = 1,2,3\} $ 作为3个BRA模块的输入,最终得到3个输出特征$ \{ {{\boldsymbol{B}}_i},i = 1,2,3\} $ 。2.4 损失函数
为缓解过拟合现象,获得更好性能,在损失函数中应用加权IoU(intersection over union)损失和加权二值交叉熵(binary cross-entropy, BCE)损失:
$$ L = L_{{\text{IoU}}}^{\text{W}} + L_{{\text{BCE}}}^{\text{W}} $$ IoU[26]损失在处理边界上的突出表现已在许多挑战性医学图像分割任务中得到验证。此外,息肉小目标分割为二分类问题,而二值交叉熵损失主要适用于二分类图像分割任务。
为充分训练底层特征,在经CNNs编码器底部3层特征聚合得到的全局引导特征图和上采样过程生成的3层特征图中应用深监督策略,所有预测特征图上采样到与G(ground truth)相同分辨率大小。因此,最终训练损失计算公式为
$$ {L_{{\text{total}}}} = \sum\limits_{i = 1}^3 {L({\boldsymbol{G}},{\boldsymbol{f}}_i^{{\text{up}}}} ) + L({\boldsymbol{G}},{\boldsymbol{f}}) $$ 3. 实验与结果分析
3.1 数据集
本文利用5个息肉小目标数据集(CVC-300*、CVC-ClinicDB*、Kvasir*、CVC-ColonDB*以及ETIS-LaribPolyPDB*)来验证TFB-Net的性能,具体如表1所示。为便于区分,在5个公共息肉数据集(CVC-300[27]、CVC-ClinicDB[28]、Kvasir[29]、CVC-ColonDB[30]和ETIS-LaribPolyPDB[31])名称后统一添加符号“*”分别代表5个息肉小目标数据集。
表 1 实验数据集Table 1 Experimental datasets数据集 总数量 训练集数量 测试集数量 CVC-300* 35 — 35 CVC-ClinicDB* 121 102 19 Kvasir* 77 67 10 CVC-ColonDB* 158 — 158 ETIS-LaribPolyPDB* 116 — 116 3.2 实现细节
在所有实验中,将所有图片大小统一调整为352像素×352像素。采用Res2Net[32]作为主干编码器,使网络具有强大的多尺度表示能力。本文训练TFB-Net 网络100个epoch,batch size设置为8,部署系统环境为Ubuntu 20.04、Python 3.9以及PyTorch 1.11,实验统一在单张NVIDIA GeForce GTX 3090 GPU上进行。
3.3 评价指标
为定量评价TFB-Net在息肉小目标数据集上分割性能,使用5个广泛使用的分割指标,包括Dice相似系数(dice similarity coefficient, Dice)、交并比(intersection over union, IoU)、灵敏度(sensitivity, SE)、特异性(specificity, SP)以及准确率(accuracy, ACC)。Dice系数通常用来计算2个样本之间的相似度,交并比通常用来表示预测特征图和Ground Truth之间的重叠率。计算方法分别为
$$ {I_{{\text{Dice}}}} = \frac{{2 \cdot {N_{\rm TP}}}}{{2 \cdot {N_{\rm TP}} + {N_{\rm FP}} + {N_{\rm FN}}}} $$ $$ {I_{{\text{oU}}}} = \frac{{{N_{\rm TP}}}}{{{N_{\rm TP}} + {N_{\rm FP}} + {N_{\rm FN}}}} $$ $$ {I_{{\text{SE}}}} = \frac{{{N_{\rm TP}}}}{{{N_{\rm TP}} + {N_{\rm FN}}}} $$ $$ {I_{{\text{SP}}}} = \frac{{{N_{\rm TN}}}}{{{N_{\rm TN}} + {N_{\rm FP}}}} $$ $$ {I_{{\text{Acc}}}} = \frac{{{N_{\rm TP}} + {N_{F\rm N}}}}{{{N_{\rm TP}} + {N_{\rm TN}} + {N_{\rm FP}} + {N_{\rm FN}}}} $$ 式中:NTP和NTN分别表示正确分割息肉小目标像素的数目,NFP代表将背景像素错误分割成息肉小目标的像素个数,NFN表示将息肉小目标像素错误分割为背景像素的个数。
3.4 消融研究
通过将Transformer辅助编码器、特征挖掘模块以及边界反转注意力模块按照不同组合方式替换到基准网络,分别实现了Baseline+TAE、Baseline+FMM、Baseline+BRA、Baseline+TAE+FMM、Baseline+TAE+BRA、Baseline+FMM+BRA6种方法,在5个息肉小目标数据集上进行测试。实验结果如表2所示。可视化分割效果图如图5所示,图5(a)表示输入图像,图5(b)表示Ground Truth,图5(c)表示基准网络,图5(d)~(i)依次代表上述6种分割方法,图5(j)表示TFB-Net。
表 2 消融研究结果Table 2 Ablation study results% 方法 CVC-300* CVC-ClinicDB* Kvasir* CVC-ColonDB* ETIS-LaribPolyPDB* Dice IoU Dice IoU Dice IoU Dice IoU Dice IoU Baseline 81.50 72.35 77.72 68.93 78.77 70.03 71.16 61.34 68.08 58.12 Baseline+TAE 86.15 77.70 83.56 75.22 84.06 75.84 74.30 65.62 72.96 63.77 Baseline+FMM 86.42 78.41 83.44 75.89 83.67 76.01 74.81 66.25 72.60 64.10 Baseline+BRA 85.39 77.43 80.15 72.84 81.08 73.82 74.96 66.56 71.65 63.20 Baseline+TAE+FMM 86.34 77.77 83.42 75.53 84.04 76.24 76.95 67.97 72.85 64.44 Baseline+TAE+BRA 87.05 79.90 84.27 77.26 84.94 77.90 75.52 67.67 71.88 64.32 Baseline+FMM+BRA 86.11 79.18 83.81 76.87 84.26 77.28 75.81 67.73 73.68 65.89 Baseline+TAE+FMM+BRA 88.80 81.16 87.15 79.52 87.18 79.58 78.72 70.06 76.15 67.26 从表2消融研究结果中可看出TFB-Net明显优于上述6种消融分割方法。从图5消融研究分割效果图可清晰看出TFB-Net的分割效果最佳。
单独模块有效性 将预训练金字塔Transformer作为辅助,与Res2Net共同构成编码器。从表2可以看出,添加TAE后网络性能在5个息肉小目标数据集上均有明显的提高。由于transformer更加关注全局信息,能够更准确地定位小目标息肉的位置。之后通过反转突出目标的边缘细节。在基线中添加FMM后,网络模型在CVC-ClinicDB*数据集上Dice和IoU分别提高了5.72百分点和6.96百分点。在基线中添加BRA后,其分割性能在CVC-300*数据集上Dice和IoU分别提高了3.89百分点和5.08百分点。
组合模块有效性 将FMM模块添加到Baseline+TAE中,由表2可得到在CVC-ColonDB*数据集中,Baseline+TAE+FMM比Baseline+TAE具有更好分割性能,其Dice和IoU分别提高2.65百分点和2.35百分点。同样在CVC-ColonDB*数据集中,Baseline+TAE+FMM比Baseline+FMM分割精度更高,Dice和IoU分别提高2.14百分点和1.72百分点。由表2 知相对于Baseline+TAE,Baseline+TAE+BRA在CVC-ColonDB*数据集上性能得到提升,其Dice和IoU分别提高了1.22百分点和2.05百分点。相比于Baseline+BRA,Baseline+TAE+BRA在Clinic-DB*数据集上Dice和IoU分别提高4.12百分点和4.42百分点。通过图5可直观地看出,其在CVC-300*、CVC-ClinicDB*以及Kvasir*数据集上性能表现优于Baseline+TAE和Baseline+BRA。通过表2数据对比Baseline+FMM,可发现Baseline+FMM+BRA在ETIS-LaribPolyPDB*数据集上Dice和IoU分别提高了1.08百分点和1.79百分点。此外,在Clinic-DB*数据集上,其Dice和IoU相比Baseline+BRA分别提高了3.66百分点和4.03百分点。
3.5 对比实验
将TFB-Net和10个先进网络模型进行对比,包括U-Net[6]、UNet++[7]、UNet3+[8]、PVT[14]、FAT-Net[16]、Transfuse[17]、MNFE-Net[18]、ACSNet[19]、PraNet[20]以及CaraNet[21]。对比实验结果如表3所示。其可视化分割结果如图6所示,其中图6(a)代表输入图像,图6(b)表示Ground Truth,图6(c)~(l)依次代表U-Net、UNet++、UNet3+、Transfuse、PVT、FAT-Net、MNFE-Net、ACSNet、PraNet以及CaraNet,图6(m)表示TFB-Net。绿色和红色轮廓曲线分别代表Ground Truth和分割结果。
表 3 对比实验结果Table 3 Comparative experimental results% 数据集 指标 U-Net UNet++ UNet3+ Transfuse PVT FAT-Net MNFE-Net ACSNet PraNet CaraNet TFB-Net
CVC-300*Dice 53.46 70.93 72.51 84.58 75.59 79.82 85.04 84.74 86.72 84.62 88.80 IoU 44.97 62.23 63.41 75.57 65.13 70.22 77.75 78.05 77.89 74.87 81.16 ACC 98.81 99.17 99.26 99.39 98.98 99.38 99.31 99.44 99.44 99.35 99.56 SE 47.67 72.98 68.78 96.37 85.58 80.41 97.07 93.46 97.57 97.83 96.81 SP 99.93 99.65 99.86 99.44 99.27 99.72 99.36 99.59 99.47 99.36 99.62 Clinic-DB* Dice 61.35 65.37 64.67 68.94 72.43 64.80 75.83 72.55 75.23 77.69 87.15 IoU 56.59 59.62 58.66 61.62 63.42 58.30 67.20 66.21 67.52 68.42 79.52 ACC 99.21 99.24 99.19 98.88 98.98 99.19 98.86 99.34 99.23 99.23 99.53 SE 59.40 63.01 63.18 74.51 79.37 62.57 87.51 77.15 80.48 87.80 93.86 SP 99.92 99.85 99.81 99.32 99.34 99.81 99.13 99.73 99.63 99.41 99.66 Kvasir* Dice 63.58 61.03 64.98 78.81 72.03 59.52 80.14 83.45 78.73 78.74 87.18 IoU 55.54 54.99 56.72 71.97 63.09 52.74 73.52 75.47 71.41 69.47 79.58 ACC 98.97 98.66 99.05 99.17 98.98 99.01 99.25 99.36 99.20 99.25 99.52 SE 56.42 60.35 58.43 80.89 78.39 54.87 80.04 80.39 81.57 88.23 93.29 SP 99.74 99.43 99.75 99.51 99.36 99.77 99.58 99.70 99.54 99.43 99.65 Colon-DB* Dice 28.19 34.99 31.86 66.88 60.06 46.30 73.01 67.26 71.08 76.51 78.72 IoU 23.23 29.89 26.78 57.86 50.33 39.87 63.11 59.55 61.46 66.12 70.06 ACC 98.77 98.92 98.88 99.19 98.72 99.13 99.01 99.30 99.27 99.30 99.40 SE 24.77 33.63 28.86 72.63 65.71 43.81 84.61 68.81 75.48 84.77 82.99 SP 99.94 99.86 99.94 99.50 99.22 99.87 99.19 99.73 99.61 99.50 99.67 ETIS-LaribPolyPDB* Dice 28.14 35.58 28.12 57.66 52.55 28.89 60.86 63.25 63.38 73.8 76.15 IoU 23.74 29.76 23.58 47.36 43.43 24.63 51.74 56.19 54.86 63.35 67.26 ACC 99.13 99.13 99.12 98.83 98.63 99.14 98.52 99.42 99.27 99.26 99.37 SE 25.79 35.84 26.49 74.51 59.41 26.74 81.00 68.13 72.25 84.38 82.66 SP 99.93 99.80 99.92 99.06 99.11 99.90 98.68 99.71 99.52 99.43 99.60 从表3可以看出,在5个息肉小目标数据集上,TFB-Net在Dice和IoU指标上均获得最高值。TFB-Net在CVC-300*数据集上Dice值比PraNet高2.08百分点,在Kvasir*数据集上超过ACSNet3.73百分点,在CVC-ClinicDB*、CVC-ColonDB*和ETIS-LaribPolyPDB*数据集中,分别超越CaraNet网络9.46百分点、2.21百分点和2.35百分点。 从图6对比实验分割效果图可看出TFB-Net具有最佳分割效果,尤其是在CVC-300*、CVC-ClinicDB*以及ETIS-LaribPolyPDB*数据集上对比效果尤为明显。此外,本文另外添加3个网络参数,分别是参数数量(parameters, Params)、浮点运算次数(floating-point operations, FLOPs)以及每秒传输帧数(frames per second, FPS),用于对比TFB-Net和其他网络在模型大小、复杂度和推理速度方面的性能,具体如表4所示,TFB-Net不仅在5个息肉小目标数据集上Dice和IoU指标均有显著提升,并且降低了模型复杂度。
表 4 不同先进模型大小、复杂度和推理速度统计情况Table 4 Statistics of size, complexity and inference speed of different advanced models指标 U-Net UNet++ UNet3+ Transfuse PVT FAT-Net MNFE-Net ACSNet PraNet CaraNet TFB-Net Params/106 31.04 47.18 26.97 26.17 25.11 29.62 58.16 29.45 30.50 44.59 69.78 FLOPs/109 103.49 377.45 377.64 8.65 10.02 42.80 9.42 21.75 13.15 21.75 31.55 FPS/(f/s) 99.72 38.38 29.13 169.56 71.73 176.36 104.93 74.77 116.81 80.26 64.20 4. 结束语
本文提出融合Transformer和边界反转注意力的特征挖掘息肉小目标分割网络,称为TFB-Net。与传统卷积神经网络编码器不同,TFB-Net以Transormer辅助CNNs编码。相比于普通卷积操作,Transormer不仅利用强大的自注意力机制扩大感受野范围,还能捕获丰富全局上下文信息。此外,为获取更全面特征,利用特征挖掘模块进一步学习稳定特征表示。为减少特征冗余,设计反转注意力模块,加强对特征边界的关注。本文在5个息肉小目标数据集(CVC-300*、CVC-ClinicDB*、Kvasir*、CVC-ColonDB*以及ETIS-LaribPolyPDB*)上进行广泛实验,实验结果表明,相比于10种先进的分割网络,TFB-Net具有出色的分割能力。在医学图像分割领域,息肉小目标分割俨然仍是当前一项富有挑战性的难题。未来的工作将仍致力于这一任务的研究,并尝试使用其他方法降低模型的复杂度,提高模型运行速度。
-
表 1 实验数据集
Table 1 Experimental datasets
数据集 总数量 训练集数量 测试集数量 CVC-300* 35 — 35 CVC-ClinicDB* 121 102 19 Kvasir* 77 67 10 CVC-ColonDB* 158 — 158 ETIS-LaribPolyPDB* 116 — 116 表 2 消融研究结果
Table 2 Ablation study results
% 方法 CVC-300* CVC-ClinicDB* Kvasir* CVC-ColonDB* ETIS-LaribPolyPDB* Dice IoU Dice IoU Dice IoU Dice IoU Dice IoU Baseline 81.50 72.35 77.72 68.93 78.77 70.03 71.16 61.34 68.08 58.12 Baseline+TAE 86.15 77.70 83.56 75.22 84.06 75.84 74.30 65.62 72.96 63.77 Baseline+FMM 86.42 78.41 83.44 75.89 83.67 76.01 74.81 66.25 72.60 64.10 Baseline+BRA 85.39 77.43 80.15 72.84 81.08 73.82 74.96 66.56 71.65 63.20 Baseline+TAE+FMM 86.34 77.77 83.42 75.53 84.04 76.24 76.95 67.97 72.85 64.44 Baseline+TAE+BRA 87.05 79.90 84.27 77.26 84.94 77.90 75.52 67.67 71.88 64.32 Baseline+FMM+BRA 86.11 79.18 83.81 76.87 84.26 77.28 75.81 67.73 73.68 65.89 Baseline+TAE+FMM+BRA 88.80 81.16 87.15 79.52 87.18 79.58 78.72 70.06 76.15 67.26 表 3 对比实验结果
Table 3 Comparative experimental results
% 数据集 指标 U-Net UNet++ UNet3+ Transfuse PVT FAT-Net MNFE-Net ACSNet PraNet CaraNet TFB-Net
CVC-300*Dice 53.46 70.93 72.51 84.58 75.59 79.82 85.04 84.74 86.72 84.62 88.80 IoU 44.97 62.23 63.41 75.57 65.13 70.22 77.75 78.05 77.89 74.87 81.16 ACC 98.81 99.17 99.26 99.39 98.98 99.38 99.31 99.44 99.44 99.35 99.56 SE 47.67 72.98 68.78 96.37 85.58 80.41 97.07 93.46 97.57 97.83 96.81 SP 99.93 99.65 99.86 99.44 99.27 99.72 99.36 99.59 99.47 99.36 99.62 Clinic-DB* Dice 61.35 65.37 64.67 68.94 72.43 64.80 75.83 72.55 75.23 77.69 87.15 IoU 56.59 59.62 58.66 61.62 63.42 58.30 67.20 66.21 67.52 68.42 79.52 ACC 99.21 99.24 99.19 98.88 98.98 99.19 98.86 99.34 99.23 99.23 99.53 SE 59.40 63.01 63.18 74.51 79.37 62.57 87.51 77.15 80.48 87.80 93.86 SP 99.92 99.85 99.81 99.32 99.34 99.81 99.13 99.73 99.63 99.41 99.66 Kvasir* Dice 63.58 61.03 64.98 78.81 72.03 59.52 80.14 83.45 78.73 78.74 87.18 IoU 55.54 54.99 56.72 71.97 63.09 52.74 73.52 75.47 71.41 69.47 79.58 ACC 98.97 98.66 99.05 99.17 98.98 99.01 99.25 99.36 99.20 99.25 99.52 SE 56.42 60.35 58.43 80.89 78.39 54.87 80.04 80.39 81.57 88.23 93.29 SP 99.74 99.43 99.75 99.51 99.36 99.77 99.58 99.70 99.54 99.43 99.65 Colon-DB* Dice 28.19 34.99 31.86 66.88 60.06 46.30 73.01 67.26 71.08 76.51 78.72 IoU 23.23 29.89 26.78 57.86 50.33 39.87 63.11 59.55 61.46 66.12 70.06 ACC 98.77 98.92 98.88 99.19 98.72 99.13 99.01 99.30 99.27 99.30 99.40 SE 24.77 33.63 28.86 72.63 65.71 43.81 84.61 68.81 75.48 84.77 82.99 SP 99.94 99.86 99.94 99.50 99.22 99.87 99.19 99.73 99.61 99.50 99.67 ETIS-LaribPolyPDB* Dice 28.14 35.58 28.12 57.66 52.55 28.89 60.86 63.25 63.38 73.8 76.15 IoU 23.74 29.76 23.58 47.36 43.43 24.63 51.74 56.19 54.86 63.35 67.26 ACC 99.13 99.13 99.12 98.83 98.63 99.14 98.52 99.42 99.27 99.26 99.37 SE 25.79 35.84 26.49 74.51 59.41 26.74 81.00 68.13 72.25 84.38 82.66 SP 99.93 99.80 99.92 99.06 99.11 99.90 98.68 99.71 99.52 99.43 99.60 表 4 不同先进模型大小、复杂度和推理速度统计情况
Table 4 Statistics of size, complexity and inference speed of different advanced models
指标 U-Net UNet++ UNet3+ Transfuse PVT FAT-Net MNFE-Net ACSNet PraNet CaraNet TFB-Net Params/106 31.04 47.18 26.97 26.17 25.11 29.62 58.16 29.45 30.50 44.59 69.78 FLOPs/109 103.49 377.45 377.64 8.65 10.02 42.80 9.42 21.75 13.15 21.75 31.55 FPS/(f/s) 99.72 38.38 29.13 169.56 71.73 176.36 104.93 74.77 116.81 80.26 64.20 -
[1] XI Yue, XU Pengfei. Global colorectal cancer burden in 2020 and projections to 2040[J]. Translational oncology, 2021, 14(10): 101174. doi: 10.1016/j.tranon.2021.101174 [2] KOLLIGS F T. Diagnostics and epidemiology of colorectal cancer[J]. Visceral medicine, 2016, 32(3): 158−164. doi: 10.1159/000446488 [3] DAI Ying, CHEN Weimin, XU Xuanfu, et al. Factors affecting adenoma risk level in patients with intestinal polyp and association analysis[J]. Journal of healthcare engineering, 2022: 9479563. [4] LI Zewen, LIU Fan, YANG Wenjie, et al. A survey of convolutional neural networks: analysis, applications, and prospects[J]. IEEE transactions on neural networks and learning systems, 2022, 33(12): 6999−7019. doi: 10.1109/TNNLS.2021.3084827 [5] SARVAMANGALA D R, KULKARNI R V. Convolutional neural networks in medical image understanding: a survey[J]. Evolutionary intelligence, 2022, 15(1): 1−22. doi: 10.1007/s12065-020-00540-3 [6] RONNEBERGER O, FISCHER P , BROX T. U-Net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2015: 234−241. [7] ZHOU Zongwei, RAHMAN SIDDIQUEE M M, TAJBAKHSH N, et al. UNet++: a nested U-net architecture for medical image segmentation[C]//Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Cham: Springer, 2018: 3−11. [8] HUANG Huimin, LIN Lanfen, TONG Ruofeng, et al. UNet 3: a full-scale connected UNet for medical image segmentation[C]//2020 IEEE International Conference on Acoustics, Speech and Signal Processing. Barcelona: IEEE, 2020: 1055−1059. [9] ZHANG Zhengxin, LIU Qingjie, WANG Yunhong. Road extraction by deep residual U-net[J]. IEEE geoscience and remote sensing letters, 2018, 15(5): 749−753. doi: 10.1109/LGRS.2018.2802944 [10] JHA D, SMEDSRUD P H, RIEGLER M A, et al. ResUNet: an advanced architecture for medical image segmentation[C]//2019 IEEE International Symposium on Multimedia. San Diego: IEEE, 2019: 225−2255. [11] TOMAR N K, JHA D, BAGCI U. DilatedSegNet: a deep dilated segmentation network for Polyp segmentation[C]//MultiMedia Modeling. Cham: Springer, 2023: 334−344. [12] TOMAR N K, SRIVASTAVA A, BAGCI U, et al. Automatic polyp segmentation with multiple kernel dilated convolution network[C]//2022 IEEE 35th International Symposium on Computer-Based Medical Systems. Shenzen: IEEE, 2022: 317−322. [13] ZHANG Wenchao, FU Chong, ZHENG Yu, et al. HSNet: a hybrid semantic network for polyp segmentation[J]. Computers in biology and medicine, 2022, 150: 106173. doi: 10.1016/j.compbiomed.2022.106173 [14] DONG Bo, WANG Wenhai, FAN Dengping, et al. Polyp-PVT: polyp segmentation with pyramid vision transformers[J]. CAAI artificial intelligence research, 2023: 9150015. [15] LI Weisheng, ZHAO Yinghui, LI Feiyan, et al. MIA-Net: multi-information aggregation network combining transformers and convolutional feature learning for polyp segmentation[J]. Knowledge-based systems, 2022, 247: 108824. doi: 10.1016/j.knosys.2022.108824 [16] WU Huisi, CHEN Shihuai, CHEN Guilian, et al. FAT-Net: feature adaptive transformers for automated skin lesion segmentation[J]. Medical image analysis, 2022, 76: 102327. doi: 10.1016/j.media.2021.102327 [17] ZHANG Yundong, LIU Huiye, HU Qiang. TransFuse: fusing transformers and CNNs for medical image segmentation[C]//Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2021: 14−24. [18] LIU Guoqi, WANG Jiajia, LIU Dong, et al. A multiscale nonlocal feature extraction network for breast lesion segmentation in ultrasound images[J]. IEEE transactions on instrumentation and measurement, 2023, 72: 1−12. [19] ZHANG Ruifei, LI Guanbin, LI Zhen, et al. Adaptive context selection for polyp segmentation[C]//Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2020: 253–262. [20] FAN Dengping, JI Gepeng, ZHOU Tao, et al. PraNet: parallel reverse attention network for polyp segmentation[C]//Medical Image Computing and Computer Assisted Intervention. Cham: Springer, 2020: 263−273. [21] LOU Ange, GUAN Shuyue, KO H, et al. CaraNet: context axial reverse attention network for segmentation of small medical objects[C]//Medical Imaging 2022: Image Processing. San Diego: SPIE, 2022: 81−92. [22] DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: transformers for image recognition at scale[EB/OL]. (2020–10–22)[2023−6−13]. http://arxiv.org/abs/2010.11929. [23] ZHENG Sixiao, LU Jiachen, ZHAO Hengshuang, et al. Rethinking semantic segmentation from a sequence-to-sequence perspective with transformers[C]//2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Nashville: IEEE, 2021: 6877−6886. [24] TOUVRON H, CORD M, DOUZE M, et al. Training data-efficient image transformers & distillation through attention[EB/OL]. (2020–12–23)[2023−06−13]. https://arxiv.org/abs/2012.12877. [25] 汪鹏程, 张波涛, 顾进广. 融合多尺度门控卷积和窗口注意力的结肠息肉分割[J]. 计算机系统应用, 2024, 33(6): 70−80. WANG Pengcheng, ZHANG Botao, GU Jinguang. Colon polyp segmentation fusing multi-scale gate convolution and window attention[J]. Computer systems and applications, 2024, 33(6): 70−80. [26] QIN Xuebin, ZHANG Zichen, HUANG Chenyang, et al. BASNet: boundary-aware salient object detection[C]//2019 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Long Beach: IEEE, 2019: 7471−7481. [27] VÁZQUEZ D, BERNAL J, SÁNCHEZ F J, et al. A benchmark for endoluminal scene segmentation of colonoscopy images[J]. Journal of healthcare engineering, 2017: 4037190. [28] BERNAL J, SÁNCHEZ F J, FERNÁNDEZ-ESPARRACH G, et al. WM-DOVA maps for accurate polyp highlighting in colonoscopy: validation vs. saliency maps from physicians[J]. Computerized medical imaging and graphics, 2015, 43: 99−111. doi: 10.1016/j.compmedimag.2015.02.007 [29] POGORELOV K, KRISTIN R R, GRIWODZ C, et al. Kvasir: A multi-class image dataset for computer aided gastrointestinal disease detection[C]//Proceedings of the 8th ACM on Multimedia Systems Conference. Taipei: ACM, 2017: 164−169. [30] BERNAL J, SÁNCHEZ J, VILARIÑO F. Towards automatic polyp detection with a polyp appearance model[J]. Pattern recognition, 2012, 45(9): 3166−3182. doi: 10.1016/j.patcog.2012.03.002 [31] SILVA J, HISTACE A, ROMAIN O, et al. Toward embedded detection of polyps in WCE images for early diagnosis of colorectal cancer[J]. International journal of computer assisted radiology and surgery, 2014, 9(2): 283−293. doi: 10.1007/s11548-013-0926-3 [32] GAO Shanghua, CHENG Mingming, ZHAO Kai, et al. Res2Net: a new multi-scale backbone architecture[J]. IEEE transactions on pattern analysis and machine intelligence, 2021, 43(2): 652−662. doi: 10.1109/TPAMI.2019.2938758


 下载:
下载: