Tumor segmentation method for breast ultrasound images incorporating CNN and ViT
-
摘要: 针对乳腺超声图像肿瘤区域形状大小差异大导致分割困难,卷积神经网络(convolutional neural networks, CNN)建模长距离依赖性和空间相关性方面存在局限性,视觉Transformer(vision Transformer, ViT)要求数据量巨大等问题,提出一种融合CNN和ViT的分割方法。使用改进的Swin Transformer模块和基于可形变卷积的CNN编码器模块分别提取全局特征和局部细节特征,设计使用交叉注意力机制融合这两种尺度的特征表示,训练过程采取二元交叉熵损失混合边界损失函数,有效提高分割精度。在两个公共数据集上的实验结果表明,与现有经典算法相比所提方法的分割结果有显著提升,dice系数提升3.841 2%,验证所提方法的有效性和可行性。Abstract: A segmentation method that fuses CNN and ViT is proposed to address the problems of large differences in shape and size of tumor regions of breast ultrasound images that lead to difficulty in segmentation, limitations in long-range dependency and spatial correlation in convolutional neural network (CNN) modeling, and the huge amount of data required by vision Transformer (ViT). Global and local detail features were extracted using a modified Swin Transformer module and a CNN encoder module based on deformable convolution, respectively. The design uses a cross-attention mechanism to fuse the feature representations of the two scales, and the training process adopts a binary cross-entropy loss combined with a boundary loss function. This approach effectively improves the segmentation accuracy. Experimental results on two public datasets show that the segmentation findings of the proposed method have been significantly improved compared with those of the existing classical algorithms, with a 3.8412% improvement in the dice coefficient. This outcome verifies the effectiveness and feasibility of the proposed method.
-
根据我国国家癌症中心2022年2月发布的最新一期全国癌症统计数据显示[1],乳腺癌是我国女性发病率第一且死亡率第四的癌症,分别占比29.05%和6.39%。在我国,乳腺癌的发病率和死亡率呈逐年上升趋势[2]。乳腺超声成像由于其无创、无放射性、低成本的特点[3]被广泛用于临床检测乳腺肿瘤,也成为资源匮乏国家和地区进行大规模乳腺癌筛查和诊断的最合适方法。而即使是有经验的放射科医师也难以准确和快速地标记乳腺超声图像的病变区域。随着人工智能和深度学习技术的发展,乳腺超声图像的计算机辅助诊断(computer aided diagnosis, CAD)技术也应运而生,而乳腺超声图像CAD系统中的一个重要内容就是乳腺肿瘤区域的精确分割。
近年来兴起的深度学习分割方法将图像的分割转换为逐像素的分类问题[4],可以获得更好的分割结果[5]。卷积神经网络(convolutional neural networks, CNN)被广泛应用于医学图像分割的各个方面[6]。U-Net[7]是比较流行的基于CNN的体系结构。Almajalid等[8]首次使用U-Net从超声图像中分割乳腺病变,与以往的基于图的和基于模糊c均值聚类的分割方法对比有更好的分割结果。与一般图像相比,由于乳腺超声图像肿瘤区域纹理、形状、大小差异大[9],而且乳腺超声图像中有许多与乳腺肿瘤病变距离较远但外观相似的正常像素[10],直接采用U-Net进行分割得不到理想的结果。为了提高分割精度,Zhuang等[11]提出了残差扩张注意力门U-Net(residual dilated attention gate UNet, RDAU-Net),它使用扩张的残差块和注意力门分别替换U-Net中的基本块和原始跳跃连接,提高了肿瘤分割的整体灵敏度和准确性。虽然基于CNN的网络结构在乳腺超声图像分割任务中取得了显著成功,但由于其有限的感受野和固有的归纳偏置[12],在图像中建立全局上下文和长距离依赖存在限制,导致医学影像任务的分割效果也受限。
最近,在自然语言处理领域中表现出色的Transformer的激励下,视觉Transformer(vision transformer, ViT)[13]被提出。ViT可以利用多头注意力机制,有效地构建长距离依赖关系并捕获全局上下文。最典型的是Valanarasu等[14]提出的医用Transformer(medical Transformer, MedT)中的门控轴向注意层,用于构建多头注意力模块。和CNN相比,ViT的缺点是必须在大数据集上进行预训练。而且ViT很难处理高分辨率图像,因为其自注意力的计算复杂度和输入图像大小成平方增长关系。此外,当ViT用于图像处理领域时,二维图像被切片并作为一维序列送入模型[13],仅关注全局上下文,低分辨率特征不能通过直接上采样到全分辨率来有效恢复详细的定位信息,从而导致分割结果粗糙。
为了提高基于ViT的方法对小数据集的适应性,一些研究人员已经进行了相关尝试。比如提出混合CNN和ViT的方法对图像进行分割,结合CNN的局部特征表示能力和ViT的全局特征表示能力。例如Chen等[15]提出的TransUnet,通过跳跃连接来自CNN和ViT块的局部和全局特征图,构成类似U-Net的模型结构。Zhang等[16]提出了TransFuse,采取并行分支结构和BiFusion模块结合CNN和ViT的特征信息。但这些方法也存在着不能有效融合信息并保持特征一致性[17]、对于乳腺超声图像肿瘤区域的细节信息学习不够、分割精度不高等问题。
因此,为了进一步提高乳腺超声图像肿瘤区域的分割效果,本文提出一种新的乳腺超声图像肿瘤区域分割模型,采用多尺度的Swin Transformer混合CNN结构,引入交叉注意力机制融合粗粒度和细粒度的特征表示。在公共数据集BUSI[18]和Dataset B[19]上的实验结果验证了本文提出算法的有效性。
1. 本文模型结构
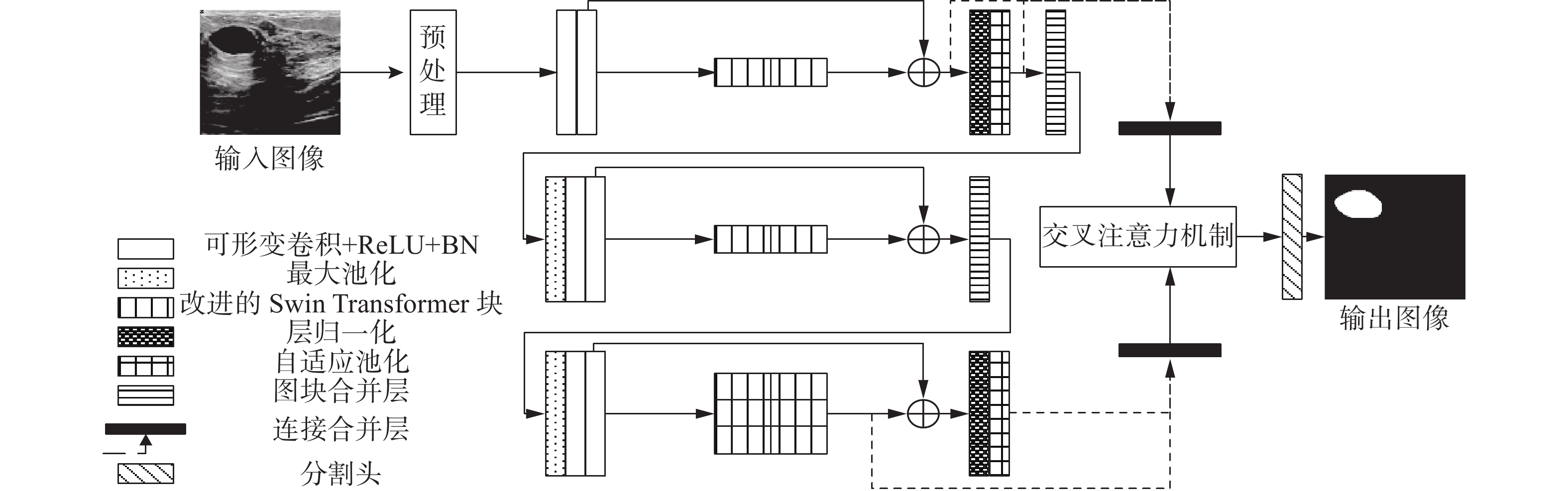
为了解决乳腺超声图像分割的难点,本文提出了融合CNN和Swin Transformer架构的乳腺肿瘤分割模型,结构如图1所示。首先对乳腺超声图像进行预处理,再送入CNN学习局部特征表示,接着使用改进的Swin Transformer块学习全局上下文表示,创造性地提出了三层级残差连接架构,将CNN和Swin Transformer信息交互,之后将学习到的高级特征和低级特征使用交叉注意力机制融合,最后由分割头生成分割掩膜图。
1.1 基于可形变卷积的CNN结构
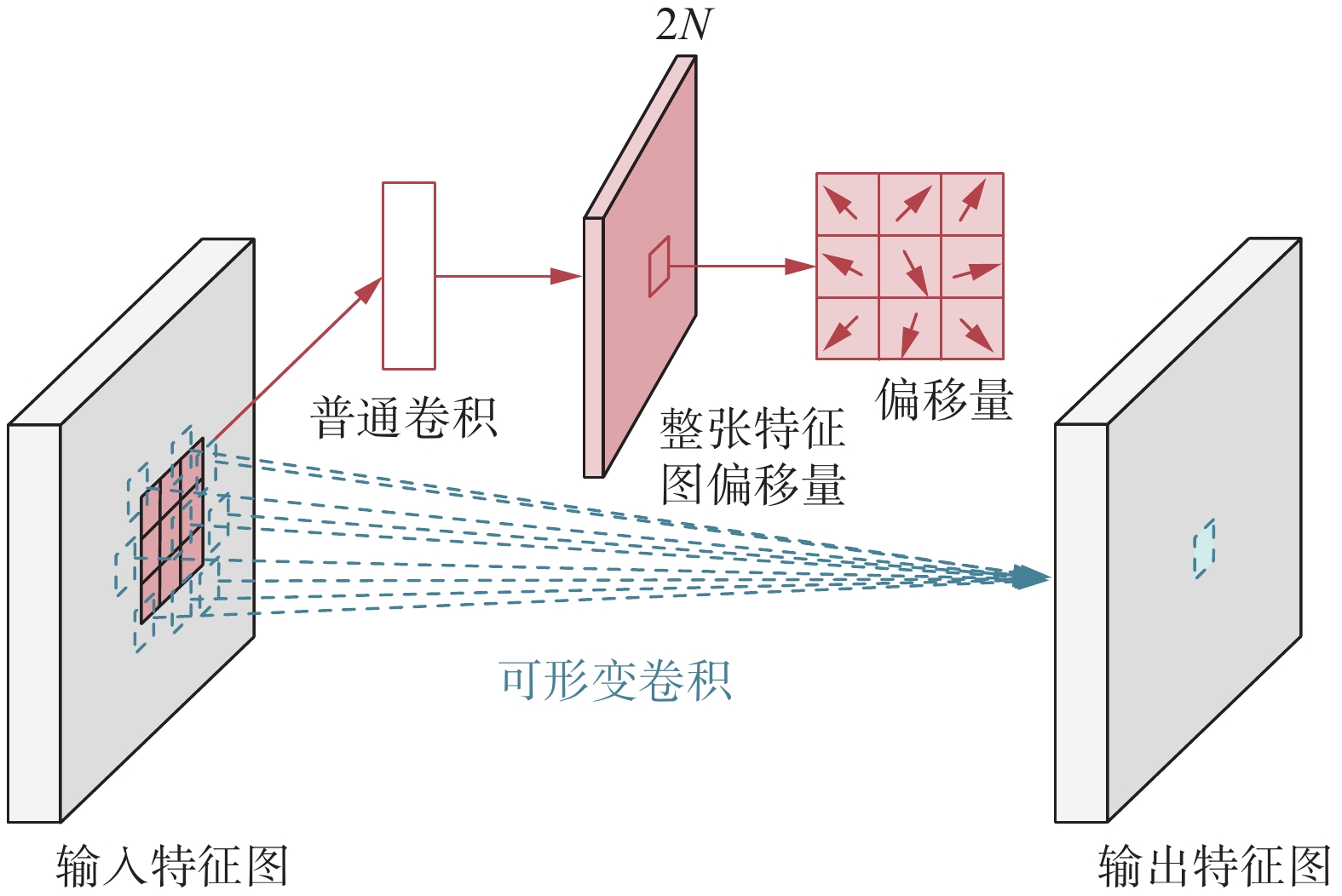
本文采用U-Net网络的编码器层代表CNN的典型结构,即输入层、卷积池化层、全连接层、输出层。所有卷积操作后都会伴随ReLU激活和批归一化(batch normalization, BN),来保障每层网络的输入都是相同的分布。肿瘤区域形状不规则,和背景区域的亮度差异不大,区分边界和背景区域非常困难。而可形变卷积[20]具有较强的空间适应性,在普通卷积的基础上引入了一组偏移量和权重值共同学习,大幅度提高网络在学习细节信息上的表现,因此引入了可形变卷积代替普通卷积作为CNN特征提取结构,可形变卷积的原理如图2所示。本文所设计的CNN特征提取结构具有三层卷积操作,前两次卷积操作后由最大池化将CNN和Swin Transformer块残差连接的特征图进行下采样。
1.2 改进的Swin Transformer结构
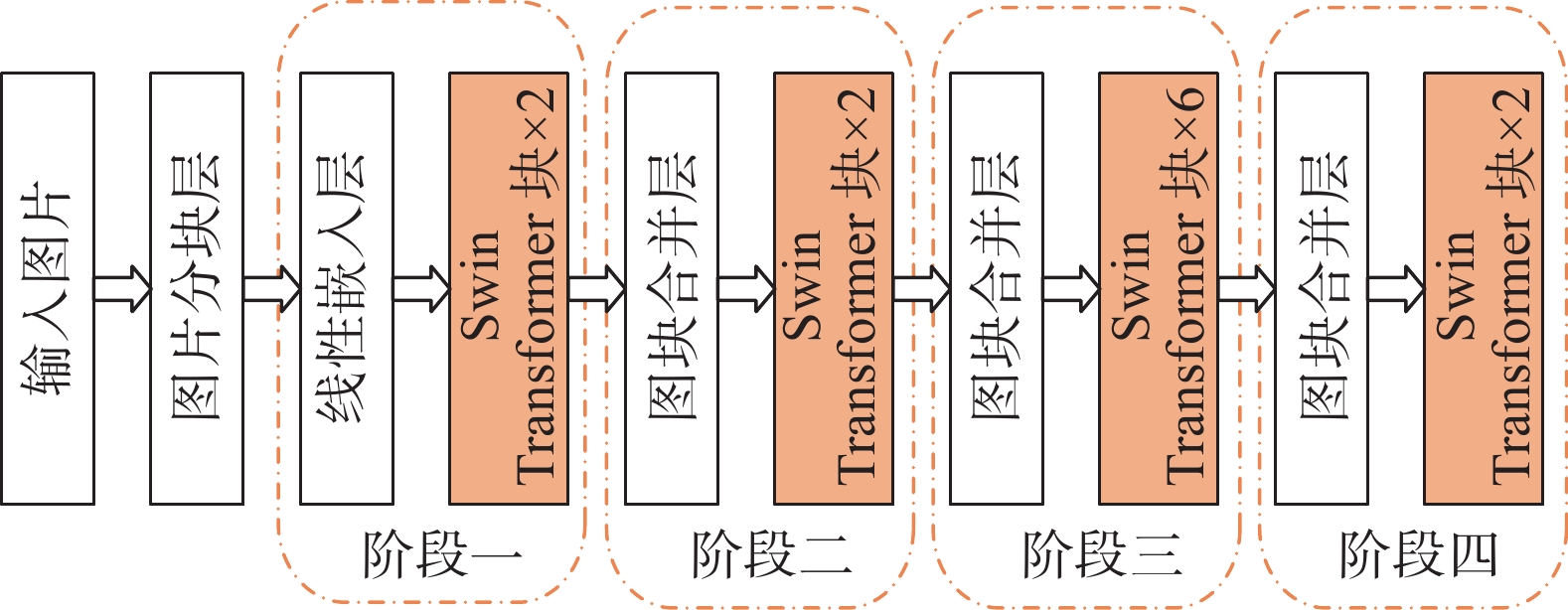
和原始的ViT始终对图片全局做自注意力计算不同,Swin Transformer是对窗口做自注意力计算[21],大大减少了计算量,把计算量从平方量级变成线性量级,因此引入经典的Swin Transformer结构作为典型ViT结构。Swin Transformer采用四阶段金字塔结构,结构如图3所示。
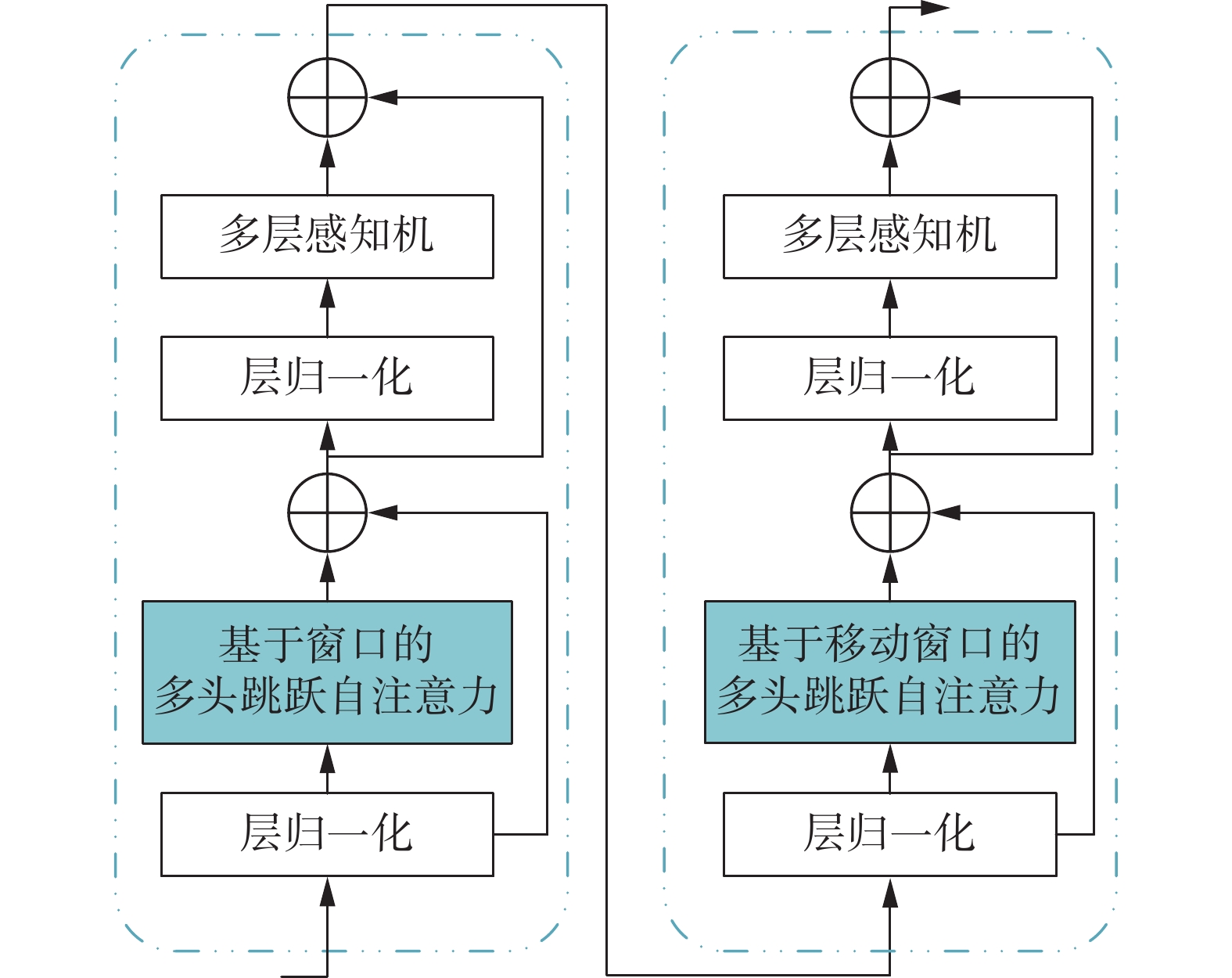
Swin Transformer包括两个连续的Swin Transformer块。用基于窗口的多头自注意力(window multi-head self attention, W-MSA)和基于移位窗口的多头自注意力(shifted window multi-head self attention, SW-MSA)来代替原始的ViT中的多头自注意力(multi-head self attention, MSA)。在W-MSA中,自注意力将应用于大小为
$M$ ×$M$ 的局部窗口。然而,由于没有跨窗口的连接,它的建模能力有限。为了缓解这种情况,引入了SW-MSA,其利用与W-MSA模块的输入相比移位的窗口配置,这是为了确保具有跨窗口连接。该过程用如下公式表示:$$ A({\boldsymbol{Q}},{\boldsymbol{K}},{\boldsymbol{V}}) = {\text{softmax}}\left(\frac{{{\boldsymbol{Q}}{{({\boldsymbol{K}})}^{\text{T}}}}}{{\sqrt d }} + B\right){\boldsymbol{V}} $$ (1) 其中:
${\boldsymbol{Q}}$ 、${\boldsymbol{K}}$ 、${\boldsymbol{V}}$ 分别为每个像素点的查询(query)、键(key)、值(value)进行注意力$A$ 的计算,$B$ 是Swin Transformer中的相对位置偏差。为了更好地利用来自不同阶段的特征映射,将Swin Transformer中的注意力机制从MSA改进为多头跳跃注意力(multi-head skip self attention, MSKA)。MSA只是计算与自身的相似度,很难有效地激活一个注意头中的单个类别。而利用MSKA计算语义相同的两个不同特征映射的相似度,更好地利用了注意力机制,能够激活一个注意头中的单个类别。使用前一阶段的输出
$M$ 作为键、值,使用特征映射$F$ 作为查询。基于窗口的多头跳跃注意力(window multi-head skip self attention, W-MSKA)设置为$$ A({{\boldsymbol{Q}}_{\text{F}}},{{\boldsymbol{K}}_{\text{M}}},{{\boldsymbol{V}}_{\text{M}}}) = {\text{softmax}}\left(\frac{{{{\boldsymbol{Q}}_{\text{F}}}{{({{\boldsymbol{K}}_{\text{M}}})}^{\text{T}}}}}{{\sqrt d }} + B\right){{\boldsymbol{V}}_{\text{M}}} $$ (2) 其中:
${{\boldsymbol{Q}}_{\text{F}}}$ 是跳跃注意力的查询,是${\text{F}}$ 的线性变换;${{\boldsymbol{K}}_{\text{M}}}$ 和${{\boldsymbol{V}}_{\text{M}}}$ 是键和值,都是${\text{M}}$ 的线性变换。改进后的Swin Transformer块的结构如图4所示。1.3 基于交叉注意力机制的特征融合模块
为了有效融合改进Swin Transformer编码器传递的嵌入信息,引入交叉注意力机制[22]融合每个级别的特征。具体地说,在融合之前,两个级别的类标记被交换,这意味着一个级别的类标记与另一个级别的标记连接。然后,每个新的嵌入被单独地馈送通过用于融合的模块,并且最终被反向投影到其自身的级别。这种与其他级别标记的交互使类标记能够与它们的跨级别标记共享丰富的信息。
根据这种思想提出的特征融合模块如图5所示,用
${P^l}$ 和${P^s}$ 分别表示来自CNN和来自Swin Transformer块的不同分辨率的特征图。由Transformer编码器输出的最高分辨率视觉标记和高分辨率关键点标记,和来自CNN的低分辨率标记重新组合生成新的视觉标记和关键点标记,作为多尺度交叉注意力模块的输入。为了避免关键点标记的冗余和多次融合关键点标记,模块采用了移动关键点标记策略。该策略的具体做法:在输入到该模块前,高、低分辨率视觉标记分别拼接固定的高、低分辨率关键点标记,将包含关键点和视觉特征的高分辨率标记作为键和值,低分辨率标记作为查询,然后进行交叉注意力计算。当进行下一次交互时,该关键点标记和其他的高、低分辨率视觉标记重新拼接。1.4 混合损失函数
根据在预测标签和真实标签之间每个像素存在的偏差,损失函数用来在训练期间矫正网络参数。医学成像中常用的分割损失函数是交叉熵和dice得分。由于乳腺超声图像中肿瘤的像素所占比例很小,肿瘤分割存在严重的数据不平衡问题。因此,该模型将主要学习具有大量像素的非肿瘤样本的特征。二元交叉熵损失函数被广泛用于解决上述问题。交叉熵测量图像中每个像素的预测概率和校正概率的对数值。对于图像二值分割的问题,二元交叉熵在像素级分类上表现良好。二元交叉熵损失函数的数学表达式为
$$ {L_{{\text{BCE}}}} = - \frac{1}{n}\sum\limits_{i = 1}^n {\left[ {{y_i}\log p({y_i})} \right.\left. { + (1 - {y_i})\log (1 - p({y_i}))} \right]} $$ (3) 式中
$ {y_i} $ 是分割图中每个像素值的预测,取值为0或1。在医学图像中,边界是不同组织之间区别较为明显的地方,在人工分割时是区分不同目标的重要参考。然而在卷积神经网络中,边界作为不同类别相交位置,与网络分割区域的特征如形态、强度相似性等不同,属于高频信息,如果不加以一定约束,特征在不断卷积的过程中不断被抽象,容易丢失这些细节信息导致网络分割错误。为了提升网络分割精度,有必要对网络施加额外的边界约束以提高网络对边界信息的利用提升网络的分割性能。同时,依靠设置交叉熵损失权重来缓解数据分布不平衡,通过对目标施加额外的监督损失,边界损失[23]也能更进一步解决分割精度不高的问题。边界损失能在不增加网络复杂性的同时充分发掘区域和边界互补信息辅助区域分割。二元边界损失函数的数学表达式为
$$ P = \frac{1}{{\left| {{B_p}} \right|}}\sum\limits_{x \in {B_p}} {\left[\kern-0.15em\left[ {d(x,{B_g}) < \theta } \right]\kern-0.15em\right]} $$ (4) $$ R = \frac{1}{{\left| {{B_g}} \right|}}\sum\limits_{x \in {B_g}} {\left[\kern-0.15em\left[ {d(x,{B_p}) < \theta } \right]\kern-0.15em\right]} $$ (5) $$ {L_{{\text{Boundary}}}} = 1 - \frac{{2P \cdot R}}{{P + R}} $$ (6) 式中:
$ {B_g} $ 和$ {B_p} $ 分别为真实标签和分割预测结果的边界,$ d(\cdot) $ 是以像素为单位测量的欧几里得距离,$ \theta $ 是预定义的阈值。采用混合损失函数的方法会获得更好的分割结果,因此本文在实验训练过程中使用混合交叉熵损失和边界损失计算预测的分割结果损失,损失函数的定义为$$ L = \alpha {L_{{\text{BCE}}}} + (1 - \alpha ){L_{{\text{Boundary}}}} $$ (7) 其中
$\alpha $ 为超参数,需要进行实验确定适合值。2. 实验仿真及结果分析
实验所使用的计算机系统环境为Ubuntu20.04,采用Python编程语言,Pycharm作为集成开发环境(IDE),Pytorch1.11作为深度学习框架,在10 GB显存的RTX3080 GPU上进行训练。输入图像大小为224像素×224像素,训练使用AdamW优化器,初始学习率为0.0001,权重衰减为0.000 05,数据批处理大小设置为4,最大训练80 000轮次。
2.1 数据集和数据预处理
使用公开的乳腺超声图像数据集BUSI[18]和Dataset B[19]进行对比实验,来验证本文提出算法的可行性和有效性。每个数据集都包含原始乳腺超声图像和由专业医师标注好的肿瘤区域。其中,BUSI数据集包含133张正常超声图像、437张良性肿瘤图像和210张恶性肿瘤图像。Dataset B数据集包含110张良性肿瘤图像和53张恶性肿瘤图像。为了更直观地评估本文方法在恶性肿瘤和良性肿瘤上的分割性能,实验只在BUSI和Dataset B中的良性肿瘤和恶性肿瘤图像上进行训练和测试,不使用正常超声图像进行训练。
考虑到数据集过小的问题,本文使用数据增广方法来扩充数据,包括平移、随机水平翻转、随机裁剪等。将BUSI扩张为3 235张图像,将Dataset B扩张为815张图像,按照4∶1随机划分为训练集、测试集。实验使用双线性插值技术将数据集中的所有图像大小统一为224像素×224像素,并进行3×3中值滤波器处理。同时,使用最近邻插值技术将所有样本对应的真实标签(Ground Truth)大小也统一为224×224。
2.2 评价指标
在每个实验中,为了定量分析分割效果,本文采用了常用的5个评价指标
$I$ ,包括dice系数、Jc指数、95%Hausdorff距离(Hd95)、准确度和召回率。dice系数可以计算分割结果和真实标签之间的相似度,取值范围是[0,1],取值越大证明分割结果越好。计算公式为
$$ {I_{{\text{dice}}}} = \frac{{2{N_{{\text{TP}}}}}}{{2{N_{{\text{TP}}}} + {N_{{\text{FP}}}} + {N_{{\text{FN}}}}}} $$ (8) 其中:
${N_{{\text{TP}}}}$ 表示图像中正常且被模型预测正常的像素点数量,${N_{{\text{FP}}}}$ 表示图像中肿瘤但被模型预测正常的像素点数量,${N_{{\text{FN}}}}$ 表示图像中肿瘤且被模型预测肿瘤的像素点数量。杰卡德指数(Jc指数)也可以计算分割结果和真实标签的相似度,作为辅助评价指标使用,取值越大证明分割结果越好。
$$ {I_{{\text{Jc}}}} = \frac{{{N_{{\text{TP}}}}}}{{{N_{{\text{TP}}}} + {N_{{\text{FP}}}} + {N_{{\text{FN}}}}}} $$ (9) Hausdorff距离可以计算分割结果和真实标签边界的最大距离差异,主要用于衡量分割边界的准确性,取值越小证明分割结果越好,计算公式为
$$ {I_{{\text{Hd}}}}(A,B) = \max (h(A,B),h(B,A)) $$ (10) $$ h(A,B) = \mathop {\max }\limits_{a \in A} \{ \mathop {\min }\limits_{b \in B} \left\| {a - b} \right\|\} $$ (11) $$ h(B,A) = \mathop {\max }\limits_{b \in B} \{ \mathop {\min }\limits_{a \in A} \left\| {b - a} \right\|\} $$ (12) 其中:
$ h $ 是计算的Hausdorff距离,$ {I_{{\text{Hd}}}} $ 是最终的Hausdorff距离结果,$ A $ 是分割结果图,$ B $ 是真实标签图。但在实际计算中往往不是取最大距离,而是取前5%距离,可以排除一些离群点距离对分割结果的影响。准确率(precision)可以计算分割结果中真正的肿瘤区域的比例,取值越大证明分割结果越好。
$$ {I_{{\text{precision}}}} = \frac{{{N_{{\text{TP}}}}}}{{{N_{{\text{TP}}}} + {N_{{\text{FP}}}}}} $$ (13) 召回率(recall)可以计算所有肿瘤区域被预测正确的比例,取值越大证明分割结果越好。
$$ {I_{{\text{recall}}}} = \frac{{{N_{{\text{TP}}}}}}{{{N_{{\text{TP}}}} + {N_{{\text{FN}}}}}} $$ (14) 2.3 超参数设置
对本文提出的混合损失函数中的超参数
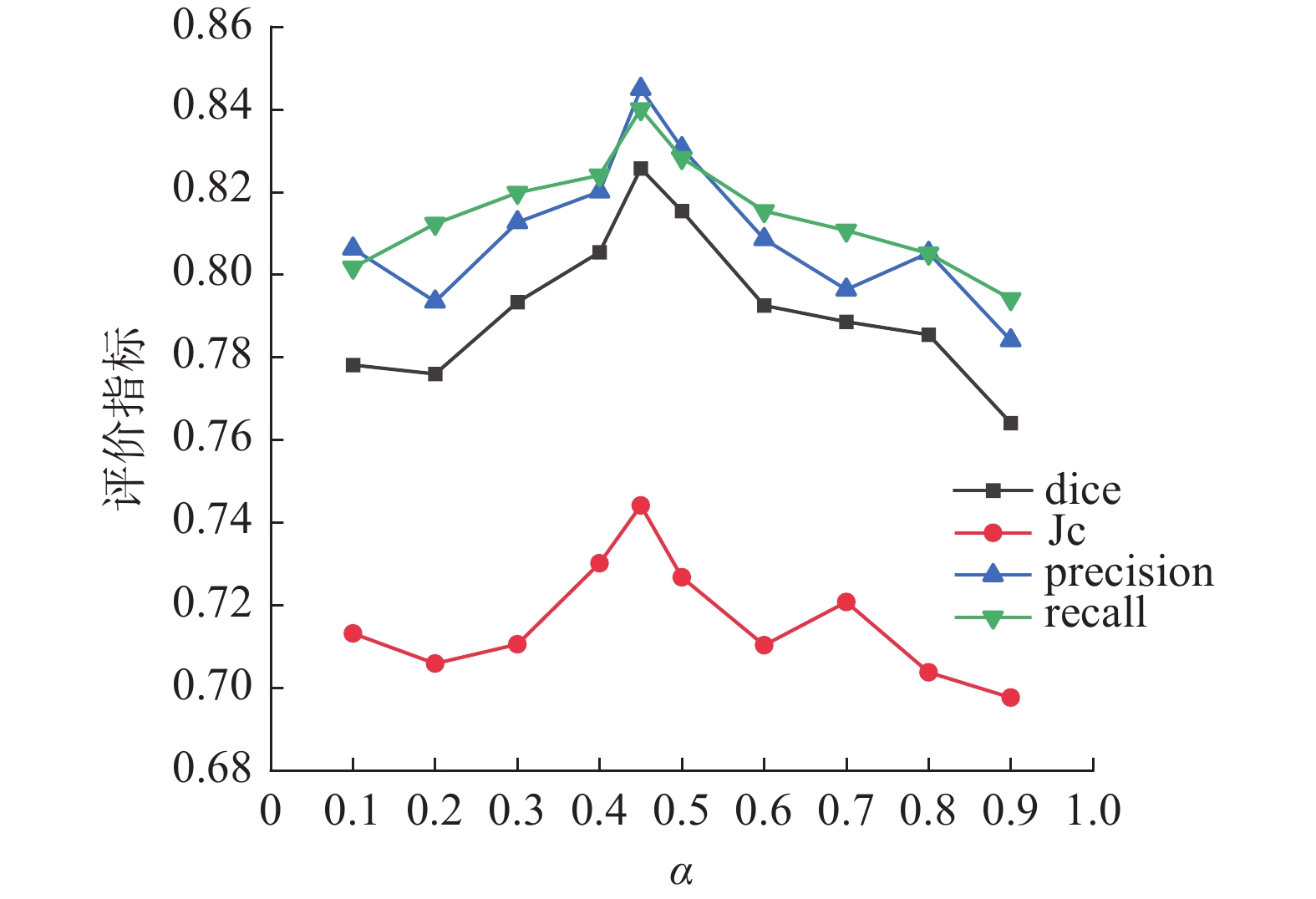
$\alpha $ 采用数据量较大的BUSI进行实验,对$\alpha $ 取值0.1~0.9,得出0.4、0.5是评价指标最大的两个值,最后取0.45进行实验验证,得出各评价指标的峰值。实验结果如图6所示。2.4 对比实验
为了验证所提出的算法的有效性,本文选择以下6个在医学图像分割方面具有代表性的网络结构与本文提出的算法进行对比。其中U-Net是经典CNN结构,Swin Transformer是纯ViT结构,其余则都是混合CNN-ViT结构。
1) U-Net[4]:通过跳跃连接收缩路径和扩展路径,可以有效地融合低分辨率和高分辨率特征。
2) Swin Transformer[21]:利用ViT提取全局特征,通过基于窗口的多头注意力计算,可以实现跨窗口信息交互和线性计算复杂度。
3) TransUnet[15]:通过跳跃连接来自CNN和ViT的局部和全局特征图,构成类似U-Net的模型结构。
4) TransDeeplab[24]:利用带有移位窗口的Swin Transformer块扩展deeplabv3+,并对空洞空间金字塔池化模块建模。
5) SwinUnet[25]:对照Swin Transformer块的图块拼接层设计了一个图块扩展层,组成类似U-Net型的编码器−解码器结构。
6) TransNorm[26]:将ViT整合到U-Net编码器和跳跃连接中,利用两级注意力机制自适应地重校准跳跃连接路径。
在两个数据集Dataset BUSI和Dataset B中分别进行分割实验。由于考虑到不同硬件环境下的计算时间不同,为了提高泛化性,所以改用对比各个方法的参数量来进行间接说明计算时间。实验中采用的算法参数量如表1所示,实验中测试集平均定量结果如表2和表3所示,表中加粗字体为每列的最优值。
表 1 不同算法的参数量对比Table 1 Comparison of parameter quantities of different algorithms算法 参数量/106 U-Net 28.05 Swin Transformer 29.43 TransUnet 105.28 TransDeeplab 21.14 SwinUnet 27.17 TransNorm 117.63 本文算法 25.51 表 2 不同算法在Dataset BUSI上的分割结果Table 2 Segmentation results of different algorithms on Dataset BUSI算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.787 320 0.703 703 56.255 785 0.815 321 0.807 724 Swin Transformer 0.781 523 0.702 417 63.498 802 0.795 067 0.813 038 TransUnet 0.797 532 0.714 024 53.049 520 0.828 517 0.815 834 TransDeeplab 0.803 847 0.717 159 55.842 163 0.813 009 0.838 311 SwinUnet 0.804 722 0.720 576 59.828 857 0.817 851 0.836 466 TransNorm 0.799 316 0.719 628 51.095 965 0.827 717 0.814 157 本文算法 0.825 732 0.744 134 33.203 520 0.844 956 0.840 086 表 3 不同算法在Dataset B上的分割结果Table 3 Segmentation results of different algorithms on Dataset B算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.755 240 0.705 914 47.186 216 0.806 826 0.811 233 Swin Transformer 0.750 100 0.674 348 51.348 517 0.777 199 0.774 130 TransUnet 0.807 067 0.743 791 24.456 953 0.801 030 0.819 393 TransDeeplab 0.811 323 0.716 784 24.501 722 0.799 767 0.853 850 SwinUnet 0.806 981 0.700 275 30.370 696 0.783 342 0.818 952 TransNorm 0.781 180 0.700 323 24.035 719 0.799 767 0.853 850 本文算法 0.825 857 0.740 428 23.125 318 0.827 233 0.859 421 经过参数量对比,可以看出本文所提出的算法参数量少于除了TransDeeplab的其他经典算法。由于本文主要解决的问题是乳腺超声图像的分割精度问题,所以本文算法的运算量虽不是最优但在可接受的范围。
对比结果表明本文算法使用交叉注意力机制融合改进的CNN和ViT,对乳腺超声图像的分割结果都优于其他算法,各个指标相较于其他算法都有明显提升,和经典的U-Net算法相比dice系数提升了3.841 2%,这表明本文算法的有效性。在两个数据集上进行的实验结果可以证明本文提出算法的泛化性能,相较于其他算法更具备实际应用价值。实验中部分测试集的分割结果如图7所示,也可以看出采用混合边界损失对肿瘤边界的分割更准确。
2.5 消融实验
为了进一步说明本算法的有效性,使用数据量较大的Dataset BUSI进行5组消融实验,实验结果如表4所示。可以看出,单独使用U-Net的分割dice结果仅为0.787 320,接续添加可形变卷积(deformable convolution, DC)、混合损失函数(hybrid loss, HL)、交叉注意力机制(cross attention, CA)、多头跳跃注意力(multi-head skip self attention, MSKA)4个模块后分割结果各方面都有所提升,dice系数、Jc指数、准确度和召回率分别提升了3.841 2%、4.043 1%、2.963 5%和3.236 2%,其中DC模块的提升效果最明显,表明了DC、HL、CA、MSKA等4个模块对乳腺超声图像肿瘤分割的可行性。
表 4 在Dataset BUSI上的消融实验分割结果Table 4 Ablation experimental segmentation results on Dataset BUSI算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.787320 0.703703 56.255785 0.815321 0.807724 U-Net+DC 0.803680 0.722510 37.643451 0.829810 0.816059 U-Net+DC+HL 0.809869 0.726249 38.527125 0.840414 0.811717 U-Net+DC+HL+CA 0.819256 0.738401 34.681035 0.834446 0.808971 U-Net+DC+HL+CA+MSKA(本文算法) 0.825732 0.744134 33.203520 0.844956 0.840086 3. 结束语
本文提出一种融合CNN和ViT的乳腺超声图像肿瘤分割方法,该方法采用可形变卷积提升了CNN提取特征的能力。训练过程中采用二元交叉熵损失混合边界损失函数来优化提出的算法模型,对乳腺超声图像肿瘤区域的边界分割性能有所提升。对Swin Transformer的改进主要是将注意力机制从MSA改进为MSKA,从计算和自身的注意力转变为计算两个相邻特征图的注意力,更好地利用了注意力机制。最后使用交叉注意力机制建立来自CNN的局部特征和来自ViT的全局特征的有效融合。通过对比实验和消融实验验证了所提出方法的有效性,并且在各个指标中都优于现有的经典算法,在智能医疗辅助诊断中具有发展前景。本文存在的不足在于对于恶性肿瘤的分割效果不如良性肿瘤,而且若没有Ground Truth情况下,只能依靠专业医师进行主观评价。以后的研究工作可以考虑对肿瘤进行分类后再分割,无监督分割,或者进行多任务处理等。
-
表 1 不同算法的参数量对比
Table 1 Comparison of parameter quantities of different algorithms
算法 参数量/106 U-Net 28.05 Swin Transformer 29.43 TransUnet 105.28 TransDeeplab 21.14 SwinUnet 27.17 TransNorm 117.63 本文算法 25.51 表 2 不同算法在Dataset BUSI上的分割结果
Table 2 Segmentation results of different algorithms on Dataset BUSI
算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.787 320 0.703 703 56.255 785 0.815 321 0.807 724 Swin Transformer 0.781 523 0.702 417 63.498 802 0.795 067 0.813 038 TransUnet 0.797 532 0.714 024 53.049 520 0.828 517 0.815 834 TransDeeplab 0.803 847 0.717 159 55.842 163 0.813 009 0.838 311 SwinUnet 0.804 722 0.720 576 59.828 857 0.817 851 0.836 466 TransNorm 0.799 316 0.719 628 51.095 965 0.827 717 0.814 157 本文算法 0.825 732 0.744 134 33.203 520 0.844 956 0.840 086 表 3 不同算法在Dataset B上的分割结果
Table 3 Segmentation results of different algorithms on Dataset B
算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.755 240 0.705 914 47.186 216 0.806 826 0.811 233 Swin Transformer 0.750 100 0.674 348 51.348 517 0.777 199 0.774 130 TransUnet 0.807 067 0.743 791 24.456 953 0.801 030 0.819 393 TransDeeplab 0.811 323 0.716 784 24.501 722 0.799 767 0.853 850 SwinUnet 0.806 981 0.700 275 30.370 696 0.783 342 0.818 952 TransNorm 0.781 180 0.700 323 24.035 719 0.799 767 0.853 850 本文算法 0.825 857 0.740 428 23.125 318 0.827 233 0.859 421 表 4 在Dataset BUSI上的消融实验分割结果
Table 4 Ablation experimental segmentation results on Dataset BUSI
算法 dice系数 Jc指数 Hd95 准确度 召回率 U-Net 0.787320 0.703703 56.255785 0.815321 0.807724 U-Net+DC 0.803680 0.722510 37.643451 0.829810 0.816059 U-Net+DC+HL 0.809869 0.726249 38.527125 0.840414 0.811717 U-Net+DC+HL+CA 0.819256 0.738401 34.681035 0.834446 0.808971 U-Net+DC+HL+CA+MSKA(本文算法) 0.825732 0.744134 33.203520 0.844956 0.840086 -
[1] ZHENG Rongshou, ZHANG Siwei, ZENG Hongmei, et al. Cancer incidence and mortality in China, 2016[J]. Journal of the national cancer center, 2022, 2(1): 1–9. doi: 10.1016/j.jncc.2022.02.002 [2] 高艳多, 阎炯, 赵胜, 等. 1990—2019年中国女性乳腺癌发病和死亡趋势的年龄−时期−队列模型分析[J]. 中国预防医学杂志, 2022, 23(12): 909–916. GAO Yanduo, YAN Jiong, ZHAO Sheng, et al. Trends in incidence and mortality of female breast cancer in China from 1990 to 2019 using age-period-cohort analysis model[J]. Chinese preventive medicine, 2022, 23(12): 909–916. [3] XIAN Min, ZHANG Yingtao, CHENG H D, et al. Automatic breast ultrasound image segmentation: a survey[EB/OL]. (2017–04–04)[2023–04–24]. http://arxiv.org/abs/1704.01472. [4] 苏丽, 孙雨鑫, 苑守正. 基于深度学习的实例分割研究综述[J]. 智能系统学报, 2022, 17(1): 16–31. SU Li, SUN Yuxin, YUAN Shouzheng. A survey of instance segmentation research based on deep learning[J]. CAAI transactions on intelligent systems, 2022, 17(1): 16–31. [5] 施俊, 汪琳琳, 王珊珊, 等. 深度学习在医学影像中的应用综述[J]. 中国图象图形学报, 2020, 25(10): 1953–1981. doi: 10.11834/jig.200255 SHI Jun, WANG Linlin, WANG Shanshan, et al. Applications of deep learning in medical imaging: a survey[J]. Journal of image and graphics, 2020, 25(10): 1953–1981. doi: 10.11834/jig.200255 [6] 张宇, 梁凤梅, 刘建霞. 融合类激活映射和视野注意力的皮肤病变分割[J/OL]. 计算机工程与应用, 2022: 1–10. (2022–10–12) [2023–04–24]. https://kns.cnki.net/kcms/detail/11.2127.tp.20221011.1633.008.html. ZHANG Yu, LIANG Fengmei, LIU Jianxia. Skin lesion segmentation based on classification activation mapping and visual field attention[J/OL]. Computer engineering and applications, 2022: 1–10. (2022–10–12) [2023–04–24]. https://kns.cnki.net/kcms/detail/11.2127.tp.20221011.1633.008.html. [7] RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2015: 234–241. [8] ALMAJALID R, SHAN Juan, DU Yaodong, et al. Development of a deep-learning-based method for breast ultrasound image segmentation[C]//2018 17th IEEE International Conference on Machine Learning and Applications. Orlando: IEEE, 2018: 1103–1108. [9] CHEN Gongping, DAI Yu, ZHANG Jianxun. RRCNet: refinement residual convolutional network for breast ultrasound images segmentation[J]. Engineering applications of artificial intelligence, 2023, 117: 105601. doi: 10.1016/j.engappai.2022.105601 [10] HE Qiqi, YANG Qiuju, XIE Minghao. HCTNet: a hybrid CNN-transformer network for breast ultrasound image segmentation[J]. Computers in biology and medicine, 2023, 155: 106629. doi: 10.1016/j.compbiomed.2023.106629 [11] ZHUANG Zhemin, LI Nan, JOSEPH RAJ A N, et al. An RDAU-NET model for lesion segmentation in breast ultrasound images[J]. PLoS One, 2019, 14(8): e0221535. doi: 10.1371/journal.pone.0221535 [12] D’ASCOLI S, TOUVRON H, LEAVITT M L, et al. ConViT: improving vision transformers with soft convolutional inductive biases[J]. Journal of statistical mechanics: theory and experiment, 2022, 2022(11): 114005. doi: 10.1088/1742-5468/ac9830 [13] DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: transformers for image recognition at scale[EB/OL]. (2020–10–22) [2023–04–24]. http://arxiv.org/abs/2010.11929. [14] VALANARASU J M J, OZA P, HACIHALILOGLU I, et al. Medical transformer: gated axial-attention for medical image segmentation[C]//International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer, 2021: 36–46. [15] CHEN Jieneng, LU Yongyi, YU Qihang, et al. TransUNet: transformers make strong encoders for medical image segmentation[EB/OL]. (2021–02–08)[2023–04–24]. http://arxiv.org/abs/2102.04306. [16] ZHANG Yundong, LIU Huiye, HU Qiang. TransFuse: fusing transformers and CNNs for medical image segmentation[C]//Medical Image Computing and Computer Assisted Intervention-MICCAI 2021: 24th International Conference. Strasbourg: ACM, 2021: 14–24. [17] HEIDARI M, KAZEROUNI A, SOLTANY M, et al. HiFormer: hierarchical multi-scale representations using transformers for medical image segmentation[C]//2023 IEEE/CVF Winter Conference on Applications of Computer Vision. Waikoloa: IEEE, 2023: 6191–6201. [18] AL-DHABYANI W, GOMAA M, KHALED H, et al. Dataset of breast ultrasound images[J]. Data in brief, 2020, 28: 104863. doi: 10.1016/j.dib.2019.104863 [19] YAP M H, GOYAL M, OSMAN F, et al. Breast ultrasound region of interest detection and lesion localisation[J]. Artificial intelligence in medicine, 2020, 107: 101880. doi: 10.1016/j.artmed.2020.101880 [20] DAI Jifeng, QI Haozhi, XIONG Yuwen, et al. Deformable convolutional networks[C]//2017 IEEE International Conference on Computer Vision. Venice: IEEE, 2017: 764–773. [21] LIU Ze, LIN Yutong, CAO Yue, et al. Swin Transformer: hierarchical Vision Transformer using Shifted Windows[C]//2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021: 9992–10002. [22] CHEN C F R, FAN Quanfu, PANDA R. CrossViT: cross-attention multi-scale vision transformer for image classification[C]//2021 IEEE/CVF International Conference on Computer Vision. Montreal: IEEE, 2021: 347–356. [23] BOKHOVKIN A, BURNAEV E. Boundary loss for remote sensing imagery semantic segmentation[C]//International Symposium on Neural Networks. Cham: Springer, 2019: 388–401. [24] AZAD R, HEIDARI M, SHARIATNIA M, et al. TransDeepLab: convolution-free transformer-based DeepLab v3+ for medical image segmentation[C]//International Workshop on PRedictive Intelligence In MEdicine. Cham: Springer, 2022: 91–102. [25] CAO Hu, WANG Yueyue, CHEN J, et al. Swin-unet: unet-like pure transformer for medical image segmentation[C]//Computer Vision – ECCV 2022 Workshops. Tel Aviv: ACM, 2022: 205–218. [26] AZAD R, AL-ANTARY M T, HEIDARI M, et al. TransNorm: transformer provides a strong spatial normalization mechanism for a deep segmentation model[J]. IEEE access, 2022, 10: 108205–108215. doi: 10.1109/ACCESS.2022.3211501

 下载:
下载: