Prediction of glioma IDH1 mutation status based on AEViT and prior knowledge
-
摘要: 针对目前预测胶质瘤异柠檬酸脱氢酶1(isocitrate dehydrogenase1, IDH1)突变状态存在的数据不足、准确率较低等问题,提出一种基于AEViT(auto-encoder vision Transformer)与先验知识的胶质瘤IDH1突变状态预测方法。首先使用改进的K-Means聚类算法为无IDH1突变状态标签的胶质瘤磁共振成像(magnetic resonance imaging,MRI)数据标注伪标签,并采用ViT(vision Transformer)网络对伪标签进行修正,得到最终的胶质瘤IDH1突变状态。为避免不准确的伪标签数据影响模型精度,采用果蝇优化算法为伪标签数据赋予最优权重;然后提出基于Auto-Encoder和ViT的分类模型AEViT,利用Auto-Encoder提取胶质瘤MRI特征;再将特征输入ViT中对胶质瘤IDH1突变状态进行分类;最后将基于胶质瘤位置信息的先验知识加入模型,达到99.01%的预测准确率。结果表明该方法优于其他现有模型,能够实现胶质瘤数据扩增和术前无创、准确地预测胶质瘤IDH1突变状态,从而辅助诊疗过程。
-
关键词:
- 胶质瘤 /
- 异柠檬酸脱氢酶1 /
- K-Means聚类算法 /
- 伪标签 /
- Auto-Encoder /
- vision Transformer /
- 果蝇优化算法 /
- 先验知识
Abstract: Aiming at the problems of insufficient data and low accuracy in predicting the mutation status of brain glioma isocitrate dehydrogenase1 (IDH1), a prediction method for IDH1 mutation status of brain glioma is proposed based on auto-encoder vision Transformer (AEViT) and priori knowledge. Firstly, an improved K-Means clustering algorithm was used to label the pseudo-labels for MRI data of glioma without IDH1 status labels, and vision Transformer (ViT) network was used to modify the pseudo labels to obtain the final glioma IDH1 mutation status. In order to avoid inaccurate pseudo-label data that affect accuracy of the model, the fruit fly optimization algorithm was used to assign the optimal weight to the pseudo-label data. Secondly, a classification model AEViT based on Auto-Encoder and Vision Transformer was proposed, and Auto-Encoder was used to extract MRI features of glioma, and then the features were input into ViT to classify the IDH1 mutation status of glioma. Finally, the prior knowledge based on glioma location information was added to the model, which achieved a prediction accuracy of 99.01%. The experimental results show that this method is superior to other existing models, and can realize glioma data augmentation and non-invasive and accurate preoperative prediction of glioma IDH1 mutation status, thereby assisting the diagnosis and treatment process. -
胶质瘤[1]是一种常见的中枢神经系统肿瘤,是由于神经胶质细胞过度增殖所导致的[2]。胶质瘤分子标志物的表达水平对于胶质瘤的病理诊断至关重要[3-4],异柠檬酸脱氢酶1(isocitrate dehydrogenase1, IDH1)突变状态在胶质瘤分型中具有重要地位[5]。有研究表明[6-7]IDH1突变型患者治疗效果、总体生存率和预后比IDH1野生型患者更好,故在术前对IDH1突变状态进行精确的预测,对临床治疗和预后判断有重要价值。目前针对胶质瘤IDH1的预测多采用有创的方法,不仅会对患者身心造成不必要的伤害,而且容易得到假阴性的结果,导致患者错过最佳治疗时机[8]。因此术前依据医学影像无创分析胶质瘤基因突变状态具有重要意义。近年来深度学习已经被广泛用于脑肿瘤的辅助诊疗,Choi等[9]利用具有注意力机制的卷积长短期记忆模型来预测 IDH1基因型;多数研究需要首先进行胶质瘤分割,再对IDH1状态进行分类[10-11]。另一方面,具有IDH1突变状态标签的胶质瘤影像数据获取难度较高,而深度学习模型需要大量的真实数据进行训练以防止由于数据不足导致的模型过拟合。Safdar等[12]对低级别胶质瘤患者的磁共振(magnetic resonance,MR)图像依次进行垂直翻转、水平翻转、180°旋转、90°旋转、模糊、噪声、剪切、裁剪和缩放操作进行数据扩增。Liu等[13]使用基于生成对抗网络(generative adversarial network, GAN)进行胶质瘤数据扩增,预测IDH1状态的准确率得到提高。Li等[14]提出的TumorGAN可以从有限的配对数据中生成高质量的图像对,有效地提高了肿瘤分割效果。现有的基于深度学习的IDH1突变状态预测方法大多数都需要手动预分割肿瘤,这限制了临床的可行性;目前使用的数据扩增方法大多比较复杂。针对以上问题,本文提出了一种基于半监督AEViT(auto-encoder vision Transformer)的胶质瘤IDH1突变状态预测方法,以期实现术前无创、准确预测胶质瘤IDH1突变状态,避免数据浪费,进一步提高医生的工作效率,辅助诊疗过程。
1. 相关理论
1.1 K-Means聚类算法
K-Means聚类算法是目前应用最为广泛的聚类算法,其主要思想是:在给定K值和K个初始质心的情况下,通过计算样本与质心的距离把每个样本分到离其最近的质心所代表的类簇中,所有样本分配完毕之后,根据一个类簇内的所有样本重新计算该类簇的质心(取平均值),并迭代完成采样与质心更新,直到质心不再发生显著改变或达到预定迭代次数,迭代结束。对于样本集D={x1, x2 , ···, xm},K-Means聚类算法就是针对聚类划分C={C1, C2,···, Ck}最小化平方误差。
$$ E = \sum\limits_{i = 1}^k {\sum\limits_{\boldsymbol{x} \in {C_i}} {\left\| {\boldsymbol{x} - {\boldsymbol{u}_i}} \right\|_2^2} } $$ 式中簇Ci的均值向量ui为
$$ {{\boldsymbol{u}}_i} = \frac{1}{{\left| {{C_i}} \right|}}\sum\limits_{x \in {C_i}} \boldsymbol{x} $$ 1.2 伪标签
伪标签(pseudo-label)[15]是一种利用已有标注数据的信息在无标注数据上生成伪标签,进而进行模型训练的半监督学习方法。伪标签方法的基本思路是:首先利用已有标注数据上训练出一个初步模型;再利用该模型对无标注数据进行预测,并将预测结果作为伪标签添加到数据集中;最后利用带有伪标签数据的扩充数据集再次训练模型,并重复以上过程,直到模型性能收敛。该方法的优点在于可以有效地利用无标注数据进行模型训练,从而提高模型的性能和泛化能力[16]。
1.3 Auto-Encoder
Auto-Encoder是一种利用反向传播算法使得输出值等于输入值的神经网络,在图像去噪和模型效率方面表现出良好的实用性和鲁棒性,非常适合作为医学图像处理过程中的特征提取器。使用Auto-Encoder能够学习到数据中重要的特征。它包含编码器(encoder)和解码器(decoder)[17]2个主要的部分。先利用编码器将输入压缩成潜在的空间表征,再利用解码器来重构来自潜在空间表征的输入,不断优化使输入和输出尽可能相似。
1.4 ViT
Transformer[18]是一种使用自注意力机制来替代传统卷积操作的新型神经网络。Dosovitskiy等[19]提出的ViT(vision Transformer)是一种基于 Transformer的网络架构。ViT将 Transformer应用于图像识别任务,并在图像分类和物体检测等任务中取得了显著的成绩。ViT的核心流程包括图像分块处理、图像块嵌入与位置编码、Transformer编码和多层感知机(multilayer perceptron,MLP)分类处理。
2. 基于半监督AEViT的胶质瘤IDH1突变状态预测模型
2.1 基于改进K-Means聚类算法与ViT的伪标签标注
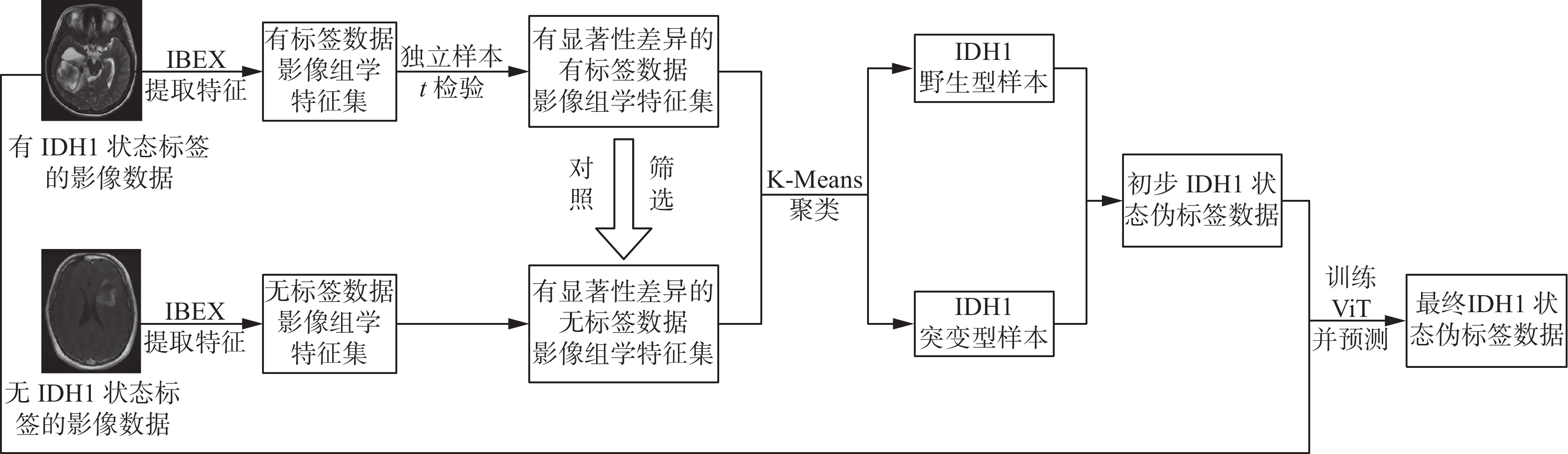
对深度神经网络的训练必须有足够多的标注样本,但目前可获得的医学影像数据缺少标注,现有的数据集未被全面利用。因此为避免数据浪费,本文利用人工智能技术构建了基于改进K-Means聚类算法与ViT的伪标签标注方法,为无标签的胶质瘤影像数据添加伪标签[20]。
首先将胶质瘤影像T1c序列导入IBEX[21]软件,提取影像组学特征,包括基于灰度共生矩阵[22]和灰度游程矩阵[23]的特征。利用SPSS软件对上述特征进行独立样本t检验,剔除不具有显著性差异的特征。在此基础上,对照筛选出有显著性差异的无标签数据影像组学特征集。本文通过一种改进的K-Means聚类算法,将所有特征集聚成2类。将IDH1突变型和野生型样本作为初始质心,进行迭代聚类,与突变型质心聚为一类的样本被预测为突变型,与野生型质心聚为一类的样本则被预测为野生型。初步预测的伪标签可能会存在一定的误差,因此使用ViT模型对通过聚类得到的伪标签进行修正,将有标签数据和初步伪标签数据共同送入ViT中训练并做进一步预测,得到最终的IDH1状态伪标签数据,如图1所示。
2.2 基于果蝇优化算法的伪标签数据权重优化
2.2.1 果蝇优化算法
果蝇优化算法[24](fruit fly optimization algorithm, FOA)是一种基于仿生学的优化算法,模拟果蝇在寻找食物的过程中的行为,以寻找最优解。该算法在多个优化问题中取得了良好的优化效果[25-26],并且具有较高的鲁棒性和可靠性[27]。果蝇优化算法的具体步骤如下:
1)初始化种群:随机生成一组果蝇个体的位置和速度,每个个体代表一个潜在解。
2)评估适应度:对于每个个体,根据其解的质量评估其适应度。适应度函数通常是目标函数,其值越小表示解越优。
3)更新果蝇位置:通过模拟果蝇在食物附近的活动,更新每个个体的位置。这可以通过随机生成新的位置,或通过调整当前位置来实现。
4)计算局部最优解:对于每个果蝇,计算其周围的果蝇的适应度,并选择其中适应度最好的个体作为其局部最优解。
5)计算全局最优解:选择所有果蝇中适应度最好的个体作为全局最优解。
6)更新种群:根据全局最优解和每个果蝇的局部最优解来更新种群,以便下一代果蝇可以更好地探索解空间。
7)判断终止条件:如果达到预设的终止条件,则算法停止,否则返回步骤3)。
通过重复上述步骤,果蝇优化算法可以寻找最优解。
2.2.2 果蝇算法优化伪标签数据权重
本研究利用半监督学习为无标签胶质瘤数据标注的IDH1突变状态伪标签可能存在误差,为降低不准确标签对模型的预测能力产生的不良影响,本研究采用果蝇优化算法,使模型能够自适应地调整训练期间伪标签数据的权重,避免不准确伪标签数据影响模型精度和稳定性。在模型训练过程中,使用交叉熵误差(cross entropy error,CEE)作为损失函数,表示为
$$ {L_{\text{c}}} = - [{y_i}\log (f({x_i}) + (1 - {y_i})\log (1 - f({x_i}))] $$ 假设胶质瘤影像数据集共有N个数据,包括Nk个有标签数据,即
$$ {X_k} = \{ ({x_1},{y_1}),({x_2},{y_2}), \cdots ,({x_{{N_k}}},{y_{{N_k}}})\} $$ 以及(N - Nk)个伪标签数据,即
$$ {X_l} = \{ ({x_{{N_{k + 1}}}},{y_{{N_{k + 1}}}}),({x_{{N_{k + 2}}}},{y_{{N_{k + 2}}}}), \cdots ,({x_N},{y_N})\} $$ 则训练期间的损失函数表示为
$$ {L_{}} = \sum\limits_{i = 1}^N {{L_c}(f({x_i}),{y_i})} $$ 通过固定真实标签数据的权重为1,为伪标签数据施加不同的权重来调整损失函数。由于有标签数据和伪标签数据具有不同重要度,因此损失函数可以重新表示为
$$ {L'_{}} = \sum\limits_{i = 1}^{{N_k}} {{L_c}(f({x_i}),{y_i})} + \sum\limits_{i = {N_k} + 1}^{{N_{}}} {{L_c}(\alpha f({x_i}),{y_i})} $$ 式中
$ \alpha $ 为施加在伪标签数据上的权重。本研究利用果蝇优化算法寻找训练过程中伪标签数据的最优权重,计算
$ \alpha $ 的最优解。基于果蝇优化算法的伪标签数据权重优化算法如算法1所示。算法1 基于果蝇优化算法的伪标签数据权重优化算法
输入 损失函数
$ {L'_{}} $ ,果蝇初始位置Xaxis、Yaxis,果蝇种群规模(popsize)P,最大迭代次数(maxgen)M,果蝇飞行半径R,优化变量个数D;输出 最佳味道浓度bsmell,伪标签数据权重
$ \alpha $ 的最优解。1)Xaxis = rand(1, 1),Yaxis = rand(1, 1);
2)根据
$ {L'_{}} $ 计算味道浓度函数,并在味道浓度Smell中记录;3)[bsmell bindex]=min(smell)S;
4)Sbest = bsmell;
5)Xaxis = X(bindex),Yaxis = Y(bindex);
6)
$ \alpha $ = 1/Dist(Xaxis , Yaxis);7)for i in range((maxgen)M) do
8)for j in range((popsize)P) do
[bsmell bindex]=min(smell)S;
9) if Sbest < bsmell then
10)X(bindex)→Xaxis;
11)Y(bindex)→Yaxis;
12)Sbest→bsmell;
13)
$ \alpha $ = 1/Dist(Xaxis, Yaxis);14)end if;
15)end;
16)return bsmell,
$ \alpha $ 。2.3 基于AEViT与先验知识的胶质瘤IDH1突变状态预测模型
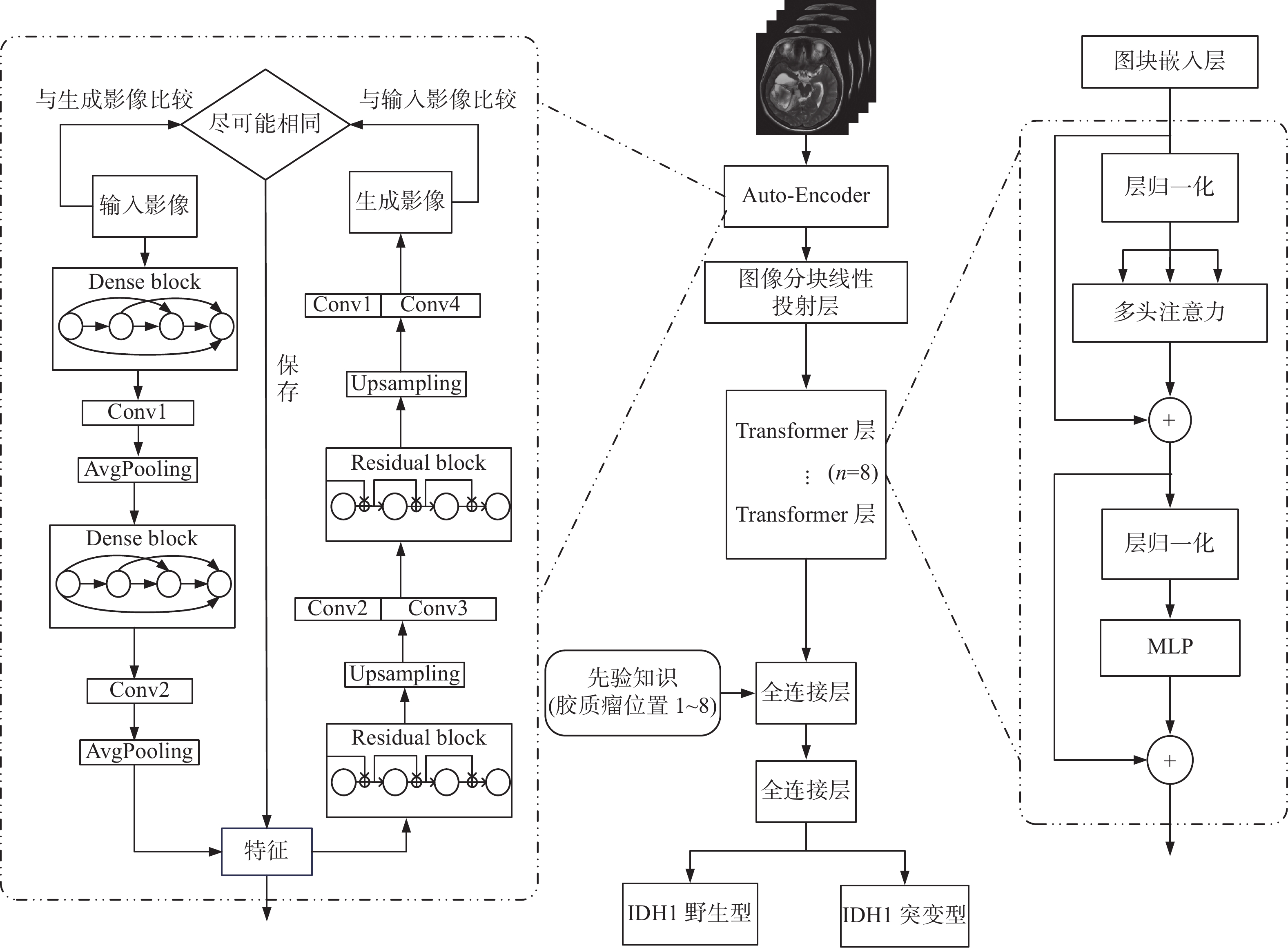
近年来,ViT展现了出色的图像分类性能,ViT的自注意机制在图像特征提取方面有着明显的优越性。ViT将图片拆分成很多小块,然后将小块排列成矩阵送入Transformer encoder模块中计算,可以捕获长距离依赖关系。但是ViT更加关注全局信息,从而会不可避免地丢失一些低级特征。而具有优异特征提取性能的Auto-Encoder更关注局部特征,它能够自适应地获得更重要的特征,实现高级特征和低级特征的融合,在一定程度上避免了信息丢失,提高了模型的准确性。Auto-Encoder的优点是可以比较生成的图像和原始的图像,两者越是相像,则说明编码器部分提取到的特征越具有代表性。因此本研究将Auto-Encoder与ViT融合,构建了基于AEViT与先验知识的胶质瘤IDH1突变状态分类模型,如图2。
首先使用Auto-Encoder进行特征提取,然后将提取出的特征图输入到ViT中进行分类预测,以弥补ViT在关注局部特征方面的不足;再将胶质瘤位置信息作为先验知识加入全连接层;最后进行IDH1突变状态预测。本研究提出的AEViT模型结合了Auto-Encoder和ViT共2种结构的优势,Auto-Encoder能够提取出更具有代表性的特征,ViT能够充分利用自注意力机制加强模型的特征提取能力。而加入先验知识可以使模型学习到更为关键的特征,从而实现更准确的胶质瘤IDH1突变状态预测。
首先使用Auto-Encoder对胶质瘤图像进行特征提取,它由基于Dense block的编码器和基于Residual block的解码器组成。编码器由2个Dense block组成,模块之间使用1×1卷积层和2×2 AvgPooling层进行连接并完成下采样。使用Dense block可以帮助减少参数数量,同时增强特征重用以加强有效特征的传播。解码器由2个Residual block组成并且依靠上采样层来增加特征图的尺寸,然后将其拼接到编码器对应特征图上。Residual block的使用可以有效加深网络的深度,同时避免梯度消失,从而获得更好的图像重建效果。本研究将胶质瘤图像传入编码器提取特征,再经由解码器将其还原。生成的图像是通过对编码器提取的特征进行解码而获得的,因此将生成影像与原始输入图像进行像素级比较,最终生成的影像与原始图像越相似,损失越小,证明提取的特征越具有代表性。
接下来将Auto-Encoder提取出的特征图输入到ViT中,对特征图进行分块处理,展平每个patch,并对展平后的patch做线性变换,将其映射到D维嵌入空间。然后将输入向量传递给Transformer层,其中的多头注意力机制不会限制专注一个或多个特定位置而影响对其他同等重要位置的注意的能力,它通过赋予注意力层不同的表征子空间来实现,可以用来提高自注意层的表现。
深度神经网络是个黑盒子,虽然能够自动学习到一些可区分度好的特征,但是往往会拟合到一些非重要特征,如果给模型加入先验知识就会让模型学习到更加关键的特征,因此本研究引入了基于胶质瘤位置信息的先验知识。目前已有研究[28-29]证明了胶质瘤位置与IDH1之间的相关性,例如IDH1突变的胶质瘤多数位于额叶,IDH1野生型肿瘤多数位于右半球的基底节区。本研究将胶质瘤的占位划分为8种位置[30],以1~8的形式将先验知识输入模型的全连接层,再进行IDH1状态预测。
3. 实验结果与分析
3.1 数据集
本研究使用的数据集来自当地医院,图像均为患者术前扫描的T1c增强胶质瘤磁共振成像(ma-gnetic resonance imaging,MRI),格式为DICOM。最终数据集包括422名患者的
7596 张影像,其中有IDH1突变状态标签的有313名患者的5634 张影像。另外109名患者的1962 张影像无IDH1突变状态标签。本实验对图像进行了归一化和重采样处理,尺寸调整为256 像素×256 像素。训练过程中,训练集和测试集按8∶2进行划分。3.2 基于K-Means聚类算法与ViT的伪标签标注结果
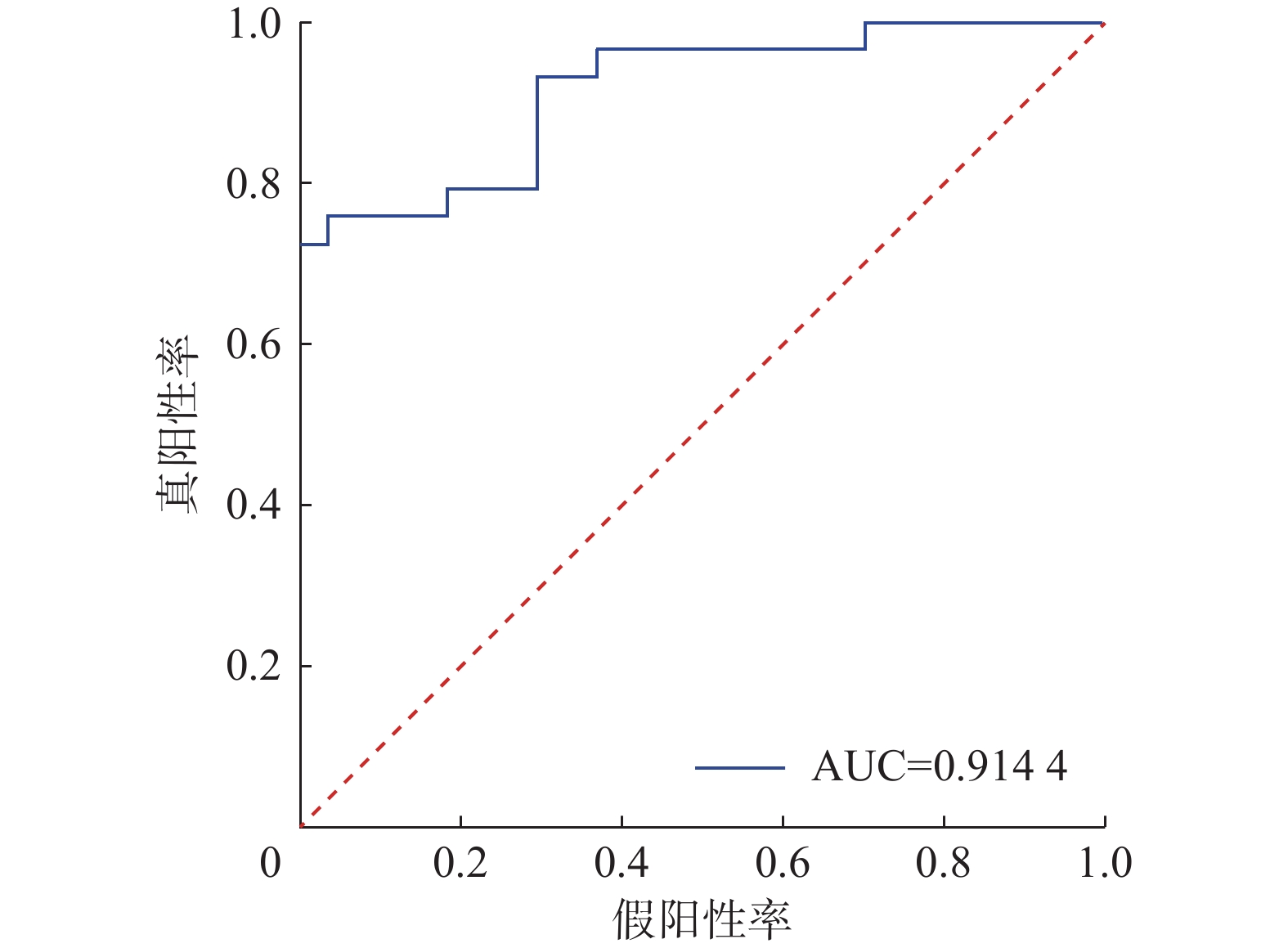
本研究利用IBEX软件对胶质瘤影像T1c序列进行特征提取,最终得到363个影像组学特征,包括330个基于灰度共生矩阵的特征、33个基于灰度游程矩阵的特征。对上述特征进行独立样本t检验,当P(不接受原假设的最小的显著性水平)<0.05时,证明该特征具有显著性差异。从而剔除了不具有显著性差异的特征。在此基础上,对照筛选出有显著性差异的94个特征,再将有标签的数据的特征集和无标签数据特征集共同进行K-Means聚类。最后使用ViT模型来校正聚类得到的伪标签,伪标签预测准确率达到83.93%,灵敏度70.96%,特异度96.83%,AUROC为
0.9144 (95%CI),其受试者工作特征(receiver operating characteristic,ROC)曲线如图3所示。最终得到了109例无IDH1状态标签数据的伪标签,包括97例IDH1野生型和12例IDH1突变型。使用本研究提出的基于K-Means聚类算法与ViT的伪标签标注方法更好地利用了有限的胶质瘤图像数据集,从而实现更高质量的胶质瘤IDH1突变状态预测。3.3 基于果蝇优化算法的伪标签数据权重优化结果
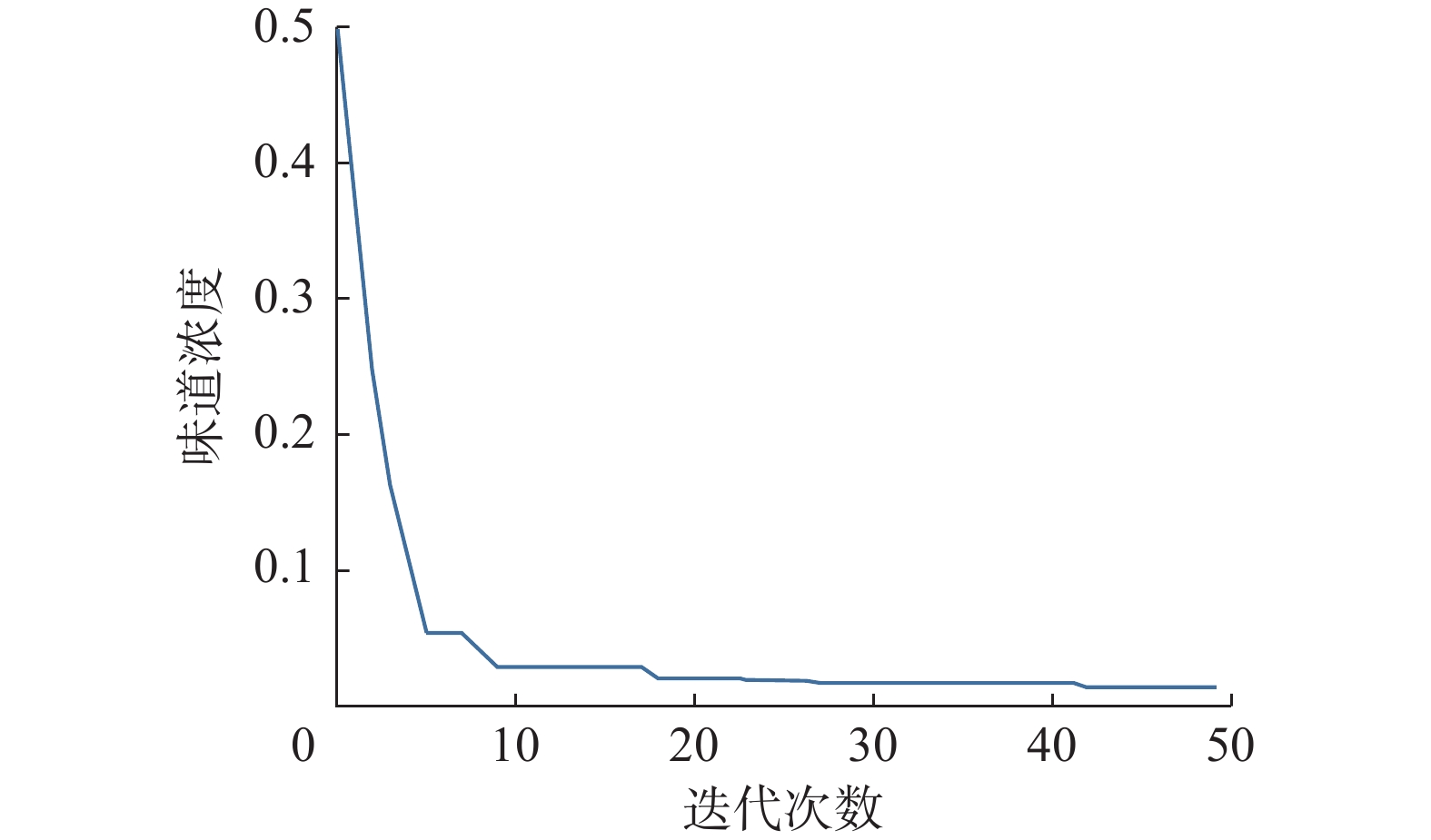
本实验固定真实标签数据的权重为1,通过果蝇优化算法调整伪标签数据的权重。在果蝇优化算法寻优过程中,将果蝇种群规模(popsize)P设为5,迭代次数(maxgen)M设为50,半径R设为1,优化变量个数D设为1。将
$ {L'_{}} $ 作为输入以不断优化$ \alpha $ 的值,最终当味道浓度值达到最优时结束优化。最终味道浓度值为0.014,此时伪标签数据的权重达到最优,$ \alpha $ 值为0.12。果蝇优化算法的寻优过程如图4所示。3.4 基于AEViT与先验知识的胶质瘤IDH1突变状态预测结果
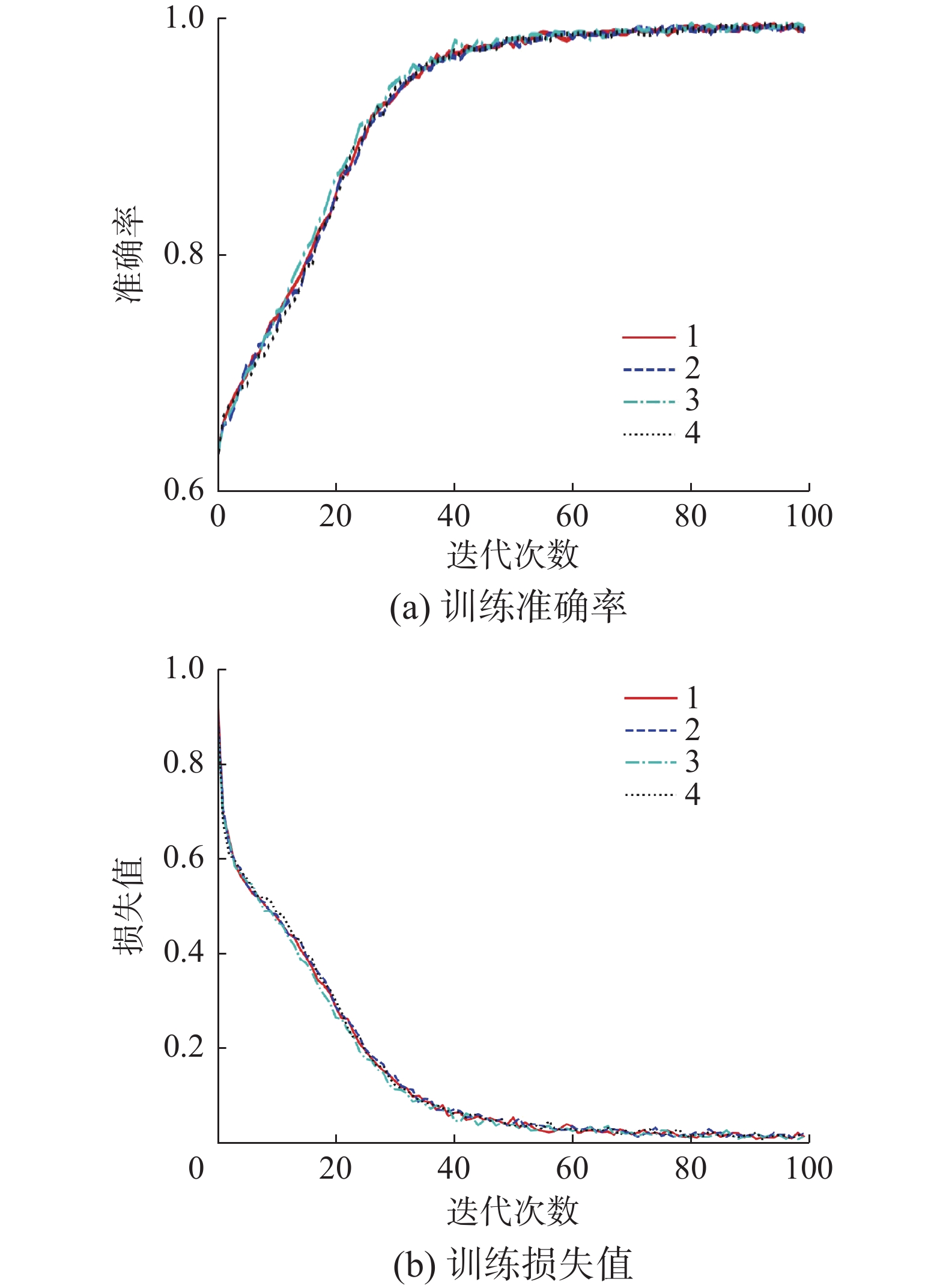
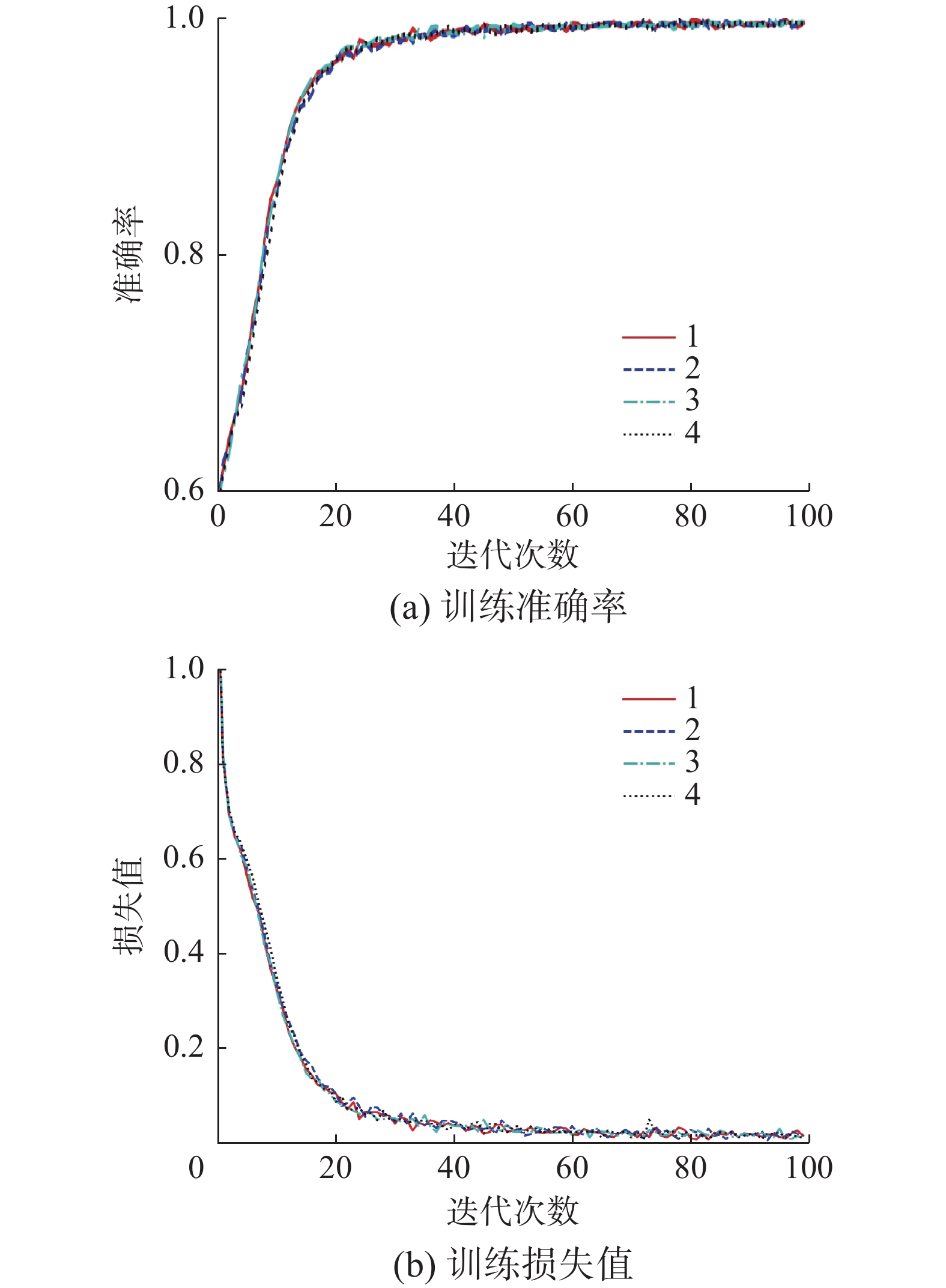
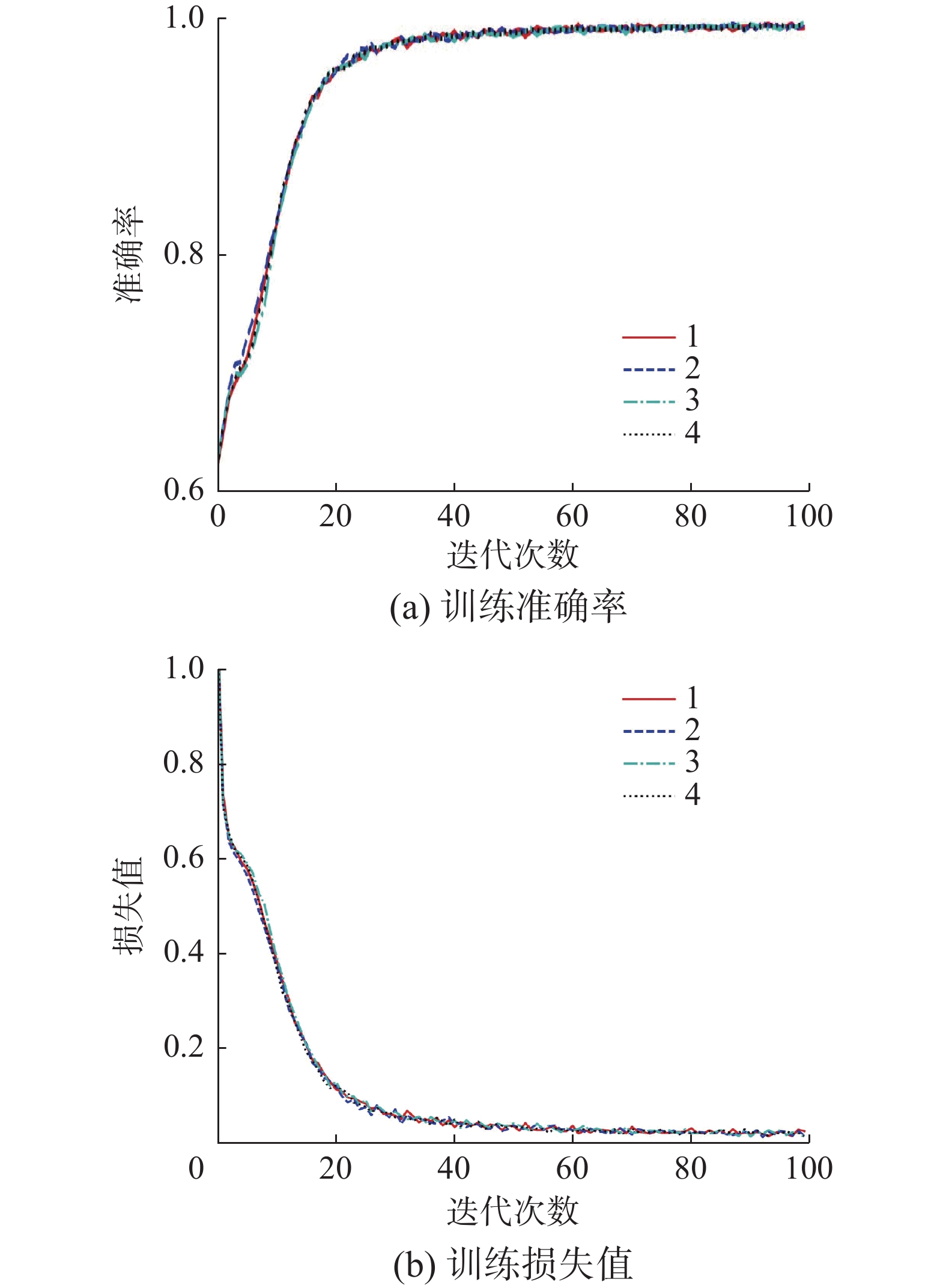
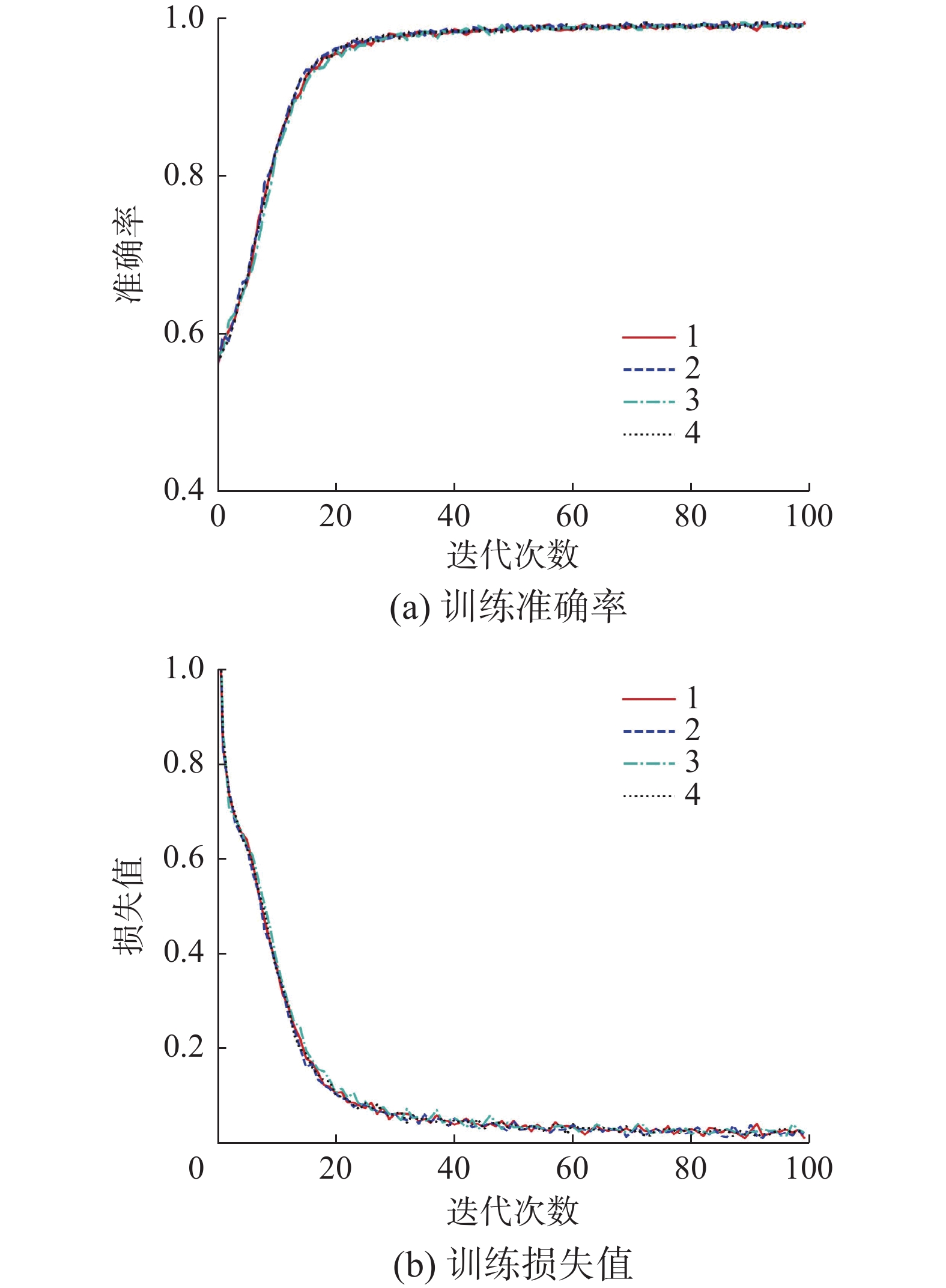
本实验训练了4种不同的模型来证明所提方法的优越性,首先是使用含伪标签数据的数据集训练,同时使用果蝇优化算法进行优化,并加入先验知识的AEViT模型,然后依次去除先验知识、果蝇算法和伪标签数据获得其他3种模型,训练过程如图5~8所示。
本实验将上述4种模型进行训练,并利用20%的有IDH1状态标签的影像数据进行测试,每个实验重复4次,并计算出平均值和标准差,结果如表1所示。实验结果表明:使用含伪标签数据的数据集训练,同时使用果蝇优化算法进行优化,并加入先验知识的AEViT模型,其胶质瘤IDH1突变状态分类平均准确率为99.01%;使用“含伪标签数据的数据集”训练和果蝇优化算法的AEViT模型,其分类平均准确率为98.82%;仅使用含伪标签数据集训练的AEViT模型,其分类平均准确率为98.05%;使用真实标签数据的数据集训练的AEViT模型,其分类平均准确率为97.96%。可以看出,使用含伪标签数据的数据集训练,同时使用果蝇优化算法进行优化,并加入基于胶质瘤位置信息的先验知识的AEViT模型精度最高。采用果蝇优化算法进行伪标签数据权重优化大大加快了模型收敛速度,提高模型分类速度的同时也能提高模型准确率。
表 1 4种模型测试结果Table 1 Test results of the four models模型 准确率/% t/s 含伪标签数据+果蝇+先验知识-AEViT 99.01±0.33 634 含伪标签数据+果蝇-AEViT 98.82±0.72 612 含伪标签数据-AEViT 98.05±0.48 585 真实标签数据-AEViT 97.96±0.58 526 3.5 模型性能比较
本文使用多个经典的分类模型对胶质瘤IDH1突变状态进行预测,展示了不同模型在测试集上预测IDH1突变状态的结果,如表2所示。可以看出,本文提出的模型在训练时间与其他模型相似甚至更短的情况下,分类准确率更高。
表 2 不同模型预测IDH1突变状态结果对比Table 2 Comparison of different models for predicting IDH1 mutation status模型 准确率/% t/s VGG 69.23 527 ResNet 75.58 541 DenseNet 77.83 557 CRNN 77.23 616 GoogleNet+SVM 81.11 646 U-shaped+ResNet 86.96 911 FCNN+LSTM 88.05 1029 ViT 83.93 765 AEViT(真实标签数据) 97.96 526 AEViT(含伪标签数据) 98.05 585 AEViT(含伪标签数据+果蝇优化算法) 98.82 612 AEViT(含伪标签数据+果蝇优化算法+
先验知识)99.01 634 4. 结束语
本文首先使用基于改进K-Means聚类算法与ViT的伪标签标注方法进行胶质瘤数据扩增;然后使用果蝇优化算法对伪标签数据的权重进行优化;最后使用基于AEViT与先验知识的胶质瘤IDH1突变状态分类模型进行预测。实验结果显示,本文提出的基于AEViT与先验知识的胶质瘤IDH1突变状态预测模型达到了99.01%的分类准确率。本文提出方法的分类性能优于其他经典的分类模型,能够更加精准地术前预测胶质瘤IDH1突变状态,对胶质瘤的诊疗具有重要的指导意义。
-
表 1 4种模型测试结果
Table 1 Test results of the four models
模型 准确率/% t/s 含伪标签数据+果蝇+先验知识-AEViT 99.01±0.33 634 含伪标签数据+果蝇-AEViT 98.82±0.72 612 含伪标签数据-AEViT 98.05±0.48 585 真实标签数据-AEViT 97.96±0.58 526 表 2 不同模型预测IDH1突变状态结果对比
Table 2 Comparison of different models for predicting IDH1 mutation status
模型 准确率/% t/s VGG 69.23 527 ResNet 75.58 541 DenseNet 77.83 557 CRNN 77.23 616 GoogleNet+SVM 81.11 646 U-shaped+ResNet 86.96 911 FCNN+LSTM 88.05 1029 ViT 83.93 765 AEViT(真实标签数据) 97.96 526 AEViT(含伪标签数据) 98.05 585 AEViT(含伪标签数据+果蝇优化算法) 98.82 612 AEViT(含伪标签数据+果蝇优化算法+
先验知识)99.01 634 -
[1] FU Jie, SINGHRAO K, ZHONG Xinran, et al. An automatic deep learning-based workflow for glioblastoma survival prediction using preoperative multimodal MR images: a feasibility study[J]. Advances in radiation oncology, 2021, 6(5): 100746. doi: 10.1016/j.adro.2021.100746 [2] ZHANG Maolei, HUANG Nunu, YANG Xuesong, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J]. Oncogene, 2018, 37(13): 1805–1814. doi: 10.1038/s41388-017-0019-9 [3] LOUIS D N, PERRY A, REIFENBERGER G, et al. The 2016 world health organization classification of tumors of the central nervous system: a summary[J]. Acta neuropathologica, 2016, 131(6): 803–820. doi: 10.1007/s00401-016-1545-1 [4] CHEN Xi, YAN Yuanliang, ZHOU Jianhua, et al. Clinical prognostic value of isocitrate dehydrogenase mutation, O-6-methylguanine-DNA methyltransferase promoter methylation, and 1p19q co-deletion in glioma patients[J]. Annals of translational medicine, 2019, 7(20): 541. doi: 10.21037/atm.2019.09.126 [5] LOUIS D N, ARIE P, PIETER W, et al. The 2021 WHO classification of tumors of the central nervous system: a summary[J]. Neuro-oncology, 2021, 23(8): 1231–1251. doi: 10.1093/neuonc/noab106 [6] MILLER J J, SHIH H A, ANDRONESI O C, et al. Isocitrate dehydrogenase-mutant glioma: evolving clinical and therapeutic implications[J]. Cancer, 2017, 123(23): 4535–4546. doi: 10.1002/cncr.31039 [7] 李锐, 马林. IDH基因突变与胶质瘤相关性的研究进展[J]. 中国医学影像学杂志, 2020, 28(2): 142–145. doi: 10.3969/j.issn.1005-5185.2020.02.017 LI Rui, MA Lin. Research progress on the correlation between IDH gene mutation and glioma[J]. China journal of medical imaging, 2020, 28(2): 142–145. doi: 10.3969/j.issn.1005-5185.2020.02.017 [8] WANG Shuo, SHI Jingyun, YE Zhaoxiang, et al. Predicting EGFR mutation status in lung adenocarcinoma on computed tomography image using deep learning[J]. European respiratory journal, 2019, 53(3): 1800986. doi: 10.1183/13993003.00986-2018 [9] CHOI K S, CHOI S H, JEONG B. Prediction of IDH genotype in gliomas with dynamic susceptibility contrast perfusion MR imaging using an explainable recurrent neural network[J]. Neuro-oncology, 2019, 21(9): 1197–1209. doi: 10.1093/neuonc/noz095 [10] BANGALORE Y C, SHAH B R, VEJDANI-JAHROMI M, et al. A novel fully automated MRI-based deep-learning method for classification of IDH mutation status in brain gliomas[J]. Neuro-oncology, 2020, 22(3): 402–411. doi: 10.1093/neuonc/noz199 [11] CHOI Y S, BAE S, CHANG J H, et al. Fully automated hybrid approach to predict the IDH mutation status of gliomas via deep learning and radiomics[J]. Neuro-oncology, 2021, 23(2): 304–313. doi: 10.1093/neuonc/noaa177 [12] SAFDAR M F, ALKOBAISI S S, ZAHRA F T. A comparative analysis of data augmentation approaches for magnetic resonance imaging (MRI) scan images of brain tumor[J]. Acta informatica medica, 2020, 28(1): 29–36. doi: 10.5455/aim.2020.28.29-36 [13] LIU Sidong, SHAH Z, SAV A, et al. Isocitrate dehydrogenase (IDH) status prediction in histopathology images of gliomas using deep learning[J]. Scientific reports, 2020, 10(1): 7733. doi: 10.1038/s41598-020-64588-y [14] LI Qingyun, YU Zhibin, WANG Yubo, et al. TumorGAN: a multi-modal data augmentation framework for brain tumor segmentation[J]. Sensors, 2020, 20(15): 4203. doi: 10.3390/s20154203 [15] LEE D. Pseudo-Label : the simple and efficient Semi-Supervised learning method for deep neural networks[C]//ICML 2013 Workshop : Challenges in Representation Learning. Atlanta: ACM, 2013, 3(2): 896. [16] XIE Qizhe, LUONG M T, HOVY E, et al. Self-training with noisy student improves imageNet classification[C]// 2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition. Seattle: IEEE, 2020: 10684−10695. [17] CHEN Dongdong, YUAN Lu, LIAO Jing, et al. Explicit filterbank learning for neural image style transfer and image processing[J]. IEEE transactions on pattern analysis and machine intelligence, 2021, 43(7): 2373–2387. doi: 10.1109/TPAMI.2020.2964205 [18] VASWANI Ashish, SHAZEER Noam, PARMAR Niki, et al. Attention is all you need[EB/OL]. (2017−06−12)[2022−07−21]. https://arxiv.org/abs/1706.03762. [19] DOSOVITSKIY A, BEYER L, KOLESNIKOV A, et al. An image is worth 16x16 words: transformers for image recognition at scale[EB/OL]. (2020−10−22)[2022−07−21]. https://arxiv.org/abs/2010.11929. [20] 徐华畅, 许倩, 赵钰琳, 等. 基于改进EfficientNetV2的脑胶质瘤IDH1突变状态预测方法[J]. 山东大学学报(理学版), 2023, 58(7): 60–66. XU Huachang, XU Qian, ZHAO Yulin, et al. Prediction method of IDH1 mutation status of glioma based on improved EfficientNetV2[J]. Journal of Shandong University (science edition), 2023, 58(7): 60–66. [21] ZHANG Lifei, FRIED D V, FAVE X J, et al. Ibex: an open infrastructure software platform to facilitate collaborative work in radiomics[J]. Medical physics, 2015, 42(3): 1341–1353. doi: 10.1118/1.4908210 [22] 刘光宇, 黄懿, 曹禹, 等. 基于灰度共生矩阵的图像纹理特征提取研究[J]. 科技风, 2021(12): 61–64. LIU Guangyu, HUANG Yi, CAO Yu, et al. Research on image texture feature extraction based on grayscale cooccurrence matrix[J]. Technology wind, 2021(12): 61–64. [23] 王铭, 田为中, 张继, 等. 基于TIRM序列的游程矩阵纹理特征联合ADC值预测乳腺癌ki-67表达水平[J]. 放射学实践, 2021, 36(12): 1520–1525. WANG Ming, TIAN Weizhong, ZHANG Ji, et al. Prediction of ki-67 expression level in breast cancer based on run-length matrix texture feature combined with ADC value based on TIRM sequence[J]. Radiology practice, 2021, 36(12): 1520–1525. [24] PAN Wen-tsao. A new fruit fly optimization algorithm: taking the financial distress model as an example[J]. Knowledge-based systems, 2012, 26: 69–74. doi: 10.1016/j.knosys.2011.07.001 [25] 陈璟, 虞继敏. 基于果蝇—广义回归神经网络优化的wsn节点定位算法[J]. 南京师大学报(自然科学版), 2017, 40(2): 31–38. CHEN Jing, YU Jimin. A wsn node localization algorithm based on drosophila-generalized regression neural network optimization[J]. Journal of Nanjing Normal University (natural science edition), 2017, 40(2): 31–38. [26] 彭启伟, 罗旺, 冯敏, 等. 改进二维otsu法和果蝇算法结合的图像分割方法[J]. 计算机应用, 2017, 37(S2): 193–197. PENG Qiwei, LUO Wang, FENG Min, et al. Improved image segmentation method by combining two-dimensional otsu method and fruit fly algorithm[J]. Computer applications, 2017, 37(S2): 193–197. [27] ZHU Hong, HE Hanzhi, XU Jinhui, et al. Medical image segmentation using fruit fly optimization and density peaks clustering[J]. Computational and mathematical methods in medicine, 2018, 2018: 1–11. [28] WIJNENGA M, VOORT S, FRENCH P J, et al. Differences in spatial distribution between WHO 2016 low-grade glioma molecular subgroups[J]. Neuro-oncology advances, 2019, 1(1): z1. [29] SONODA Y, SHIBAHARA I, KAWAGUCHI T, et al. Association between molecular alterations and tumor location and MRI characteristics in anaplastic gliomas[J]. Brain tumor pathology, 2015, 32(2): 99–104. doi: 10.1007/s10014-014-0211-3 [30] 徐芊芊, 许倩, 徐华畅, 等. 基于CnViT的胶质瘤IDH1突变状态智能预测方法[J]. 山东大学学报(工学版), 2023, 53(2): 127–134. XU Qianqian, XU Qian, XU Huachang, et al. Intelligent prediction of glioma IDH1 mutation status based on CnViT[J]. Journal of Shandong University (engineering edition), 2023, 53(2): 127–134.



 下载:
下载: