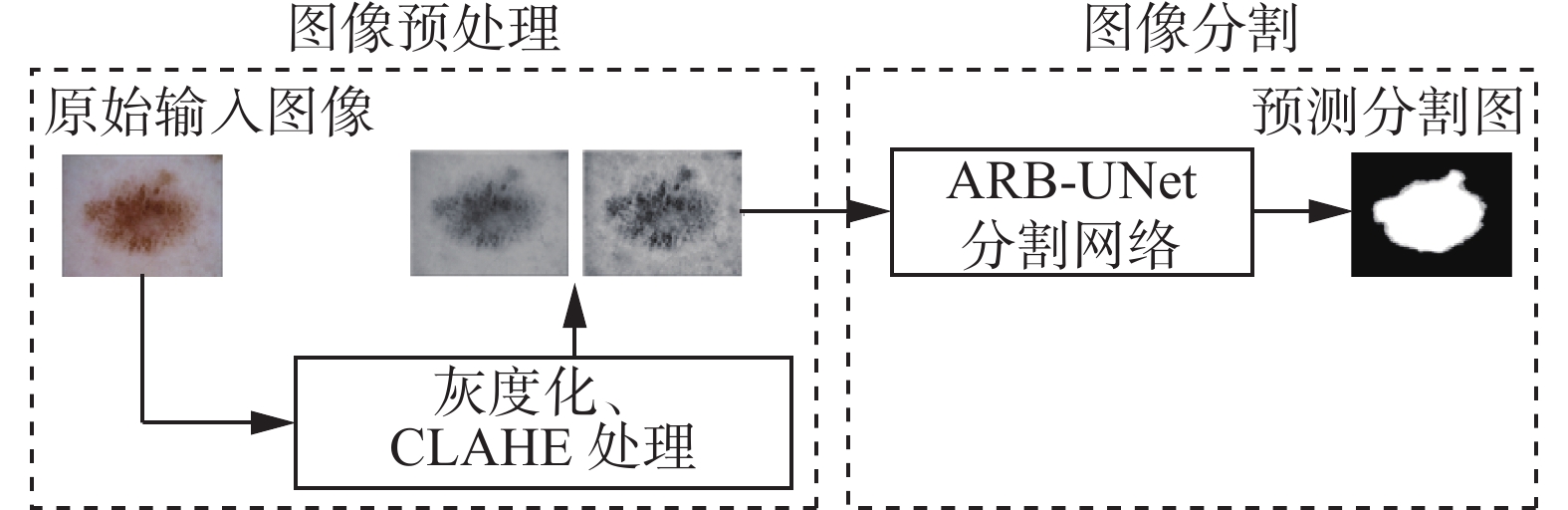
Dermoscope image segmentation method based on ARB-UNet
-
摘要: 针对皮肤镜图像类内差异性、类间相似性、数据集不平衡等问题,本文提出了一种基于注意力残差U-Net(attention residual block-UNet,ARB-UNet)的皮肤镜图像分割方法。将卷积块注意力机制模块(convolutional block attention module,CBAM)引入到U-Net模型的“跳过连接”中;同时将CBAM模块集成到残差模块DRB(dilated residual networks)中得到注意力残差结构(attention residual block, ARB);且选取Focal Tversky Loss作为该模型的损失函数;在ISIC2016数据集上对所提ARB-UNet模型进行训练和测试,并与传统方法和U-Net等经典方法进行了对比实验,实验结果中灵敏度(sensitivity,SE)达到了92.9%,特异性(specificity,SP)达到了94.1%,Dice相似指数(dice similarity cofficient,DSC)达到了92.1%,整体上均优于其他对比方法,从而验证了本文方法是有效的和可行的。
-
关键词:
- 图像分割 /
- 皮肤镜 /
- 卷积神经网络 /
- 注意力残差 U-Net /
- 注意力机制 /
- 卷积块注意力机制模块 /
- 深度学习 /
- 残差网络
Abstract: Aiming at the problems of intra-class difference, inter-class similarity, and dataset imbalance in dermoscopic images, a dermoscopic image segmentation method based on attention residual block-UNet (ARB-UNet) is proposed. Firstly, the convolutional block attention module (CBAM) is introduced into the “skip connection” of U-Net model; at the same time, the CBAM module is integrated into the residual module DRB (dilated residual networks) to obtain the attention residual block(ARB); Focal Tversky loss is selected as the loss function of the model; Finally, the proposed ARB-UNet model is trained and tested on ISIC2016 data set, and compared with traditional methods and classical methods such as U-Net. The experimental results show that the sensitivity (SE), specificity (SP), and dice similarity index (DSC) have reached 92.9%, 94.1%, and 92.1%, respectively, which are all better than other comparative methods in overall. Thus, the feasibility and effectiveness of the method in this paper are verified -
黑色素瘤是一类死亡率较高,极其危险的皮肤癌[1-2]。美国每年就涉及100000多例新增病例,其中死亡病例超过9000例[3]。治疗黑色素瘤最好的方法是尽早治疗,这样患者的存活率可超过95%。皮肤镜技术是一种非侵入性的皮肤成像技术,它是检测皮肤病的主要手段,可以帮助皮肤科医生辨别良性病变和恶性病变,特别在黑色素瘤的诊断上。然而靠医生的人工诊断比较浪费时间,效率较低,且带有部分主观看法,所以利用计算机来辅助医生在黑色素瘤的诊断中有着很高的价值,给皮肤科医生提供定量和客观的评估,而皮肤镜图像的分割是整个辅助诊断过程中一个重要的阶段。
皮肤镜图像分割是从病变区域中提取出关键的特征,以便在接下来的病变分类任务中更好地区分病变类型。然而,由于病变的不规则性、不同的形状和颜色,还有诸如毛发等噪声的干扰,皮肤病变的分割具有很大的挑战性。
在较早的研究中,皮肤镜图像分割大多采用传统的分割算法,如阈值分割[4-6]、区域生长[7-8]和边缘检测[9]。这些方法依赖于从颜色[10]、形状[11]和纹理[12]等特征中获取的手工特征,实现起来较麻烦,比较费时间,而且鲁棒性较差,不能获得满意的结果。为达到准确分割的目标,目前大多数学者采用卷积神经网络对图像进行分割。Shelhamer等 [13]创建了全卷积神经网络(fully convolutional networks, FCN),将原始应用于图像分类的模型转移到图像分割任务中,显著提高了图像分割的性能。Ronneberger等[14]创建了U-Net模型,该模型分为2个阶段,先向下采样,后向上采样。通过下采样路径提取高级语义信息,并结合跳跃连接和上采样路径来恢复详细信息。经过多次实验论证,U-Net模型有着较好的性能,特别是在医学图像分割方面。Zhou等[15]提出了U-Net++网络,通过在下采样阶段和上采样阶段之间增加Dense block与卷积层来提高网络的分割性能。Oktay等[16]提出在U-Net跳跃式连接中加入注意门(attention gate,AG),以提高胰腺分割协议的预测准确性和敏感性。Sarker等[17]介绍了SLSDeep,结合了跳跃连接、扩张残差和金字塔网络。在该模型中,编码器网络依赖于扩张的残余网络层和金字塔池化网络,增强了从皮肤镜图像中学习特征的能力。Zhang等[18]利用FCN学习的特征,将FCN生成的特征与浅层卷积神经网络相结合,提出了一种自动分割皮损区域的框架,以提高模型的整体分割性能。Baghersalimi等[19]提出了具有编码器−解码器范式的DermoNet,处理早期层的高分辨率特征以及更深层次的高语义特征,以将每个密集块的输出与相应的解码器联系起来。
以上文献增加了网络结构复杂性,不能克服图像边界模糊、数据集不平衡和ROI区域较小等问题。为此,本文提出一种ARB-UNet模型,引入有效的CBAM注意力机制,并选用Focal tversky loss作为模型的损失函数,以提高模型的分割性能。
1. ARB-UNet算法原理
ARB-UNet算法流程图如图1所示。首先对皮肤镜图像预处理,包括灰度化和限制对比度自适应直方图均衡化(contrast limited adaptive histogram equalization,CLAHE)[20]处理;然后,进行图像分割,将经过预处理的图像经过ARB-UNet模型进行训练和测试,从而得到皮肤镜图像的分割图。
1.1 图像预处理
图像预处理是图像分割任务中的第1步,也是较为关键的一个阶段,好的预处理方法能显著提升模型的分割效率。下面介绍所提算法采用的图像预处理方法。
本文训练集中的部分图像如图2所示,为了降低后续处理过程中的运算量,首先对其进行灰度化处理。与RGB图像相比,灰度图像可以更好地显示病变区域,对比度较强,并且灰度化处理可以减少数据量,使运算速度大幅度提高,得到的结果如图3所示。
从图3中可以看出,将彩色图像转变为了灰度图像,但是由于图中的背景与要分割出的病变区域区别不够明显,图像的对比度有待提升。
在提升图像对比度的方法中,较常用的是CLAHE算法。自适应直方图均衡化(adaptive histogram equalization,AHE)算法通过求所有像素邻域的变换函数来增强每个像素。CLAHE基于自适应直方图均衡化,其中直方图是针对像素的上下文区域计算的,然后将像素强度转换为显示范围内的值,在局部强度直方图中,该值与像素强度的等级成比例。CLAHE算法可以解释为
$$ p\left( {{r_k}} \right) = \frac{{{n_k}}}{n} $$ (1) 式中:
${n_k}$ 为具有灰度值${r_k}$ 的像素数,$n$ 为像素总数,$p\left( {{r_k}} \right)$ 为具有灰度值${r_k}$ 的像素总数。灰度值变换为
$$ s = T\left( r \right) $$ (2) 式中
$T\left( r \right)$ 为$0 \leqslant r \leqslant 1$ 的单调递增函数。最后对上述灰度图像进行CLAHE处理,得到的结果如图4所示。
从图4可以看出,经过CLAHE处理得到的皮肤镜图像的局部对比度明显提升,与原图相比病变区域更加明显,充分体现了皮肤镜图像的细节信息,有利于下一步进行皮肤镜图像的分割。
1.2 ARB-UNet
本文所提的ARB-UNet模型是U-Net模型的改进版。ARB-UNet模型的详细结构如图5所示。
从图5可以看出,ARB-UNet由收缩路径和扩展路径这2个部分构成。左侧的收缩路径向下采样,用于提取图像局部的特征,右侧的扩展路径根据上下文信息对图像的特征精准定位。受注意力机制最近在图像处理领域得到广泛应用的启发,在该模型中,引入了卷积块注意力模块(convolutional block attention module,CBAM)[21],通过关注图像的细节特征来增强特征的表达。一方面,本文将CBAM模块应用到原U-Net模型的“跳过连接”中,为收缩路径中的每个特征映射赋予权重,而不是像原U-Net网络中将它们平等地复制到对应的扩展路径中;另一方面,考虑到特征通道之间的关系,本文将CBAM注意力机制模块应用到残差结构(double residual block,DRB)中,并将其命名为注意力残差结构(attention residual block,ARB)。整个网络模型的结构来源于U-Net,U-Net的卷积块被ARB替代。收缩路径包含4个模块,每一个模块包括ARB结构和规格为2×2的最大池化层,扩展路径同样包含4个模块,每一个模块包括规格为2×2的转置卷积,并与通过CBAM模块从收缩路径加权的特征映射连接,然后通过ARB模块之后,最后经过卷积层和sigmoid函数得到模型的分割图。
1.3 注意力残差模块ARB
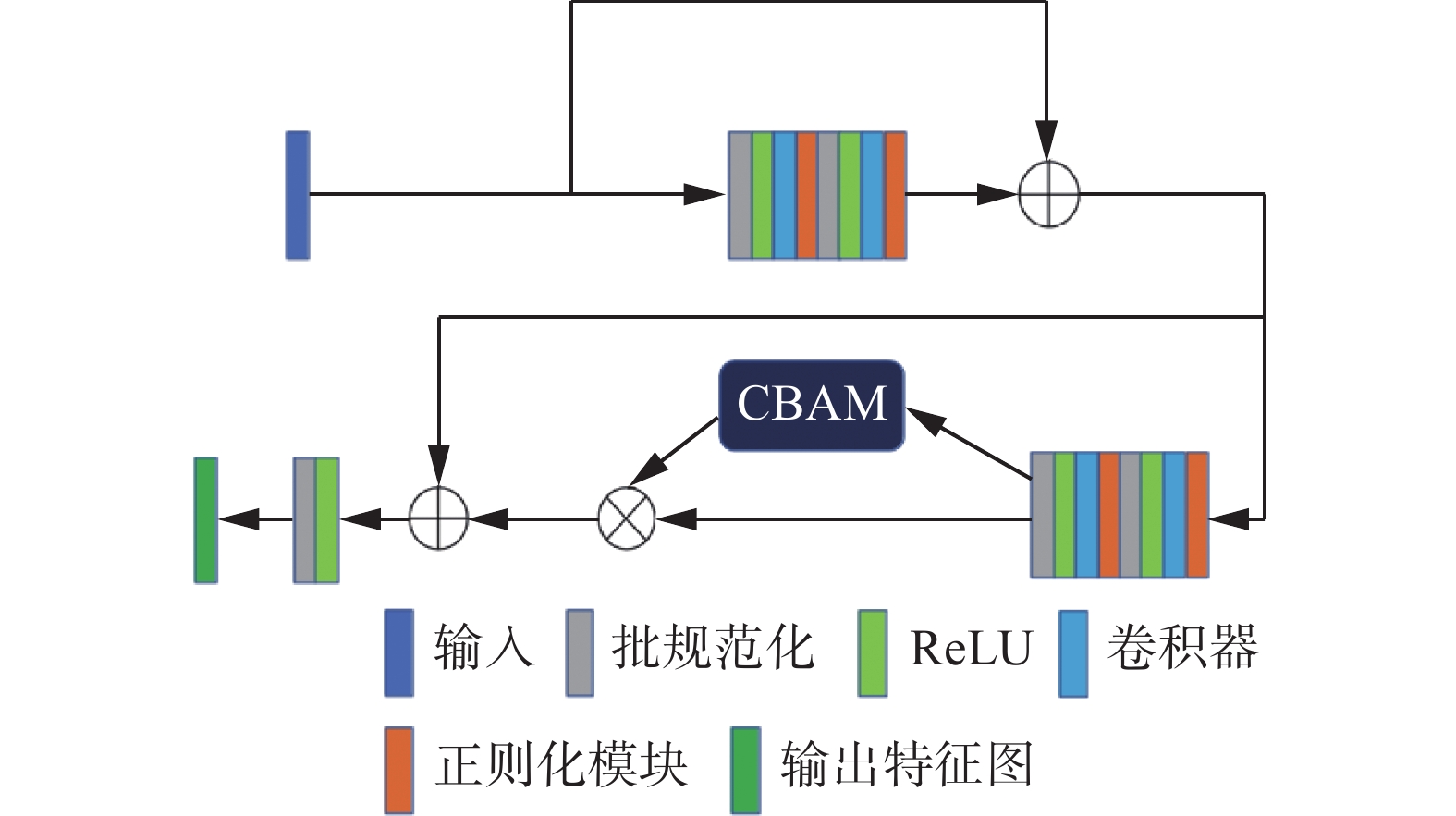
为了充分提取图像中的高级特征,DRNet[22]提出了残差模块DRB, 本文将CBAM模块集成到残差模块DRB中,提出注意力残差模块ARB,ARB模块的结构图如图6所示。
从图6可以看出,注意力残差结构ARB中引入了批规范化(BN)优化算法[23],该算法通过规范化操作使得每批训练数据的均值和方差分别为0和1,提高了网络的收敛速度,还能减小“梯度分散”现象对网络的影响,同时优化了模型的训练效果。批规范化算法的实现步骤较简单,首先计算批数据的均值
$ \mu $ 和方差$ \sigma $ ,接着由$ \mu $ 和$ \sigma $ 对该批数据进行规范化计算,最后通过参数$ \gamma $ 和$ \beta $ 推导尺度变换和偏移。批规范化算法流程如下所示。输入 批处理数据:
$\left\{ {{x_1},{x_2},\cdots,{x_m}} \right\}$ 。1)计算批数据的均值:
$\mu = \dfrac{1}{m}\displaystyle\sum_{i = 1}^m {{x_i}}$ ;2)计算批数据的方差:
$\sigma = \dfrac{1}{m}\displaystyle\sum_{i = 1}^m {\left( {{x_i} - \mu } \right)}$ ;3)根据
$ \mu $ 和$ \sigma $ 进行规范化计算:${\hat x_i} = \dfrac{{{x_i} - \mu }}{{\sqrt {{\sigma ^2} + \in } }}$ ;4)推导尺度变换和偏移:
${y_i} = {\hat \gamma} {{{\hat x}_l}} + \beta$ ;5)返回参数
$ \gamma $ 和$ \beta $ 。输出 规范后的结果
$ y $ :$\left\{ {{y_i} = {N_{\gamma ,\beta }}\left( {{x_i}} \right)} \right\}$ 。为防止网络出现过拟合,在上述的注意力残差结构ARB中添加了Dropblock层[24],Dropblock是一种结构化的Dropout形式,它比Dropout更有效地随机丢弃局部块区域和正则化卷积层,可以让模型获得更好的精度。Dropblock通过随机丢弃特征图中一部分相邻的块状区域,迫使网络利用其他部位语义信息进行决策分类,使用伯努利分布来生成二进制掩码矩阵,只得到0和1两个值,其中值为0的元素作为基点,然后通过将特征图和该掩码矩阵进行哈达玛积计算来获得输出特征图。其计算为
$$ \gamma = \dfrac{1-p}{a^2}\dfrac{s^2}{(s - a+ 1)^2} $$ (3) 式中:
$ \gamma $ 为丢弃基点的概率;$a$ 为随机丢弃区域的大小;$p$ 为神经元保留概率;$(s- a + 1)^2$ 为有效种子采样区域大小;$s$ 表示输入特征图大小。1.4 注意力机制
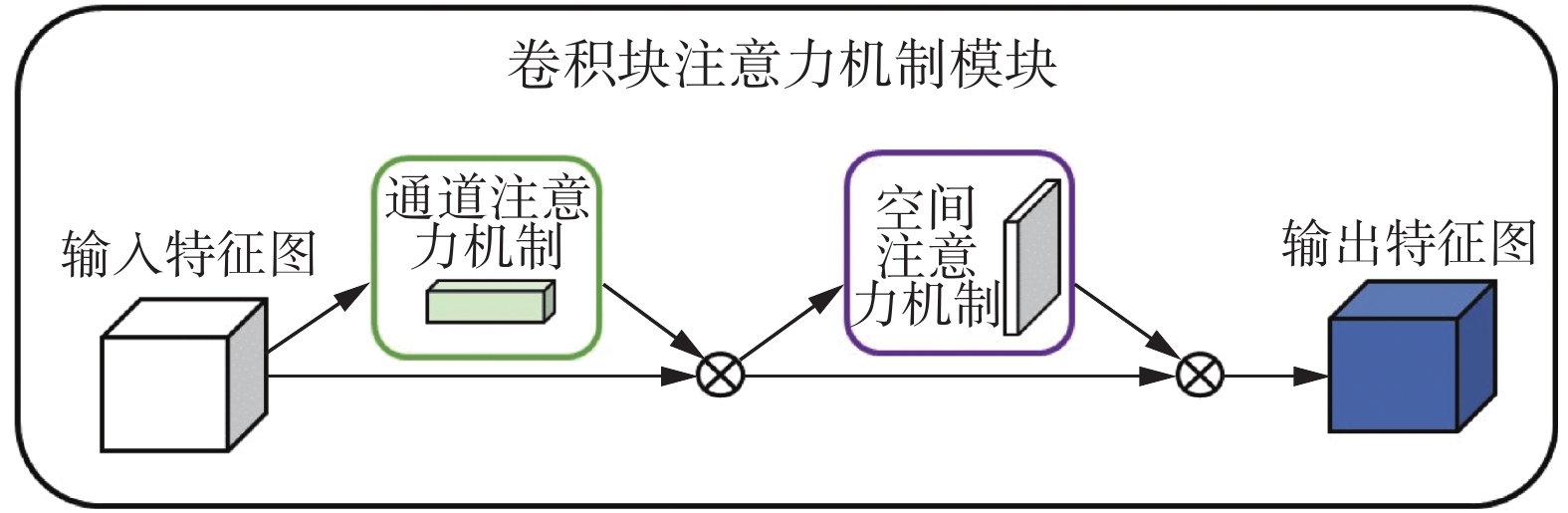
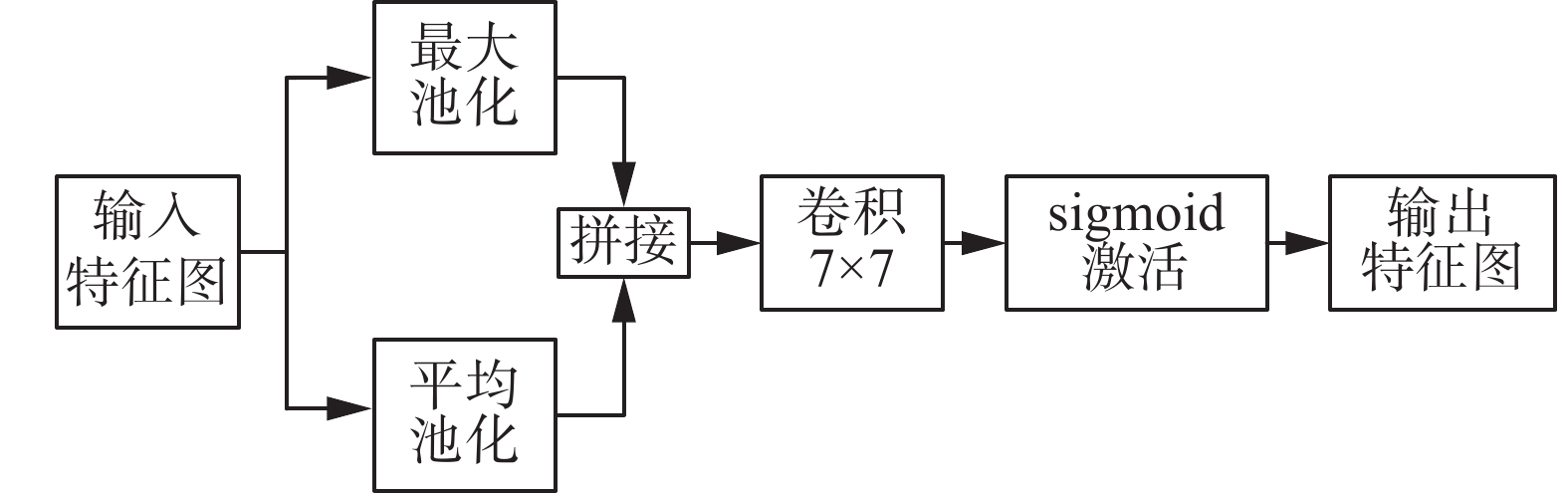
CBAM是一种轻量级的注意力机制,适用于任何卷积神经网络模型,大大提高了模型的性能。图7是CBAM的模型结构图。
CBAM模块将注意力映射分别注入通道维度与空间维度,然后将注意力映射与输入特征映射相乘,以达到自适应优化。设定
${\boldsymbol{F}} \in {{\bf{R}}^{C \times H \times W}}$ 作为输入特征图,CBAM先后映射出通道注意力特征图${{\boldsymbol{M}}_c} \in {{\bf{R}}^{C \times 1 \times 1}}$ 和空间注意力特征图${{\boldsymbol{M}}_s} \in {{\bf{R}}^{1 \times H \times W}}$ 。整个注意力过程可以概括为$$ {\boldsymbol{F}}' = {{\boldsymbol{M}}_c}\left( {\boldsymbol{F}} \right) \otimes {\boldsymbol{F}} $$ (4) $$ {\boldsymbol{F}}'' = {{\boldsymbol{M}}_s}\left( {{\boldsymbol{F}}'} \right) \otimes {\boldsymbol{F}}' $$ (5) 式中
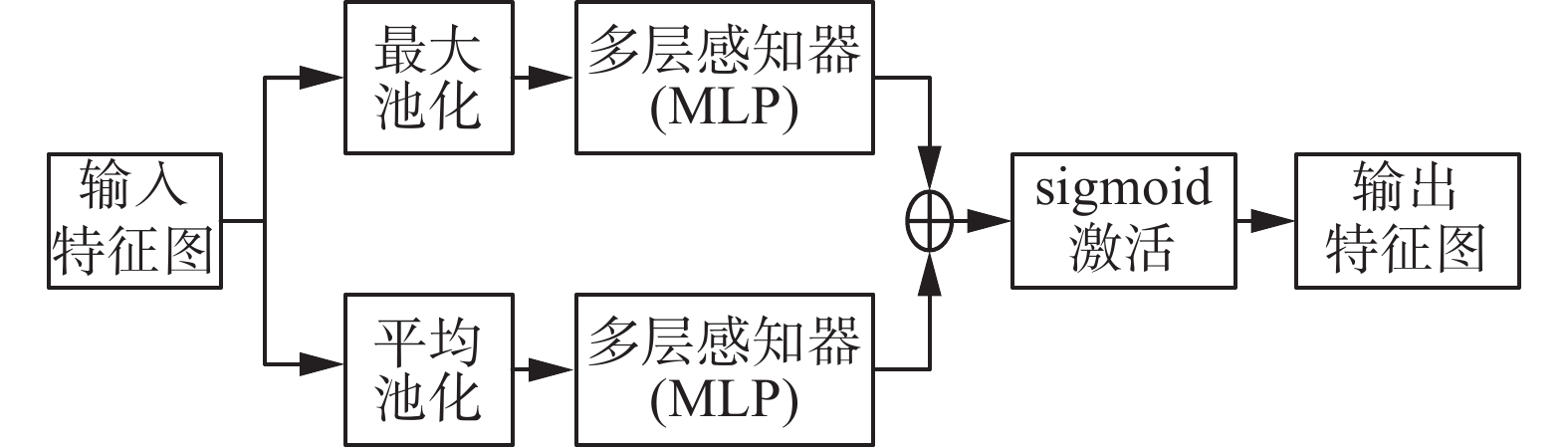
$ \otimes $ 表示为element-wise乘法。在乘法过程中,注意力值会根据特征的重要性分配不同的权重,通道注意力值和空间注意力值会沿着彼此的维度进行分配,提取图像的细节特征。${\boldsymbol{F}}''$ 是最终的输出特征图。如图8所示,通道注意模块通过输入,然后分别通过多层感知器(multilayer perceptron,MLP),对MLP输出的特征逐个元素求和,生成最终的通道注意特征图。对其进行乘法运算并输入特征图,生成空间模块所需的输入特征,表示为
$$\begin{gathered} {{\boldsymbol{M}}_c}\left( {\boldsymbol{F}} \right) = {\rm{sigmoid}}\left( {\rm{MLP}}\left( {{\rm{AvgPool}}\left( {\boldsymbol{F}} \right)} \right) +\right. \\ \left.{\rm{MLP}}\left( {{\rm{MaxPool}}\left( {\boldsymbol{F}} \right)} \right) \right) \end{gathered}$$ (6) 如图9所示,空间注意力机制的输入特征图是上述通道注意力机制的输出特征图。接着将输入特征分别进行最大池化操作和平均池化操作,接着进行特征拼接,然后通过卷积层提取特征,最后经过sigmoid函数生成输出特征,可以表示为
$$ {{\boldsymbol{M}}_s}({\boldsymbol{F}}) = {\rm{sigmoid}}\left({f^{7 \times 7}}\left(\left[ {{\rm{AvgPool}}({\boldsymbol{F}});{\rm{MaxPool}}\left( {\boldsymbol{F}} \right)} \right]\right)\right) $$ (7) 式中
$ {f^{7 \times 7}} $ 为大小为7×7的卷积核。1.5 损失函数
由于ISIC2016官方皮肤数据集存在数据不平衡问题,所以本文应用了基于Tversky指数的Focal Tversky Loss[25]来评估模型预测的质量。在医学图像分割领域,Dice相似指数(dice score coefficient)被广泛用于评估图像预测值和真实值之间的相似性:
$$ {{{D}}_c} = \dfrac{{\displaystyle\sum_{i = 1}^N {{p_{ic}}{g_{ic}} + \varepsilon } }}{{\displaystyle\sum_{i = 1}^N {{p_{ic}} + {g_{ic}} + \varepsilon } }} $$ (8) 式中:
$ {p_{ic}} \in \left[ {0,1} \right] $ 为预测标签;$ {g_{ic}} \in \left[ {0,1} \right] $ 为真相标签;N为图像的总像素;$ \varepsilon $ 为很小的参数,避免被零除。Dice Loss表示预测图像和实际标签图像之间重叠的最小值,定义为
$$ {L}_c = \displaystyle\sum_c {1 - {D}_c} $$ (9) Dice Loss最明显的第1个缺点是对于假阳性(false positive,FP)和假阴性(false negative,FN)分配的权重是相等的,使得分割图的精度较高但其召回率较低。在这一点上,指数Tversky优化了Dice Loss,其数学表达式为
$$ {{{T}}_c} = \dfrac{{\displaystyle\sum_{i = 1}^N {{p_{ic}}{g_{ic}} + \varepsilon } }}{{\displaystyle\sum_{i = 1}^N {{p_{ic}}{g_{ic}} + \alpha \displaystyle\sum_{i = 1}^N {{p_{i\overline c }}{g_{ic}} + \beta \displaystyle\sum_{i = 1}^N {{p_{ic}}{g_{i\overline c }} + \varepsilon } } } }} $$ (10) 式中:
$ {p_{ic}} $ 为像素$i$ 属于目标区域类别$c$ 的概率,$ {p_{i\overline c }} $ 为像素$i$ 属于背景类别$c$ 的概率,$ {g_{ic}} $ 为像素i属于目标区域类别c的标签值,$ {g_{i\overline c }} $ 为像素i属于背景类别c的标签值。在数据集不平衡的情况下,通过调整超参数
$\alpha $ 和$\beta $ 来控制FP和FN的平衡,提高召回率。通过最小化$\displaystyle\sum_c {1 - {{{T}}_c}}$ 来作为Tversky Loss损失函数。Dice Loss第2个缺点是很难分割较小的感兴趣区域(region of interest,ROI)区域,因为小的ROI区域对损失的影响不大,Dice Loss就不适合这样的分割任务,影响了模型的分割性能。为了克服这个缺点,提出了一种含参数
$ \gamma $ 的Focal Tversky Loss来控制背景简单且难度较大的ROI区域分割,该损失函数中焦点参数将交叉熵损失指数化,以便关注以较低概率检测到的样本。定义Focal Tversky Loss为$$ {{{F}}}_{c}={\displaystyle \sum _{c}}(1-{{{T}}}_{c})^{1/{\gamma} } $$ (11) 式中:
${{{T}}_c}$ 是Tversky系数,$ \gamma \in \left[ {1,3} \right] $ 。2. 实验分析
本文实验选择在ISIC2016公共数据集上进行验证和测试,该数据集来自国际皮肤病图像协会(international skin imaging collaboration,ISIC)官方网站。ISIC2016数据集的训练集共有900张图片,测试集共有379张图片,以及对应的真实标签图片。本文将该数据集的训练集以8∶2的比例分为训练集和验证集。
如表1所示,混淆矩阵是一种评估和分析图像分割的方法。本文采用了灵敏度(sensitivity,SE)、特异性(specificity,SP)和Dice相似指数(dice similarity cofficient,DSC)这3项指标来评估所提算法的效果。3项指标定义为
表 1 混淆矩阵Table 1 Confusion matrix标签 实际预测结果 正样本 负样本 正样本 T E 负样本 W R $$ S = \frac{{T}}{{T + W}} $$ (12) $$P = \frac{{R}}{{R + E}} $$ (13) $$ {{D }}= \frac{{2T}}{{2T + W + E}} $$ (14) 式中:S为所有正样本中被预测为正样本的比重;P为被预测为负样本的占所有负样本的比重;D为集合之间相似的评估指标,计算真实值和预测值的相似比例,最大值为1,此时分割结果最好,最小值为0,此时分割结果最差。
本文的实验是基于谷歌colab平台上进行的,整个算法的实现是通过Python语言进行编写,基于Tensorflow框架。本文在ISIC2016官方皮肤数据集上训练提出的ARB-UNet模型,为了保持较小的参数量,设定第1个卷积层之后的通道数为16,并使用收敛速度较快的Adam优化器和上述介绍的Focal Tversky Loss损失函数优化本文的模型。为了让模型的性能达到最佳,经过多次实验论证,训练时的迭代轮次设置为100,批次大小(batch size)设置为2,学习率设置为1×10−5。
ISIC2016数据集中的图像分辨率是不统一的,为了保持输入数据分辨率的一致性,防止训练过程中出现梯度爆炸问题,需要将数据集中的图片调整为统一分辨率。本文使用3种不同分辨率的图片对分割结果进行对比,分别是192像素×192像素、256像素×256像素、512像素×512像素,实验结果如表2所示。
表 2 不同图像分辨率的实验结果对比Table 2 Comparison of experimental results with different image resolutions分辨率/像素×像素 S D P 192×192 0.919 0.917 0.932 256×256 0.925 0.920 0.938 512×512 0.929 0.921 0.941 从表2可以看出,相比于其他2种分辨率图片,512像素×512像素的图片在所有指标上获得了最优的结果,更能被充分提取特征。因此将数据集图片的分辨率统一调整为512像素×512像素。
为了验证Focal Tversky Loss损失函数对模型的提升效果,本文选取了交叉熵损失函数和Focal Tversky Loss损失函数进行了实验对比,结果如表3所示。
表 3 不同损失函数对比实验Table 3 Comparative experiment of different loss functions损失函数 S D P 交叉熵损失 0.908 0.903 0.912 Focal Tversky Loss 0.929 0.921 0.941 从表3可以看出,相比于交叉熵损失函数,Focal Tversky Loss损失函数明显提升了模型的分割效果,其中S提升了2.1%,D提升了1.8%,P提升了2.9%,表明了Focal Tversky Loss损失函数更加适用于类别不均衡的数据集,在一定程度上避免了数据量较小且不均衡对分割结果的影响。
ARB-UNet模型训练过程中的准确率和损失曲线如图10和图11。
从图10可以看出,横坐标表示训练次数,纵坐标表示准确率,ARB-UNet模型在前20个epoch的训练过程中,准确率曲线在逐渐上升,其中训练集曲线和验证集曲线之间距离在拉大,表明了模型处于学习和训练的过程中,且在不断完善;20个epoch之后模型的准确率上升幅度减小,保持在较小的幅度内变化,最后趋于稳定,且训练集和验证集之间的差距较大,体现出所提模型的泛化能力较弱。从图11中可以看出,横坐标表示训练次数,纵坐标表示损失值,模型的损失曲线随着epoch次数的增加在下降,最终在较小的幅度范围内收敛。
本文方法与大津算法(Otsu algorithm,OTSU)和遗传算法(genetic algorithm,GA)的实验结果对比图如图12所示,其中,图12 (a)表示数据集原始图像,图12 (b)表示OTSU算法分割得到的图像,图12 (c)表示GA算法分割得到的图像,图12 (d)表示本文方法分割得到的图像。
由图12可知,OTSU算法分割出的图形不够完整,孔洞较多,且边缘的毛刺很多,不够光滑,基于GA的分割算法整体效果要优于OTSU算法,病变区域的轮廓更加完整,但是仍存在孔洞较多的问题,且对第2幅和第4幅图像的分割效果较差,而本文算法分割出的病变区域轮廓完整、光滑,没有出现大面积的缺失。综上所述,本文方法的分割效果要远好于以OTSU算法、GA遗传算法为代表的传统方法,能够适应多种不同深浅颜色的病变区域的分割,满足分类识别的要求。
本文方法的分割结果和真实标签值的对比图如图13所示。
从图13可以看出,本文所提出的模型的最终分割图与真实值很相似,对图像的分割较为精准。但当图像含有毛发干扰的情况下,分割结果存在一定的误差,这和本文的预处理过程较简单有关系,这也是本文方法需要改进的地方。
分别采用ARB-UNet模型、U-Net模型、结合注意力机制的Attention-UNet模型对ISIC2016数据集进行图像分割,这3种网络的分割结果如表4所示。
表 4 不同网络在ISIC2016数据集上的实验结果Table 4 Experimental results of different networks on ISIC2016 dataset方法 S D P U-Net 0.906 0.882 0.918 Attention-UNet 0.913 0.897 0.938 本文方法 0.929 0.921 0.941 由表4中的数据可知,本文提出的方法取得了令人满意的效果,各个指标较其他2种方法都有所提高。这表明ARB-UNet与其他2种模型相比在皮肤镜图像分割上有显著的改进。
为了验证本文运用注意力机制(CBAM)的有效性,在ISIC2016数据集上进行了消融实验,通过不加入CBAM模块与本文方法对比,实验结果如表5所示。
表 5 加入CBAM模块前后的实验结果对比Table 5 Comparison of experimental results before and after adding CBAM module方法 S D P 未将CBAM模块加入到
U-Net的跳过连接中0.901 0.898 0.917 未将CBAM模块应用到
DRB残差结构中0.914 0.907 0.923 本文方法 0.929 0.921 0.941 从表5可以看出,加入注意力机制模块(CBAM)后,模型的性能得到了全面提升。其中将CBAM模块加入到U-Net的跳过连接后,3个指标S、D和P分别提升了2.8%、2.3%和1.8% ;将CBAM模块应用到DRB残差结构得到ARB模块后,3个指标S、D和P分别提升了1.5%、1.4%和2.1%。验证了注意力机制模块(CBAM)可以关注图像的细节特征,使模型能够充分提取图像的特征,提高了模型的分割性能,也证实了所提算法是有效的。
为了更进一步证明本文方法是有优势的,选取了在ISIC2016皮肤病变检测挑战赛中成绩排在前5名的参赛团队的实验结果与本文方法的实验结果进行比较,结果如表6所示。
表 6 本文方法与ISIC2016挑战赛前5名实验结果对比Table 6 Proposed method compared with the top 5 experimental results of ISIC2016 challenge方法 S D P Team-EXB 0.910 0.910 0.965 Team-CUMED 0.911 0.897 0.957 Team-Rahman 0.880 0.895 0.969 Team-SFU 0.915 0.885 0.955 Team-TMU 0.832 0.888 0.987 本文方法 0.929 0.921 0.941 分析表6得知,ARB-UNet模型的前2项评估指标均高出其他参赛团队,相比于Team-EXB参赛团队,S、D两项指标分别提升了1.9%、1.1%。本文方法的最后一项指标P稍低于其他方法,一方面是因为所提模型的局限性;另一方面是因为数据集的数据量较小且不均衡,模型没有充分学习到皮肤镜图像的特征。这2个方面的不足将是后续研究工作中要着重解决的问题。表6的实验结果综合表明了本文方法在提升皮肤镜图像的分割性能方面是可行的,并且具有一定的优势。
3. 结束语
本文提出了一种注意力残差U-Net (ARB-UNet)网络模型来分割皮肤镜图像,该方法采用CBAM注意力模块来提升网络的区分能力。并且本文采用了Focal Tversky Loss损失函数来优化本文提出的模型,其适用于类别不平衡的数据集,提高了模型的性能。模型的改进首先是在U-Net的跳过连接中增加CBAM注意力机制模块,将权重分配给来自收缩路径的特征图,充分关注图像的细节特征;另一方面将CBAM注意力机制模块应用到DRB残差结构中得到注意力残差结构ARB,来构造网络的收缩路径和扩张路径。通过实验证明,所提出的ARB-UNet模型在灵敏度SE、特异性SP、Dice相似指数DSC这3项评价指标中优于一些现有的经典方法,证明了本文方法对皮肤镜图像的分割是有效的和可行的,在辅助医学诊断中有一定的前景。针对本文方法存在的不足,在未来的研究工作中,首先要继续改进和优化模型的结构,其次增加图像预处理算法以适应小数据集或在数据量较大的数据集上训练,使模型能够充分学习图像的特征,使模型的分割能力得到全方面提升。
-
表 1 混淆矩阵
Table 1 Confusion matrix
标签 实际预测结果 正样本 负样本 正样本 T E 负样本 W R 表 2 不同图像分辨率的实验结果对比
Table 2 Comparison of experimental results with different image resolutions
分辨率/像素×像素 S D P 192×192 0.919 0.917 0.932 256×256 0.925 0.920 0.938 512×512 0.929 0.921 0.941 表 3 不同损失函数对比实验
Table 3 Comparative experiment of different loss functions
损失函数 S D P 交叉熵损失 0.908 0.903 0.912 Focal Tversky Loss 0.929 0.921 0.941 表 4 不同网络在ISIC2016数据集上的实验结果
Table 4 Experimental results of different networks on ISIC2016 dataset
方法 S D P U-Net 0.906 0.882 0.918 Attention-UNet 0.913 0.897 0.938 本文方法 0.929 0.921 0.941 表 5 加入CBAM模块前后的实验结果对比
Table 5 Comparison of experimental results before and after adding CBAM module
方法 S D P 未将CBAM模块加入到
U-Net的跳过连接中0.901 0.898 0.917 未将CBAM模块应用到
DRB残差结构中0.914 0.907 0.923 本文方法 0.929 0.921 0.941 表 6 本文方法与ISIC2016挑战赛前5名实验结果对比
Table 6 Proposed method compared with the top 5 experimental results of ISIC2016 challenge
方法 S D P Team-EXB 0.910 0.910 0.965 Team-CUMED 0.911 0.897 0.957 Team-Rahman 0.880 0.895 0.969 Team-SFU 0.915 0.885 0.955 Team-TMU 0.832 0.888 0.987 本文方法 0.929 0.921 0.941 -
[1] CIĄŻYŃSKA M, KAMIŃSKA-WINCIOREK G, LANGE D, et al. The incidence and clinical analysis of non-melanoma skin cancer[J]. Scientific reports, 2021, 11: 4337. doi: 10.1038/s41598-021-83502-8 [2] LI Yuexiang, SHEN Linlin. Skin lesion analysis towards melanoma detection using deep learning network[J]. Sensors, 2018, 18(2): 556. doi: 10.3390/s18020556 [3] SIEGEL R L, FEDEWA S A, MILLER K D, et al. Cancer statistics for hispanics/latinos, 2015[J]. CA:a cancer journal for clinicians, 2015, 65(6): 457–480. doi: 10.3322/caac.21314 [4] 徐国雄, 张骁, 胡进贤, 等. 基于阈值分割和轮廓提取的图像边缘检测算法[J]. 计算机技术与发展, 2015, 25(12): 64–67,71. XU Guoxiong, ZHANG Xiao, HU Jinxian, et al. Medical cell edge detection algorithm based on threshold segmentation and contour extraction[J]. Computer technology and development, 2015, 25(12): 64–67,71. [5] RIYANTO I. Morphology based melanoma skin segmentation method[J]. Helix, 2021, 11(3): 7–13. doi: 10.29042/2021-11-3-7-1 [6] 尹雨山, 王李进, 尹义龙, 等. 回溯搜索优化算法辅助的多阈值图像分割[J]. 智能系统学报, 2015, 10(1): 68–74. doi: 10.3969/j.issn.1673-4785.201410008 YIN Yushan, WANG Lijin, YIN Yilong, et al. Backtracking search optimization algorithm assisted multilevel threshold for image segmentation[J]. CAAI transactions on intelligent systems, 2015, 10(1): 68–74. doi: 10.3969/j.issn.1673-4785.201410008 [7] 孔令军, 王茜雯, 包云超, 等. 基于深度学习的医疗图像分割综述[J]. 无线电通信技术, 2021, 47(2): 121–130. doi: 10.3969/j.issn.1003-3114.2021.02.001 KONG Lingjun, WANG Qianwen, BAO Yunchao, et al. A survey on medical image segmentation based on deep learning[J]. Radio communciations technology, 2021, 47(2): 121–130. doi: 10.3969/j.issn.1003-3114.2021.02.001 [8] STARVIN M S. A novel and efficient approach for the classification of skin melanoma[J]. Journal of ambient intelligence and humanized computing, 2021, 12(12): 10435–10459. doi: 10.1007/s12652-020-02648-x [9] LAKSHMI B V, SRIDEVI K. A qualitative approach on de-noising and segmentation algorithms for melanoma images[J]. Annals of the romanian society for cell biology, 2021: 1384-1393. [10] FAN Haidi. Automatic segmentation of dermoscopy images using saliency combined with Otsu threshold[J]. Computers in biology and medicine, 2017, 85: 75–85. doi: 10.1016/j.compbiomed.2017.03.025 [11] XIE Fengying, FAN Haidi, LI Yang, et al. Melanoma classification on dermoscopy images using a neural network ensemble model[J]. IEEE transactions on medical imaging, 2017, 36(3): 849–858. doi: 10.1109/TMI.2016.2633551 [12] GLAISTER J, WONG A, CLAUSI D A. Segmentation of skin lesions from digital images using joint statistical texture distinctiveness[J]. IEEE transactions on bio-medical engineering, 2014, 61(4): 1220–1230. doi: 10.1109/TBME.2013.2297622 [13] SHELHAMER E, LONG J, DARRELL T. Fully convolutional networks for semantic segmentation[C]//IEEE Transactions on Pattern Analysis and Machine Intelligence. Piscataway: IEEE, 2016: 640−651. [14] RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[M]. Cham: Springer International Publishing, 2015: 234−241. [15] ZHOU Zongwei, RAHMAN SIDDIQUEE M M, TAJBAKHSH N, et al. UNet++: A nested U-net architecture for medical image segmentation[M]. Cham: Springer International Publishing, 2018: 3−11. [16] OKTAY O, SCHLEMPER J, FOLGOC L L, et al. Attention U-net: learning where to look for the pancreas[EB/OL]. (2018-04-11)[2022-01-18].https://arxiv.org/abs/1804.03999. [17] SARKER M M K, RASHWAN H A, AKRAM F, et al. SLSDeep: skin lesion segmentation based on dilated residual and pyramid pooling networks[M]. Cham: Springer International Publishing, 2018: 21−29. [18] ZHANG Lei, YANG Guang, YE Xujiong. Automatic skin lesion segmentation by coupling deep fully convolutional networks and shallow network with textons[J]. Journal of medical imaging, 2019, 6(2): 024001. [19] BAGHERSALIMI S, BOZORGTABAR B, SCHMID-SAUGEON P, et al. DermoNet: densely linked convolutional neural network for efficient skin lesion segmentation[J]. EURASIP journal on image and video processing, 2019, 2019(1): 71. doi: 10.1186/s13640-019-0467-y [20] SELLA VELUSWAMI J R, EZHIL PRASANTH M, HARINI K, et al. Melanoma skin cancer recognition and classification using deep hybrid learning[J]. Journal of medical imaging and health informatics, 2021, 11(12): 3110–3116. doi: 10.1166/jmihi.2021.3898 [21] LIU Jing, CHEN Aibin, ZHOU Guoxiong, et al. Dermatoscopic image melanoma recognition based on CFLDnet fusion network[J]. Multimedia tools and applications, 2021, 80(17): 25477–25494. doi: 10.1007/s11042-021-10920-1 [22] GUO Changlu, SZEMENYEI M, YI Yugen, et al. Dense residual network for retinal vessel segmentation[C]//2020 IEEE International Conference on Acoustics, Speech and Signal Processing. Piscataway: IEEE, 2020: 1374−1378. [23] KAUR R, GHOLAMHOSSEINI H, SINHA R, et al. Melanoma classification using a novel deep convolutional neural network with dermoscopic images[J]. Sensors (Basel, Switzerland), 2022, 22(3): 1134. doi: 10.3390/s22031134 [24] WANG Hailing, YANG Jingdong. FBUNet: full convolutional network based on fusion block architecture for biomedical image segmentation[J]. Journal of medical and biological engineering, 2021, 41(2): 185–202. doi: 10.1007/s40846-020-00583-y [25] CHEN Ziming, WANG Shengsheng. The NL-SC net for skin lesion segmentation[M].Pattern Recognition and Computer Vision. Cham: Springer International Publishing, 2021: 313−324.

 下载:
下载: