抑郁症(major depressive disorder, MDD)是一种非常常见的精神类疾病,对患者的生活质量和身体健康有着显著的危害。据世界卫生组织统计,目前,全球有超3亿人患有抑郁症[1],抑郁症患者的自残、自杀等行为对患者及其家属造成的痛苦、对社会造成的损失不容忽视。抑郁症的临床诊断主要依据精神疾病诊断手册,缺乏定量的生理指标,目前常用的诊断标准有《国际疾病分类》第10版(ICD-10)[2]和美国《精神障碍诊断与统计手册》第4版 (DSM-IV)[3],但这种方式过于依赖医生的主观判断,正确诊断率低,延误了最佳治疗时间,导致病情迁延或加重。因此,寻找一个客观且有效的诊断方法,例如根据常见的脑部医学影像来诊断抑郁症的方法,是非常必要的。
结构磁共振成像(structural magnetic resonance imaging, sMRI)[4]是当下流行的医学成像方式,具有无创、对比度高等优势,广泛用于抑郁症的诊断研究[5-7]。随着人工智能和大数据时代的到来,深度学习[8-10]作为人工智能领域的核心技术,与传统算法相比,在图像领域取得了突破性的成绩[11-12]。因此,越来越多的学者将深度学习方法应用到医疗图像领域的研究课题中[13-15]。
深度卷积神经网络(convolutional neural networks, CNN)通过使用反向传播算法来调整其内部参数,叠加了多层神经元,用来发现大数据集中更深层次的特征。文献[16]表明,网络层数对网络的最终性能的影响巨大,越深层的网络往往越能提高其泛化能力。CNN网络的发展也符合这一规律,自AlexNet[11]的7层发展到了VGG[17]的16层,甚至19层,后来发展到GoogLeNet[18]的22层。但随着层数的增加,训练网络所需的计算能力和时间成本也随之增加,而且一味地增加层数,并不总是能够提升网络性能,网络达到一定深度后再加深,反而会导致网络收敛速度变慢,分类准确率也变差。排除数据集过小带来的模型过拟合等问题,分类准确率会在增加网络层数的过程中逐渐趋于饱和,继续加深网络会出现准确率下降的现象,这种现象被称为退化问题,网络层数越深,网络的分类性能退化越快。
针对这一问题,He等[19]在2016年提出了残差网络(residual network, ResNet),通过引入残差模块,建立相邻层之间的短路连接,有助于训练过程中梯度的反向传播,将网络层数做到了152层以上,一定程度上解决了梯度消失和网络退化问题。
基于此,Huang等[20]在2017年提出了密集连接网络(densely connected convolutional networks, DenseNet),其基本思路与ResNet一致,但是该网络建立了前面所有层与后面层的连接,实现特征重用,在参数量和计算成本均比ResNet少的情况下,DenseNet在多个公开大型数据集上表现出了比ResNet更优的性能。
虽然深度学习在计算机视觉领域大放异彩,但目前的深度学习网络,大多只能处理如ImageNet等二维的自然图像数据,对于三维数据涉及较少,如何将深度学习网络应用在抑郁症结构磁共振影像的分类任务上,成为一个研究热点和难点。基于此,本文将DenseNet网络模型进行三维(three dimensional, 3D)化,并引入迁移学习方法,进一步优化了实验结果。本文主要贡献概括如下:
1)提出一种三维密集连接网络(3D-DenseNet),将密集连接的思想扩展到三维,以充分挖掘三维sMRI数据中的层间结构信息,实现对抑郁症患者与健康对照者sMRI数据的准确分类;
2)引入迁移学习,精心设计了一个迁移学习工作流程,用于目标领域同类型、同部位的数据进行预训练,然后将模型迁移至抑郁症sMRI数据,并进行再训练,提高了分类精度,证明了迁移学习的有效性,通过与其他迁移学习方法做对比,证明了提出的迁移学习方法的优越性;
3)与多组先进的二维、三维网络进行对比实验,证明了提出的3D-DenseNet网络在抑郁症sMRI数据分类任务中具有优越性和有效性。
1 实验准备 1.1 数据库本研究共包含174名被试者,其中99名重症抑郁症患者和75名年龄、性别、受教育程度匹配的健康对照者(heathy control, HC)。重症抑郁症患者是从首都医科大学附属安定医院招募的,健康对照组是从报纸广告上招募的。所有重症抑郁症患者都达到了美国精神障碍诊断与统计学手册第4版(DSM-IV)对抑郁症的诊断标准,所有正常人都通过了DSM-IV的非病人版结构化临床访谈面试,在实验前,所有被试者都签署了知情同意书。2组被试者的临床统计学分析结果如表1所示。
| 表 1 被试者统计学分析结果 Tab.1 Statistical analysis results of subjects |
表1中:P为MDD和HC的双样本T检验值;HAMD表示汉密尔顿忧郁量表(Hamilton depression scale);HAMA表示汉密尔顿焦虑量表(Hamilton anxiety scale)。
1.2 sMRI数据采集所有sMRI图像都是在北京师范大学认知神经科学与学习国家重点实验室用一台Tesla3.0扫描所得,扫描时采用三维磁化快速梯度回波成像(magnetization prepared rapid gradient echo,MPRAGE),扫描参数如下:射频重复时间(repetition time, TR) = 2530 ms,回波时间(echo time, TE) = 3.39 ms,翻转角度(flip angle, FA) = 7°,成像视野(field of view, FOV) = 256 mm×256 mm,体素大小为1 mm×1 mm×1.33 mm,层厚为1.33 mm,扫描层数为128。
1.3 数据预处理sMRI数据预处理工作使用基于Matlab的SPM12工具包[21]实现。考虑到灰质区域对抑郁症诊断的重要影响[22],本文只用灰质部分进行实验。预处理主要内容包括:去除非脑组织、空间标准化、灰质分割、空间平滑。处理后每个被试者sMRI数据的尺寸为121×145×121体素。数据预处理流程如图1所示。
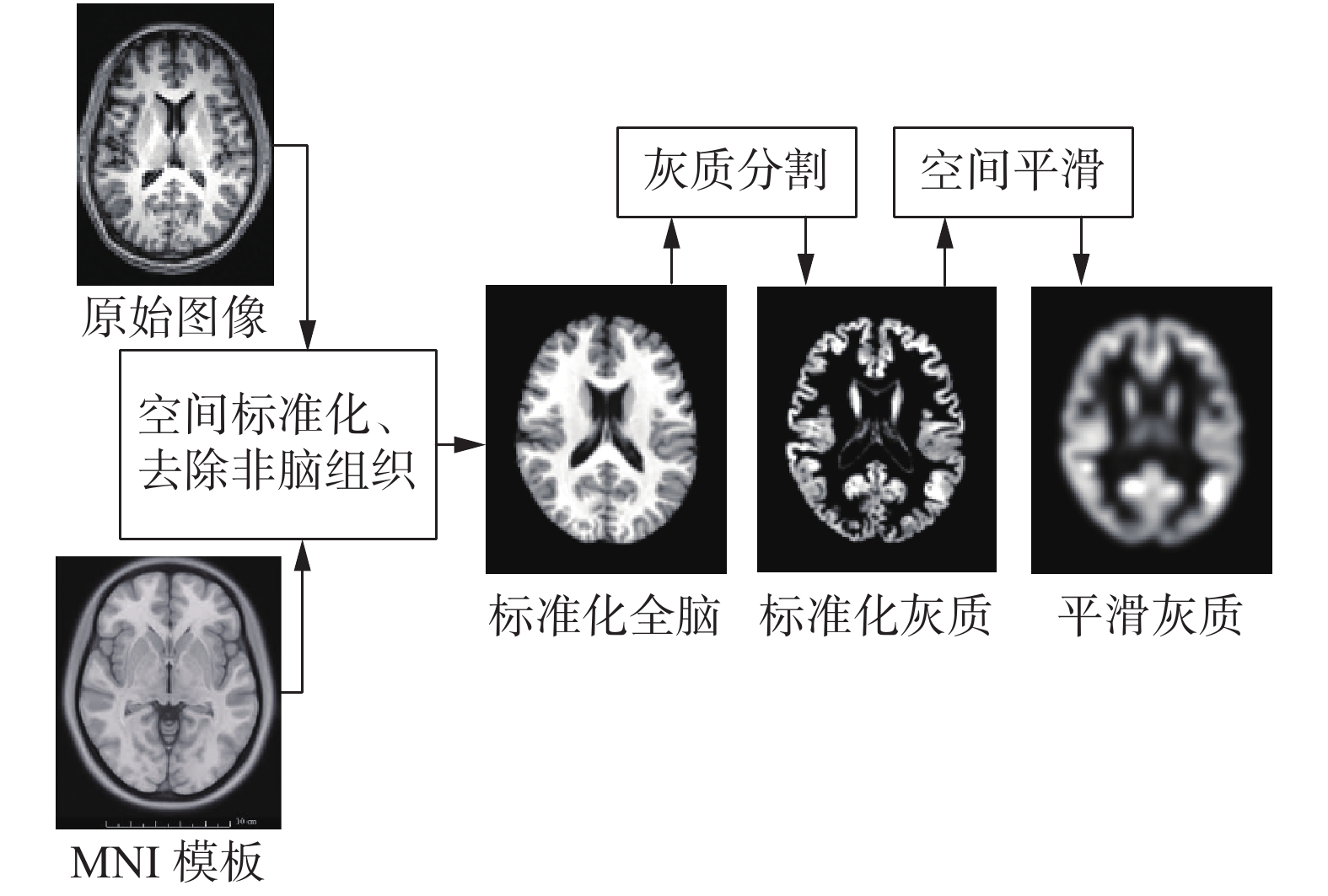
|
Download:
|
| 图 1 数据预处理流程 Fig. 1 Data preprocessing flowchart | |
尽管DenseNet在很多2D自然图片数据集上取得了卓越的成绩,但在医学图像领域却鲜有建树,原因是2D网络中的卷积核、池化核等都是二维的矩阵,只能在二维平面图像的高度H和宽度W两个方向上移动,自然也只能提取二维的特征。而sMRI等医学图像数据大多是三维的立体数据,使用2D网络处理时只能将三维图像分层输入,或将其中一个维度当作channel维度,但2种方法都不能很好地利用数据的层间结构信息。基于此,本文将2D网络中的卷积核、池化核等过滤器增加一个深度维度D,扩展为三维矩阵,这样过滤器就可以在sMRI数据所有3个方向(H、W、D)上移动,也就可以充分挖掘出数据的空间信息,每个过滤器的输出同样是一个3D的数据。若其中一个三维卷积核的尺寸为k×k×k×channel,数量为n,输入数据尺寸为h×w×d,由于本文使用的sMRI数据类似灰度图,channel维度为1,故该卷积核的输出尺寸为
| $(h - k + 1) \times (w - k + 1) \times (d - k + 1) \times n$ | (1) |
通过类似的方法可以扩展DenseNet中的池化层、批归一化层,从而构建出一个三维密集连接网络(3D-DenseNet),以更好地从三维sMRI数据中提取有代表性的特征,并提高抑郁症的sMRI数据分类的精度。
一个121层3D-DenseNet结构如图2所示。其中3D-DenseBlock 1包含6层,3D-DenseBlock 2包含12层,3D-DenseBlock 3包含24层,3D-DenseBlock 4包含16层。每一层都包含一个1×1×1三维卷积层(3D-Conv)、一个3×3×3三维卷积层,2个批量归一化(batch normalization, BN)[23]层,以及2个线性整流函数(ReLU)[24]层。一个6层的3D-DenseBlock结构如图3所示。
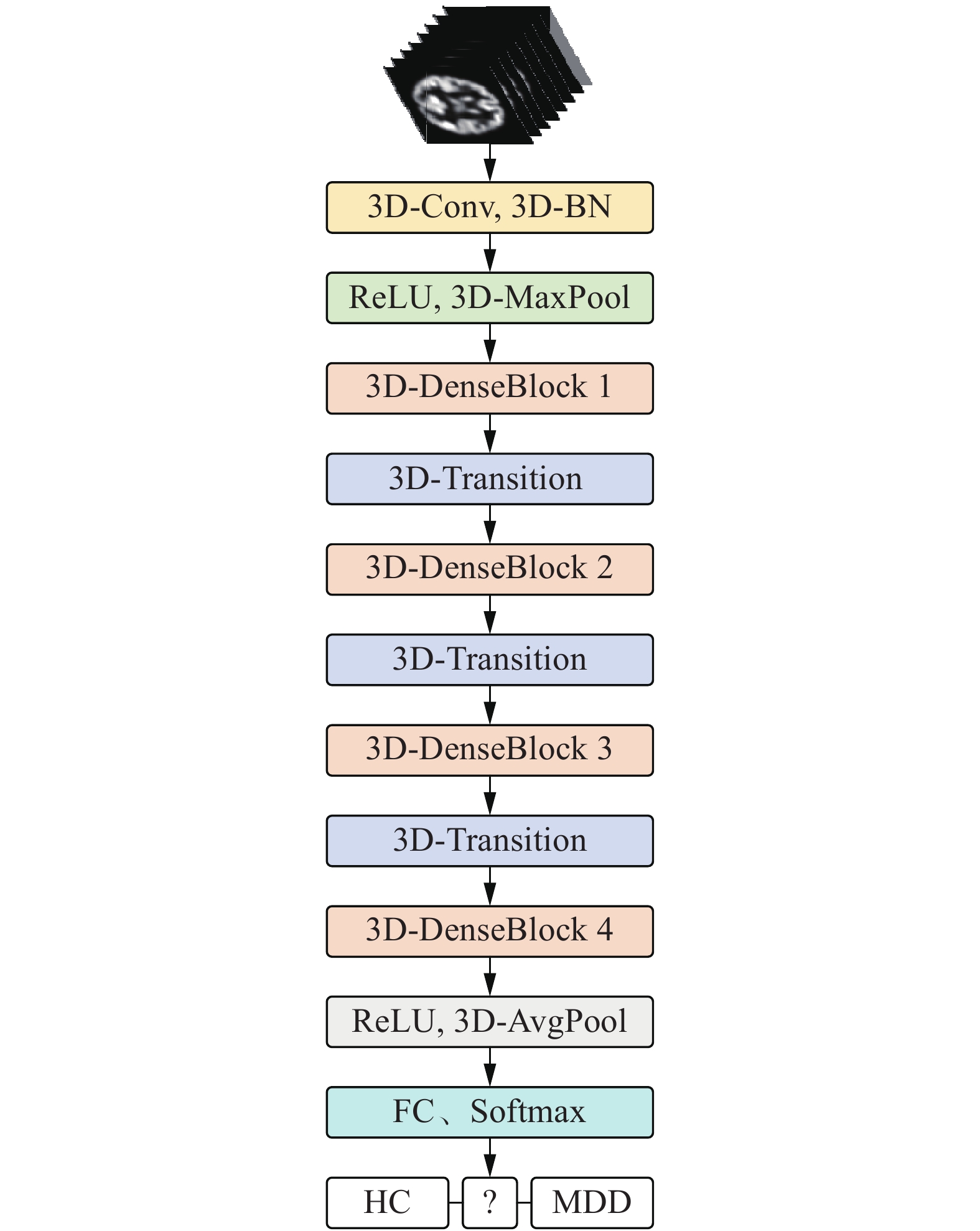
|
Download:
|
| 图 2 3D-DenseNet的121层网络结构 Fig. 2 Structure of 3D-DenseNet 121 | |

|
Download:
|
| 图 3 一个6层3D-DenseBlock结构 Fig. 3 A 6-layer 3D-DenseBlock | |
每一层的密集连接(dense connectivity)都可表示为
| ${{{x}}_l} = {H_l}([{x_0}\;{x_1}\; \cdots \;{x_{l - 1}}])$ | (2) |
式中:
式(2)中的密集连接操作在特征图大小不一致时是不可行的,所以在每2个3D-DenseBlock之间都加入一个过渡层3D-Transition,包含一个BN层、一个ReLU层、一个1×1×1卷积层,以及一个均匀池化层(AvgPool),用来进行特征图降维。最后,一个3D-DenseBlock后面连接一个ReLU层、一个AvgPool层、一个全连接层(FC),以及一个分类器层(Softmax),用来实现最后的特征降维和分类。
一个121层的3D-DensNet网络的具体参数和架构如表2所示,其中每一个Conv都代表一个BN-ReLU-Conv顺序流程。
| 表 2 3D-DenseNet 121网络架构 Tab.2 Parameters and architecture of the 3D-DenseNet 121 |
卷积神经网络使用反向传播算法进行训练,其中每一层的参数在迭代中不断更新,以最小化特定的损失函数值。通常,训练之前会用一组随机值将参数初始化。值得注意的是,网络层数的增加会导致网络参数量的增加,这就需要更多的训练数据,以使反向传播算法更好地收敛,如果数据量不够,很容易造成过拟合,使算法陷入局部最小值,导致分类性能欠佳。为解决这个问题,一个可行的方法是采取迁移学习。在迁移学习中,网络参数的初始值并不是随机的,而是从已经在更大数据集上训练过的网络模型中复制的,随后再对网络进行微调。
由于医学影像数据普遍数据量不大且难以获取,很多学者探索将迁移学习引入医学图像领域。Tajbakhsh等[25]深入地讨论和比较了在医学图像分析领域中,从零开始的训练网络和引入迁移学习,并进行微调的结果。研究表明,在大多数情况下,引入迁移学习并进行微调,比从零开始的训练网络效果更好。目前为止,迁移学习已被应用于阿尔兹海默症[26]、脑瘤[27]和肺结节[28]等疾病的医学图像分类或分割研究当中,并表现出了优秀的效果。据我们所知,目前没有任何关于抑郁症医学图像分类的研究使用迁移学习,但所有上述研究都证明,将迁移学习应用于医学成像领域具有巨大的价值,并且与从无到有的训练相比,在训练数据集较小的情况下,迁移学习的引入有可能提高抑郁症sMRI图像分类的精度。
Chen等[29]收集了包括肝脏、心脏等8种不同部位的3D医学图像分割数据集,在训练过程中,8组数据共享编码器,并分别使用8个解码器,最后只迁移共用的编码器部分用于肺结节CT图像的分类研究。受Chen等[29]的启发,本文设计了一个新颖的三维sMRI数据迁移学习框架。在数据选择方面,只收集同一部位(大脑)和同一类型(sMRI)的数据进行预训练,并只考虑分类任务。在这3个限制条件下,选择使用开源的老年痴呆症数据集ADNI(https://ida.loni.usc.edu)作为预训练数据。为了实现本文的迁移学习,设计了1个3步处理工作流,如图4所示。

|
Download:
|
| 图 4 迁移学习架构 Fig. 4 Framework of the proposed transfer learning | |
1)从ADNI数据库中挑选出合适的sMRI数据,包含阿兹海默患者(alzheimer’s disease, AD)、轻度认知障碍(mild cognitive impairment, MCI),以及健康对照组(heathy control, HC)3类,共656个被试者,并用与本文中相同的数据预处理步骤,对挑选出来的数据进行预处理;
2)用所选数据训练1个三分类的3D-DenseNet网络,让网络学习sMRI数据的特征;
3)将训练好的网络模型中的主干网络(图4中红框部分)部分迁移到抑郁症sMRI数据的分类任务中,并添加二分类层,使用本文的抑郁症sMRI数据继续进行训练,以增强网络的分类性能。
之所以只从脑部sMRI数据集中选择数据作预训练,是因为如果选择的源域和目标域的相似度太小,很可能会导致负迁移,从而导致性能下降,即分类准确率不提高反而降低。相反,2个数据集越相似,2个数据集的高层特征就越相似,这将产生更好的代表性特征和更适合目标域的预训练模型,以提高目标域任务的分类性能。为了进行对比实验,在第3步中用相同的过程和相同的数据训练三维化的ResNet。此外,本文使用较小的学习速率对迁移过后的主干网络进行微调,用较大的学习速率来训练未经过预训练的分类层,以使传输的网络能够从MDD数据中提取一些新的特征,提高分类性能。
3 实验与结果分析为了验证本文提出方法的有效性,本文精心设计了一系列实验,探讨不同深度学习网络以及三维化相应网络对分类精度的影响,验证所设计的迁移学习流程的有效性和优越性。
3.1 模型评价指标本文分类问题为二分类问题,也就是将样本分成MDD患者和HC两类,规定MDD患者为正类(positive),HC为负类(negative),分类算法对测试数据集的预测有正确或错误之分,具体包括将正类预测为正类数(true positive, TP)、将正类预测为负类数(false negative, FN)、将负类预测为正类数(false positive, FP)、将负类预测为负类数(true negative, TN)4种情况。针对研究课题的特殊性,本文选取了正确率和召回率2个指标来评价模型的分类性能。正确率定义为Accuracy = (TP + TN)/(TP + FN + FP + TN),反映了分类器对所有样本的判定能力;召回率定义为Recall = TP/(TP+FN),反映了被正确判定的MDD患者占患者总数的比重。
3.2 训练设置所有网络在训练时都使用交叉熵损失函数与Adam优化算法[30],weight decay值设置为0.001,将所有数据按80%、10%、10%的比例划分训练、验证、测试集,并采用五折交叉验证,epoch值设置为100。由于GPU内存容量有限,在训练2D网络时batch size设置为64,在训练3D网络时batch size设置为8。当不使用迁移学习时学习率初始值设置为0.01,当使用迁移学习时,未迁移部分的初始学习率保持原有的0.01,迁移部分的初始学习率部分设为原有的0.001倍,当验证集loss值连续10个epoch不下降时,学习率变为原来的0.1倍。所有训练都是在一台带有一块NVIDIA TITAN Xp GPU、四核Intel Xeon E3-1230 v5 3.41GHz处理器的win10专业版系统服务器上进行的,编程语言为Python3.6,深度学习框架为PyTorch1.0。
3.3 不同深度的2D网络对比实验在进行2D网络实验时,将预处理过后的sMRI数据分层输入网络,输入尺寸为121×145,并采用投票算法,即对于每个被试者,测试结果为正类的层数超过一半判定为正类,否则判定为负类。通过对比几个不同深度的网络,以证明网络深度对分类性能的影响和DenseNet的相对优越性,实验结果见表3。
| 表 3 2D网络实验结果 Tab.3 2D network experimental results |
从表3中的数据可以看出,随着网络层数的增多,网络的分类准确率和召回率大体上逐步升高,这表明网络的加深可以提供更好的非线性表达能力,学习更加复杂的知识,从而可以拟合更加复杂的特征输入;而DenseNet在层数大致相同的情况下比ResNet表现得更好,表现最好的DenseNet264达到了69.96%的准确率和76.32%的召回率,这表明DenseNet中的密集连接思想在本研究中优于ResNet的残差思想,故接下来的实验主要以DenseNet为主。
3.4 3D网络与2D网络的对比实验接下来,将部分2D网络三维化,并进行了对比实验,以证明3D网络的优越性,实验结果见表4。
| 表 4 2D网络与3D网络实验结果对比 Tab.4 Comparison of experimental results between 2D and 3D networks |
从表4中的数据可以看出,对网络进行三维化操作后,分类准确率和召回率有了显著提升,例如3D-DenseNet264的分类准确率比二维的DenseNet264提升了7.46%;而层数相似的3D-DenseNet要比3D-ResNet效果好,例如3D-DenseNet201的分类精度为76.53%,3D-ResNet200的分类精度为74.81%。这表明抑郁症sMRI数据的层间信息非常丰富,而本文提出的三维密集连接网络3D-DenseNet恰恰能够挖掘出这些有效信息,相比一般的DenseNet以及其他传统的二维网络提供更多的有效特征,从而提高分类性能。
3.5 迁移学习对比实验本文将3D-DenseNet系列中表现最好的3D-DenseNet264用ADNI数据库做了预训练,并进行迁移学习(以下称ADNI-Transfer),与直接用抑郁症数据训练(以下称None)的实验结果进行对比。因为Chen等[29]使用的迁移学习方法(以下称Med3D-Transfer)只在3D-ResNet系列网络做了实验,并且只释放出了预训练好的模型,并未提供训练数据的获取渠道,故为了证明本文提出的ADNI-Transfer方法的优越性,将3D-ResNet系列中表现最好的3D-ResNet200也做了ADNI-Transfer,并与进行Med3D-Transfer的3D-ResNet200网络作对比,实验结果见表5。
| 表 5 迁移学习实验结果对比 Tab.5 Comparison of experimental results of transfer learning |
从表5可以看出,引入迁移学习后,网络的分类性能有了显著提升,例如3D-DenseNet264在经过ADNI-Transfer操作之后,分类准确率提升了6.95%,这说明迁移学习能够将其他领域的知识引入到抑郁症sMRI数据的分类任务中,一定程度上解决了样本不充足问题,从而加快模型训练的效率,提升模型最终的泛化能力。相比于Med3D-Transfer方法,本文提出的ADNI-Transfer迁移学习方法在准确率和召回率上均有更优表现,例如进行ADNI-Transfer的3D-ResNet200网络的分类准确率要比进行Med3D-Transfer的3D-ResNet200网络高出2.83%,召回率高出3.54%,这说明网络从与目标领域数据相同部位、相同类型的源领域数据中提取信息对目标任务更有价值,从而这样的做法也更能提高目标任务的评价指标,也就是更能够提升抑郁症sMRI数据的分类精准确率和召回率。由本文提出的3D-DenseNet264网络和ADNI-Transfer迁移学习方法组合后得到的实验结果明显优于其他方法,证明了本文提出方法的有效性和优越性。
4 结束语本文提出一种三维密集连接深度学习网络3D-DenseNet,并首次将它用于三维抑郁症sMRI数据的分类研究中。通过对DenseNet进行三维化改进,使之能够更好地提取三维信息,在与多个计算机视觉领域先进的网络对比实验中发现,本文设计的网络均表现出更优的性能,并且随着网络深度的增加,网络的分类性能也随之提升。除此之外,引入迁移学习,针对目标任务数据的特殊性,精心设计了一个迁移学习工作流程ADNI-Transfer,并与当下领域内流行的迁移学习方法作了比较,实验结果证明了本文提出的迁移学习方法的有效性和优越性。同前沿方法相比,本文提出的方法能够有效提高抑郁症与健康对照者的sMRI数据分类准确率和召回率,继而辅助医生完成诊断,充分验证了提出方法的有效性和可行性,具有重要的研究价值。
| [1] |
世界卫生组织. 抑郁症[EB/OL]. (2019−08−29)[2019−12−04] https://www.who.int/zh/news-room/fact-sheets/detail/depression. World Health Organization. Depression fact sheets[EB/OL]. (2019−08−29)[2019−12−04] https://www.who.int/zh/news-room/fact-sheets/detail/depression. (  0) 0)
|
| [2] |
BRANDT W A, LOEW T, VON HEYMANN F, et al. How does the ICD-10 symptom rating (ISR) with four items assess depression compared to the BDI-II? A validation study[J]. Journal of affective disorders, 2015, 173: 143-145. DOI:10.1016/j.jad.2014.10.070 ( 0) 0)
|
| [3] |
MASKE U E, HAPKE U, RIEDEL-HELLER S G, et al. Respondents’ report of a clinician-diagnosed depression in health surveys: comparison with DSM-IV mental disorders in the general adult population in Germany[J]. BMC psychiatry, 2017, 17(1): 39. DOI:10.1186/s12888-017-1203-8 ( 0) 0)
|
| [4] |
GIEDD J N. Structural magnetic resonance imaging of the adolescent brain[J]. Annals of the New York academy of sciences, 2004, 1021(1): 77-85. DOI:10.1196/annals.1308.009 ( 0) 0)
|
| [5] |
GAO Shuang, CALHOUN V D, SUI Jing. Machine learning in major depression: from classification to treatment outcome prediction[J]. CNS neuroscience & therapeutics, 2018, 24(11): 1037-1052. ( 0) 0)
|
| [6] |
HILBERT K, LUEKEN U, MUEHLHAN M, et al. Separating generalized anxiety disorder from major depression using clinical, hormonal, and structural MRI data: a multimodal machine learning study[J]. Brain and behavior, 2017, 7(3): e00633. DOI:10.1002/brb3.633 ( 0) 0)
|
| [7] |
SANKAR A, ZHANG Tianhao, GAONKAR B, et al. Diagnostic potential of structural neuroimaging for depression from a multi-ethnic community sample[J]. BJPsych open, 2016, 2(4): 247-254. DOI:10.1192/bjpo.bp.115.002493 ( 0) 0)
|
| [8] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 ( 0) 0)
|
| [9] |
马世龙, 乌尼日其其格, 李小平. 大数据与深度学习综述[J]. 智能系统学报, 2016, 11(6): 728-742. MA Shilong, WUNIRI Qiqige, LI Xiaoping. Deep learning with big data: state of the art and development[J]. CAAI transactions on intelligent systems, 2016, 11(6): 728-742. (  0) 0)
|
| [10] |
刘帅师, 程曦, 郭文燕, 等. 深度学习方法研究新进展[J]. 智能系统学报, 2016, 11(5): 567-577. LIU Shuaishi, CHENG Xi, GUO Wenyan, et al. Progress report on new research in deep learning[J]. CAAI transactions on intelligent systems, 2016, 11(5): 567-577. (  0) 0)
|
| [11] |
KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks[C]//Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, USA, 2012: 1097−1105.
( 0) 0)
|
| [12] |
DONAHUE J, HENDRICKS L A, ROHRBACH M, et al. Long-term recurrent convolutional networks for visual recognition and description[J]. IEEE transactions on pattern analysis and machine intelligence, 2017, 39(4): 677-691. DOI:10.1109/TPAMI.2016.2599174 ( 0) 0)
|
| [13] |
KERMANY D S, GOLDBAUM M, CAI Wenjia, et al. Identifying medical diagnoses and treatable diseases by image-based deep learning[J]. Cell, 2018, 172(5): 1122-1131, e1-e2. DOI:10.1016/j.cell.2018.02.010 ( 0) 0)
|
| [14] |
吕鸿蒙, 赵地, 迟学斌. 基于增强AlexNet的深度学习的阿尔茨海默病的早期诊断[J]. 计算机科学, 2017, 44(S1): 50-60. LV Hongmeng, ZHAO Di, CHI Xuebin. Deep learning for early diagnosis of Alzheimer's disease based on intensive AlexNet[J]. Computer science, 2017, 44(S1): 50-60. (  0) 0)
|
| [15] |
LITJENS G, KOOI T, BEJNORDI B E, et al. A survey on deep learning in medical image analysis[J]. Medical image analysis, 2017, 42: 60-88. DOI:10.1016/j.media.2017.07.005 ( 0) 0)
|
| [16] |
SZEGEDY C, LIU Wei, JIA Yangqing, et al. Going deeper with convolutions[C]//IEEE Conference on Computer Vision and Pattern Recognition. Boston, USA, 2015: 1−9.
( 0) 0)
|
| [17] |
SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition[J]. Computer science, 2014, 18(3): 178-182. ( 0) 0)
|
| [18] |
SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision[C]//IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas, USA, 2016: 2818−2826.
( 0) 0)
|
| [19] |
HE Kaiming, ZHANG Xiangyu, REN Shaoqing, et al. Deep residual learning for image recognition[C]//IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas, USA, 2016: 770−778.
( 0) 0)
|
| [20] |
HUANG Gao, LIU Zhuang, VAN DER MAATEN L, et al. Densely connected convolutional networks[C]//IEEE Conference on Computer Vision and Pattern Recognition. Honolulu, USA, 2017: 2261−2269.
( 0) 0)
|
| [21] |
ASHBURNER J, BARNES G, CHEN Chunchuan, et al. SPM12 manual[EB/OL]. (2019−01−13)[2020−08−29]. https://www.fil.ion.ucl.ac.uk/spm/software/spm12.
( 0) 0)
|
| [22] |
ARNONE D, MCKIE S, ELLIOTT R, et al. State-dependent changes in hippocampal grey matter in depression[J]. Molecular psychiatry, 2013, 18(12): 1265-1272. DOI:10.1038/mp.2012.150 ( 0) 0)
|
| [23] |
IOFFE S, SZEGEDY C. Batch normalization: accelerating deep network training by reducing internal covariate shift[C]//Proceedings of the 32nd International Conference on International Conference on Machine Learning. Lille, France, 2015: 448−456.
( 0) 0)
|
| [24] |
GLOROT X, BORDES A, BENGIO Y. Deep sparse rectifier neural networks[C]//Proceedings of the Fourteenth International Conference on Artificial Intelligence and Statistics. Fort Lauderdale, USA, 2011: 315−323.
( 0) 0)
|
| [25] |
TAJBAKHSH N, SHIN J Y, GURUDU S R, et al. Convolutional neural networks for medical image analysis: full training or fine tuning[J]. IEEE transactions on medical imaging, 2016, 35(5): 1299-1312. DOI:10.1109/TMI.2016.2535302 ( 0) 0)
|
| [26] |
HON M, KHAN N M. Towards Alzheimer’s disease classification through transfer learning[C]//IEEE International Conference on Bioinformatics and Biomedicine. Kansas City, USA, 2017: 1166−1169.
( 0) 0)
|
| [27] |
LIU Renhao, HALL L O, GOLDGOF D B, et al. Exploring deep features from brain tumor magnetic resonance images via transfer learning[C]//International Joint Conference on Neural Networks. Vancouver, Canada, 2016: 235−242.
( 0) 0)
|
| [28] |
DA NÓBREGA R V M, PEIXOTO S A, DA SILVA S P P, et al. Lung nodule classification via deep transfer learning in CT lung images[C]//IEEE 31st International Symposium on Computer-Based Medical Systems. Karlstad, Sweden, 2018: 244−249.
( 0) 0)
|
| [29] |
CHEN S, MA K, AND ZHENG Y. Med3D: transfer learning for 3D medical image analysis[EB/OL].(2019−04−09)[2019−09−025] https://arxiv.org/abs/1904.00625.
( 0) 0)
|
| [30] |
DIEDERIK P K, JIMMY B. Adam: a method for stochastic optimization[J/OL]. (2017-1-30)[2019-9-29] https://arxiv.org/abs/1412.6980v5.
( 0) 0)
|
 2021, Vol. 16
2021, Vol. 16


