文章信息
- 胡艳, 黄智平, 周维, 张红卫, 俞雷
- Hu Yan, Huang Zhiping, Zhou Wei, Zhang Hongwei, Yu Lei
- miR-605-3p通过靶向ZIK1抑制肝细胞癌迁移和侵袭
- miR-605-3p inhibits hepatocellular carcinoma cell migration and invasion by targeting ZIK1
- 实用肿瘤杂志, 2020, 35(1): 30-36
- Journal of Practical Oncology, 2020, 35(1): 30-36
-
作者简介
- 胡艳(1973-), 女, 上海人, 副主任医师, 从事肿瘤分型及靶点识别研究.
-
通信作者
- 俞雷, E-mail:yuulei@hotmail.com
-
文章历史
- 收稿日期:2018-09-20
2. 广州军医广州总医院肝脏外科, 广东 广州 510010
2. Department of Liver Surgery, General Hospital of Guangzhou Military Command of PLA, Guangzhou 510010, China
肝细胞癌(hepatocellular carcinoma, HCC)是全球范围内最常见的恶性肿瘤之一,在亚洲和非洲的发展中国家中发病率最高[1]。虽然临床上肝癌治疗技术和手段不断发展和提高,但肝癌患者整体预后仍不理想,10年总生存率为22%~35%[2]。由于肝癌的发生过程涉及众多的癌基因和肿瘤抑制基因[3-4], 导致HCC确切的致癌机制仍有待进一步探索[5-6]。
microRNAs(miRNAs)是由19~25个高度保守核苷酸组成的小非编码RNAs,能够调控众多编码基因[7-8]。miRNAs可通过直接与目标mRNA的3′非翻译区域(3′-untranslated region,3′-UTR)结合导致蛋白合成的转录后抑制。许多miRNAs参与包括细胞凋亡、增殖、分化、干细胞更新、应激反应和代谢在内的多种细胞过程[9-13]。近年来有关miRNAs与肿瘤关系的研究日益增多。miR-605家族是目前研究较多的miRNA家族之一。miR-605首先被证明通过抑制鼠双微体2的表达活化p53。越来越多的证据表明,miR-605的异常表达参与多种肿瘤细胞的癌变过程,如胃癌[14]、前列腺癌[15]和肺癌[16]等。研究发现,miR-605在肝内胆管癌中异常低表达并且通过靶向抑制非ATP酶蛋白酶体26S亚基蛋白表达抑制肝内胆管癌细胞增殖和转移[17]。但对其在肝细胞癌中的生物作用和临床意义研究甚少。miR-605-3p是miR-605重要成员之一。本研究组发现,在肝细胞癌组织中miR-605-3p的低水平表达与患者预后不良相关,因此重点研究miR-605-3p在肝细胞癌进展中的作用和相应机制。
1 材料与方法 1.1 临床标本采集及处理收集广州军医广州总医院肝胆外科2009年1月1日至2013年5月31日收治的80例肝细胞癌患者的肝细胞癌组织和配对癌旁组织,用于实时定量PCR分析。所有样本通过组织病理学检查证实为肿瘤或非肿瘤组织。组织样本冻存于-80 ℃以备后用。本研究得到广州军医广州总医院机构审查委员会的批准。参与研究的所有患者均签署知情同意书。患者年龄45~81岁,中位年龄56.5岁;男性72例,女性8例。中位随访时间3(0.5~7)年。
1.2 细胞株和细胞培养人肝癌细胞株(Huh7、HCC-LM3、Hep3B以及SMMC-7721)和肝细胞株(THLE-3)均购自中国上海生命科学研究所细胞资源中心。所有细胞株均在5% CO2、37℃湿润的环境中培养于含10%胎牛血清(Life Technologies,美国)和1%的青/链霉素(Life Technologies, 美国)的DMEM (HyClone, 美国)培养液中。
1.3 RNA提取﹑反转录和实时定量PCRRNA抽提试剂盒购自美国Qiagen公司,依照说明书提取组织或细胞中总RNA。根据cDNA合成试剂盒使用说明书反转录mRNA和miRNA。在CFX96系统上使用iQ SYBR Green进行互补DNA的扩增和定量。以U6或β-actin作为内参。以2-ΔΔCt方法计算基因的相对表达。定量PCR试剂盒购自美国Qiagen公司,检测目的基因的表达。所有试验均重复3次。PCR实验按以下条件进行:30次循环由94℃变性30 s;56℃退火30 s;72℃延伸30 s。
1.4 划痕试验划痕试验参考文献[18]:6孔板每孔种植1×105个细胞;细胞融合后在培养板中间划痕同时用无血清培养液取代之前培养液进行培养;孵育24 h后检测细胞愈合面积。
1.5 Transwell试验Transwell试验参考文献[18],实验采用Millicell 24孔培养插入板(Millipore, 美国)和孔径大小为8 μm的聚碳酸酯膜。插入板中加入0.5 mL的DMEM在5% CO2、37℃条件下放置1 h,用含10%胎牛血清的0.5 mL DMEM替换置于下室。上室加入含5×104细胞的无血清DMEM 400 μL。孵育24 h后, 使用PBS清洗插入板且刮掉膜上表面的细胞。用Giemsa染色对膜下细胞进行染色并在显微镜下计数。
1.6 RNA寡核苷酸的合成和瞬时转染在线软件预测miR-605-3p与ZIK1之间的结合位点(http://www.targetscan.org/vert_7.1/)。miR-605-3p模拟物(miR-605-3p mimic)、模拟物对照(NC mimic)、miR-605-3p抑制剂(miR-605-3p inhibitor)、抑制剂对照(NC inhibitor)均合成自上海吉玛有限责任公司。ZIK1过表达载体及NC载体来源于上海吉凯有限公司。后续转染操作根据说明进行。
1.7 细胞增殖试验采用细胞计数Kit-8(cell counting kit-8,CCK-8)测定细胞增殖。转染24 h后,以3 000个细胞/孔种植于96孔板中。种植72 h后加入CCK-8试剂(上海三工生物科技有限公司)。用酶标仪检测450 nm处吸光度(absorbance,A)。
1.8 Western blot分析用10%硫酸十二烷基钠聚丙烯酰胺凝胶分离细胞蛋白裂解物, 电泳转移到聚偏二氟乙烯膜上, 用兔源一抗ZIK1单克隆抗体(Abcam公司,美国)进行检测。使用实验室工作图像采集和分析软件进行条带密度的定量。
1.9 荧光素酶活性分析在pGL3/荧光素酶载体下游扩增和克隆ZIK1的3′ UTR。以pGL3/luc-ZIK1 3′UTR为模板扩增ZIK1的突变体3′ UTR,并在pGL3/Luc载体下游克隆。荧光素酶报告试验中, 将SMMC-7721细胞与miR-605-3p模拟物或对照物,pGL3/luc-ZIK1 3′ UTR或突变体3′ UTR及对照物共转染。转染48 h后, 根据说明书使用RIPA缓冲液裂解细胞, 用F-4500荧光分光光度计测定荧光素酶强度。
1.10 统计学分析采用GraphPad 5.0软件进行数据分析。所有实验重复3次。计数资料采用均数±标准差( x±s)表示,两组间比较采用配对t检验。计量资料采用频数(百分比)表示,组间比较采用χ2检验。采用Kaplan-Meier法绘制生存曲线,生存率比较采用Log-rank检验。以P < 0.05为差异具有统计学意义。
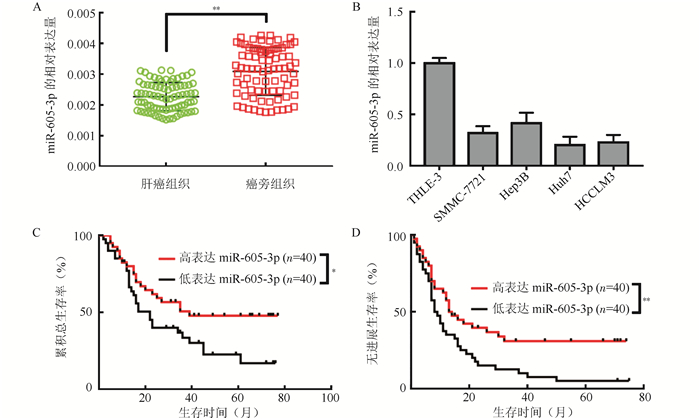
2 结果 2.1 肝癌中miR-605-3p的表达及与患者预后的关系定量PCR检测显示,肝细胞癌组织中miR-605-3p较癌旁组织表达降低(P < 0.01,图 1A), Huh7、HCC-LM3、Hep3B以及SMMC-7721肝癌细胞株中miR-605-3p的表达均较肝细胞株THLE-3降低(P < 0.05,图 1B)。以miR-605-3p在肝癌肿瘤组中位表达量分为高表达和低表达,高表达40例,低表达40例。Kaplan-Meier曲线显示,miR-605-3p在肝癌组织低表达患者5年总生存率(21.6% vs 48.3%,P < 0.05,图 1C)和无进展生存率均低于高表达患者(9.5% vs 30.8%,P < 0.05,图 1D)。
|
| 注 A:定量PCR检测80例配对的肝癌组织和癌旁组织中miR-605-3p的转录水平(U6 RNA为标准化内参);B:人肝癌细胞株和正常肝细胞株中miR-605-3p转录水平;C:miR-605-3p高表达和低表达患者总生存曲线;D:miR-605-3p高表达和低表达患者无进展生存曲线;*P < 0.05;**P < 0.01 图 1 miR-605-3p在肝细胞癌中表达下调且与肝细胞癌患者预后相关 Fig.1 miR-605-3p is down-regulated in hepatocellular carcinoma and correlated with the prognosis of HCC patients |
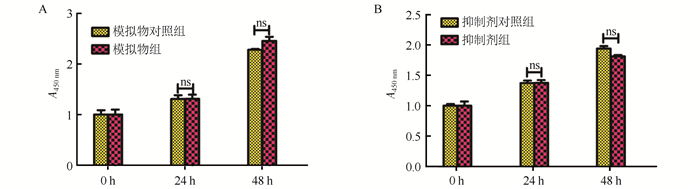
为研究肝细胞癌中miR-605-3p的生物学功能, 选择SMMC-7721细胞进行功能学研究。SMMC-7721细胞分别转染miR-605-3p模拟物(过表达miR-605-3p质粒)和miR-605-3p抑制剂。定量PCR检测验证过表达和抑制miR-605-3p的效率。转染模拟物组较模拟物对照组miR-605-3p的表达增高53.2倍,差异具有统计学意义(P < 0.01);抑制剂对照组较抑制剂组miR-605-3p表达高9.8倍,差异具有统计学意义(P < 0.01)。细胞增殖实验显示,miR-605-3p过表达或抑制表达均不影响HCC细胞的增殖能力(均P>0.05,图 2A~2B)。
|
| 注 A:CCK8试验中转染72 h后miR-605-3p模拟物组和模拟物对照组细胞生长情况; B:CCK8试验中转染72 h后miR-605-3p抑制剂组和抑制剂对照组细胞生长情况比较; ns: P > 0. 05 图 2 miR-605-3p对肝癌细胞株SMMC-7721生长的影响 Fig.2 Effect of miR-605-3p on the growth of HCC cell SMMC-7721 |
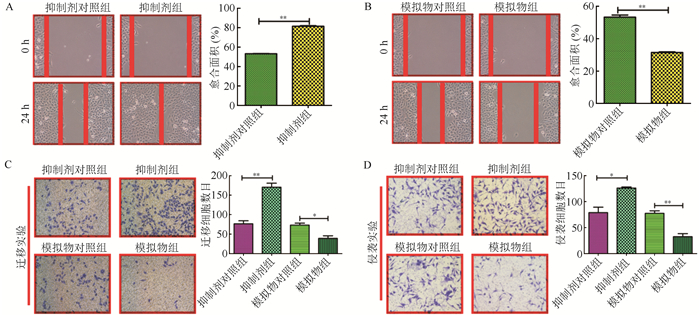
在划痕实验中,0 h和24 h镜检显示,抑制剂组细胞较抑制剂对照组细胞迁移能力强(P < 0.05,图 3A)。模拟物组较模拟物对照组细胞迁移能力下降(P < 0.05,图 3B)。Transwell实验发现,与其他组比较,抑制剂组细胞具有更强的迁移和侵袭能力(P < 0.05), 而模拟物组细胞表现出较弱的迁移和侵袭能力(P < 0.05,图 3C~D)。
|
| 注 A:miR-605-3p抑制剂组和抑制剂对照组细胞划痕实验结果; B:miR-605-3p模拟物组和模拟物对照组细胞划痕实验结果; C:转染72 h后transwell转移实验结果; D:转染72 h后transwell侵袭实验结果; 在100倍镜下随机3个视野进行细胞计数; *P < 0. 05;**P < 0. 01 图 3 miR-605-3p对肝癌细胞株SMMC-7721迁移和侵袭具有抑制作用 Fig.3 Inhibition effect of miR-605-3p in the migration and invasion of HCC cell SMMC-7721 in vitro |
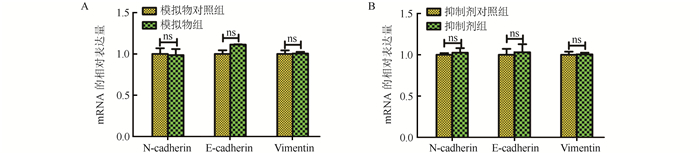
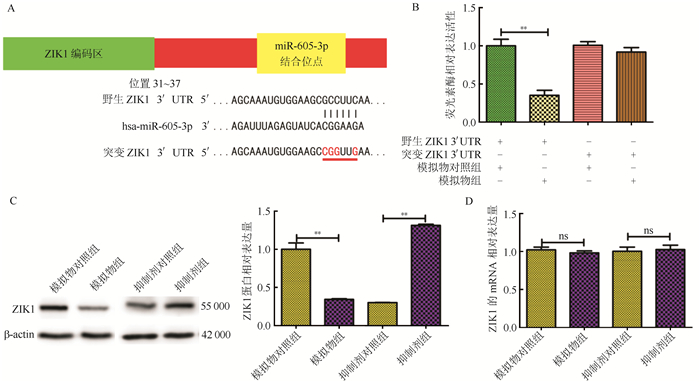
实时PCR检测结果显示,miR-605-3p对E-cadherin、N-cadherin和vimentin等上皮细胞-间充质转化(epithelial-mesenchymal transition, EMT)相关基因表达水平无影响(均P>0.05,图 4)。为探索miR-605-3p在肝细胞癌细胞迁移和侵袭中可能调控的靶基因,使用TargetScan (http://www.targetscan.org)预测到ZIK1的3′UTR有miR-605-3p的潜在结合位点,采用荧光素酶报告实验验证ZIK1是否是miR-605-3p的直接靶点, 为此构建ZIK1 3′UTR的野生型(wild type, WT)和突变型(mutant type, MUT,图 5A)。结果显示,miR-605-3p过表达抑制野生报告质粒的荧光素酶活性,而突变型报告质粒则无影响(图 5B)。此外,miR-605-3p过表达后ZIK1蛋白表达水平降低;miR-605-3p敲低后ZIK1蛋白表达水平升高(图 5C)。ZIK1 mRNA表达水平无明显变化(图 5D)。
|
| 注 A:miR-605-3p模拟物组和模拟物对照组转染72 h后RT-PCR检测N-cadherin、E-cadherin和vimentin mRNA水平; B:miR-605-3p抑制剂组和抑制剂对照组转染72 h后RT-PCR检测N-cadherin、E-cadherin和vimentin mRNA水平; ns:P > 0. 05 图 4 miR-605-3p对肝癌细胞株SMMC-7721中上皮细胞-间充质转化相关基因表达水平的影响 Fig.4 Effect of miR-605-3p on the expression levels of epithelial-mesenchymal transformation-related genes in HCC cell SMMC-7721 |
|
| 注 A:序列显示, ZIK1 3′ UTR上预测的miR-605-3p结合位点和突变miR-605-3p-结合位点是通过定点诱变产生的; B:在SMMC-7721细胞中, 双荧光素酶报告实验显示, 只有当miR-605-3p与野生型ZIK1 3′ UTR配对时才会显著抑制荧光素酶活性; C:Western blot分析SMMC-7721细胞转染miR-605-3p后ZIK1蛋白的表达情况; D:qRT-PCR检测SMMC-7721细胞转染miR-605-3p后ZIK1mRNA表达; **P < 0. 01 图 5 miR-605-3p与ZIK1 3′UTR直接结合 Fig.5 Direct binding of miR-605-3p to 3′UTR of ZIK1 2.4 |
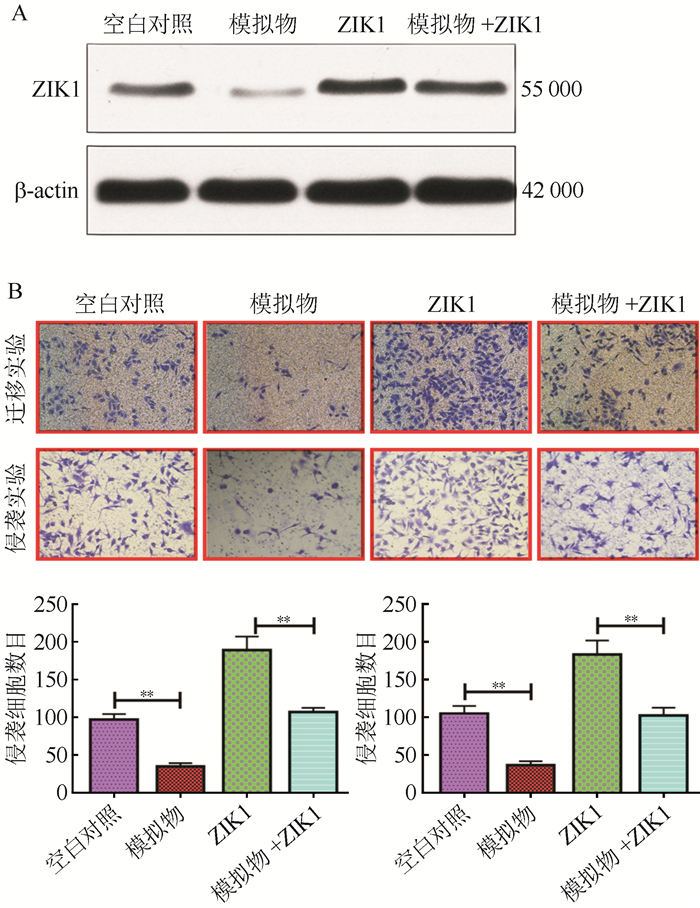
在SMMC-7721细胞中进行功能学拯救实验,将pcDNA3.1- ZIK1质粒转染至SMMC-7721细胞内(图 6A)。过表达的ZIK1减弱miR-605-3p对肝细胞癌细胞迁移和侵袭能力的抑制作用(图 6B)。
|
| 注 A:Western blot检测ZIK1质粒转染SMMC-7721细胞后ZIK1蛋白表达量; B:Transwell迁移实验和侵袭实验结果; **P < 0. 01 图 6 过表达ZIK1逆转miR-605-3p对肝癌细胞SMMC-7721迁移和侵袭能力的抑制作用 Fig.6 Overexpression of ZIK1 reverses the inhibitory effect of miR-605-3p on the migration and invasion of HCC cell SMMC-7721 |
虽然有许多参与肝癌发生的生长因子、致癌基因和抑癌基因在不断被发现[19-20], 但导致肝癌发生的确切分子机制尚不清楚。研究表明,microRNAs可通过靶向癌基因或肿瘤抑制基因在肝癌发生和发展中发挥重要作用[21-22]。本研究比较miR-605-3p在肝癌组织中与癌旁正常组织中的表达情况,证明miR-605-3p在肝癌组织中异常低表达,提示miR-605-3p在肝细胞癌的发生和发展中发挥重要作用。进一步的生存曲线分析证明miR-605-3p低表达的肝癌患者临床预后较差,提示miR-605-3p可能是临床上肝癌治疗的潜在治疗靶点和评估患者预后的临床肿瘤标志物。既往研究表明,miR-605在肝内胆管癌中异常低表达并且通过靶向抑制PSMD表达抑制肝内胆管癌细胞增殖和转移[17],然而本研究的细胞增殖实验提示,miR-605-3p对肝细胞癌的增殖能力没有显著影响,提示miR-605家族不同成员在不同肿瘤类型发挥的生物功能作用不同,这也符合肿瘤异质性的特性。
本研究探讨miR-605-3p对肝细胞癌迁移和侵袭的影响及其分子生物学机制。通过生物信息学和分子实验学手段, 确定ZIK1基因可能是miR-605-3p一个新的作用靶点。ZIK1最初被认为是一种与细胞核核糖核酸蛋白粒子K蛋白结合的转录抑制因子[23-24],在胃肠化生中表现高度甲基化[24]。ZIK1异常表达与肾细胞癌[21]、弥漫性大B细胞淋巴瘤[25]、食管癌[26]、结直肠癌和胃癌的发生有关[27]。本研究通过荧光素酶报告基因检测,实时PCR和Western blot验证miR-605-3p通过与其3′ UTR相互作用直接靶向ZIK1。这些数据表明,miR-605-3p可以通过靶向ZIK1部分抑制肝癌细胞迁移和侵袭,不仅为HCC的进展和转移提供新的线索,也为癌症的预防和治疗提供一个潜在的靶点。
综上所述,本研究发现,miR-605-3p在肝癌组织中下调, 可作为肝癌患者预后的标志物。本研究结果提示,miR-605-3p可以通过直接抑制ZIK1的表达来抑制HCC细胞的迁移和侵袭, miR-605-3p能否成为肝细胞癌早期诊断和治疗的新靶点有待进一步研究。
| [1] |
Ye JZ, Wang YY, Bai T, et al. Surgical resection for hepatocellular carcinoma with portal vein tumor thrombus in the asia-pacific region beyond the barcelona clinic liver cancer treatment algorithms:A review and update[J]. Oncotarget, 2017, 8(54): 93258-93278. |
| [2] |
何明燕, 夏景林. 肝癌精准医学之路:面临的挑战[J]. 实用肿瘤杂志, 2018, 33(1): 34-37. |
| [3] |
Ren FH, Yang H, He RQ, et al. Analysis of microarrays of mir-34a and its identification of prospective target gene signature in hepatocellular carcinoma[J]. BMC Cancer, 2018, 18(1): 12. DOI:10.1186/s12885-017-3941-x |
| [4] |
Geng M, Xin X, Bi LQ, et al. Molecular mechanism of hepatitis B virus x protein function in hepatocarcinogenesis[J]. World J Gastroenterol, 2015, 21(38): 10732-10738. DOI:10.3748/wjg.v21.i38.10732 |
| [5] |
Novikova MV, Khromova NV, Kopnin PB. Components of the hepatocellular carcinoma microenvironment and their role in tumor progression[J]. Biochemistry (Mosc), 2017, 82(8): 861-873. DOI:10.1134/S0006297917080016 |
| [6] |
Yoo S, Wang W, Wang Q, et al. A pilot systematic genomic comparison of recurrence risks of hepatitis B virus-associated hepatocellular carcinoma with low- and high-degree liver fibrosis[J]. BMC Med, 2017, 15(1): 214. DOI:10.1186/s12916-017-0973-7 |
| [7] |
Wang G, Fang X, Han M, et al. MicroRNA-493-5p promotes apoptosis and suppresses proliferation and invasion in liver cancer cells by targeting vamp2[J]. Int J Mol Med, 2018, 41(3): 1740-1748. |
| [8] |
Lin H, Ewing LE, Koturbash I, et al. MicroRNAs as biomarkers for liver injury:Current knowledge, challenges and future prospects[J]. Food Chem Toxicol, 2017, 110: 229-239. DOI:10.1016/j.fct.2017.10.026 |
| [9] |
Singaravelu R, Quan C, Powdrill MH, et al. MicroRNA-7 mediates cross-talk between metabolic signaling pathways in the liver[J]. Sci Rep, 2018, 8(1): 361. |
| [10] |
Zhou SJ, Liu FY, Zhang AH, et al. MicroRNA-199b-5p attenuates TGF-beta1-induced epithelial-mesenchymal transition in hepatocellular carcinoma[J]. Br J Cancer, 2017, 117(2): 233-244. DOI:10.1038/bjc.2017.164 |
| [11] |
Hu D, Hu Y, Xu W, et al. Mir203 inhibits the expression of collagenrelated genes and the proliferation of hepatic stellate cells through a smad3 dependent mechanism[J]. Mol Med Rep, 2017, 16(2): 1248-1254. DOI:10.3892/mmr.2017.6702 |
| [12] |
Riazalhosseini B, Mohamed R, Apalasamy YD, et al. Circulating microRNA as a marker for predicting liver disease progression in patients with chronic hepatitis B[J]. Rev Soc Bras Med Trop, 2017, 50(2): 161-166. DOI:10.1590/0037-8682-0416-2016 |
| [13] |
Wang S, Wang JQ, Lv XW. Exosomal mirnas as biomarkers in the diagnosis of liver disease[J]. Biomark Med, 2017, 11(6): 491-501. DOI:10.2217/bmm-2017-0011 |
| [14] |
Zhang MW, Jin MJ, Yu YX, et al. Associations of lifestyle-related factors, hsa-mir-149 and hsa-mir-605 gene polymorphisms with gastrointestinal cancer risk[J]. Mol Carcinog, 2012, 51(Suppl 1): E21. |
| [15] |
Huang SP, Lévesque E, Guillemette C, et al. Genetic variants in microRNAs and microRNA target sites predict biochemical recurrence after radical prostatectomy in localized prostate cancer[J]. Int J Cancer, 2015, 135(11): 2661-2667. |
| [16] |
Yin Z, Li H, Cui Z, et al. Polymorphisms in pre-mirna genes and cooking oil fume exposure as well as their interaction on the risk of lung cancer in a chinese nonsmoking female population[J]. Onco Targets Ther, 2016, 9: 395. |
| [17] |
Li J, Tian F, Li D, et al. Mir-605 represses psmd10/gankyrin and inhibits intrahepatic cholangiocarcinoma cell progression-Febs Letters[J]. Febs Letters, 2014, 588(18): 3491-3500. DOI:10.1016/j.febslet.2014.08.008 |
| [18] |
Wang RY, Chen L, Chen HY, et al. Muc15 inhibits dimerization of EGFR and PI3K-AKT signaling and is associated with aggressive hepatocellular carcinomas in patients[J]. Gastroenterology, 2013, 145(6): 1436-1448. DOI:10.1053/j.gastro.2013.08.009 |
| [19] |
Lv X, Li J, Yang B. Clinical effects of miR-101 on prognosis of hepatocellular carcinoma and carcinogenic mechanism of anti-miR-101[J]. Oncol Rep, 2016, 36(4): 2184-2192. DOI:10.3892/or.2016.4980 |
| [20] |
Murakami Y, Kubo S, Tamori A, et al. Comprehensive analysis of transcriptome and metabolome analysis in intrahepatic cholangiocarcinoma and hepatocellular carcinoma[J]. Sci Rep, 2015, 5: 16294. DOI:10.1038/srep16294 |
| [21] |
Liu Y, Yang Z, Du F, et al. Molecular mechanisms of pathogenesis in hepatocellular carcinoma revealed by rnasequencing[J]. Mol Med Rep, 2017, 16(5): 6674-6682. DOI:10.3892/mmr.2017.7457 |
| [22] |
Morimoto A, Kannari M, Tsuchida Y, et al. An HNF4alpha-microRNA-194/192 signaling axis maintains hepatic cell function[J]. J Biol Chem, 2017, 292(25): 10574-10585. DOI:10.1074/jbc.M117.785592 |
| [23] |
Denisenko ON, O'Neill B, Ostrowski J, et al. Zik1, a transcriptional repressor that interacts with the heterogeneous nuclear ribonucleoprotein particle K protein[J]. J Biol Chem, 1996, 271(44): 27701-27706. DOI:10.1074/jbc.271.44.27701 |
| [24] |
Ota T, Suzuki Y, Nishikawa T, et al. Complete sequencing and characterization of 21, 243 full-length human cdnas[J]. Nat Genet, 2004, 36(1): 40-45. DOI:10.1038/ng1285 |
| [25] |
Liu P, Jiang W, Zhao J, et al. Integrated analysis of genome-wide gene expression and DNA methylation microarray of diffuse large B-cell lymphoma with tet mutations[J]. Mol Med Rep, 2017, 16(4): 3777-3782. DOI:10.3892/mmr.2017.7058 |
| [26] |
Oka D, Yamashita S, Tomioka T, et al. The presence of aberrant DNA methylation in noncancerous esophageal mucosae in association with smoking history:A target for risk diagnosis and prevention of esophageal cancers[J]. Cancer, 2009, 115(15): 3412-3426. DOI:10.1002/cncr.24394 |
| [27] |
Mihara M, Yoshida Y, Tsukamoto T, et al. Methylation of multiple genes in gastric glands with intestinal metaplasia[J]. Am J Pathol, 2006, 169(5): 1643-1651. DOI:10.2353/ajpath.2006.060552 |
 2020, Vol. 35
2020, Vol. 35


