文章信息
- 江淑珍, 连辉, 熊远芳, 罗开金, 苏小青, 陈世品
- JIANG Shuzhen, LIAN Hui, XIONG Yuanfang, LUO Kaijin, SU Xiaoqing, CHEN Shipin
- 锥栗β-淀粉酶基因家族的全基因组鉴定与分析
- Genome-wide identification and expression analysis of the β-amylase gene family in Castanea henryi
- 森林与环境学报,2021, 41(5): 545-553.
- Journal of Forest and Environment,2021, 41(5): 545-553.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2021.05.013
-
文章历史
- 收稿日期: 2021-06-07
- 修回日期: 2021-07-18
2. 福建农林大学国家林业和草原局兰花保护与利用重点实验室, 福建 福州 350002
2. Key Laboratory of National Forestry and Grassland Administration for Orchid Conservation and Utilization, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
淀粉是碳水化合物最重要的储存形式,能被一系列酶降解,释放初级化学能和有机物,用于植物生长发育及响应非生物胁迫[1]。研究发现,α-淀粉酶(α-amylase,AMY)、普鲁兰酶、β-葡萄糖苷酶和α-葡聚糖磷酸化酶在活组织,如拟南芥[(Arabidopsis thaliana (L.) Heynh.)]叶[2-3]的短暂淀粉分解中起着次要的作用,β-淀粉酶(β-amylase,BAM,EC3.2.1)才是主要的降解酶[4],其主要负责储藏型淀粉的水解和过渡型淀粉的降解,从而产生β-极限糊精和β-麦芽糖。
到目前为止,全基因组分析已经确定了拟南芥9个BAM基因,水稻(Oryza sativa L.)10个、玉米(Zea mays L.)13个、二穗短柄草[Brachypodium distachyon (Linnaeus) P. Beauvois]11个、高粱[Sorghum bicolor (L.) Moench]10个、粟[Setaria italica var. germanica(Mill.) Schred]10个[5]、香蕉(Musa nana Lour.)16个[4]、马铃薯(Solanum tuberosum L.)10个和枳(Citrus trifoliata L.)8个[6]等。大多数关于BAM家族在体内功能的研究都是在模式植物拟南芥中展开的。对陆地植物BAM基因保守内含子位点的分析发现,该家族包含两个亚家族。一个亚家族包含BAM1/3/9,另一个亚家族包含BAM2/4/8[7]。在拟南芥中,BAM1与质体局部的AMY3启动子中都包含脱落酸响应元件基序,由脱落酸转录诱导,共同响应渗透胁迫[8]。枳中的PtrBAM1通过调节可溶性糖水平抵御寒冷[9]。研究表明在许多物种中没有BAM4,如经济作物香蕉[10]。有研究[8]分析了每个拟南芥个体BAM对植物生长和叶片淀粉降解的影响,仅BAM3就足以完全降解叶片淀粉,而BAM1只能部分降解叶片淀粉。相反,BAM2、BAM5和BAM6没有检测到对淀粉降解或植物生长有影响。MATTHIAS et al[10]研究发现一种新的植物BAM基因,命名为BAM10,存在于大多数被子植物、苏铁(Cycas revoluta Thunb.)、银杏(Ginkgo biloba L.)和百岁兰(Welwitschia mirabilis Hook. f.)中,但不存在于十字花科(Brassicaceae)、蕨类(Fern)和松科(Pinaceae)植物中,其具体功能也还有待挖掘。
锥栗[Castanea henryi (Skan) Rehd. et Wils.]属壳斗科(Fagaceae)栗属(Castanea),是中国南方著名的木本粮食及果材兼用树种。锥栗果实淀粉含量高达47%~57%,其味道可口,风味独特,可以作为主食[11]。锥栗果实发育和种子萌发过程伴随着淀粉的合成与降解[12-13],BAM基因可能在其中起着重要调控作用,但其如何参与和影响淀粉代谢、控制果实发育和种子萌发目前尚不清楚。本研究从锥栗全基因组中鉴定出9个锥栗ChBAM基因。从基因蛋白理化性质、染色体位置、基因结构、保守基序、结构域比对及系统进化方面对ChBAM基因家族进行分析。此外,还通过ChBAM基因家族蛋白互作、启动子元件分析和表达分析,挖掘ChBAM基因家族的功能与进化特征。该结果为今后研究ChBAM基因家族的结构和功能,以及识别和表征其他物种的BAM基因家族提供了一定的参考,也为进一步研究锥栗果实发育和种子萌发的分子机制提供了理论基础。
1 材料与方法 1.1 ChBAM基因家族鉴定与理化性质分析从TAIR网站(https://www.arabidopsis.org/)上下载拟南芥BAM基因家族氨基酸序列,以拟南芥BAM蛋白序列作为索引在锥栗基因组中进行本地序列比对。将获得的候选序列提交到NCBI-CDD网站(https://www.ncbi.nlm.nih.gov/cdd)和HMMER网站(https://www.ebi.ac.uk/Tools/hmmer/search/hmmsearch),验证是否含有Glyco_hydro_14(PF01373)保守结构域。在ExPASy(https://web.expasy.org/compute_pi)里得到氨基酸数目、分子量、等电点和亲疏水性均值。利用在线工具WoLF PSORT(https://wolfpsort.hgc.jp/)对ChBAM基因家族成员编码蛋白进行亚细胞定位预测。
1.2 BAM蛋白序列比对BAM葡糖基水解酶结构域的序列比对由具有默认设置的ClustalX程序执行,并由jalview(http://www.jalview.org/)将序列进行设置、筛选与展示。
1.3 ChBAM家族染色体分布、共线性和系统进化分析基因在染色体上的定位信息来自基因组中GFF3数据,采用TBtools工具绘制示意图并进行基因共线性分析。Rice Genome Annotation Project(http://rice.plantbiology.msu.edu/)中下载水稻BAM蛋白序列,Banana Genome Hub(http://banana-genome-hub.southgreen.fr/)中下载香蕉BAM氨基酸序列。从Spud DB potato genomic resource(http://solanaceae.plantbiology.msu.edu/index.shtml)中下载马铃薯BAM氨基酸序列。从NCBI网站(https://www.ncbi.nlm.nih.gov/)下载枳BAM蛋白序列,使用ClustalX2.1的默认设置对所有序列进行对齐,并使用MEGA-7(http://www.megasoftware.net/mega-7)邻接(neighbour joining, NJ)法进行系统发育树构建。进行1 000次bootstrap抽样,其它参数为默认值。
1.4 ChBAM基因结构域与保守基序分析利用GSDS网站(http://gsds.cbi.pku.edu.cn)在线分析锥栗BAM基因结构。MEME在线工具(http://meme-suite.org/tools/meme)对ChBAM基因家族的保守基序进行识别,设置6~50个基序宽度和最多12个基序。采用TBtools软件进行基因结构和保守基序可视化分析。
1.5 BAM蛋白互作与启动子元件分析将BAM蛋白作为研究对象,选定模式植物拟南芥作为物种参数,在STRING(http://string-db.org/)网站进行蛋白互作网络分析。确定ChBAM家族基因起始密码子上游约1 500 bp的序列作为调控启动子区域数据库。使用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)分析ChBAM家族基因启动子顺式作用元件。利用TBtools软件进行热图制作。
1.6 ChBAM基因表达分析锥栗品种“油榛”种植在福建省建瓯市(29°45′N,106°22′E)。取3棵树龄为12~15 a,同一方位的新鲜、健康、无损坏的雌花、雄花、叶柄、壳斗、幼果、中果、成熟果样品立即冷冻在液氮中,保存于-80 ℃冰箱中。锥栗种子在福建农林大学国家林业和草原局兰花保护与利用重点实验室的温室里萌发。在播种后0、10、20和30 d(处理编号分别为S1、S2、S3和S4),剥除壳斗、果皮,取种仁部位,清洗后放入冻存管液氮速冻,将样品送至华大基因采用第2代测序技术,基于Illumina HiSeq测序平台,进行转录组测序,另外取种仁部位对其淀粉和可溶性糖含量进行测定,具体参照LIU et al[13]的方法。试验均进行3次重复。从转录组数据中获取ChBAM的表达丰度(fragments per kilobase per million,FPKM)值,通过log2(FPKM+1)公式计算表达差异程度,使用TBtools软件中HeatMap程序绘制基因表达量热图。
2 结果与分析 2.1 ChBAM基因家族的鉴定与理化性质分析在锥栗基因组中鉴定了9个非冗余的ChBAM基因。将锥栗BAM基因家族成员与模式植物拟南芥进行同源比对分析后,按照最匹配的结果对锥栗BAM基因家族成员进行命名(表 1),有研究表明,马铃薯StBAM4和香蕉MaBAM3a基因属于BAM10同源基因[10],而Che028807与StBAM4基因有较高序列同源性(图 3),所以将其命名为ChBAM10。ChBAM基因序列长度为301~691个氨基酸,预测的分子量和等电点分别介于33.23~77.53 kDa和5.25~9.12之间。根据ChBAM的亲疏水性指数(负值表示亲水性,正值表示疏水性),所有蛋白都是亲水性蛋白。ChBAM1/3/6/9基因预测出来的亚细胞定位与拟南芥不同[10]。
| 基因ID Gene ID |
基因名称 Gene name |
氨基酸数 Number of amino acids |
蛋白分子质量 Molecular weight /kDa |
理论等电点 Isoelectric point |
亲疏水性均值 Grand average of hydropathicity |
亚细胞定位 Subcellular location |
| Che008525 | ChBAM1 | 580 | 64.07 | 6.05 | -0.372 | 细胞外Extracellular |
| Che000225 | ChBAM2 | 547 | 61.80 | 5.33 | -0.345 | 叶绿体Chloroplast |
| Che002888 | ChBAM3 | 546 | 61.34 | 8.21 | -0.474 | 线粒体Mitochondrion |
| Che011937 | ChBAM4 | 398 | 45.36 | 8.80 | -0.251 | 叶绿体Chloroplast |
| Che015653 | ChBAM6 | 568 | 64.43 | 5.28 | -0.425 | 细胞质Cytoplasm |
| Che000224 | ChBAM7 | 691 | 77.53 | 5.35 | -0.435 | 细胞核Nucleus |
| Che019386 | ChBAM8 | 682 | 76.06 | 5.46 | -0.375 | 细胞核Nucleus |
| Che019166 | ChBAM9 | 301 | 33.23 | 5.25 | -0.523 | 细胞核Nucleus |
| Che028807 | ChBAM10 | 501 | 56.71 | 9.12 | -0.375 | 叶绿体Chloroplast |
基于已知的大豆β-淀粉酶GmBMY1的葡糖基水解酶结构[14-15],对9个锥栗BAM、1个马铃薯StBAM4和1个香蕉MaBAM3a保守葡糖基水解酶结构域进行了比对。图 1中相应标示出了大豆GmBMY1的21个底物结合残基(黑色箭头)、两个催化残基Glu-186和Glu-380(红色箭头)、外环和内环(红线)。ChBAM1、ChBAM3和ChBAM6的催化活性显示出所有已知参与催化的区域都具有高度保守的氨基酸基序,表明ChBAM1、ChBAM3和ChBAM6是具有活性的酶。Ⅱ组的ChBAM2,同样在这些区域显示出保守的基序,说明BAM2是一种活性酶。相反,Ⅱ组的ChBAM7和ChBAM8有许多氨基酸突变,外环、内环、催化残基Glu-380及其周围的氨基酸的保守性很差,推测这两个是催化无活性的。ChBAM4和ChBAM9在关键残基、外环以及内环中有大量突变,则Group Ⅳ的ChBAM可能也是无活性的蛋白质。ChBAM10、StBAM4和MaBAM3a同属于BAM10,3个同源BAM10的外环和内环的氨基酸发生较多突变,所以BAM10可能是无活性的酶。
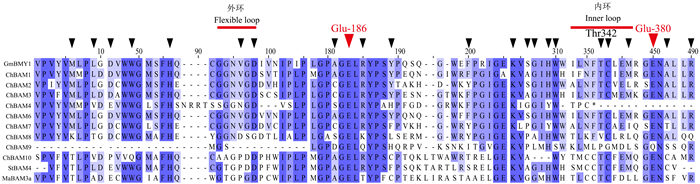
|
图 1 BAM基因家族的葡糖基水解酶结构域比对 Fig. 1 Alignment of the conserved glucosyl hydrolase domains from BAM gene family |
染色体位置信息如图 2所示。9个ChBAM基因成员不均匀分散到4条染色体上。通过对ChBAM基因家族进行物种内共线性分析,发现有1对基因发生串联复制,即ChBAM2和ChBAM7。
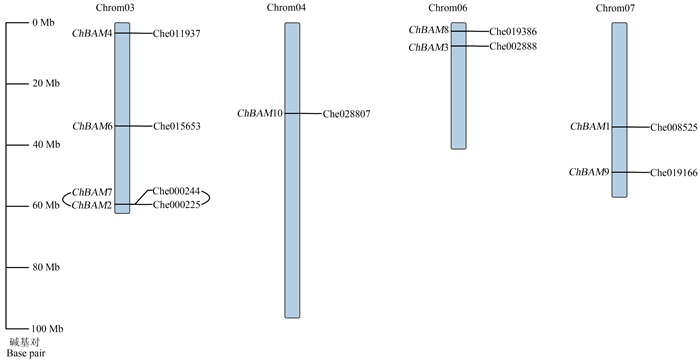
|
图 2 ChBAM基因在染色体上的分布 Fig. 2 Chromosome distribution of the ChBAM genes |
为了研究ChBAM与其他植物BAM之间的进化关系,结合9个拟南芥AtBAM蛋白序列[4]、10个水稻OsBAM蛋白序列、9个马铃薯StBAM蛋白序列、16个香蕉MaBAM蛋白序列和8个枳CtBAM蛋白序列[4],对锥栗9个ChBAM蛋白全长序列进行系统进化分析。锥栗9个ChBAM分为4个组(图 3)。Ⅰ组包含ChBAM1/3/10;Ⅱ组包含ChBAM2/7/8;Ⅲ组包含ChBAM6;Ⅳ组包含ChBAM4/9。总的来说,比起其他4个物种,锥栗ChBAM家族在进化关系上更接近枳CtBAM家族。
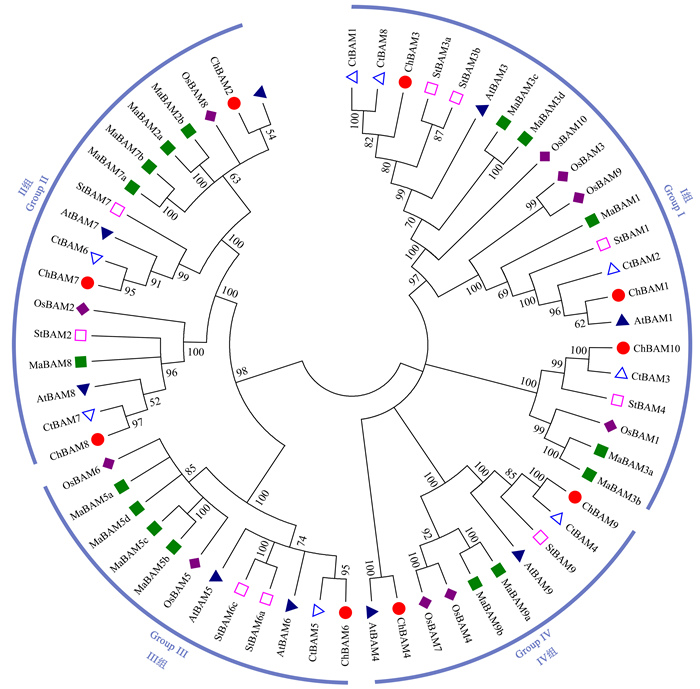
|
图 3 BAM基因家族邻接树 Fig. 3 NJ tree of BAM gene family |
基因结构和保守基序多样性是促进基因家族进化的一种机制。通过研究外显子-内含子的结构和保守基序,更深入地了解ChBAM家族的进化特征。ChBAM基因家族蛋白系统发育关系显示[图 4(a)],9个ChBAM家族蛋白分成4组,与图 3结果一致。ChBAM基因家族外显子有4~9个[图 4(b)]。在锥栗ChBAM基因家族中鉴定了12个保守基序[图 4(c)]。ChBAM蛋白有几个共同保守基序,表明这些基序是ChBAM蛋白序列的重要组成部分。只有Ⅱ组的ChBAM7和ChBAM8包含motif12。ChBAM4和ChBAM9相比,ChBAM4蛋白的序列C端缺少3个motifs,即motif 2/7/10,ChBAM9蛋白的序列N端缺少4个motifs,即motif 8/3/11/1。总体而言,同一组ChBAM成员相似的基因结构和保守的基序组成都有力地支持了系统进化分析中该家族分组的可靠性。
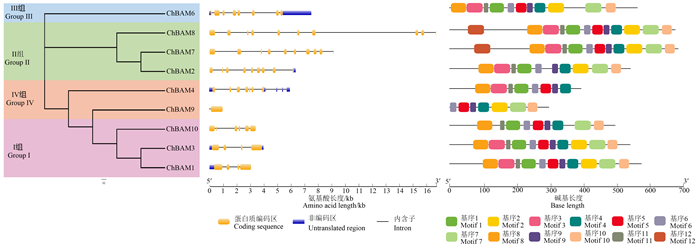
|
图 4 ChBAM基因家族的系统发育、基因结构和保守基序 Fig. 4 Phylogeny, gene structures and conserved motifs of ChBAM family genes |
采用STRING在线软件对ChBAM家族成员之间的蛋白互作进行预测(图 5)。相连接的蛋白具有紧密的功能联系,不同的线条颜色代表了关联的证据类型。ChBAM1和ChBAM2、ChBAM3之间关联紧密,且与其他淀粉水解酶蛋白α-淀粉酶(AMY)、4-α-葡聚糖转移酶(4-α-glucan transferase, DPE)、淀粉异构酶(starch isoamylase, ISA)和糖基水解酶家族31号蛋白(AT5G11270,EC: 3.2.1.20)相互作用。
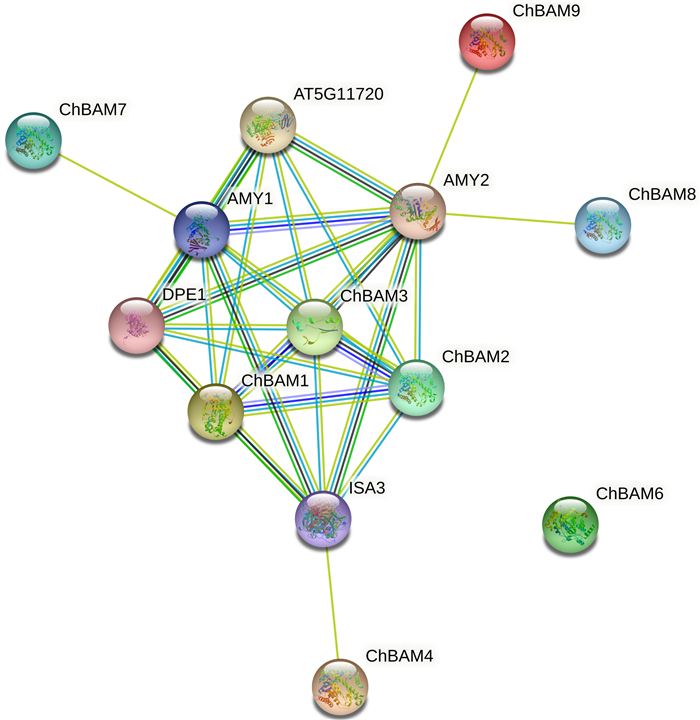
|
图 5 ChBAM基因家族蛋白互作网络 Fig. 5 Protein interaction network of ChBAM family genes |
启动子区域中包含的特异性顺式作用元件可以调节基因表达。分析了9个ChBAM基因上游大小为1.5 kb的DNA序列,并确定了潜在的顺式作用元件(图 6)。其中与激素反应相关的顺式作用元件有5种,包括脱落酸响应元件、生长素响应元件、茉莉酸甲酯响应元件、赤霉素响应元件以及水杨酸响应元件。茉莉酸甲酯响应元件最多,有30个,其次是脱落酸响应元件,有13个。ChBAM基因家族中,除了ChBAM4,其它8个ChBAM基因都含有茉莉酸甲酯响应元件,表明ChBAM家族基因启动子上的茉莉酸甲酯响应元件可能对锥栗生长发育起到调控作用。ChBAM基因家族胁迫相关元件共有6种28个,分别是干旱诱导响应元件、缺氧响应元件、低温响应元件、防御和应激响应元件、脱水响应元件和厌氧响应元件。总之,顺式作用元件分析表明,ChBAM家族基因与不同的非生物胁迫联系紧密。
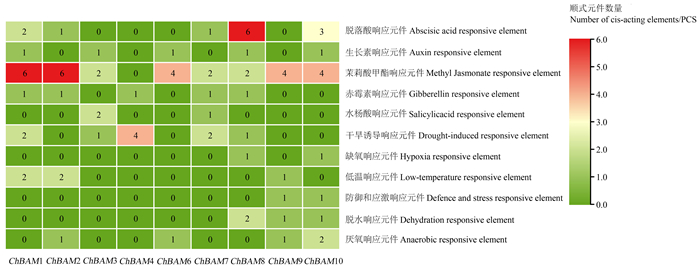
|
图 6 ChBAM基因家族启动子顺式作用元件的种类和数量 Fig. 6 Species and number of cis-acting elements of the promoter of ChBAM family genes |
锥栗种子萌发S1~S2时期,淀粉含量从50.66%升高至60.12%,可能是因为锥栗种子在萌发初期受到某些胁迫,为了响应胁迫,某些物质迅速转化为淀粉。这种现象在水稻[16]、麻风树种子[17]以及黄瓜种子[18]萌发初期也存在。在S2~S4时期,淀粉含量逐渐下降,淀粉被酶降解以满足种子萌发能量所需。在种子萌发的初始阶段,胚轴突破种皮向下生长延伸需要消耗大量的贮藏物质,可溶性糖为种子的生长发育提供主要营养最先被利用,所以其含量在S1~S2时期从20.87%急剧下降至11.30 %。在S2~S4时期缓慢下降(图 7)。
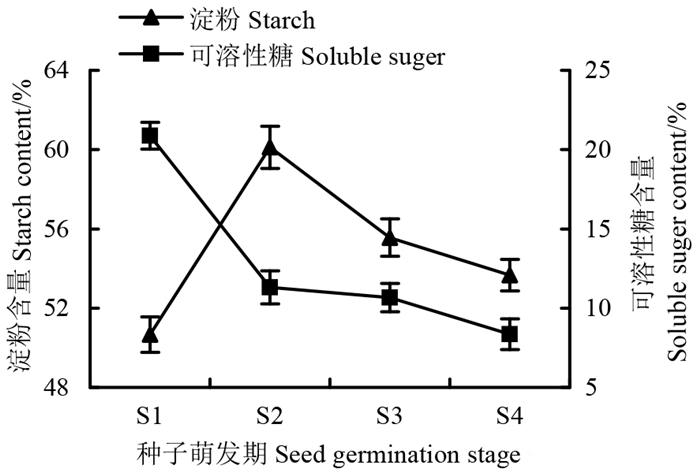
|
图 7 锥栗种子萌发期间淀粉含量和可溶性糖含量的变化 Fig. 7 Changes in the starch content and soluble sugar content in the seeds of Castanea henryi during germination |
对锥栗ChBAM家族基因的转录组数据进行表达分析,发现其在锥栗不同器官或不同的生物过程具有表达差异性(图 8)。ChBAM2/7/10无论是在各器官、果实发育期还是种子萌发期都表达痕量或不表达。ChBAM3在叶柄部位和种子萌发S1时期表达量较高。果实发育期间,ChBAM1表达量先下调后上调,ChBAM6表达量上调,ChBAM4/9表达量下调。种子萌发期间,ChBAM4/6/9基因表达量上调,ChBAM1/3基因表达量下调,其中ChBAM1基因表达量在S2时期下调,可能是S2时期锥栗种子淀粉急剧增加从而抑制了淀粉降解酶ChBAM1的表达。

|
注:高表达水平为红色,低表达水平为蓝色。 Note: high expression levels are shown in red, and low expression levels are shown in blue. 图 8 ChBAM基因的表达模式 Fig. 8 Expression patterns of ChBAM genes |
本研究通过与拟南芥、水稻、马铃薯、香蕉和枳的BAM基因家族进行系统发育分析,将9个ChBAM基因分为4个组,这与其他植物BAM基因家族分组结果一致。锥栗ChBAM氨基酸序列中催化残基的保存程度与拟南芥相似,其中第2个催化残基(Glu-186)即使在催化无活性的ChBAM蛋白中也很保守,说明Glu-186催化残基不仅有催化功能,可能还具备其他功能。Ⅳ组的ChBAM2和ChBAM7发生复制,而基因复制事件在植物基因家族的扩增和进化中起着重要作用。ChBAM家族基因结构和基序特征与拟南芥、水稻[5]、马铃薯[6]、香蕉[4]等物种的同源BAM蛋白相似,说明BAM基因家族在结构上高度保守。蛋白互作网络表明,ChBAM蛋白与α-淀粉酶(AMY)、4-α-葡聚糖转移酶(DPE)和淀粉异构酶(ISA)等联系紧密,暗示其与这些淀粉降解酶相互作用共同调控淀粉代谢。茉莉酸甲酯作为一种化学信号,影响植物新陈代谢,参与种子萌发、开花开颖和病虫抗逆性形成等植物生理过程[19]。ChBAM基因家族启动子激素相关元件中茉莉酸甲酯响应元件最多,在8个ChBAM基因的启动子区都存在,香蕉MaBAM基因家族茉莉酸甲酯元件数量也最多[4]。推测BAM家族基因可能参与了由茉莉酸甲酯响应元件调控的多种生长发育过程。
拟南芥AtBAM4已被证明参与淀粉降解过程[20],ChBAM4在锥栗种子萌发S1~S4时期表达量不断上调,推测其可能在锥栗种子淀粉降解过程中起作用。人们对BAM6的功能研究甚少,有研究初步认定BAM6位于质体中,可能是质体转运肽,但BAM6质体定位的概率比其他质体BAMs更低[21-22]。本文通过亚细胞定位预测ChBAM6位于细胞质中(表 1),葡糖基水解酶结构域比对显示它具有催化活性。MONROE et al[23]研究推断AtBAM6在生长3周的拟南芥中不发挥作用,而在拟南芥生长至6周才发挥作用。转录组表达分析发现ChBAM6在锥栗果实发育的幼果期几乎不表达,但在中果期和成熟果期表达明显。因此,推断BAM6可能在较成熟的植物中起重要作用。
锥栗ChBAM基因家族中只有ChBAM7和ChBAM8蛋白含有motif12,即BZR1/BES1型结构域。有研究表明BZR1-BAM7/8蛋白在拟南芥中充当转录因子,控制幼苗的生长和发育[24]。ChBAM7和ChBAM8基因在锥栗种子萌发和果实发育中痕量表达,它们可能和拟南芥AtBAM7/8功能相同,主要作为转录因子提供一个代谢信号来影响植物生长的速度或模式,而不直接参与催化淀粉降解。ChBAM1/9无论是在锥栗各组织器官、果实生长和种子萌发过程表达量都较高,马铃薯低温糖化即淀粉降解过程中StBAM1/9也有较高的表达[6],拟南芥AtBAM9不与淀粉结合,而是与其他分子结合调控淀粉代谢,且试验证明AtBAM9作用于BAM1和BAM3,调控这两个基因的活性。马铃薯StBAM9被证明与StBAM1互作于淀粉粒[6]。因此,锥栗ChBAM9可能与ChBAM1结合,进而调控淀粉代谢。在番茄中,同源BAM10广泛表达于淀粉合成组织中[25],然而ChBAM10在锥栗果实发育即淀粉合成过程几乎不表达,表明BAM10在不同植物上的功能具有差异性。
总之,目前除了对模式植物拟南芥BAM基因家族有深入研究外,其他植物的研究较浅, 尤其是木本植物。本研究基于锥栗全基因组测序数据,鉴定筛选了锥栗ChBAM基因家族成员,并深入分析其结构与功能,为进一步研究ChBAM基因家族在锥栗生长发育过程中的作用提供理论基础。
| [1] |
ZEEMAN S C, DELATTE T, MESSERLI G, et al. Starch breakdown: recent discoveries suggest distinct pathways and novel mechanisms[J]. Functional Plant Biology, 2007, 34(6): 465-473. DOI:10.1071/FP06313 |
| [2] |
LLOYD J R, KOSSMANN J, RITTE G. Leaf starch degradation comes out of the shadows[J]. Trends in Plant Science, 2005, 10(3): 130-137. DOI:10.1016/j.tplants.2005.01.001 |
| [3] |
YU T S, ZEEMAN S C, THORNEYCROFT D, et al. α-amylase is not required for breakdown of transitory starch in Arabidopsis leaves[J]. Journal of Biological Chemistry, 2005, 280(11): 9773-9779. DOI:10.1074/jbc.M413638200 |
| [4] |
MIAO H X, SUN P G, MIAO Y L, et al. Genome-wide identification and expression analysis of the β-amylase genes strongly associated with fruit development, ripening, and abiotic stress response in two banana cultivars[J]. Frontiers of Agricultural Science and Engineering, 2016, 3(4): 346-356. DOI:10.15302/J-FASE-2016127 |
| [5] |
MASON-GAMER R J. The β-amylase genes of grasses and a phylogenetic analysis of the Triticeae (Poaceae)[J]. American Journal of Botany, 2005, 92(6): 1045-1058. DOI:10.3732/ajb.92.6.1045 |
| [6] |
侯娟. 马铃薯低温糖化相关淀粉酶基因的功能鉴定及机制解析[D]. 武汉: 华中农业大学, 2017.
|
| [7] |
MONROE J D, BREAULT J S, POPE L E, et al. Arabidopsis β-amylase 2 is a K+-requiring, catalytic tetramer with sigmoidal kinetics[J]. Plant Physiology, 2017, 175(4): 1525-1535. DOI:10.1104/pp.17.01506 |
| [8] |
THALMANN M T, PAZMINO D, SEUNG D, et al. Regulation of leaf starch degradation by abscisic acid is important for osmotic stress tolerance in plants[J]. The Plant Cell, 2016, 28(8): 1860-1878. DOI:10.1105/tpc.16.00143 |
| [9] |
PENG T, ZHU X F, DUAN N, et al. PtrBAM1, a β-amylase-coding gene of Poncirus trifoliata, is a CBF regulon member with function in cold tolerance by modulating soluble sugar levels[J]. Plant, Cell & Environment, 2014, 37(12): 2754-2767. |
| [10] |
THALMANN M, COIRO M, MEIER T, et al. The evolution of functional complexity within the β-amylase gene family in land plants[J]. BMC Evolutionary Biology, 2019, 19(1): 66. DOI:10.1186/s12862-019-1395-2 |
| [11] |
郑诚乐, 吴少华, 佘文琴, 等. 锥栗果实营养成分分析与品质的模糊评判[J]. 福建林学院学报, 2003, 23(4): 293-296. |
| [12] |
范晓明, 袁德义, 段经华, 等. 锥栗种仁发育期叶片与果实矿质元素含量的变化[J]. 园艺学报, 2014, 41(1): 44-52. DOI:10.3969/j.issn.0513-353X.2014.01.006 |
| [13] |
LIU B, LIN R Q, JIANG Y T, et al. Transcriptome analysis and identification of genes associated with starch metabolism in Castanea henryi seed (Fagaceae)[J]. International Journal of Molecular Sciences, 2020, 21(4): 1431. DOI:10.3390/ijms21041431 |
| [14] |
MIKAMI B, HEHRE E J, SATO M, et al. The 2.0-A resolution structure of soybean beta-amylase complexed with alpha cyclodextrin[J]. Biochemistry, 1993, 32(27): 6836-6845. DOI:10.1021/bi00078a006 |
| [15] |
MIKAMI B, DEGANO M, HEHRE E J, et al. Crystal structures of soybean β-amylase reacted with β-maltose and maltal: active site components and their apparent roles in catalysis[J]. Biochemistry, 1994, 33(25): 7779-7787. DOI:10.1021/bi00191a005 |
| [16] |
DE GUZMAN M K, PARWEEN S, BUTARDO V M, et al. Investigating glycemic potential of rice by unraveling compositional variations in mature grain and starch mobilization patterns during seed germination[J]. Scientific Reports, 2017, 7(1): 5854. DOI:10.1038/s41598-017-06026-0 |
| [17] |
ALENCAR N L M, GADELHA C G, GALLÃO M I, et al. Ultrastructural and biochemical changes induced by salt stress in Jatropha curcas seeds during germination and seedling development[J]. Functional Plant Biology, 2015, 42(9): 865-874. DOI:10.1071/FP15019 |
| [18] |
BU R F, XIAO X M, LIAO W B, et al. Exogenous Si alleviation of autotoxicity in cucumber (Cucumis sativus L.) seed germination is correlated with changes in carbohydrate metabolism[J]. Journal of Plant Growth Regulation, 2018, 37(3): 784-793. DOI:10.1007/s00344-017-9773-8 |
| [19] |
张知晓, 泽桑梓, 户连荣, 等. 茉莉酸甲酯生物活性研究进展[J]. 河南农业科学, 2018, 47(11): 1-7. |
| [20] |
LI J, FRANCISCO P, ZHOU W X, et al. Catalytically-inactive β-amylase BAM4 required for starch breakdown in Arabidopsis leaves is a starch-binding-protein[J]. Archives of Biochemistry and Biophysics, 2009, 489(1/2): 92-98. |
| [21] |
EMANUELSSON O, NIELSEN H, BRUNAK S, et al. Predicting subcellular localization of proteins based on their N-terminal amino acid sequence[J]. Journal of Molecular Biology, 2000, 300(4): 1005-1016. DOI:10.1006/jmbi.2000.3903 |
| [22] |
SCHEIN A I, KISSINGER J C, UNGAR L H. Chloroplast transit peptide prediction: a peek inside the black box[J]. Nucleic Acids Research, 2001, 29(16): e82. DOI:10.1093/nar/29.16.e82 |
| [23] |
MONROE J D, STORM A R, BADLEY E M, et al. β-amylase1 and β-amylase 3 are plastidic starch hydrolases in Arabidopsis that seem to be adapted for different thermal, pH, and stress conditions[J]. Plant Physiology, 2014, 166(4): 1748-1763. DOI:10.1104/pp.114.246421 |
| [24] |
REINHOLD H, SOYK S, ŠIMKOVÁ K, et al. β-amylase-like proteins function as transcription factors in Arabidopsis, controlling shoot growth and development[J]. The Plant Cell, 2011, 23(4): 1391-1403. DOI:10.1105/tpc.110.081950 |
| [25] |
The Tomato Genome Consortium. The tomato genome sequence provides insights into fleshy fruit evolution[J]. Nature, 2012, 485(7400): 635-641. DOI:10.1038/nature11119 |

 2021, Vol. 41
2021, Vol. 41


