文章信息
- 王利美, 邓敏捷, 张艳芳, 程智远, 李熙尧, 范国强
- WANG Limei, DENG Minjie, ZHANG Yanfang, CHENG Zhiyuan, LI Xiyao, FAN Guoqiang
- 泡桐丛枝病相关microRNAs测序及其靶基因预测
- Identification of the microRNAs and their target genes related to Paulownia witches' broom phytoplasma
- 森林与环境学报,2021, 41(2): 180-187.
- Journal of Forest and Environment,2021, 41(2): 180-187.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2021.02.009
-
文章历史
- 收稿日期: 2020-12-07
- 修回日期: 2021-02-10
泡桐(Paulownia spp.)是泡桐科落叶乔木,原产于中国,具有速生、耐旱、易繁殖等优良特性,不但能够防风固沙,而且可制作乐器和航空器材,是重要的经济林木,且具有较高的生态价值[1]。然而,由泡桐丛枝植原体(Paulownia witches′ broom phytoplasma)侵染引起的泡桐丛枝病(Paulownia witches′ broom,PaWB)可导致幼树死亡,成年树生长缓慢、蓄材量严重减少,已成为影响泡桐栽培和生产的制约因素,限制了泡桐产业的健康可持续发展[2]。前期研究发现,白花泡桐(P. fortunei)、毛泡桐(P. tomentosa)和豫杂一号(P. fortunei×P. tomentosa)的丛枝病苗经适宜浓度的甲基磺酸甲酯(methyl methanesulfonate,MMS)试剂处理后,可使其恢复至健康的生长状态,同时在试剂处理后的植物体内也检测不到植原体的存在[3]。
microRNAs(miRNA)以转录后调控的方式参与植物响应植原体的侵染。比如,部分miRNAs在植原体感染墨西哥来檬(Citrus aurantifolia L.)、枣树(Ziziphus jujuba)和鲁桑(Morus multicaulis Perr.)前后,表达量发生显著变化[4-6]。有关泡桐感染泡桐丛枝植原体过程中miRNAs的作用也有报道,FAN et al[7]通过转录组、miRNAs和降解组测序对毛泡桐健康苗、丛枝病苗及其经处理恢复健康形态的丛枝病苗进行分析,鉴定得到62个保守miRNAs和32个新miRNAs,其中24个miRNAs与泡桐丛枝病发生相关,参与代谢通路、次生代谢物生物合成和植物病原互作等代谢途径。CAO et al[8]对3种丛枝病泡桐苗和恢复健康状态的泡桐苗miRNAs进行研究, 发现76个miRNAs及其靶基因通过影响光合作用、植物防御反应及植物能量相关途径参与泡桐丛枝病发生。研究还发现miR156、miR169和miR172等家族在泡桐丛枝病发生过程中发挥重要作用[7-9];在毛果杨中过表达miR156f后,植株出现丛枝症状[9]。为了阐明泡桐丛枝病发生相关的miRNAs及其靶基因,以白花泡桐、毛泡桐和豫杂一号泡桐健康苗、丛枝病苗及MMS处理的丛枝病苗为试材,分别构建了小RNAs文库和降解组文库,经高通量测序与分析,筛选泡桐丛枝病发生特异相关的miRNAs及其靶基因,为今后揭示泡桐丛枝病发生机理和培育抗病品种奠定基础。
1 材料与方法 1.1 材料与处理研究所用的白花泡桐、毛泡桐和豫杂一号泡桐取自河南农业大学泡桐生物技术实验室。9个样品分别为:白花泡桐健康苗(PF)、丛枝病苗(PFI)和MMS(浓度为60 mg·L-1)处理的丛枝病苗(PFI60);毛泡桐健康苗(PT)、丛枝病苗(PTI)和MMS(浓度为60 mg·L-1)处理的丛枝病苗(PTI60);豫杂一号泡桐健康苗(PTF)、丛枝病苗(PTFI)和MMS(浓度为30 mg·L-1)处理的丛枝病苗(PTFI30)。样品培养条件和MMS处理方法参见文献[7]。
1.2 小RNAs文库的构建与测序采用Trizol试剂分别提取9个样品的总RNA,小RNAs文库构建和测序方法参见文献[8]。其中,PF、PFI、PT、PTI、PTF和PTFI测序结果参见文献[8],测序策略为单末端50 bp(测序工作由深圳华大基因科技服务有限公司完成)。
1.3 miRNAs鉴定测序得到的原始数据经过滤后得到有效序列,有效序列经比对和软件预测分别得到泡桐9个文库的保守miRNAs和新miRNAs。具体方法参见文献[8]。
1.4 丛枝病相关miRNAs鉴定将miRNAs测序序列标准化,在此基础上,以miRNAs在样品间差异倍数为|log1.2(处理组/对照组)|≥1,且卡方(2×2)检验中阈值P<0.05为标准,寻找样品间显著差异表达miRNAs。首先,在白花泡桐组(PFI vs. PF和PFI60 vs. PFI)、毛泡桐组(PTFI vs. PTF和PTI60 vs. PTI)和豫杂一号泡桐组(PTI vs. PT和PTFI30 vs. PTFI)分别筛选组内共同显著差异表达miRNAs,再取3种泡桐共有的显著差异表达miRNAs, 即为与泡桐丛枝病发生密切相关的miRNAs。
1.5 miRNAs靶基因鉴定及其功能注释首先,对miRNAs的靶基因进行预测,再通过降解组测序对预测到的靶基因进行验证。预测的靶基因和降解组序列文件(mRNAs)通过结合运算取交集,筛选出miRNAs的靶基因。功能注释参见文献[7-8]。
1.6 miRNAs-靶基因互作网络图构建为筛选得到与靶基因编码的蛋白存在相互作用的蛋白,使用在线数据库STRING 11.0(https: //string-db.org/)对靶基因分析后,利用Cytoscape 3.5.1软件(https: //cytoscape.org/)构建与泡桐丛枝病发生相关miRNAs及其靶基因的蛋白互作网络图。
1.7 差异表达miRNAs及其靶基因的qRT-PCR验证随机挑选两个miRNAs和两个靶基因进行qRT-PCR验证高通量测序结果的准确度。选用U6 snRNA和18 S rRNA分别作为miRNAs和靶基因的内参。具体方法参见文献[8]。
2 结果与分析 2.1 小RNA文库构建与分类通过Solexa/Illumina测序,从9个小RNAs文库中共得到160 576 064条原始序列,剔除低质量及接头等序列,得到有效序列80 866 126条;对数据进行统计,获得31 209 953条唯一有效序列,占唯一原始序列总数的81.38%(表 1)。对有效序列进行长度统计可知,其长度分布在18~25 nt,约85%的序列介于20~24 nt之间,24 nt所占比例最高,其次为21 nt(图 1),这与泡桐前期研究结果类似[8]。
| 样品 Sample |
原始序列数量 Raw read counts |
有效序列数量 Valid read counts |
唯一原始序列数量 Unique raw read counts |
唯一有效序列数量 Unique valid read counts |
| PF | 14 755 106 | 11 378 626 | 4 647 738 | 4 308 335 |
| PFI | 15 263 318 | 10 311 261 | 4 312 727 | 3 929 696 |
| PFI60 | 14 591 856 | 10 505 310 | 4 325 134 | 3 974 673 |
| PT | 23 033 611 | 7 283 265 | 3 688 854 | 2 641 512 |
| PTI | 17 608 489 | 8 660 319 | 4 694 782 | 3 539 947 |
| PTI60 | 17 931 331 | 9 473 428 | 4 735 159 | 3 851 088 |
| PTF | 23 314 360 | 10 376 326 | 4 600 250 | 3 669 368 |
| PTFI | 16 562 154 | 6 791 333 | 3 882 880 | 2 825 081 |
| PTFI30 | 17 515 839 | 6 086 258 | 3 463 707 | 2 470 253 |
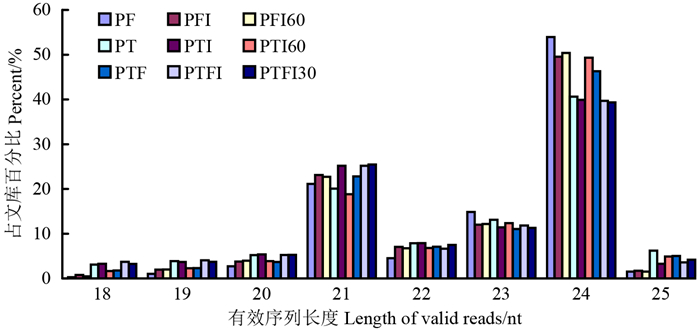
|
图 1 有效序列长度分布 Fig. 1 Length distribution of valid reads |
利用生物信息学软件、白花泡桐基因组和数据库miRBase 21.0进行泡桐miRNAs的鉴定,从3种泡桐苗中共获得546个miRNAs,包括332个保守miRNAs和214个新miRNAs。其中,白花泡桐组中鉴定得到419个miRNAs(259个保守miRNAs,160个新miRNAs);毛泡桐组中鉴定得到408个miRNAs(289个保守miRNAs,119个新miRNAs);豫杂一号泡桐组中鉴定得到407个miRNAs(292个保守miRNAs,115个新miRNAs)。泡桐文库中保守miRNAs的数量212~280个;新miRNAs数量67~127个(表 2)。
| 样品 Sample |
保守miRNAs数量 Conserved miRNA counts |
总保守miRNAs数量 Total conserved miRNA counts |
新miRNAs数量 Novel miRNA counts |
总新 miRNAs数量 Total novel miRNA counts |
差异保守 miRNAs数量 Different conserved miRNA counts |
差异新 miRNAs数量 Different novel miRNA counts |
共有差异 miRNAs数量 Common different miRNA counts |
| PF | 241 | 259 | 127 | 160 | 89 | 37 | |
| PFI | 240 | 114 | |||||
| PFI60 | 212 | 119 | |||||
| PT | 274 | 289 | 67 | 119 | 119 | 21 | 11 |
| PTI | 267 | 87 | |||||
| PTI60 | 280 | 104 | |||||
| PTF | 280 | 292 | 107 | 115 | 139 | 24 | |
| PTFI | 275 | 68 | |||||
| PTFI30 | 272 | 69 |
对鉴定到的miRNAs进行差异表达分析显示,在PFI60 vs. PFI、PTI60 vs. PTI和PTFI30 vs. PTFI比对组中分别获得182(保守130;新52)、185(保守138;新47)和217(保守178;新39)个显著差异表达的miRNAs。根据比对组方案,结合CAO et al[8]之前的研究,白花泡桐组共有显著差异表达的保守miRNAs 89个和新miRNAs 37个;毛泡桐组共有显著差异表达的保守miRNAs 119个和新miRNAs 21个;豫杂一号泡桐组共有显著差异表达的保守miRNAs 139个和新miRNAs 24个(表 2)。3个泡桐种共有显著差异表达miRNAs 11个(表 2),其中9个为保守miRNAs,2个为新miRNAs(表 3)。结果表明,不同种类泡桐在响应泡桐丛枝植原体侵染时,由不同的miRNAs参与,且不同的miRNAs在泡桐丛枝病发生过程中可能发挥着不同的调控作用;3个种共有的11个显著差异表达miRNAs可能在泡桐丛枝病发生过程中起重要作用。
| miRNA名称 miRNA name |
靶基因Target gene | |
| 编号ID | 注释Annotation | |
| miR399e | PAU004923.1 | 泛素连接酶E3 KEG E3 ubiquitin-protein ligase KEG(KEG) |
| miR169j | PAU021305.2 | Rab3 GTP激活蛋白催化亚基Rab3 GTPase-activating protein catalytic subunit(Rab3-GAP) |
| miR391 | PAU006287.1 | NADPH依赖型羰基还原酶1 Carbonyl reductase [NADPH] 1 (CBR1) |
| miR4414a-5p | PAU001964.1 | 环核苷酸门控的离子通透性通道2 Cyclic nucleotide gated channel 2(CNGC2) |
| PAU020236.1 | F-box蛋白家族F-box family protein | |
| miR6424 | PAU001306.1 | R3h结构域蛋白R3h domain-containing protein 2(R3HDM2) |
| PAU012878.1 | SQUAMOSA启动子结合蛋白16 SQUAMOSA promoter binding protein-like 16(SPL16) | |
| miR156d | PAU002457.1 | SQUAMOSA启动子结合蛋白6 SQUAMOSA promoter binding protein-like 6(SPL6) |
| PAU023019.1 | 镁螯合酶亚基ChlI Magnesium-chelatase subunit ChlI(CHLI) | |
| PAU017804.1 | 假定蛋白Hypothetical protein | |
| mir20-5p | PAU017667.1 | 质体运动受损2 Plastid movement impaired 2(PMI2) |
| PAU012968.1 | CBL互作蛋白激酶9 CBL-interacting serine/threonine-protein kinase 9(CIPK9) | |
| miR319d-5p | ||
| miR5368j-3p | ||
| miR5368k-3p | ||
| mir48-5p | ||
通过软件预测,白花泡桐、毛泡桐和豫杂一号泡桐组分别得到1 801、1 975和1 944个靶基因。对与泡桐丛枝病发生相关的11个miRNAs进行靶基因预测,7个miRNAs预测到34个靶基因。
降解组测序共得到179 553 407条原始序列,通过数据处理获得有效序列177 811 491(99.03%)条,用于生成降解组序列文件。与预测结果进行整合后发现,白花泡桐235个miRNAs鉴定到1 385个靶基因;毛泡桐238个miRNAs鉴定到1 498个靶基因;豫杂一号泡桐215个miRNAs鉴定到1 463个靶基因。通过降解组测序,泡桐丛枝病发生相关的7个miRNAs(miR169j、miR399e、miR4414a-5p、miR6424、miR391、miR156d和mir20-5p)共鉴定到29个靶基因,其中,miR169j、miR399e、miR4414a-5p和miR391均只有1个靶基因,miR6424有2个靶基因,mir20-5p有3个靶基因。MiR156d鉴定到的靶基因多达20个,且多数编码SPL家族转录因子,与已报道的其他物种miR156家族的靶基因同源[10],其另一靶基因PAU024966.1,编码富亮氨酸重复类受体激酶TDR。
为研究miRNAs的靶基因在泡桐丛枝病发生过程中可能发挥的功能,对鉴定得到的29个靶基因编码的蛋白进行基因本体论功能注释和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)代谢通路分析。结果发现,这些miRNAs的靶基因主要参与细胞、细胞组分、分子结合、蛋白催化活性、代谢过程和细胞过程等(表 4),有的靶基因还参与信号转导和应激反应等生物学过程,表明这些miRNAs参与的生物学过程可能在泡桐响应泡桐丛枝植原体过程中起重要作用。KEGG分析发现这些靶基因共参与15条代谢通路,包括植物的信号转导、病原互作和激素代谢等。
| 本体论分类 Gene ontology classification |
本体论条目 Gene ontology term |
靶基因数目 Number |
靶基因百分比 Percent/% |
| 细胞组分Cellular component | 细胞组成Cell composition | 15 | 60 |
| 细胞Cell | 15 | 60 | |
| 细胞器Organelle | 14 | 56 | |
| 膜Membrane | 2 | 8 | |
| 蛋白复合体Protein-containing complex | 1 | 4 | |
| 转运活性Transporter activity | 2 | 8 | |
| 催化活性Catalytic activity | 4 | 16 | |
| 分子功能Molecular function | 分子调节功能Molecular function regulator | 2 | 8 |
| 结合Binding | 23 | 92 | |
| 细胞过程Cellular process | 3 | 12 | |
| 代谢过程Metabolic process | 4 | 16 | |
| 定位Localization | 2 | 8 | |
| 生物进程Biological process | 生物调节Biological regulation | 1 | 4 |
| 生物调节过程Regulation of biological process | 1 | 4 | |
| 信号通路Signaling pathway | 1 | 4 | |
| 应激反应Response to stimulus | 1 | 4 |
为了进一步探究与泡桐丛枝病发生相关的miRNAs及其靶基因间是否存在相互作用,对miRNAs及其靶基因蛋白进行互作关系的预测,并使用Cytoscape进行互作网络图的构建(图 2)。由图 2可知,miR6424-PAU001306.1(R3HDM2)与miR169j-PAU021305.2(Rab3-GAP)通过DMP2、PAB7B和GDI等参与光合作用;miR169j-PAU021305.2(Rab3-GAP)与miR4414a-5p-PAU001964.1(CNGC2)通过与REP、RGTA1/2和ERA1等互作参与植物防御抗病过程;miR399e-PAU004923.1(KEG)、mir20-5p-PAU012968.1(CIPK9)以及miR156d-PAU012878.1(SPL16)通过与AT2G20050、CBL1/2/3/9/SOS3/SNF4和AKRP互作参与防御反应;miR156d-PAU002457.1以及miR4414a-5p-PAU001964.1的靶基因通过与R蛋白(SNC1/K9E15.1)、TPR蛋白(T21F11.18)互作参与植物免疫反应。综上所述,与泡桐丛枝病发生相关的miRNAs相互协作,在泡桐丛枝病发生过程中影响植物光合作用和植物病原互作等多个生物学过程。
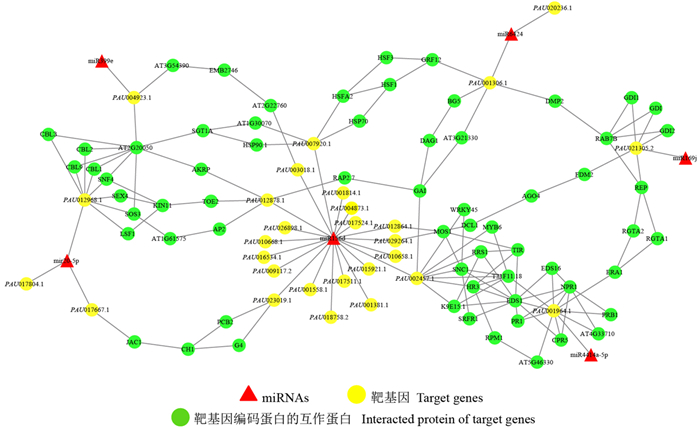
|
图 2 miRNAs-靶基因互作网络 Fig. 2 Interaction network of miRNA target genes associated with PaWB |
为验证泡桐miRNAs响应丛枝病的动态表达模式和其与靶基因的相关性,在11个miRNAs中选取两个miRNAs及其靶基因进行qRT-PCR定量分析(图 3)。定量分析表明,miRNAs表达量变化趋势和测序结果基本相同,miR156d与其靶基因PAU024966.1(TDR)在3种泡桐中均为负调控关系,miR391与其靶基因PAU006287.1在白花泡桐和豫杂一号泡桐中表现为负调控关系,说明测序结果真实可靠。
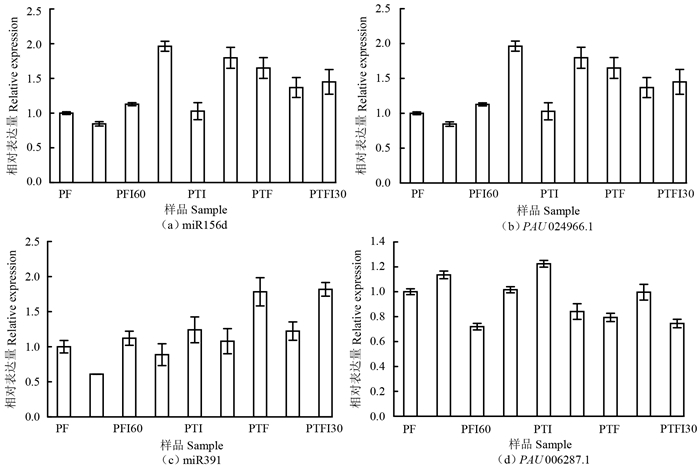
|
图 3 泡桐差异表达miRNAs及其靶基因qRT-PCR验证 Fig. 3 The qRT-PCR validation of differentily expressed miRNAs and their target genes in Paulownia |
目前,植原体病害已成为影响粮食作物和蔬菜产量及林木生产的严重病害,也已成为植物病理学和分子生物学亟待解决的重点和难点问题[11]。学者从转录组、蛋白组和代谢组等角度对泡桐丛枝病进行了研究[12-14],但对于泡桐丛枝病发生的分子调控机制研究尚未揭示,因此, 利用小分子RNA测序和降解组测序技术在3种泡桐健康苗、丛枝病苗和处理恢复健康状态的丛枝病苗中鉴定得到了多个泡桐丛枝病发生相关的miRNAs及其靶基因,蛋白互作网络分析发现这些miRNAs及其靶基因在泡桐丛枝病发生过程中协同作用,参与多个生物学过程和代谢通路。这些结果丰富了对泡桐丛枝病发生中miRNAs功能的认识。
前期研究发现,miR156和miR169家族成员参与泡桐丛枝病发生过程,本研究也发现这两个家族成员在泡桐感染泡桐丛枝植原体后表达水平显著变化,其变化趋势与前人研究报道的枣树[5]、桑树[6]感染植原体以及水稻[15]感染病原菌时的结果一致。此外,miR4414在胁迫响应中的作用也值得关注,如,在辣椒(Capsicum annuum L.)中miR4414b-3p受辣椒疫霉孢子诱导后表达量显著上调[16]; 地黄(Rehmannia glutinosa L.)在连作条件下miR4414过量表达[17]。地黄miR4414家族靶向负调控钙离子浓度的钙离子逆向运输交换器基因[16-17],本研究中miR4414a-5p靶向一个钙离子通道蛋白相关基因,即环核苷酸门控的离子通透性通道CNGC2(PAU001964.1), 这表明泡桐丛枝植原体侵染泡桐可能引起钙离子和CNGC共同参与的免疫反应[18]。
miR399存在于植物韧皮部,在调节磷酸盐稳态的长距离信号中发挥重要作用[19],同时有相关报道显示它也参与植物免疫反应。在稻瘟病菌感染水稻的研究中发现,miR399是水稻免疫反应的负调节因子,过表达miR399f导致水稻对稻瘟病菌敏感性明显增强[20]。ZHANG et al[21]发现miR399可能参与梨树对苹果茎痘病毒的免疫反应,梨树感染病毒后其表达量显著升高。本研究中,3种泡桐感染泡桐丛枝植原体后,miR399e表达量显著升高,与上述梨树中的研究结果一致,而且,植原体和miR399e均存在于植物韧皮部,据此,推测该miRNAs可能在韧皮部作为泡桐响应泡桐丛枝植原体胁迫的信号分子,参与泡桐丛枝病发生过程。泡桐miR399e靶向泛素连接酶E3 KEG(PAU004923.1)。有研究发现,KEG泛素化蛋白激酶CIPK26及其下游转录因子,进而泛素化产物通过26 s蛋白酶体途径被降解[22]。此外,本研究中泡桐mir20-5p靶向蛋白激酶CIPK9(PAU012968.1)。在根结线虫感染水稻根系的研究中发现,CIPK9基因表达量发生变化,其可能参与植物免疫反应[23],先前研究报道CIPK26与CIPK9同源,在拟南芥中可归为同一个分支[24]。因此,推测泡桐丛枝病发生中,miR399e和mir20-5p可能通过调控CIPK家族成员,参与泡桐丛枝病发生中的免疫反应过程。
在白花泡桐、毛泡桐和豫杂一号泡桐病健苗及MMS处理的丛枝病泡桐苗中鉴定到了与泡桐丛枝病发生相关的miRNAs及其靶基因。发现miR156、miR4414、miR169和miR391等保守miRNAs家族成员响应泡桐对丛枝植原体的侵染,这些结果与之前的研究相似[7-9],同时,还发现miR399e和泡桐新的miRNAs可能共同参与泡桐丛枝病形成的过程,这些miRNAs可能通过调控其靶基因的表达,进而影响植物体内生物学过程和相关代谢通路的变化,参与泡桐丛枝病发生的过程。后续将开展与泡桐丛枝病相关miRNAs的深入研究,进一步了解miRNAs在泡桐丛枝病发生中的具体功能。
| [1] |
蒋建平. 泡桐栽培学[M]. 北京: 中国林业出版社, 1990.
|
| [2] |
CAO X B, FAN G Q, ZHAO Z L, et al. Morphological changes of Paulownia seedlings infected phytoplasmas reveal the genes associated with witches' broom through AFLP and MSAP[J]. PLoS One, 2014, 9(11): e112533. DOI:10.1371/journal.pone.0112533 |
| [3] |
曹喜兵. 甲基磺酸甲酯对丛枝病泡桐基因表达影响研究[D]. 郑州: 河南农业大学, 2014.
|
| [4] |
EHYA F, MONAVARFESHANI A, FARD E M, et al. Phytoplasma-responsive microRNAs modulate hormonal, nutritional, and stress signalling pathways in Mexican lime trees[J]. PLoS One, 2013, 8(6): e66372. DOI:10.1371/journal.pone.0066372 |
| [5] |
SHAO F J, ZHANG Q, LIU H W, et al. Genome-wide identification and analysis of microRNAs involved in witches'-broom phytoplasma response in Ziziphus jujuba[J]. PLoS One, 2016, 11(11): e0166099. DOI:10.1371/journal.pone.0166099 |
| [6] |
GAI Y P, ZHAO H N, ZHAO Y N, et al. MiRNA-seq-based profiles of miRNAs in mulberry phloem sap provide insight into the pathogenic mechanisms of mulberry yellow dwarf disease[J]. Scientific Reports, 2018, 8(1): 812. DOI:10.1038/s41598-018-19210-7 |
| [7] |
FAN G Q, CAO X B, NIU S Y, et al. Transcriptome, microRNA, and degradome analyses of the gene expression of Paulownia with phytoplamsa[J]. BMC Genomics, 2015, 16(1): 896. DOI:10.1186/s12864-015-2074-3 |
| [8] |
CAO X B, ZHAI X Q, ZHANG Y F, et al. Comparative analysis of microRNA expression in three Paulownia species with phytoplasma infection[J]. Forests, 2018, 9(6): 302-318. DOI:10.3390/f9060302 |
| [9] |
王哲. 泡桐丛枝病发生相关ceRNA研究[D]. 郑州: 河南农业大学, 2020.
|
| [10] |
ZHENG C F, YE M X, SANG M M, et al. A regulatory network for miR156-SPL module in Arabidopsis thaliana[J]. International Journal of Molecular Sciences, 2019, 20(24): 6166. DOI:10.3390/ijms20246166 |
| [11] |
ERMACORA P, OSLER R. Symptoms of phytoplasma diseases[J]. Methods in Molecular Biology, 2019, 1875: 53-67. |
| [12] |
LIU R N, DONG Y B, FAN G Q, et al. Discovery of genes related to witches broom disease in Paulownia tomentosa×Paulownia fortunei by a De Novo assembled transcriptome[J]. PLoS One, 2013, 8(11): e80238. DOI:10.1371/journal.pone.0080238 |
| [13] |
CAO Y B, FAN G Q, WANG Z, et al. Phytoplasma-induced changes in the acetylome and succinylome of Paulownia tomentosa provide evidence for involvement of acetylated proteins in witches' broom disease[J]. Molecular & Cellular Proteomics, 2019, 18(6): 1210-1226. |
| [14] |
CAO Y B, ZHAI X Q, DENG M J, et al. Relationship between metabolites variation and Paulownia witches' broom[J]. Scientia Silvae Sinicae, 2017, 53(6): 85-93. |
| [15] |
LI Y, ZHAO S L, LI J L, et al. Osa-miR169 negatively regulates rice immunity against the blast fungus Magnaporthe oryzae[J]. Frontiers in Plant Science, 2017, 8: 2. |
| [16] |
孟纯阳, 魏小春, 赵艳艳, 等. 12个辣椒microRNA的靶标预测及表达分析[J]. 分子植物育种, 2018, 16(12): 3820-3829. |
| [17] |
曹晓风, 杨艳会, 冯法节, 等. 地黄植株体内miRNAs与其连作障碍关系的研究进展[J]. 中国中药杂志, 2017, 42(6): 1104-1108. |
| [18] |
MA W. Roles of Ca2+ and cyclic nucleotide gated channel in plant innate immunity[J]. Plant Science, 2011, 181(4): 342-346. DOI:10.1016/j.plantsci.2011.06.002 |
| [19] |
PANT B D, BUHTZ A, KEHR J, et al. MicroRNA399 is a long-distance signal for the regulation of plant phosphate homeostasis[J]. The Plant Journal, 2008, 53(5): 731-738. DOI:10.1111/j.1365-313X.2007.03363.x |
| [20] |
CAMPOS-SORIANO L, BUNDÓ M, BACH-PAGES M, et al. Phosphate excess increases susceptibility to pathogen infection in rice[J]. Molecular Plant Pathology, 2020, 21(4): 555-570. DOI:10.1111/mpp.12916 |
| [21] |
ZHANG Q L, ZHANG Y, WANG S N, et al. Characterization of genome-wide microRNAs and their roles in development and biotic stress in pear[J]. Planta, 2019, 249(3): 693-707. DOI:10.1007/s00425-018-3027-2 |
| [22] |
LYZENGA W J, SULLIVAN V, LIU H X, et al. The kinase activity of calcineurin B-like interacting protein kinase 26(CIPK26) influences its own stability and that of the ABA-regulated ubiquitin ligase, keep on going (KEG)[J]. Frontiers in Plant Science, 2017, 8: 502. |
| [23] |
ZHOU Y, ZHAO D, SHUANG L, et al. Transcriptome analysis of rice roots in response to root-knot nematode infection[J]. International Journal of Molecular Sciences, 2020, 21(3): 848. DOI:10.3390/ijms21030848 |
| [24] |
TANG R J, ZHAO F G, GARCIA V J, et al. Tonoplast CBL-CIPK calcium signaling network regulates magnesium homeostasis in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(10): 3134-3139. DOI:10.1073/pnas.1420944112 |

 2021, Vol. 41
2021, Vol. 41


