文章信息
- 孙华乐, 翟晓巧, 曹喜兵, 邓敏捷, 赵振利, 范国强
- SUN Huale, ZHAI Xiaoqiao, CAO Xibing, DENG Minjie, ZHAO Zhenli, FAN Guoqiang
- 白花泡桐二四倍体的蛋白质组差异分析
- Proteomic analysis of the diploid and autotetraploid of Paulownia fortunei
- 森林与环境学报,2020, 40(6): 619-626.
- Journal of Forest and Environment,2020, 40(6): 619-626.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2020.06.008
-
文章历史
- 收稿日期: 2020-08-03
- 修回日期: 2020-09-23
2. 河南省林业科学研究院, 河南 郑州 450008
2. Forestry Academy of Henan, Zhengzhou, Henan 450008, China
通过基因组加倍、多倍化在物种形成及植物进化中都起着十分重要的作用。多倍体通常表现出诸多优良性状,如生物量增加、抗逆性增强、对病虫害抗性增强等,这些优良性状在植物育种中具有重要意义[1]。白花泡桐[Paulownia fortunei (Seem.) Hemsl.]为泡桐科泡桐属植物,原产于中国,是重要的庭院绿化、速生用材以及防护树种,具有重要的生态意义和经济价值,在世界范围内广泛种植[2]。范国强等[3]用二倍体白花泡桐叶片,利用秋水仙素成功诱导出其同源四倍体,扩大了泡桐的种质资源。与二倍体相比,白花泡桐四倍体具有更优良的光合特性和更强的抗逆性[4-5]。然而,目前四倍体优良性状在蛋白质水平的分子机制尚不清楚,有关白花泡桐二四倍体间的蛋白质组变化仍有待探讨。
文中利用同位素标记相对定量和绝对定量(isobaric tags for relative and absolute quantitation, iTRAQ)技术对白花泡桐二四倍体的蛋白质组进行定量研究,分析两者之间蛋白质表达水平的差异,并对差异表达蛋白质进行功能注释,结合转录组分析筛选与四倍体形成特异蛋白质,为进一步了解四倍体泡桐优良特性提供参考。
1 材料与方法 1.1 试验材料与处理试验材料来源于河南农业大学泡桐研究所培养的二倍体及其同源四倍体白花泡桐,培养温度为(25±2)℃,光照强度为130 μmol·m-2·s-1,光照时间为16 h·d-1。分别剪取上述两种培养30 d,长约1.5 cm,长势一致的组培苗顶芽,用液氮冷冻后迅速置于-80℃冰箱内。样品标记为:二倍体白花泡桐(B2、B2_1);四倍体白花泡桐(B4、B4_1)。每个样品各两个生物学重复。
1.2 蛋白质提取、iTRAQ标记与预分离使用丙酮沉淀法提取蛋白质,分别从B2和B4两种样品中提取100 μg蛋白质并用iTRAQ试剂标记获得肽段,根据QIAO et al[6]使用的方法将蛋白质消化并用iTRAQ技术标记,将标记后的各组肽段依次进行混合、分离、除盐和抽干。具体操作步骤详见文献[7]。
1.3 肽段分离与液相串联质谱分析将抽干得到的组分用缓冲液A(5%乙腈,0.1%甲酸)重悬, 用液相色谱仪(型号LC-20 AD,日本京都岛津)和分离柱对样品进行分离, 分离后传输至AB SCIEX Triple TOF 5600质谱系统(美国),动态参数设置详见文献[8]。
1.4 蛋白质鉴定与定量分析使用Mascot 2.3.02软件进行蛋白质的鉴定,数据库来源于白花泡桐转录组数据,相关测序数据已上传到美国国家生物技术信息中心(National Center of Biotechnology Information, NCBI)的序列片段归档数据库(https://www.ncbi.nlm.nih.gov/sra, SRA),登录号为SRP032166[9]。对鉴定到的蛋白质在基因本体论(gene ontology,GO)、京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)数据库中进行功能注释。蛋白质的差异标准为差异倍数>1.2或<0.83,且P<0.05。
1.5 实时荧光定量聚合酶链式反应验证采用植物总RNA提取试剂盒提取B2和B4中的总RNA。随机挑选8个差异表达的蛋白质,用Taq SYBR Green qPCR Premix试剂进行实时荧光定量聚合酶链式反应(quantitative real-time-polymerase chain reaction,qRT-PCR),在美国伯乐公司的CFX96TM Real-TimeSystem荧光定量聚合酶链式反应检测系统上完成。参照DONG et al[10]方法进行PCR扩增,每个样品3次重复。使用18 S rRNA作为内参,数据统计采用2-△△Ct法。引物序列见表 1。
| 蛋白质编号 Protein ID |
正义引物 Forward primer |
反义引物 Reverse primer |
| m.13192 | TTGATAAGCCGTCTGATG | CAACCAAGGATAACCAAGT |
| m.29938 | TGTTACTATTCCTCCATTGG | AACGGCTACTTAGACCTT |
| m.24953 | GCTAGGCTCTACATCATA | CTGCTATTTACACTCTAAGG |
| m.40032 | TGGAATCTTAGGCACTGG | CAATATGGAGCTTGTCAACA |
| m.32969 | GCTGTCGTTCTCATCCAT | TTCTCTAAGGAGCGGTATTC |
| m.39316 | GCCCAACCAAGAATCTAA | TTCCACTAATCCACCTGT |
| m.9824 | TGAGGAATTGTGAAGTGG | TGGTGGAGAGGTATATGTAT |
| m.16063 | GAGGAATTTATGACAAGAGG | CATCAATCCAAGTGACAC |
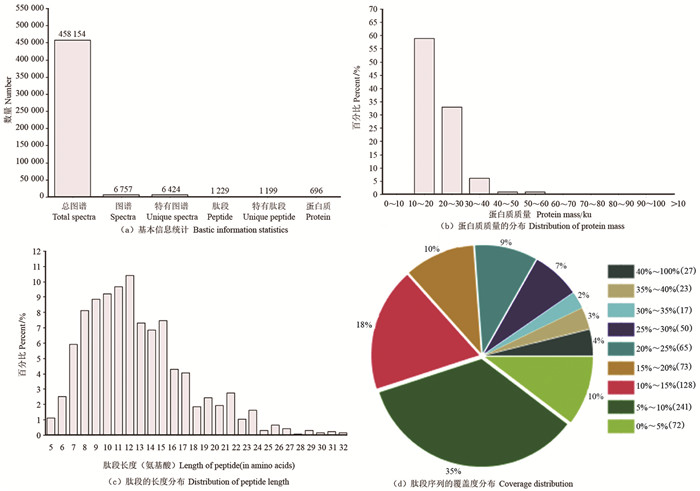
为了解白花泡桐二四倍体蛋白质组的整体信息,对提取的蛋白质进行质量检测。两个样品蛋白质含量符合试验要求(表 2)。共鉴定到458 154个图谱,Mascot搜库后,匹配到6 757个图谱,6 424个特有图谱,肽段和特有肽段分别为1 229个和1 199个,最终鉴定到696个蛋白质[(图 1(a)],这些蛋白质的相对分子质量主要集中在10~60 ku,其中占较大比例的范围为10~20 ku[(图 1(b)],肽段长度主要集中在7~15个氨基酸[(图 1(c)],肽段序列覆盖度在5%~10%的蛋白质占比最多[(图 1(d)]。
| 样品 Sample |
浓度 Concentration /(μg·μL-1) |
体积 Volume /μL |
总蛋白质 Total protein /μg |
| B2 | 1.77 | 180 | 318.6 |
| B2_1 | 2.38 | 250 | 595.0 |
| B4 | 1.83 | 180 | 329.4 |
| B4_1 | 3.67 | 250 | 917.5 |
|
注:图 1(d)中括号内的数字代表肽段序列覆盖在每个区间中蛋白质的数量。 Note:the numbers in brackets in figure 1 (d) represent the number of proteins in each interval covered by the peptide sequence. 图 1 总蛋白质的鉴定与分析 Fig. 1 Identification and analysis of total proteins |
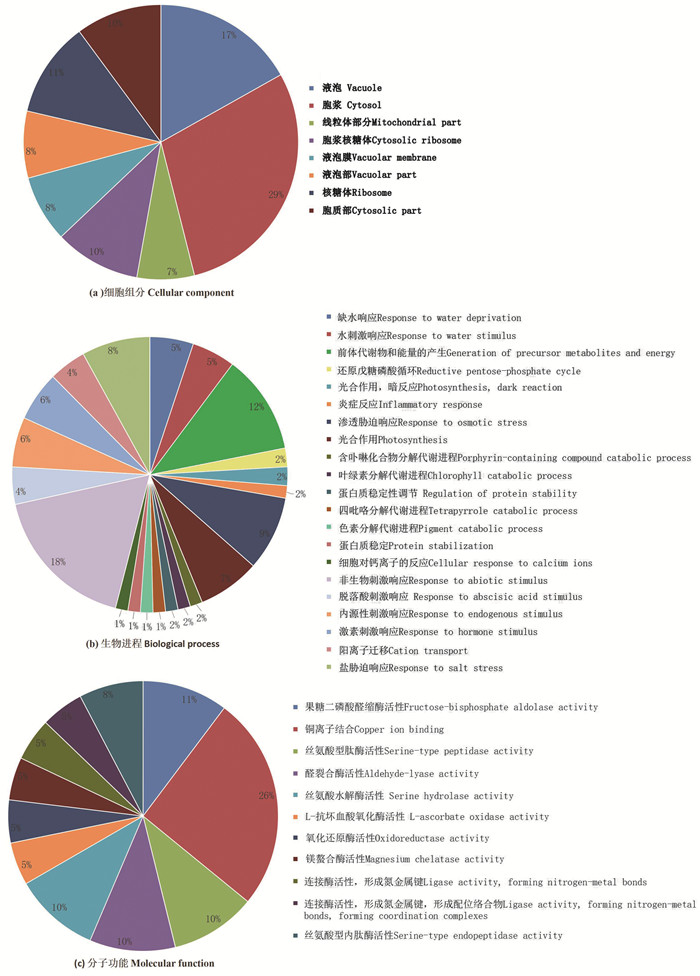
为进一步了解四倍体优良特性相关的特异蛋白质,对B2和B4两样本鉴定到的总蛋白质进行差异分析。结果共鉴定到75个差异表达的蛋白质,其中,39个上调,36个下调。GO富集分析结果显示,显著性富集的GO条目有40个:细胞组分主要条目有胞浆、液泡、核糖体[图 2(a)];生物进程主要条目有非生物刺激响应、前体代谢物和能量的产生、渗透胁迫响应[图 2(b)];分子功能主要条目有铜离子结合、果糖二磷酸醛缩酶活性、醛裂合酶活性[图 2(c)]。KEGG通路富集分析参与了35条代谢通路,主要包括代谢途径(29,48.33%)、糖酵解/糖异生(7,11.67%)、次生代谢产物的生物合成(17,28.33%)、光合生物的碳固定(8,13.33%)和氧化磷酸化(5,8.33%)等(表 3)。
|
图 2 差异表达蛋白质的基因本体论功能注释分类 Fig. 2 Gene ontology analysis of differentially expressed proteins |
| 代谢通路 Metabolic pathway |
具有通路注释的不同 蛋白质Different proteins withpathway annotation |
P值 P value |
通路名称 Pathway ID |
| 果糖和甘露糖代谢Fructose and mannose metabolism | 6(10.00%) | 0.001 966 | ko00051 |
| 戊糖磷酸途径Pentose phosphate pathway | 6(10.00%) | 0.003 123 | ko00030 |
| 光合生物的碳固定Carbon fixation in photosynthetic organisms | 8(13.33%) | 0.031 283 | ko00710 |
| 氧化磷酸化Oxidative phosphorylation | 5(8.33%) | 0.040 431 | ko00190 |
| 糖酵解/糖异生Glycolysis/gluconeogenesis | 7(11.67%) | 0.052 817 | ko00010 |
| 蛋白酶体Proteasome | 4(6.67%) | 0.090 594 | ko03050 |
| 抗坏血酸和醛酸代谢Ascorbate and aldarate metabolism | 3(5.00%) | 0.132 132 | ko00053 |
| 苯丙氨酸代谢Phenylalanine metabolism | 2(3.33%) | 0.152 701 | ko00360 |
| 代谢途径Metabolic pathways | 29(48.33%) | 0.156 166 | ko01100 |
| 核糖体Ribosome | 5(8.33%) | 0.254 616 | ko03010 |
| 次生代谢产物的生物合成Biosynthesis of secondarymetabolites | 17(28.33%) | 0.271 649 | ko01110 |
| 乙醛酸和二元酸代谢Glyoxylate and dicarboxylate metabolism | 4(6.67%) | 0.316 829 | ko00630 |
| 核苷酸切除修复Nucleotide excision repair | 1(1.67%) | 0.316 865 | ko03420 |
| 植物病原相互作用Plant-pathogen interaction | 2(3.33%) | 0.338 296 | ko04626 |
| 类胡萝卜素生物合成Carotenoid biosynthesis | 1(1.67%) | 0.398 677 | ko00906 |
| 蛋白输出Protein export | 1 (1.67%) | 0.398 677 | ko03060 |
| 光合作用-天线蛋白Photosynthesis -antenna proteins | 1(1.67%) | 0.398 677 | ko00196 |
| 内质网蛋白质加工Protein processing in endoplasmic reticulum | 4(6.67%) | 0.461 496 | ko04141 |
| 维生素B6代谢Vitamin B6 metabolism | 1(1.67%) | 0.470 836 | ko00750 |
| 酪氨酸代谢Tyrosine metabolism | 1(1.67%) | 0.470 836 | ko00350 |
| 淀粉和蔗糖代谢Starch and sucrose metabolism | 3(5.00%) | 0.530 786 | ko00500 |
| 其他聚糖降解Other glycan degradation | 1(1.67%) | 0.534 463 | ko00511 |
| 卟啉与叶绿素代谢Porphyrin and chlorophyll metabolism | 2(3.33%) | 0.551 062 | ko00860 |
| 苯丙酸生物合成Phenylpropanoid biosynthesis | 2(3.33%) | 0.551 062 | ko00940 |
| 甘氨酸、丝氨酸和苏氨酸代谢Glycine, serine and threonine metabolism | 2(3.33%) | 0.551 062 | ko00260 |
| RNA降解RNA degradation | 1(1.67%) | 0.590 552 | ko03018 |
| 精氨酸和脯氨酸代谢Arginine and proline metabolism | 1(1.67%) | 0.639 982 | ko00330 |
| 谷胱甘肽代谢Glutathione metabolism | 1(1.67%) | 0.683 533 | ko00480 |
| 氨基糖和核苷酸糖代谢Amino sugar and nucleotide sugarmetabolism | 2(3.33%) | 0.684 478 | ko00520 |
| 光合作用Photosynthesis | 1(1.67%) | 0.721 892 | ko00195 |
| 吞噬体Phagosome | 1(1.67%) | 0.721 892 | ko04145 |
| 过氧化物酶体Peroxisome | 1(1.67%) | 0.755 671 | ko04146 |
| 丙氨酸、天冬氨酸和谷氨酸代谢Alanine, aspartate and glutamatemetabolism | 1(1.67%) | 0.785 406 | ko00250 |
| 氰胺酸代谢Cyanoamino acid metabolism | 1(1.67%) | 0.811 576 | ko00460 |
| 剪接体Spliceosome | 1(1.67%) | 0.942 296 | ko03040 |
| 注:表中括号内的数字表示每个通路中蛋白质占所有通路的百分比。Note:the numbers in brackets in the table indicate the percentage of proteins in each pathway for all pathways. | |||
为了筛选与倍性相关的差异蛋白质,把上述得到的蛋白质数据与白花泡桐转录组数据进行关联分析。结果显示,总蛋白质关联到18 984个基因,其中75个差异蛋白质关联到1 158个基因, 在75个差异蛋白质中,有65个对应的基因在转录水平上变化不明显;有37个不差异的蛋白质,但其对应的基因在转录水平差异表达。在关联到的9个差异蛋白质中,有7个蛋白质变化趋势与其对应基因的变化趋势一致,这7个蛋白质中有4个注释到KEGG数据库中,分别是镁螯合酶H亚基(m.55631)、V型H+转运ATPase亚基A(m.33869)、细胞色素C氧化酶亚基5b(m.12004)和果糖二磷酸醛缩酶(m.24953);有两个差异表达蛋白质丰度变化趋势和其对应基因的变化趋势相反,蛋白质丰度减少,但其对应的基因均上调,这两个蛋白质只有玉米黄素环氧化酶(m.32969)注释到KEGG数据库中。
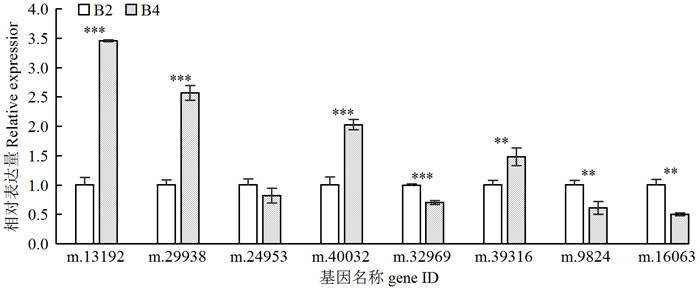
2.4 差异表达蛋白质的qRT-PCR验证为了验证测序结果的可靠性,在B2和B4文库中随机挑选m.13192(谷胱甘肽S-转移酶)、m.29938(L-抗坏血酸氧化酶)、m.24953(果糖二磷酸醛缩酶)、m.40032(铜氧化酶)、m.32969(玉米黄质环氧酶)、m.39316(蛋白二硫键异构酶A1)、m.9824(2-磷酸乙醇酸磷酸酶1)、m.16063(羧亚甲基丁烯酸酶)等8个差异蛋白质进行qRT-PCR验证(图 3)。B2表达量归一化为1,18 S rRNA作为内参。结果显示:4个基因在蛋白质水平和转录水平均上调,4个基因在蛋白质水平上调而在转录水平下调,这种结果可能是由于基因转录、蛋白质翻译后加工和修饰,或者mRNA和蛋白质的降解等因素造成的。
|
注:**表示B2和B4之间差异达0.01显著水平;***表示B2和B4之间差异达0.001显著水平。 Note: ** represents significant differences between B2 and B4(P < 0.01), and *** represents significant differences between B2 and B4(P < 0.001). 图 3 白花泡桐二四倍体差异表达蛋白质qRT-PCR表达验证 Fig. 3 mRNA expression of DEPs between B2 and B4 validated using qRT-PCR |
植物在受到生物胁迫(如植原体、病毒、真菌等)和非生物胁迫(如高低温、干旱、盐渍等)时启动信号转导通路,进而产生抗性[11]。相对于同源二倍体,多倍体植物的抗逆性更强,比如冷调节的稳定性[12]和光保护[13],多倍化有助于竞争能力的进化[14]。多倍化植物在形态和生理上有一定程度的变化,如器官增大、抗逆性增强等,另外,植物内部代谢物的产量、营养成分、内源激素以及次生代谢物质等的变化也对抗逆性有所增强。文中利用iTRAQ技术对二倍体及其同源四倍体白花泡桐的蛋白质组进行了研究,从蛋白质层面上了解二四倍体植物之间生理生化指标的差异。前人研究结果表明,泡桐同源四倍体中与胁迫响应相关的基因相对于二倍体显著上调[15-18]。此外,泡桐同源四倍体响应干旱胁迫和盐胁迫的microRNA对应的靶基因与二倍体亲本相比上调,增加了其对环境的适应性[19-21]。
与二倍体亲本相比,白花泡桐四倍体参与应对生物胁迫和非生物胁迫的蛋白质差异表达。例如,在泡桐发育和胁迫耐受性中起重要作用的果糖二磷酸醛缩酶(m.24953)和L-抗坏血酸氧化酶(m.29938)。结果表明,对同源四倍体蛋白质丰度的调节可能导致了其对环境适应性的增强。因此,与二倍体亲本相比,胁迫相关蛋白质和光合作用相关蛋白质在四倍体白花泡桐中的丰度均较高,为四倍体泡桐生长抗逆性强和生长速度快的优良特性提供了解释。
在植物中抗坏血酸氧化酶(ascorbate oxidase,AAO)属于多铜氧化酶家族,定位于植物细胞壁[22]。AAO具有重要的意义,与植物生长发育、逆境和衰老等有关[23]。AAO在四倍体白花泡桐中表达量明显高于二倍体,对同源四倍体蛋白质丰度的调节可能导致了其对环境适应性的增强。
镁螯合酶主要由H、I和D三个亚基组成[24-25],通过催化原卟啉IX和Mg2+螯合形成镁原卟啉IX,它是叶绿素合成过程中的关键酶。其中,镁螯合酶H亚基为该复合体主体,在反应中起到关键性作用,其它亚基, 如I和D亚基可能通过H亚基参与调控[26],其表达均能影响叶绿素合成[27]。通过这3种主要亚基的协调以及配合,并在ATP驱动下,Mg2+与原卟啉IX螯合,最后推动叶绿素合成。镁螯合酶H亚基在四倍体泡桐中表达量明显高于二倍体,这说明四倍体在叶绿素合成、抑制黄化等方面明显增强。
从文中的蛋白质组学和转录组学数据得出了不同的丰度/表达结果, 这可能是因为转录后调控或翻译后修饰未将基因表达差异转化为蛋白质丰度差异,或者是因为蛋白质组学水平低估了同源四倍体与二倍体亲本之间的差异,基于iTRAQ的分析,确定了差异表达蛋白质的数量估计不同样品的倍数变化。据报道,基于iTRAQ的方法可能存在准确性低的问题,因为它们可能会系统地低估比率,尤其是当比率变化较大时[28]。蛋白质丰度和mRNA表达水平之间一致性较缺乏, 主要是由以下几个因素造成:转录调控和翻译后修饰可以使蛋白质丰度和mRNA表达水平间有差异的存在, 例如,调节其靶基因表达的small RNA(包括micro RNA)在许多生物学过程中(如抗病抗旱等)发挥重要作用[29]; 不同试验技术的局限性也可能导致此问题; 由于基因表达有一定时空特异性,因此, 不同生长阶段、生长部位的组织可能会导致试验结果不同。对整个组织的蛋白质组学进行研究可能会得出更全面的信息,而对特定细胞器的研究可能会更准确地显示差异。总之,尽管相关性不是很高,但转录组和蛋白质组数据的各自及关联分析对于深刻了解多倍体至关重要。
本研究利用iTRAQ及液相串联质谱技术,对白花泡桐二倍体及其同源四倍体的蛋白质组进行定量,寻找与四倍体形成相关的蛋白质和基因。在研究中获得了二倍体和四倍体白花泡桐蛋白质图谱,鉴定到了与抗逆和光合作用等相关的差异表达蛋白质,预测铜氧化酶、L-抗坏血酸氧化酶、镁螯合酶与白花泡桐多倍化相关,指出了二倍体及其同源四倍体在蛋白质水平上的差异,为培育新的泡桐品种和扩大现有种质资源提供理论依据。
| [1] |
COMAI L. The advantages and disadvantages of being polyploid[J]. Nature Reviews Genetics, 2005, 6(11): 836-846. DOI:10.1038/nrg1711 |
| [2] |
KRIKORIAN A D. Paulownia in China:cultivation and utilization[J]. Economic Botany, 1988, 42(2): 283. DOI:10.1007/BF02858931 |
| [3] |
范国强, 曹艳春, 赵振利, 等. 白花泡桐同源四倍体的诱导[J]. 林业科学, 2007, 43(4): 31-35. |
| [4] |
张晓申, 翟晓巧, 赵振利, 等. 不同种四倍体泡桐光合特性的研究[J]. 河南农业大学学报, 2013, 47(4): 400-404. |
| [5] |
张晓申, 刘荣宁, 范国强, 等. 四倍体泡桐对干旱胁迫的生理响应研究[J]. 河南农业大学学报, 2013, 47(5): 543-547, 551. |
| [6] |
QIAO J J, WANG J X, CHEN L, et al. Quantitative iTRAQ LC-MS/MS proteomics reveals metabolic responses to biofuel ethanol in Cyanobacterial Synechocystis sp. PCC 6803[J]. Journal of Proteome Research, 2012, 11(11): 5286-5300. DOI:10.1021/pr300504w |
| [7] |
WANG Z, LIU W S, FAN G Q, et al. Quantitative proteome-level analysis of Paulownia witches' broom disease with methyl methane sulfonate assistance reveals diverse metabolic changes during the infection and recovery processes[J]. PeerJ, 2017, 5: e3495. DOI:10.7717/peerj.3495 |
| [8] |
苏亚春.甘蔗应答黑穗病菌侵染的转录组与蛋白组研究及抗性相关基因挖掘[D].福州: 福建农林大学, 2014.
|
| [9] |
ZHANG X S, DENG M J, FAN G Q. Differential transcriptome analysis between Paulownia fortunei and its synthesized autopolyploid[J]. International Journal of Molecular Sciences, 2014, 15(3): 5079-5093. DOI:10.3390/ijms15035079 |
| [10] |
DONG Y P, DENG M J, ZHAO Z L, et al. Quantitative proteomic and transcriptomic study on autotetraploid Paulownia and its diploid parent reveal key metabolic processes associated with Paulownia Autotetraploidization[J]. Frontiers in Plant Science, 2016, 7: 892. |
| [11] |
CASNEUF T, BODT S D, RAES J, et al. Nonrandom divergence of gene expression following gene and genome duplications in the flowering plant Arabidopsis thaliana[J]. Genome Biology, 2006, 7(2): R13. DOI:10.1186/gb-2006-7-2-r13 |
| [12] |
SONG X M, WANG J P, SUN P C, et al. Preferential gene retention increases the robustness of cold regulation in Brassicaceae and other plants after polyploidization[J]. Horticulture Research, 2020, 7: 20. DOI:10.1038/s41438-020-0253-0 |
| [13] |
GARCÍA-GARCÍA A L, GRAJAL-MARTÍN M J, GONZÁLEZ-RODRÍGUEZ Á M. Polyploidization enhances photoprotection in the first stages of Mangifera indica[J]. Scientia Horticulturae, 2020, 264: 109198. DOI:10.1016/j.scienta.2020.109198 |
| [14] |
CHENG J L, YANG X H, XUE L F, et al. Polyploidization contributes to evolution of competitive ability:a long term common garden study on the invasive Solidago canadensis in China[J]. Oikos, 2020, 129(5): 700-713. DOI:10.1111/oik.07095 |
| [15] |
DONG Y P, FAN G Q, DENG M J, et al. Genome-wide expression profiling of the transcriptomes of four Paulownia tomentosa accessions in response to drought[J]. Genomics, 2014, 104(4): 295-305. DOI:10.1016/j.ygeno.2014.08.008 |
| [16] |
DONG Y P, FAN G Q, ZHAO Z L, et al. Transcriptome expression profiling in response to drought stress in Paulownia australis[J]. International Journal of Molecular Sciences, 2014, 15(3): 4583-4607. DOI:10.3390/ijms15034583 |
| [17] |
XU E K, FAN G Q, NIU S Y, et al. Transcriptome-wide profiling and expression analysis of diploid and autotetraploid Paulownia tomentosa×Paulownia fortunei under drought stress[J]. PLoS One, 2014, 9(11): e113313. DOI:10.1371/journal.pone.0113313 |
| [18] |
FAN G Q, WANG L M, DENG M J, et al. Changes in transcript related to osmosis and intracellular ion homeostasis in Paulownia tomentosa under salt stress[J]. Frontiers in Plant Science, 2016, 7: 384. |
| [19] |
DONG Y P, FAN G Q, ZHAO Z L, et al. Transcriptome-wide profiling and expression analysis of two accessions of Paulownia australis under salt stress[J]. Tree Genetics & Genomes, 2017, 13(5): 97. |
| [20] |
WANG Z, ZHAO Z L, FAN G Q, et al. A comparison of the transcriptomes between diploid and autotetraploid Paulownia fortunei under salt stress[J]. Physiology and Molecular Biology of Plants, 2019, 25(1): 1-11. DOI:10.1007/s12298-018-0578-4 |
| [21] |
FAN G Q, LI X Y, DENG M J, et al. Comparative analysis and identification of miRNAs and their target genes responsive to salt stress in diploid and tetraploid Paulownia fortunei seedlings[J]. PLoS One, 2016, 11(2): e0149617. DOI:10.1371/journal.pone.0149617 |
| [22] |
SANMARTIN M, PATERAKI I, CHATZOPOULOU F, et al. Differential expression of the ascorbate oxidase multigene family during fruit development and in response to stress[J]. Planta, 2007, 225(4): 873-885. DOI:10.1007/s00425-006-0399-5 |
| [23] |
郭燕, 朱杰, 许自成, 等. 植物抗坏血酸氧化酶的研究进展[J]. 中国农学通报, 2008(3): 196-199. |
| [24] |
AXELSSON E, LUNDQVIST J, SAWICKI A, et al. Recessiveness and dominance in barley mutants deficient in Mg-chelatase subunit D, an AAA protein involved in chlorophyll biosynthesis[J]. The Plant Cell, 2006, 18(12): 3606-3616. DOI:10.1105/tpc.106.042374 |
| [25] |
ZHANG H T, LI J J, YOO J H, et al. Rice Chlorina-1 and Chlorina-9 encode ChlD and ChlI subunits of Mg-chelatase, a key enzyme for chlorophyll synthesis and chloroplast development[J]. Plant Molecular Biology, 2006, 62(3): 325-337. DOI:10.1007/s11103-006-9024-z |
| [26] |
PONTIER D, ALBRIEUX C, JOYARD J, et al. Knock-out of the magnesium protoporphyrin IX methyltransferase gene in Arabidopsis:effects on chloroplast development and on chloroplast-to-nucleus signaling[J]. The Journal of Biological Chemistry, 2007, 282(4): 2297-2304. DOI:10.1074/jbc.M610286200 |
| [27] |
PAPENBROCK J, PFÜNDEL E, MOCK H P, et al. Decreased and increased expression of the subunit CHL I diminishes Mg chelatase activity and reduces chlorophyll synthesis in transgenic tobacco plants[J]. The Plant Journal, 2000, 22(2): 155-164. DOI:10.1046/j.1365-313x.2000.00724.x |
| [28] |
KARP N A, HUBER W, SADOWSKI P G, et al. Addressing accuracy and precision issues in iTRAQ quantitation[J]. Molecular & Cellular Proteomics, 2010, 9(9): 1885-1897. |
| [29] |
HA M, LU J, TIAN L, et al. Small RNAs serve as a genetic buffer against genomic shock in Arabidopsis interspecific hybrids and allopolyploids[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(42): 17835-17840. DOI:10.1073/pnas.0907003106 |

 2020, Vol. 40
2020, Vol. 40


