文章信息
- 叶小真, 杨泽慧, 张清华, 宋漳, 陈全助
- YE Xiaozhen, YANG Zehui, ZHANG Qinghua, SONG Zhang, CHEN Quanzhu
- 桉树枯萎病菌Fusarium solani分子检测技术研究
- Molecular detection of Fusarium solani, the pathogen of Eucalyptus wilt disease
- 森林与环境学报,2019, 39(6): 629-635.
- Journal of Forest and Environment,2019, 39(6): 629-635.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.06.010
-
文章历史
- 收稿日期: 2019-06-24
- 修回日期: 2019-07-23
2. 福建农林大学林学院, 福建 福州 350002
2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
镰刀菌(Fusarium spp.)引起的桉树枯萎病是桉树(Eucalyptus spp.)人工林中的一种重要枝干病害,其典型症状表现为患病枝、干、根的横切面有深褐色的环状坏死斑,严重时木质部全变成深褐色。目前研究多认为该病是由尖孢镰刀菌(F. oxysporum)侵染引起的[1-2]。而KUMAR et al[3]曾报道茄病镰刀菌(F. solani)可侵染赤桉(Eucalyptus camaldulensis Dehnh.)引起典型的枯萎病症状。我们的前期研究也发现茄病镰刀菌是桉树枯萎病的重要病原菌。茄病镰刀菌除了侵染桉树外,还可引起杨树(Populus sp.)、印度黄檀(Dalbergia sissoo DC.)、西瓜[Citrullus lanatus (Thunb.) Matsum. & Nakai]、马铃薯(Solanum tuberosum Linn.)等多种植物的枯萎[4-10]。该菌主要从根部侵入,通过维管束输导组织向上扩展至树干、枝条,导致根部腐烂,主干、枝条干枯,叶片脱落,植株枯萎死亡[1]。2006年该病首次在我国广东省河源市发现,导致大片桉树幼林枯萎死亡,之后陆续在广西、云南等地发生,造成重大经济损失[11]。
快速、准确的检测技术对桉树枯萎病的早期诊断和科学防治具有重要指导意义。传统的植物病原真菌检测方法包括病害症状识别,病原菌形态和培养性状观察、生物学特性测定等,但这些方法存在操作繁琐、检测速度慢、结果不太可靠等局限性。近年来,随着分子生物学的不断发展,以PCR为基础的分子检测技术的普及,为病原菌快速检测带来了新的途径[12-13]。随机扩增多态性(random amplified polymorphic DNA,RAPD)标记技术是不同的碱基随机排列的寡聚核苷酸链(通常为8~10 bp)为引物,对研究对象基因组DNA进行PCR扩增,具有信息量大、费用小等优点[14-15]。但随机引物扩增所需要的退火温度较低,易出现稳定性差、重复性差、效率低的问题[16]。序列特征性扩增区域(sequence characterized amplified region,SCAR)标记是基于RAPD技术发展而来的PCR标记方法[17]:即先通过随机引物找到RAPD片段,将其克隆测序,根据序列分析结果在其两端序列设计特异性引物,进一步通过PCR特异反应,把与原RAPD片段相对应的特异位点扩增出来。SCAR标记结果更稳定,且重复性高,适用于大量样品的快速分析鉴定。
至今,SCAR分子标记已在病害防治、生理小种检测、动植物检疫及抗病育种等多方面产生重要作用[18-20]。在病害检测上,TSYGANKOVA et al[21]在研究病原菌黄单胞杆菌属(Xanthomonas)、假单胞菌属(Pseudumonas)、欧文氏菌属(Erwinia)时发现引物KRPN2能从参试的所有X. campestri菌株中特异地扩增出1条约600 bp的片段,并进一步将该特异性片段转换成稳定可靠的SCAR标记。REDDY et al[22]通过引物OPJ-14和OPK-12从F. oxysporum f. sp. ricini分别扩增出两条特异性的RAPD片段,且成功地转化成SCAR标记。此外,SCAR标记还被用于猕猴桃溃疡病(Pseudomonas syringae pv. actinidiae)、板栗疫病(Cryphonectria parasitica)、马铃薯晚疫病(Phytophthora infestans)、稻曲病(Ustilaginoidea virens)、小麦秆锈病(Puccinia graminis f. sp. tritici.)等多种植物病害的快速检测[23-27]。但桉树枯萎病菌F. solani的SCAR分子标记及其检测技术的研究尚未见报道。本文旨在利用RAPD与SCAR分子标记技术,建立一套灵敏快速的桉树枯萎病病原菌检测技术,为该病害的分子鉴定、早期防治提供技术指导。
1 材料与方法 1.1 试验材料供试菌株包括常见植物病原镰刀菌共9株,详见表 1。其中,茄病镰刀菌菌株从采集自福建南安的具有典型枯萎病症状的桉树枝干分离鉴定得到,禾谷镰刀菌及串珠镰刀菌由福建农林大学闽台作物有害生物生态防控国家重点实验室提供,其余菌株由本实验室分离并保存。
| 编号 No. |
菌株 Species |
寄主 Host |
| 1 | 茄病镰刀菌F. solani | 桉树Eucalyptus urophylla×E. grandis 3229 |
| 2 | 尖孢镰刀菌F. oxysporum | 鄂西红豆Ormosia hosiei Hemsl. et Wils |
| 3 | 禾谷镰刀菌F. graminearum | 小麦Triticum aestivum L. |
| 4 | 砖红镰刀菌F. lateritium | 鄂西红豆O. hosiei Hemsl. et Wils |
| 5 | 多隔镰刀菌F.decemcellulare | 闽楠Phoebe bournei (Hemsl.) Yang |
| 6 | 层生镰刀菌F. proliferatum | 闽楠Phoebe bournei (Hemsl.) Yang |
| 7 | 厚孢镰刀菌F. chlamydosporum | 无患子Sapindus mukorossi Gaertn. |
| 8 | 九州镰刀菌F. kyushuense | 无患子S. mukorossi Gaertn. |
| 9 | 串珠镰刀菌F. moniliforme | 玉米Zea mays L. |
将供试菌株接种至PDA(土豆200 g·L-1、葡萄糖20 g·L-1、琼脂粉15 g·L-1)平板上,28 ℃培养6 d后收集菌丝。按照真菌基因组DNA提取试剂盒(OMEGA)提取基因组DNA。
1.2.2 RAPD扩增与特异性片段克隆共合成100条大小为8~10 bp的随机引物去扩增所有供试菌株,筛选桉树枯萎病菌F. solani特异性片段。扩增体系:12.5 μL PCR mix(Premix为Taq酶、dNTPs、MgCl2、10×buffer以及上样色素的混合物),1 μL 10 μmol·L-1的引物,1 μL 40 ng·μL-1的DNA模板,10.5 μL ddH2O。反应程序为94 ℃预变性5 min,94 ℃变性1 min,36 ℃退火1 min,72 ℃延伸1 min,35个循环,最后延伸10 min,4 ℃保存。反应结束后,取5 μL扩增产物在1%琼脂糖凝胶中120 V下电泳25 min,经溴化乙锭(EB)染色后在凝胶成像系统(Bio-Rad)上观察、拍照。回收特异片段,将回收产物连接到PEASY-T1载体,再转化至大肠杆菌感受态细胞Trans-T1,转化后置于含有氨苄青霉素、IPTG和X-Gal的LA培养基上37 ℃培养过夜,挑取白斑进行PCR阳性鉴定。将阳性克隆送至广州擎科生物公司进行测序。
1.2.3 特异性引物设计根据测序结果,利用DNAMAN软件(Version6.0.3),设计特异性引物F9/R9,由福州擎科生物技术有限公司合成。
1.2.4 引物特异性验证及扩增条件优化用特异性引物F9/R9对所有供试菌株的基因组DNA进行PCR扩增,以ddH2O为阴性对照,桉树枯萎病菌F. solani为阳性对照,进一步验证引物特异性。同时利用温度梯度PCR仪,在45~65 ℃之间共设置11个温度梯度,以优化退火温度,并对引物浓度(编号1~4为5 mol·μL-1; 5~8为10 mol·μL-1; 9~12为15 mol·μL-1; 13~16为20 mol·μL-1)、DNA模板浓度(编号1、5、9、13为20 ng·μL-1; 编号2、6、10、14为40 ng·μL-1; 编号3、7、11、15为60 ng·μL-1; 编号4、8、12、16为80 ng·μL-1)进行优化。
1.2.5 引物灵敏度检测按逐级稀释法,用ddH2O将病原菌基因组DNA配置成400、40、4 ng·μL-1,400、40、4 pg·μL-1,400、40、4、0.4 fg·μL-1共10个浓度梯度,用特异性引物F9/R9进行PCR扩增,以确定最高灵敏度。同时在显微镜下挑取桉树枯萎病菌F. solani单个分生孢子,用特异性引物进行扩增。
1.2.6 发病组织及带菌土壤的检测用无菌水将培养10 d的桉树枯萎病菌F. solani分生孢子洗下,配制成浓度为1×107个·mL-1的悬浮液,分别接种于桉苗(E. urophylla × E. grandis 3229)和灭菌土壤中。用无菌剪刀剪取接种枯萎病菌3 d后的桉树枝干组织,根据真菌基因组DNA提取试剂盒(OMEGA)操作说明提取基因组DNA。采用SDS高盐法抽提含有病原菌的土壤基因组DNA[28]。以接种枯萎病菌的桉树枝干组织及土壤基因组DNA为模板,用特异性引物F9/R9进行PCR扩增,以枯萎病菌F. solani为阳性对照,ddH2O、健康桉树枝干组织及无菌土为阴性对照。
2 结果与分析 2.1 特异片段筛选以所有供试菌株的基因组DNA为模板,采用随机引物进行RAPD扩增。结果如图 1所示,引物P38(5′-AGGTGTGCCA-3′)可以产生一条大小为400 bp左右的特异性片段,此片段只有病原菌F. solani可以扩增出,其它供试菌株不能扩增出此条带,说明P38是桉树枯萎病菌特异性引物。
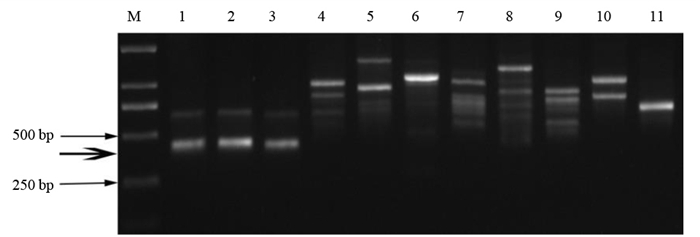
|
注:M为DL2000标记;1~3为茄病镰刀菌;4为尖孢镰刀菌;5为禾谷镰刀菌;6为砖红镰刀菌;7为多隔镰刀菌;8为层生镰刀菌;9为厚孢镰刀菌;10为九州镰刀菌;11为串珠镰刀菌。 Note: M is DL2000 marker; 1-3 are Fusarium solani; 4 is F. oxysporum; 5 is F. graminearum; 6 is F. lateritium; 7 is F.decemcellulare; 8 is F. proliferatum; 9 is F. chlamydosporum; 10 is F. kyushuense; 11 is F. moniliforme. 图 1 随机引物P38对桉树枯萎病菌及其他病原菌的扩增结果 Fig. 1 PCR products of F. solani and other pathogens amplified with the primer P38 |
将引物P38所扩增出的特异性片段回收、克隆并测序。在序列两端找到随机引物及其反向互补序列所在位置,去掉载体序列,得到大小为376 bp的特异性序列(图 2)。将序列在GenBank中在线比对,未发现同源序列。进一步利用DNAMAN软件,设计一对特异性引物F9/R9。正向引物F9(5′-TCGGTTGTCGCAAAGACT-3′),位于特异片段的第159~176位点。反向引物R9(5′-GGGAAGTTGTGTCAGCATT-3′),位于特异片段的第310~328位点,扩增目的片段大小为170 bp。

|
注:阴影部分为随机引物及其反向互补序列; 下划线部分为SCAR特异性引物正反向序列。 Note: shaded parts are the random primers and their reverse complementary sequence; underlined parts are the forward and backward sequence of SCAR specific primers. 图 2 桉树枯萎病菌特异DNA片段序列 Fig. 2 Nucleotic sequences of the specific DNA fragment of F. solani |
利用设计的特异性引物F9/R9对所有供试菌株基因组DNA进行PCR扩增。结果显示,只有桉树枯萎病菌能够扩增出一条大小为200 bp左右的条带,其他菌株和阴性对照均无条带产生,说明该引物具有特异性(图 3)。对扩增产物进行回收、克隆、测序。用DNAMAN软件将测序结果与RAPD筛选出的特异片段序列进行比对,结果显示,二者相似性达到99%,二者检测结果相同,进一步证明引物具有特异性,且RAPD标记成功转化为更稳定的SCAR标记。
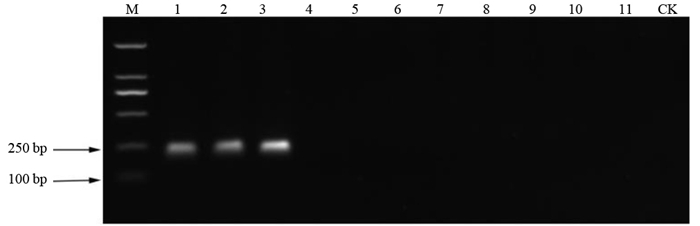
|
注:M为DL2000标记;1~3为茄病镰刀菌;4为尖孢镰刀菌;5为禾谷镰刀菌;6为砖红镰刀菌;7为多隔镰刀菌;8为层生镰刀菌;9为厚孢镰刀菌;10为九州镰刀菌;11为串珠镰刀菌;CK为阴性对照。 Note:M is DL2000 marker; 1-3 is F. solani; 4 is F. oxysporum; 5 is F. graminearum; 6 is F. lateritium; 7 is F. decemcellulare; 8 is F. proliferatum; 9 is F. chlamydosporum; 10 is F. kyushuense; 11 is F. moniliforme; CK is negative control. 图 3 引物F9/R9特异性验证结果 Fig. 3 Specificity detection of primers F9/R9 |
从退火温度、DNA模板浓度、引物浓度3个方面对扩增条件进行优化。结果表明,退火温度设置在45~61 ℃均可产生扩增条带,其中,57.3 ℃可产生清晰明亮的条带,故后续退火温度设定为57 ℃(图 4)。对模板和引物浓度优化结果见图 5,随着引物浓度增加,条带越清晰明亮,当引物浓度为5 mol·μL-1时,条带都较亮。同一引物浓度,随着DNA模板用量的增加,条带逐渐变亮。泳道11、12、15、16扩增效果相对较好,从节约角度考虑,选择12泳道即引物浓度为15 mol·μL-1,模板浓度为80 ng·μL-1为最终PCR扩增体系。因此特异性引物PCR扩增的最佳反应体系为:12.5 μL Premix;引物F9/R9(10 mol·μL-1)各1.5 μL;2 μL模板DNA(40 ng·μL-1);ddH2O补足至25 μL。反应程序为:95 ℃预变性4 min,94 ℃变性1 min,57 ℃退火1 min,72 ℃延伸1 min,共35个循环,72 ℃延伸10 min,最后4 ℃保存。
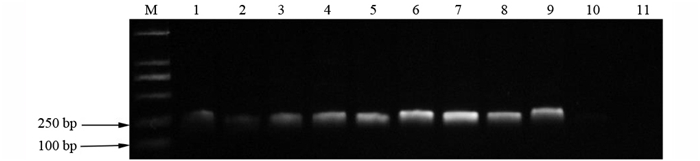
|
注:M为DL2000标记;1~11分别为45, 46.8, 49.4, 51.6, 52.9, 55.2, 57.3, 58.5, 61.1, 63.3, 65 ℃。 Note:M is DL2000 marker; 1-11 is 45, 46.8, 49.4, 51.6, 52.9, 55.2, 57.3, 58.5, 61.1, 63.3, 65 ℃, respectively. 图 4 特异性引物F9/R9退火温度筛选 Fig. 4 Optimization of annealing temperature of primers F9/R9 |
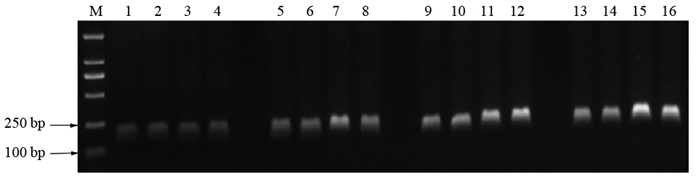
|
注:M为DL2000标记;引物浓度(编号1~4为5 mol·μL-1,5~8为10 mol·μL-1,9-12为15 mol·μL-1,13~16为20 mol·μL-1);DNA模板浓度(编号1、5、9、13为20 ng·μL-1,编号2、6、10、14为40 ng·μL-1,编号3、7、11、15为60 ng·μL-1,编号4、8、12、16为80 ng·μL-1)。 Note: M is DL2000 marker; primer concentration(No. 1-4 are 5 mol·μL-1, 5-8 are 10 mol·μL-1, 9-12 are 15 mol·μL-1, 13-16 are 20 mol·μL-1); template concentration(No. 1、5、9、13 are 20 ng·μL-1, No. 2、6、10、14 are 40 ng·μL-1, No. are 3、7、11、15 are 60 ng·μL-1, No. are 4、8、12、16 are 80 ng·μL-1). 图 5 特异性引物PCR反应体系优化 Fig. 5 Optimization of PCR reaction system of specific primers |
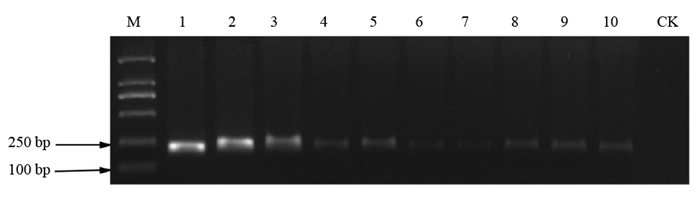
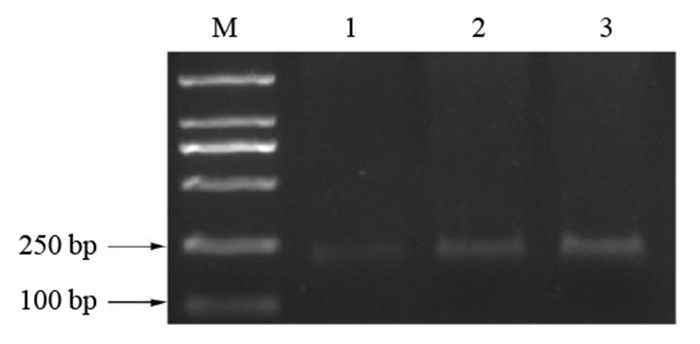
用特异性引物F9/R9分别对不同浓度的基因组DNA进行PCR扩增。结果如图 6显示,DNA浓度为0.4 fg·μL-1时,仍有条带产生,而对照无条带产生,说明该引物具有较高的灵敏度。另外,该特异性引物对单个分生孢子也能扩增出明显的条带(图 7),进一步说明该引物具有极高的灵敏度。
|
注:M为DL2000标记。1~10分别为400, 40, 4 ng·μL-1; 400, 40, 4 pg·μL-1; 400, 40, 4, 0.4 fg·μL-1。CK为阴性对照。 Note: M is DL2000 marker. 1-10 is 400, 40, 4 ng·μL-1; 400, 40, 4 pg·μL-1; 400, 40, 4, 0.4 fg·μL-1, respectively. CK is negative control. 图 6 特异性引物F9/R9灵敏度检测 Fig. 6 Sensitivity of PCR with specific primers F9/R9 |
|
注:M为DL2000标记;1~3为茄病镰刀菌单孢。 Note:M is DL2000 marker; 1-3 are single spore of F. solani. 图 7 特异性引物F9/R9单孢检测 Fig. 7 Single spore detection of PCR with specific primers F9/R9 |
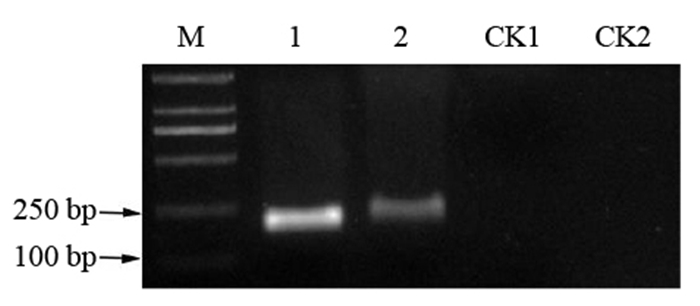
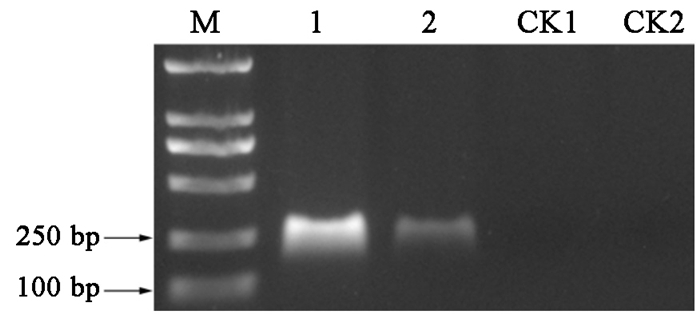
分别以接种病原菌F. solani的枝干组织和土壤基因组DNA为模板,利用特异性引物F9/R9进行PCR扩增。结果如图 8、图 9所示,病原菌、发病桉树枝干、带菌土壤均可以扩增出一条大小为170 bp的条带,而健康桉树枝干、无菌土和ddH2O均无条带产生。表明该特异性引物可以从含有枯萎病菌的桉树枝干及土壤中检测到病原菌,这为桉树枯萎病F. solani的预警与早期防治带来了新的方法。
|
注:M为DL2000标记;1为F. solani;2为接种病原菌枝干;CK1为健康植株枝干;CK2为ddH2O。 Note:M is DL2000 marker; 1 is F. solani; 2 is DNA of Eucalyptus stem after artificial inoculation; CK1 is DNA of healthy stem; CK2 is ddH2O. 图 8 桉树枝干组织特异性检测 Fig. 8 Detection of F. solani from Eucalyptus stem |
|
注:M为DL2000标记;1为F. solani;2为F. solani+无菌土;CK1为ddH2O+无菌土;CK2为ddH2O。 Note:M is DL2000 marker; 1 is F. solani; 2 is DNA of F. solani+sterile soil; CK1 is DNA of ddH2O + sterile soil; CK2 is ddH2O 图 9 土壤特异性检测 Fig. 9 Detection of F. solani from soil |
镰刀菌广泛分布在自然界中,其种类繁多、形态变异大,受温湿度、光照、pH值等环境条件的影响室内培养时培养性状容易发生明显变化,且形态鉴定本身耗时耗力,无法满足基层对镰刀菌快速准确鉴定的需求。分子标记是镰刀菌快速准确鉴定的强有力手段。其中,RAPD分子标记已被用于不同寄主专化型的茄类镰刀菌种群研究。如被用于区分大豆猝死综合症和印度黄檀枯萎病的致病茄类镰刀菌和非致病镰刀菌[29-30]。另外,GUPTA et al[31]发现相比RPLP,RAPD能更有效地将7个F. solani菌株区分开来,并于2012年开发了用以分析番石榴枯萎病菌F. oxysporum f. sp. psidii和F. solani的RAPD标记。但以往研究茄类镰刀菌时一般未将RAPD转化为SCAR标记。而RAPD标记所使用的引物较短、退火温度较低,存在不稳定的缺陷,并且RAPD多态性强,产生条带数量多且较杂乱,主观判断易出错。
本研究通过RAPD分子标记技术,从100条大小为10 bp的随机引物中筛到一条引物P38,能够特异地从桉树枯萎病菌F. solani中扩增出一条376 bp的条带,进一步将该特异片段进行回收、克隆测序,根据序列设计出一对特异性强、稳定性高的特异性引物F9/R9,从而将RAPD标记转化为SCAR标记。同时,对PCR扩增体系中的退火温度、引物及模板浓度作了梯度优化,以此提高PCR扩增效率。该特异性引物不仅能从所有供试菌株中特异性检测出桉树枯萎病菌F. solani,还能在含有致病菌F. solani的桉树枝干和土壤中检测到病原菌,在DNA含量为4×10-7 ng·μL-1时都可以检测到病原菌的存在,挑取单个孢子进行检测,该特异性引物也能检测到单个孢子的存在,说明该引物适合活体检测,且灵敏度极高。与常规病菌检测技术相比,该方法成本极低,只需准备引物和常规PCR试剂,在此基础上,1 d之内即可获得检测结果,明显快于常规方法。该方法可用于桉树枯萎病的早期诊断和预测预报,对防止该病害进一步蔓延扩展具有重要的实践意义。
| [1] |
伍慧雄, 王军, 丁志锋, 等. 桉树枯萎病的病原鉴定[J]. 广东林业科技, 2007, 23(5): 59-61. DOI:10.3969/j.issn.1006-4427.2007.05.013 |
| [2] |
汪燕, 王军, 孙思. 桉树枯萎病菌的生物学特性及对杀菌剂的敏感性试验[J]. 广东林业科技, 2010, 26(5): 75-78. DOI:10.3969/j.issn.1006-4427.2010.05.015 |
| [3] |
KUMAR A, NATH V. Fusarium solani causing wilt of Eucalyptus[J]. Current Science, 1988, 57(16): 907-908. |
| [4] |
陈世荣, 王建华, 储祥宏, 等. 杨树枯萎病的发生与防治初探[J]. 江苏林业科技, 2013, 40(4): 31-33. DOI:10.3969/j.issn.1001-7380.2013.04.009 |
| [5] |
李伶俐, 韩正敏, 吕明亮, 等. 杨树枯萎病菌茄类镰刀菌的生物学特性[J]. 林业科技开发, 2009, 23(4): 51-54. DOI:10.3969/j.issn.1000-8101.2009.04.012 |
| [6] |
BASAK A C, BASAK S R. Fusarium solani sp. dalbergiae, the wilt pathogen of Dalbergia sissoo, by Trichoderma viride and T. harzianum[J]. Journal of Tropical Forest Science, 2011, 23(4): 460-466. |
| [7] |
COLE D L, HEDGES T R, NDOWORA T. A wilt of passion fruit (Passiflora edulis f. edulis Sims) caused by Fusarium solani and Phytophthora nicotianae var. parasitica[J]. Tropical Pest Management, 1992, 38(4): 362-366. DOI:10.1080/09670879209371728 |
| [8] |
GUPTA V K. PCR-RAPD profiling of Fusarium spp. causing guava wilt disease in India[J]. Journal of Environmental Science and Health, Part B, 2012, 47(4): 315-325. DOI:10.1080/03601234.2012.640915 |
| [9] |
OMMATI F, ZAKER M. In vitro and greenhouse evaluations of Trichoderma isolates for biological control of potato wilt disease (Fusarium solani)[J]. Archives of Phytopathology and Plant Protection, 2012, 45(14): 1715-1923. DOI:10.1080/03235408.2012.702467 |
| [10] |
TURÓCZI G, POSTA K, BADENSZKY L, et al. Fusarium wilt of water melon caused by Fusarium solani in Hungary[J]. Plant Breeding and Seed Science, 2012, 63(1): 23-28. |
| [11] |
王勇智. 速生桉枯萎病的观察与研究[J]. 农家之友, 2008(12): 38-39. |
| [12] |
常有宏, 张磊, 刘邮洲, 等. 植物病原真菌PCR检测技术研究进展[J]. 江苏农业科学, 2012, 40(2): 89-93. DOI:10.3969/j.issn.1002-1302.2012.02.037 |
| [13] |
彭丹丹, 张源明, 舒灿伟, 等. 植物病原真菌分子检测技术的研究进展[J]. 基因组学与应用生物学, 2017, 36(5): 2015-2022. |
| [14] |
WELSH J, MCCLELLAND M. Fingerprinting genomes using PCR with arbitrary primers[J]. Nucleic Acids Research, 1990, 18(24): 7213-7218. DOI:10.1093/nar/18.24.7213 |
| [15] |
WILLIAMS J G K, KUBELIK A R, LIVAK K J, et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Research, 1990, 18(22): 6531-6535. DOI:10.1093/nar/18.22.6531 |
| [16] |
姜自锋, 林乃铨, 徐梅. RAPD技术及其应用中的一些问题[J]. 福建农林大学学报(自然科学版), 2002, 31(3): 356-360. |
| [17] |
PARAN I, MICHELMORE R W. Development of reliable PCR-based markers linked to downy mildew resistance genes in lettuce[J]. Theoretical and Applied Genetics, 1993, 85(8): 985-993. DOI:10.1007/BF00215038 |
| [18] |
毕波, 王瑜, 袁庆华. SCAR分子标记在植物抗病育种中的应用[J]. 安徽农业科学, 2010, 38(30): 16778-16780, 16957. DOI:10.3969/j.issn.0517-6611.2010.30.026 |
| [19] |
廖林凤, 董章勇, 王振中, 等. 香蕉枯萎病菌RAPD分析及4号生理小种的快速检测[J]. 植物病理学报, 2009, 39(4): 353-361. |
| [20] |
马丽娜, 钱路, 李健, 等. SCAR标记在植物检疫中应用的研究进展[J]. 南方农业, 2017, 11(29): 85-86. |
| [21] |
TSYGANKOVA S V, IGNATOV A N, BOULYGINA E S, et al. Genetic relationships among strains of Xanthomonas campestris pv. campestris revealed by novel rep-PCR primers[J]. European Journal of Plant Pathology, 2004, 110(8): 845-853. DOI:10.1007/s10658-004-2726-7 |
| [22] |
REDDY J M, RAOOF M A, ULAGANATHAN K. Development of specific markers for identification of Indian isolates of Fusarium oxysporum f. sp. ricini[J]. European Journal of Plant Pathology, 2012, 134(4): 713-719. DOI:10.1007/s10658-012-0047-9 |
| [23] |
陈福如, 林廷邦, 甘林, 等. 稻曲病菌的SCAR标记及其PCR检测[J]. 植物保护学报, 2013, 40(6): 481-487. |
| [24] |
陈思, 曹远银, 李天亚, 等. 小麦秆锈菌生理小种21C3CTH SCAR标记的建立[J]. 麦类作物学报, 2014, 34(6): 740-744. |
| [25] |
李新新.马铃薯晚疫病菌的长期保存与特异性SCAR标记的开发[D].哈尔滨: 东北农业大学, 2012.
|
| [26] |
麻文建.板栗疫病菌的分子检测研究[D].成都: 四川农业大学, 2015.
|
| [27] |
邵宝林, 刘瑶, 朱天辉, 等. 猕猴桃溃疡病菌的分子检测技术研究[J]. 植物病理学报, 2013, 43(5): 458-466. |
| [28] |
邵劲松, 浦牧野, 顾祝军. 3种土壤微生物基因组DNA提取方法的比较[J]. 南京晓庄学院学报, 2013(3): 64-67. DOI:10.3969/j.issn.1009-7902.2013.03.016 |
| [29] |
ACHENBACH L A, PATRICK J, GRAY L. Use of RAPD markers as a diagnostic tool for the identification of Fusarium solani isolates that cause soybean sudden death syndrome[J]. Plant Disease, 1996, 80(11): 1228-1232. DOI:10.1094/PD-80-1228 |
| [30] |
ARIF M, ZAIDI N W, HAQ Q M R, et al. Morphological and comparative genomic analyses of pathogenic and non-pathogenic Fusarium solani isolated from Dalbergia sissoo[J]. Molecular Biology Reports, 2015, 42(6): 1107-1122. DOI:10.1007/s11033-014-3849-3 |
| [31] |
GUPTA V K, JAIN P K, MISRA A K, et al. Comparative molecular analysis of Fusarium solani isolates by RFLP and RAPD[J]. Microbiology, 2010, 79(6): 772-776. DOI:10.1134/S0026261710060081 |

 2019, Vol. 39
2019, Vol. 39


