文章信息
- 殷雨晴, 任健, 陈典, 王勇
- YIN Yuqing, REN Jian, CHEN Dian, WANG Yong
- 马占相思AmCesA2基因的克隆与表达分析
- Cloning and expression analysis of AmCesA2 gene in Acacia mangium
- 森林与环境学报,2019, 39(2): 174-180.
- Journal of Forest and Environment,2019, 39(2): 174-180.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.02.009
-
文章历史
- 收稿日期: 2018-04-19
- 修回日期: 2018-05-15
2. 农业部东北地区园艺作物生物学与种质创制重点实验室, 黑龙江 哈尔滨 150030
2. Key Laboratory of Biology Improvement of Horticulture Crops Ministry of Agriculture at Northeast Region, Harbin, Heilongjiang 150030, China
马占相思(Acacia mangium Willd)为豆科金合欢属,常绿高大乔木,生长迅速,是热带地区重要的经济树种,具有较大的栽培面积[1-2]。马占相思木质坚硬,适合于造纸、人造板以及家具制造等[3]。除具有商业价值外,还常用于恢复矿区退化土地,经济效益和生态效益都十分显著[4]。作为一种速生类木材,相思树的纤维素含量是其重要的经济性状[5]。
纤维素是由D-葡萄糖以β-1,4糖苷键组成的大分子多糖,分子量在50 000~2 500 000之间,相当于300~15 000个葡萄糖基,是植物细胞壁的主要多糖类组成成分,也是众多生物不可缺少的结构组分[6-9]。它由植物纤维素合酶亚基A组成的纤维素合酶复合体和其他酶共同作用而形成的,储存在植物的初生壁和次生壁中[10-13]。其中,纤维素合酶是植物体内纤维素合成最为关键的酶[14]。纤维素合成酶基因最早从木醋杆菌[15]中分离得到,而植物中的纤维素合酶基因最早从陆地棉[16]纤维中被克隆得到。随后,其他植物中的纤维素合酶基因陆续被发现,如毛果杨[17]、松树[18]、桉树[19]、杨树[20]等。通过对这些木本类植物的纤维素合酶基因研究,发现在植物细胞壁的初生壁和次生壁形成时期,纤维素合酶基因的表达含量相对较高,尤其在次生维管组织发育的后期高表达,表明其主要参与木材次生壁纤维素的合成。近年来,有关纤维素合酶的研究也取得了许多成果。张海平等[21]人将调控木醋杆菌纤维素合成的两个主要基因acsA和acsB构建双价载体,分别转化白色棉和彩色棉,提高了棉花的纤维品质。用转基因方法特异性上调GA20-oxidase和抑制GA2-oxidase,使棉花纤维内源性GA生物活性升高,促进了纤维素的生物合成和次生壁的沉积,进而稳定纤维的形态[22]。
本研究通过RACE技术,从马占相思的幼苗中获得了1个CesA基因AmCesA2,通过分析该基因的序列,预测其编码蛋白结构,进而分析该基因在植株内的表达情况和对激素的反应,并对其功能做出了预测,从而为后续的实验研究提供参考。
1 材料与方法 1.1 植物材料马占相思的种子购自山东省济南市日出种业有限责任公司,其幼苗的培养基质为排水条件良好的砂质土壤,温室内温度保持在25~35 ℃之间。选取在温室中生长约30~40 d苗龄的马占相思幼苗用于RNA提取,用于检测基因表达的为随机选取的3组各10株40 d苗龄马占相思的根、茎、叶,分别取样,混合后用液氮速冻,于-80 ℃的条件下保存。
1.2 AmCesA2基因的克隆以硫氰酸胍-酚法提取相思树总RNA [23],以OligodT为引物进行反转录。现有的研究结果表明,生物的纤维素合酶家族具有非常保守的结构域,为此,分别就2段保守结构域设计了2条简并引物AmCesDe1和AmCesDe2 (表 1),结合3′RACE技术和5′RACE技术进行基因克隆。
| 序号 Number |
引物名称 Primer name |
引物序列(5′→3′) Primer sequence |
应用Application |
| 1 | AmCesDe1 | GGTIAT(A/C/T)TG(T/C)GA(A/G)(A/G)TITGGTT(T/C)GC | 扩增AmCesA2中间cDNA To amplify the intermediate cDNA of AmCesA2 |
| 2 | Aces2.3-3 | TGGTTGGGTCAACATGCTTGCT | |
| 3 | AmCesDe2 | TT(T/C)CCICA(A/G)(A/C)GITT(T/C)GA(T/C)GGIAT(A/C/T)GA | AmCesA2 3′ RACE |
| 4 | OligodT | GCGGTACCCTTTTTTTTTTTTTTTTTT | |
| 5 | AAP | GGCCACGCGTCGACTAGTACGGGIIGGGIIGGGIIG | Nest PCR for AmCesA2 |
| 6 | Aces2GSP1 | ATATATCAACAGCTGCTAGT | AmCesA2 5′ RACE |
| 7 | AUAP | GGCCACGCGTCGACTAGTAC | |
| 8 | Aces2GSP2 | AGATATGTTTCCCGGTTTACC | |
| 9 | Aces2.3′-1 | TGAGCTCTTCTAGCAGTTTATTCCACACT | 扩增AmCesA2全长cDNA Amplify AmCesA2 full-length cDNA |
| 10 | Aces2.5′-1 | ATCTAGACAGCTTATGGAGTCAGAAGG | |
| 11 | QAmCesA2F | AGAGGCTTTCAATGGCATCC | 实时定量基因荧光检测Rreal-time quantitative gene fluorescence detection |
| 12 | QAmCesA2R | CCAGGATCCACAGCCCTAAT | |
| 13 | ActinF | GGTAACATTGTCCTCTGTGGT | 荧光定量检测内参基因Fluorescence quantitative detection of internal reference genes |
| 14 | ActinR | CATCGTATTCTGCCTTCGA |
以反转录产物为模板,用AmCesDe2和OligodT引物进行第一步PCR扩增,PCR条件为:94℃预变性5 min,热启动; 94 ℃ 25 s, 55 ℃ 1 min, 72 ℃ 2 min, 33个循环;72 ℃ 10 min终止反应。PCR产物经琼脂糖凝胶电泳检测后,过柱回收,与pGEM-T easy载体连接,转化E.coli DH5α,筛选阳性克隆测序。根据测序结果设计基因特异引物以Aces2.3-3仍以反转录产物为模板,以Aces2.3-3、AmCesDe1进行第二步PCR扩增,条件:94 ℃预变性5 min,热启动; 94 ℃ 30 s, 53 ℃ 1 min,72 ℃ 90s,30个循环;72 ℃ 10 min终止反应。将所得产物测序后与第一次PCR所得产物组装,设计基因特异引物Aces2GSP1和Aces2GSP2,利用Takara公司5′-Full RACE Kit with TAP试剂盒进行5′RACE。RACE反应按试剂盒说明操作(Code No.6106)。所得的5′RACE片段测序后与所获得的下游序列组装,根据序列信息,设计引物Aces2.3′-1, Aces2.5′-1,进行PCR扩增得基因cDNA序列。
1.3 统计分析方法以DNAMAN6.0软件应用于序列比对;EXPASY-ProtParamtool (http://web.expasy.org/protparam/)用于对蛋白质的一级结构进分析;TMHMM Sever v.2.0 (http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构预测;PFAM (http://pfam.xfam.org/)用于对蛋白质的结构域进行分析;MEGA 5.22用于系统进化树的构建。
1.4 统计分析方法根据克隆得到的AmCesA2的cDNA,设计荧光定量PCR引物(表 1),分别提取3组相思树根、茎、叶的总RNA,反转录均衡浓度后,取1μl cDNA作为模板,利用TaKaRa公司SYBR Premix Ex Taq (TaKaRa)荧光定量试剂盒完成荧光定量实验。
1.5 AmCesA2对激素的应答反应激素处理试验体系,条件参照魏凯莉的方法并加以适当调整[24]。方法为:将40 d苗龄且生长旺盛的马占相思幼苗分成3组,每组分别喷施10 μmol · L-1赤霉素(gibberellin, GA3), 0.1 mol · L-16-BA (6-benzylaminopurine), 1 nmol · L-124-表油菜素内酯(24-epicastasterone)。每隔2 h均匀喷施1次共喷施3次,每喷施1次后立刻取其叶片并放置-80 ℃冰箱中保存。每个处理设置1个对照,喷施与激素等量的蒸馏水。每个处理设置3次重复。
2 结果与分析 2.1 相思树AmCesA2基因的克隆以AmCesDe2、OligodT为引物扩增出一条包含有预期为1.6 kb的半弥散条带[图 1(a)],回收该条带,测序后表明为纤维素同源基因的3′端部分序列,据此设计引物Aces2.3-3,以Aces2.3-3、AmCesDe1为引物进行扩增,得到了一条约1.1 kb的cDNA片段[图 1(b)],之后通过5′RACE获得一条约1.0 kb的条带[图 1(c)],3个片段进行组装,最终得到一个长度为3 643 bp的AmCesA2 (AY643520.1)基因[图 1(d)],其编码区为3 228 bp,推测编码1 075个氨基酸,5′上游区为263 bp,3′下游区为152 bp。
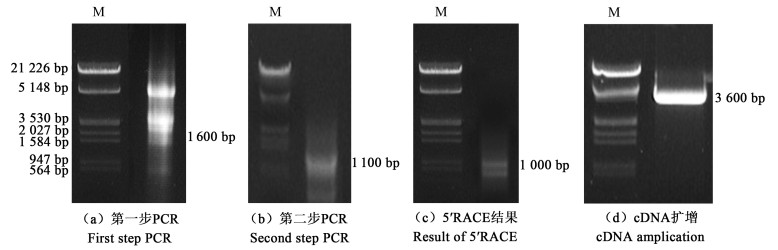
|
图 1 AmCesA2基因的扩增 Fig. 1 Amplification of AmCesA2 gene |
采用ExPASY对AmCesA2基因编码的蛋白质一级结构进行分析。分析表明,AmCesA2正电荷氨基酸残基数(Arg+Lys)的数目为119, 负电荷氨基酸残基数(Asp+Glu)的数目也为119,等电点为7.19,为碱性蛋白质。它的不稳定系数是38.56,属于稳定蛋白质。利用TMHMM Sever v.2.0进行跨膜结构预测。结果显示AmCesA2 N端存在2个跨膜区, 在C端具有6个跨膜区。利用Pfam对AmCesA2结构域进行分析,表明它存在着糖基转移酶类家族所特有的D, D, D, QxxRW结构。在整个蛋白的结构上,于N端存在一个Zinc-finger区,其后为植物纤维素合酶家族所特有的高度可变区1 (HVR-1区),之后顺序为植物特异性保守区(P-CR)和高度可变区2 (HVR-2),其中P-CR位于保守的domainA区域内,在HVR-2后为另一个纤维素合酶家族的保守区域domainB。
2.3 相思树AmCesA2同源性比对利用DNAman6.0软件将AmCesA2与拟南芥AtCesA3、陆地棉GhCesA3、毛白杨PtCesA3、银合欢LlCesA7进行序列比对分析,同时把相应的区域进行了标注(图 2)。比对结果显示,5个纤维素合酶具有较高的同源性,且在C端的同源性更高,高度可变区分别位于N端和保守的C端中部。进一步比对5个基因氨基酸水平上的相似性,发现AmCesA2与拟南芥AtCesA3的相似性为86.62%,与陆地棉GhCesA3的相似性为89.41%,与毛白杨PtCesA3的相似性为90.57%,与银合欢LlCesA7的相似性为95.81%,说明该基因家族同源性很高,进化上是非常保守的。
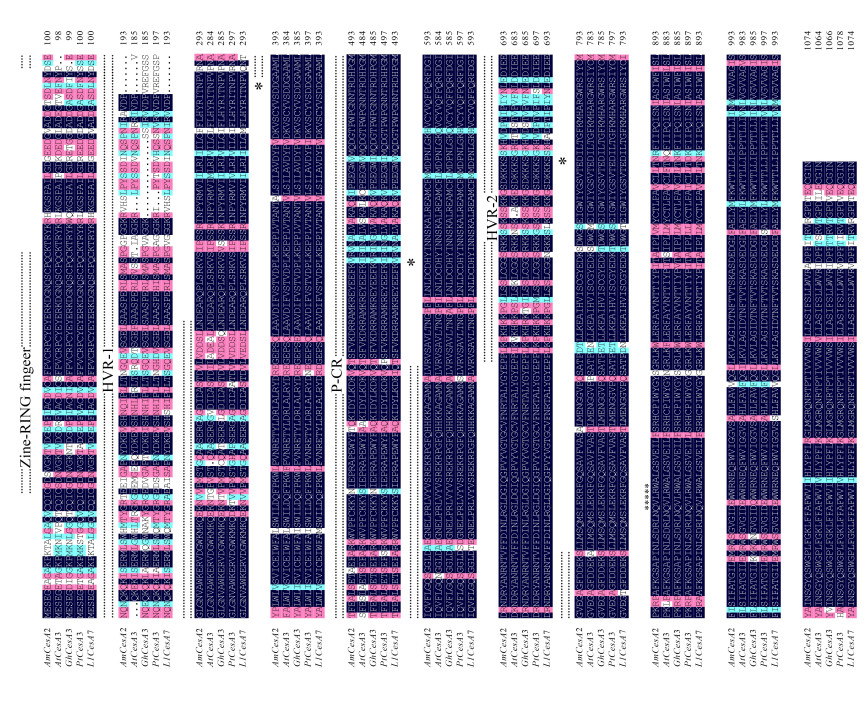
|
注:3个Asp (D)残基和QxxRW基序标有星号。顶部双点划线表示植物保守区(P-CR),可变区(VR1和-2)以及RING指状基序。 Note: 3 Asp (D) residues and the QxxRW motif that are marked by asterisks. The plant conserved region (P-CR), variable regions (VR1 and -2), and RING finger motif are also shown by overhead double-dashed lines. 图 2 AmCesA2氨基酸序列比对分析 Fig. 2 Alignment of the deduced amino acid sequences of AmCesA2 |
选取拟南芥、陆地棉、银合欢、杨树、白桦、赤桉、大豆、光皮桦、火炬松、巨桉、绿竹、马铃薯、花生、毛竹、杉木、亚麻、玉米等植物的纤维素合酶基因序列, 利用CLUSTAL1.81和MEGA5.22的NJ算法进行进化树构建,以了解植物CesA的进化特点,并选择拟南芥纤维素合酶类似蛋白(Csl)作为外类群以检验分析结果。结果表明,AmCesA2与LlCesA7、LlCesA8最为接近(图 3),这符合它们均为豆科含羞草亚科植物的特性。相思树AmCesA2进化树中并没有出现预期同木本植物聚集的情况,推测CesA的分化应早于草本植物和木本植物的分化。在与拟南芥纤维素合酶基因的比较中发现AmCesA2和拟南芥的AtCesA3比较相似,进一步比较其相似性为86.62%,说明它的功能可能与相应的拟南芥纤维素合酶异构体相近,且CesA基因与外类群基因Csl聚为两类,说明结果的可靠性较高。
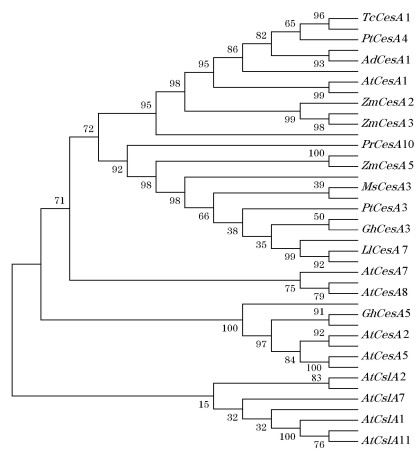
|
图 3 基于CesA的系统进化分析 Fig. 3 Phylogenetic analysis based on CesA |
AmCesA2基因广泛表达于黑木相思根、茎、叶中(图 4),其中在根和叶中的表达量相似,差异不显著(P>0.05),在茎中的表达量最高,约2倍于根中和叶中的表达量,表达丰度之间存在显著差异(P<0.05)。
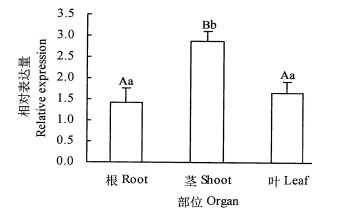
|
注:不同小写字母表示差异显著(P<0.05);不同大写字母表示差异极显著(P<0.01), Note: different lowercase letters denote significant difference(P<0.05), different uppercase letters denote highly significant difference(P<0.01). 图 4 各器官中AmcesA2的相对表达量 Fig. 4 AmcesA2 expression in different organs |
研究表明,激素能诱导CesA基因的表达[24-25]。以GA3, 6-BA, BR处理相思幼苗,观察AmCesA2对激素的反应。结果表明,在3种激素都增加了AmCesA2的表达量,且随着处理时间的延长,呈递增趋势(图 5)。AmCesA2对3种激素的响应程度不同,AmCesA2对GA3和6-BA的响应程度更高,且对GA3的响应最为强烈。GA3和6-BA具有促进细胞伸长和分裂的作用,因此推测AmCesA2可能参与细胞壁的快速形成。
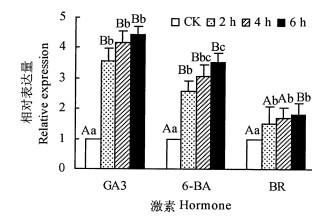
|
注:不同小写字母表示差异显著(P<0.05);不同大写字母表示差异极显著(P<0.01), Note: different lowercase letters denote significant difference(P<0.05), different uppercase letters denote highly significant difference(P<0.01). 图 5 AmCesA2对3种激素的反应 Fig. 5 AmCesA2 response to three hormones |
纤维素是植物重要的特征结构,可调控植物的形态建成,研究纤维素合成对可再生生物能源具有重要意义[26-29]。植物CesA基因以超基因家族形式存在,不同成员参与不同组织、器官或细胞壁层次纤维素合成[30]。其中大部分CesA基因参与初生壁的形成,而参与次生壁合成的CesA基因较少[31-32]。研究表明CesA基因编码的蛋白质都有类似的结构:具有8个跨膜结构域,N端有2个,C端有6个,此外在N端还有1个锌指结构域,可能和蛋白质之间的相互作用有关,CesA蛋白的保守功能域为外D, D, D, QxxRW,本研究以相思属的马占相思的幼苗为材料,从中获取了AmCesA2,其编码的蛋白具有CesA的典型特征。
纤维素合酶以亚基组成复合体的形式进行纤维素合成,在离体条件下进行基因的作用机理及功能研究具有较大的难度[33]。因此,可通过基因间的相似性,推测某一未知基因的功能[34]。通过拟南芥纤维素合酶基因(AtCesA)家族进行表达分析发现, 拟南芥纤维素合酶基因AtCesA1, 3, 6参与细胞壁初生壁的合成,AtCesA4, 7, 8细胞壁次生壁的合成,并存在明显的共表达现象[35-36]。其中, AtCesA1, 3, 6在全生育期表达, AtCesA4, 7, 8主要在根、茎和叶脉等次生壁细胞中表达。AtCesA2和AtCesA5、AtCesA6和AtCesA1以及AtCesA9和AtCesA10等基因对均有基因重复作用。从进化树分析可知,AmCesA2和拟南芥的AtCesA3在结构上比较相似,根据织特异性表达结果显示,虽然AmCesA2在相思树的3个组织器官中均能检测到表达,但其组织器官特异性差异较显著,在茎中AmCesA2的表达量最高,而快速成长的幼苗的茎中最主要的成份是发育中的木质部,这意味着AmCesA2可能既参与初级细胞壁也参与次级细胞壁的形成。通过AmCesA2对激素的反应分析,发现在不同的激素处理下存在差异,GA3处理最为显著地提高AmCesA2的表达量,6-BA次之,GA3和6-BA均具有促进细胞伸长发育的作用,因此AmCesA2有可能加快细胞壁的形成。
最近有研究表明多种植物激素和转录因子参与纤维素合酶的调控。在水稻中GA信号途径通过多级转录因子调控次生壁纤维素合酶基因表达[7],在拟南芥中生长素是花药发育过程中次生壁合成的负调控因子[37]。在转录水平上, NAC转录因子可以激活一些与次生壁合成相关的纤维素合酶基因[38],拟南芥转录因子MYB46可直接调节三种次生壁纤维素合酶AtCesA4,AtCesA7和AtCesA8 [39]。水稻转录因子OsMYB58/63直接上调水稻次生壁特异性纤维素合酶基因OsCesA7的表达[7]。但纤维素合酶是否参与其他途径,还需进一步研究,未来课题组拟通过构建AmCesA2的正义表达载体并转化模式植物,研究该基因的信号转导途径并进行功能验证,为研究相思树纤维素合酶的功能提供更丰富的理论依据,并为育成高纤维率的马占相思良种提供帮助。
| [1] | KONDA R, OHTA S, ISHIZUKA S, et al. Seasonal changes in the spatial structures of N2O, CO2, and CH4 fluxes from Acacia mangium plantation soils in Indonesia[J]. Soil Biology and Biochemistry, 2010, 42(9): 1512–1522. DOI:10.1016/j.soilbio.2010.05.022 |
| [2] | LE MAIRE G, NOUVELLON Y, CHRISTINA M, et al. Tree and stand light use efficiencies over a full rotation of single- and mixed-species Eucalyptus grandis and Acacia mangium plantations[J]. Forest Ecology and Management, 2013, 288: 31–42. DOI:10.1016/j.foreco.2012.03.005 |
| [3] | HOONG Y B, PIZZI A, TAHIR P, et al. Characterization of Acacia mangium polyflavonoid tannins by MALDI-TOF mass spectrometry and CP-MAS 13C NMR[J]. European Polymer Journal, 2010, 46(6): 1268–1277. DOI:10.1016/j.eurpolymj.2010.03.002 |
| [4] | BINI D, DOS SANTOS C A, BOUILLET J P, et al. Eucalyptus grandis and Acacia mangium in monoculture and intercropped plantations:evolution of soil and litter microbial and chemical attributes during early stages of plant development[J]. Applied Soil Ecology, 2013, 63: 57–66. DOI:10.1016/j.apsoil.2012.09.012 |
| [5] | DANISH M, HASHIM R, IBRAHIM M N M, et al. Effect of acidic activating agents on surface area and surface functional groups of activated carbons produced from Acacia mangium wood[J]. Journal of Analytical and Applied Pyrolysis, 2013, 104: 418–425. DOI:10.1016/j.jaap.2013.06.003 |
| [6] | KEEGSTRA K. Plant cell walls[J]. Plant Physiology, 2010, 154(2): 483–486. |
| [7] | NODA S, KOSHIBA T, HATTORI T, et al. The expression of a rice secondary wall-specific cellulose synthase gene, OsCesA7, is directly regulated by a rice transcription factor, OsMYB58/63[J]. Planta, 2015, 242(3): 589–600. |
| [8] | KUMAR M, TURNER S. Plant cellulose synthesis:CESA proteins crossing kingdoms[J]. Phytochemistry, 2015, 112: 91–99. DOI:10.1016/j.phytochem.2014.07.009 |
| [9] | 袁红梅, 郭文栋, 赵丽娟, 等. 亚麻纤维素合酶超基因家族的生物信息学及表达分析[J]. 中国农业科学, 2016, 49(23): 4656–4668. DOI:10.3864/j.issn.0578-1752.2016.23.018 |
| [10] | 郭涛, 杨青川, 晁跃辉, 等. 植物纤维素合酶结构与基因表达研究进展[J]. 草地学报, 2016, 24(3): 501–508. |
| [11] | KAUR S, DHUGGA K S, GILL K, et al. Novel structural and functional motifs in cellulose synthase (CesA) genes of bread wheat (Triticum aestivum L[J]. PLoS One, 2016, 11(1): e0147046. DOI:10.1371/journal.pone.0147046 |
| [12] | 李秀云, 陈晓沛, 徐英武, 等. 毛竹生长过程中纤维素合成酶基因的时空表达和功能预测[J]. 浙江农林大学学报, 2017, 34(4): 565–573. |
| [13] | 高艳, 陈光辉, 陈秀娟, 等. 植物细胞壁纤维素生物合成的调控[J]. 生物技术通报, 2014(1): 1–7. |
| [14] | MCFARLANE H E, DÖRING A, PERSSON S. The cell biology of cellulose synthesis[J]. Annual Review of Plant Biology, 2014, 65: 69–94. DOI:10.1146/annurev-arplant-050213-040240 |
| [15] | WONG H C, FEAR A L, CALHOON R D, et al. Genetic organization of the cellulose synthase operon in Acetobacter xylinum[J]. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(20): 8130–8134. DOI:10.1073/pnas.87.20.8130 |
| [16] | PEAR J R, KAWAGOE Y, SCHRECKENGOST W E, et al. Higher plants contain homologs of the bacterial celA genes encoding the catalytic subunit of cellulose synthase[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996, 93(22): 12637–12642. DOI:10.1073/pnas.93.22.12637 |
| [17] | DJERBI S, LINDSKOG M, ARVESTAD L, et al. The genome sequence of black cottonwood (Populus trichocarpa) reveals 18 conserved cellulose synthase (CesA) genes[J]. Planta, 2005, 221(5): 739–746. DOI:10.1007/s00425-005-1498-4 |
| [18] | KRAUSKOPF E, HARRIS P J, PUTTERILL J. The cellulose synthase gene PrCESA10 is involved in cellulose biosynthesis in developing tracheids of the gymnosperm Pinus radiata[J]. Gene, 2005, 350(2): 107–116. DOI:10.1016/j.gene.2005.02.018 |
| [19] | RANIK M, MYBURG A A. Six new cellulose synthase genes from Eucalyptus are associated with primary and secondary cell wall biosynthesis[J]. Tree Physiology, 2006, 26(5): 545–556. |
| [20] | SONG D L, SHEN J H, LI L G. Characterization of cellulose synthase complexes in Populus xylem differentiation[J]. New Phytologist, 2010, 187(3): 777–790. DOI:10.1111/j.1469-8137.2010.03315.x |
| [21] | 张海平, 王学德, 邵明彦, 等. 外源纤维素合酶基因对棉纤维品质的改良作用[J]. 棉花学报, 2008, 20(2): 110–115. DOI:10.3969/j.issn.1002-7807.2008.02.006 |
| [22] | BAI W Q, XIAO Y H, ZHAO J, et al. Gibberellin overproduction promotes sucrose synthase expression and secondary cell wall deposition in cotton fibers[J]. PLoS One, 2014, 9(5): e96537.. DOI:10.1371/journal.pone.0096537 |
| [23] | 范玉龙, 姜达, 李明容, 等. 适合于高通量测序的槟榔总RNA的提取方法[J]. 热带生物学报, 2015, 6(1): 87–90. DOI:10.3969/j.issn.1674-7054.2015.01.013 |
| [24] | 魏凯莉, 周厚君, 江成, 等. 杨树次生壁纤维素合酶的表达与互作模式分析[J]. 林业科学研究, 2017, 30(2): 245–253. |
| [25] | XIE L Q, YANG C J, WANG X L. Brassinosteroids can regulate cellulose biosynthesis by controlling the expression of CESA genes in Arabidopsis[J]. Journal of Experimental Botany, 2011, 62(13): 4495–4506. DOI:10.1093/jxb/err164 |
| [26] | 张保才, 周奕华. 植物细胞壁形成机制的新进展[J]. 中国科学:生命科学, 2015, 45(6): 544–556. |
| [27] | 黄成, 李来庚. 植物细胞壁研究与生物质改造利用[J]. 科学通报, 2016, 61(34): 3623–3629. |
| [28] | 张凯权, 马履一, 段劼, 等. 刺槐纤维素合酶基因(RpCesA2)克隆、表达及SNP分析[J]. 分子植物育种, 2017, 15(2): 474–482. |
| [29] | SETHAPHONG L, DAVIS J K, SLABAUGH E, et al. Prediction of the structures of the plant-specific regions of vascular plant cellulose synthases and correlated functional analysis[J]. Cellulose, 2016, 23(1): 145–161. |
| [30] | 庞景, 童再康, 黄华宏, 等. 杉木纤维素合成酶基因CesA的克隆及表达分析[J]. 浙江农林大学学报, 2015, 32(1): 40–46. |
| [31] | SUZUKI S, LI L G, SUN Y H, et al. The cellulose synthase gene superfamily and biochemical functions of xylem-specific cellulose synthase-like genes in Populus trichocarpa[J]. Plant Physiology, 2006, 142(3): 1233–1245. |
| [32] | 陈亚娟, 王宏芝, 李瑞芬, 等. 毛白杨纤维素合酶基因家族部分成员的克隆及表达[J]. 林业科学, 2011, 47(10): 70–75. DOI:10.11707/j.1001-7488.20111011 |
| [33] | HUANG D B, WANG S G, ZHANG B C, et al. A gibberellin-mediated DELLA-NAC signaling cascade regulates cellulose synthesis in rice[J]. The Plant Cell, 2015, 27(6): 1681–1696. |
| [34] | 陈英, 邵志龙, 王浩然, 等. 杨树PeAFB基因克隆及表达模式初步分析[J]. 林业科学, 2015, 51(8): 26–32. |
| [35] | ROBERT S, MOUILLE G, HÖFTE H. The mechanism and regulation of cellulose synthesis in primary walls:lessons from cellulose-deficient Arabidopsis mutants[J]. Cellulose, 2004, 11(3/4): 351–364. DOI:10.1023/B:CELL.0000046415.45774.80 |
| [36] | 江海霞, 郭栋良, 李玉环, 等. 亚麻快速生长期细胞壁形成相关基因的表达分析[J]. 中国农业科学, 2017, 50(13): 2442–2450. DOI:10.3864/j.issn.0578-1752.2017.13.004 |
| [37] | CECCHETTI V, ALTAMURA M M, BRUNETTI P, et al. Auxin controls Arabidopsis anther dehiscence by regulating endothecium lignification and jasmonic acid biosynthesis[J]. The Plant Journal, 2013, 74(3): 411–422. DOI:10.1111/tpj.2013.74.issue-3 |
| [38] | YAMAGUCHI M, MITSUDA N, OHTANI M, et al. VASCULAR-RELATED NAC-DOMAIN 7 directly regulates the expression of a broad range of genes for xylem vessel formation[J]. The Plant Journal, 2011, 66(4): 579–590. DOI:10.1111/j.1365-313X.2011.04514.x |
| [39] | KIM W C, KO J H, KIM J Y, et al. MYB46 directly regulates the gene expression of secondary wall-associated cellulose synthases in Arabidopsis[J]. The Plant Journal, 2013, 73(1): 26–36. DOI:10.1111/j.1365-313x.2012.05124.x |

 2019, Vol. 39
2019, Vol. 39


