文章信息
- 欧阳乐军, 袁玉梅, 李莉梅, 陈剀钊, 戴发
- OUYANG Lejun, YUAN Yumei, LI Limei, CHEN Kaizhao, DAI Fa
- 巨桉miR156 CRISPR/Cas9载体构建
- Construction of Eucalyptus grandis miR156 family CRISPR/Cas9 vector
- 森林与环境学报,2018, 38(4): 488-493.
- Journal of Forest and Environment,2018, 38(4): 488-493.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2018.04.016
-
文章历史
- 收稿日期: 2018-03-16
- 修回日期: 2018-06-18
CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats)基因组定向编辑技术是近几年发展起来的对基因组进行定向精确修饰的一种技术。该系统可以对基因组中的靶位点进行缺失、敲入、核苷酸修正等操作, 是继转基因技术之后人类对生物进行遗传操作的又一革命性技术[1-3]。CRISPR/Cas9系统作为一种新型的基因编辑技术, 已经在人类细胞、小鼠、斑马鱼、酵母、细菌、果蝇、线虫以及拟南芥、水稻、烟草中进行了广泛的应用研究[4]。然而, 作为一种还在不断完善的基因组定点编辑技术, CRISPR/Cas9系统在植物中的应用还有许多需要改进的地方, 如单位点突变容易造成脱靶及后续突变体检测繁琐等[5]。目前, 多数CRISPR载体都只有1个sgRNA, 只能靶向基因的1个位点, 基因突变的效率并不高, 要想设计高效率的CRISPR/Cas9系统, 要考虑到该系统表达的时间, 空间以及效率等几个方面。多个sgRNA能靶向同一基因的不同位点, 提高基因突变的频率, 也能造成较大片段的缺失突变, 也利于突变后代的PCR方法筛选检测。另外, 多个sgRNA也能靶向不同的基因, 特别是同一信号通路、同一代谢途径或同一基因家族的基因, 在基础生命科学研究中具有重要的应用价值[6]。
桉树(Eucalyptus spp.)是世界公认的速生经济林树种, 全世界桉树人工林面积超过2 000万hm2。桉树在中国引种已有120多年历史, 全国共有17个省(区)种植, 每年木材产量超过4 000万m3, 提供全国50%以上纸浆用材原材料, 是维护我国木材安全的重要造林树种[7]。巨桉(Eucalyptus grandis)具有树干通直、适应性广、耐寒性强、速生丰产等优点, 是理想的工业与实木用材原料林树种, 已在广东、广西、海南及福建等省(区)大面积栽培, 是我国栽培面积较大的一种桉树树种。利用CRISPR技术开展桉树相关基因功能研究对于桉树遗传改良以及研究体细胞再生机理具有十分重要的意义[8]。近年来, miRNA已成为生物学领域研究的一大热点[9], 巨桉miR156具有高度保守的结构域, 在植物的营养生长和生殖生长阶段具有重要的调控作用, miR156通过它的靶基因SPL转录因子调控植物的阶段发育[10]。以miR156为目标基因, pHDE/Cas9为基础载体, 通过聚合酶链式反应(polymerase chain reaction, PCR)同源重组方法, 构建了带有筛选标记的巨桉miR156家族CRISPR/Cas9载体系统, 可实现目标基因的大片段编辑, 为研究巨桉miR156功能奠定基础。
1 材料与试剂 1.1 菌种与质粒大肠杆菌感受态细胞DH5α购自鼎国生物广州公司, pHDE-mcherry质粒及pHDE/Cas9质粒由加州大学圣地亚哥分校Professor ZHAO赠送。
1.2 主要试剂卡那霉素、DNA Loading Buffer购自上海生工生物工程股份有限公司, Epicentre T5 exonuclease (T5外切酶)、NEB Phusion DNA polymerase (DNA聚合酶)、NEB Taq DNA Ligase (40 U·μL-1) (DNA连接酶)和BsaI限制性内切酶购自New England Biolabs公司(美国), 100 mmol·L-1二硫苏糖醇(dithiothreitol, DTT)购自碧云天生物技术公司(北京), Tiangen DNA marker购自天根生化(北京)科技有限公司。
1.3 研究方法运用SnapGene软件进行载体构建分析, 运用Primer Premier 5软件进行引物分析; 运用Dnastar软件进行序列分析。
2 试验方法 2.1 巨桉miR156序列分析及靶位点设计先用拟南芥miR156的mRNA序列比对Genebank巨桉所有的miRNA序列, 从而确定巨桉miR156的序列。根据Crisprscan找到目的基因的高评分靶点。利用Rgenome评估高评分靶点的脱靶情况。以巨桉miR156家族基因为靶基因, 选择靠近5′端得分最高的G-N19-NGG 23 bp序列, 其中第一个G是小RNA转录的起始信号位点, NGG是Cas9基因定位的PAM序列, 需要插入gRNA载体的是G-N19共20 bp序列, 共设计两个打靶位点的Target。
2.2 引物设计根据低脱靶、高评分的靶点设计引物。试验所有的PCR引物均由上海生工生物工程股份有限公司合成, 引物序列见表 1。
| 引物名称 Primer name |
引物序列 Primer sequence | 扩增长度 Length/bp |
| miR156A-F | CTAGAGTCGAAGTAGTGATTGAAGAGCACGATGGAGAAGCGTTTTAGAGCTAGAAATAGC | 634 |
| miR156A-R | TGCTATTTCTAGCTCTAAAACTGAGTGGGTTCTTCATGTGCAATCTCTTAGTCGACTCTA | 634 |
| miR156B-F | CTAGAGTCGAAGTAGTGATTGCTACCCCGTGGTTGATAATGTTTTAGAGCTAGAAATAGC | 634 |
| miR156B-R | TGCTATTTCTAGCTCTAAAACATATCGCGTGCCATTGTCACAATCTCTTAGTCGACTCTA | 634 |
| miR156C-F | CTAGAGTCGAAGTAGTGATTGAGAGAAGAGAAGCTATTTAGTTTTAGAGCTAGAAATAGC | 634 |
| miR156C-R | TGCTATTTCTAGCTCTAAAACGACCAATATCACGCCATGTCAATCTCTTAGTCGACTCTA | 634 |
| 35S-F | TAATGCATTTTATGACTTGCAACATGGTGGAGCACGACAC | 452 |
| 35S-R | CCAAGAGCTCCGTGTCCTCTCCAAATGAAA | 452 |
| Mch-F | AGAGGACACGGAGCTCTTGGACTCCCATGT | 293 |
| Mch-R | GAATTCGTTGTCAATCAATTAATACGATAATTTATTTGAA | 293 |
以pHDE-mcherry质粒为模板, Mch-F和Mch-R为引物PCR扩增mcherry基因, 扩增体系(50 μL); 以pHDE质粒为模板, 35S-F和35S-R为引物PCR扩增35S启动子基因, 扩增体系(50 μL)。PCR结束后进行电泳分析, 确定目的条带后, 切胶回收目的条带。
2.3.2 PCR扩增35S启动子和mcherry全长片段以第一轮PCR回收的两条目的片段共同为模板, 35S-F和Mch-R为引物进行重叠PCR, PCR扩增体系(50 μL)。PCR完成后电泳, 将符合目的片段大小的条带割胶回收, 命名为“35S+Mch”。
2.3.3 35S+Mch与载体pHDE/Cas9的重组连接用MfeⅠ酶切pHDE/Cas9质粒, 酶切体系(20 μL)包括3.0 μL的pHDE-Cas9、2.0 μL的CutSmart、0.4 μL的MfeⅠ和14.6 μL的ddH2O, 反应条件为37 ℃, 60 min。
采用同源重组技术, 将回收的35S+Mch片段与经MfeⅠ酶切线性化的载体pHDE/Cas9连接, 连接组装反应体系:50 ℃反应1 h, 热激法转化DH5α感受态细胞。同源重组体系包括0.4 μL的pHDE/Cas9、浓度为20 ng· μL-1的PCR Fragment 5.0 μL, 重组酶Recombinase 3.0 μL。
2.3.4 重组子pHDE-mcherry转化后的菌液PCR鉴定将重组产物pHDE-mcherry利用热激法转化DH5α感受态大肠杆菌, 抗性培养12 h后, 挑单菌落摇床培养3~4 h, 菌液PCR鉴定, 用0.5%琼脂糖凝胶电泳分析。PCR反应条件为:94 ℃ 3 min, 30个循环(94 ℃ 30 s, 55 ℃30 s, 72 ℃ 45 s), 72 ℃ 2 min。
2.3.5 重组质粒pHDE-mcherry提取及PCR鉴定选取阳性克隆在含50 mg·mL-1的卡那霉素30 μL和30 mL液体LB培养基中, 37 ℃摇床过夜培养。提取质粒并进行质粒PCR扩增, 1%浓度的琼脂糖凝胶电泳鉴定PCR产物。
2.3.6 重组质粒pHDE-mcherry送样测序取经PCR鉴定阳性的质粒pHDE-mcherry送至上海生工生物工程股份有限公司广州分公司测序。
2.4 表达载体pHDE-mcherry-miR156构建 2.4.1 PCR扩增miR156-gRNA由含gRNA的pCBC质粒作模板, 用针对相应编码区的特异引物进行PCR扩增相应片段, PCR反应条件:95 ℃ 30 min, 30个循环(95 ℃ 30 s, 55 ℃ 30 s, 72 ℃ 45 s), 72 ℃ 2 min, 16 ℃ 2 min。1.5%琼脂糖凝胶电泳PCR产物, 切胶回收目的条带, -20 ℃冷冻20 min, 离心半径8 cm, 15 000 r·min-1低温离心20 min, 吸取上清液于PCR小管中用于后续与载体连接。
2.4.2 pHDE-mcherry质粒载体酶切及重组连接用BsaI酶切2.3.6中测序正确的pHDE-mcherry载体, 酶切体系体积20 μL, 50 ℃恒温过夜。以酶切前的pHDE质粒作对照, 配制0.5%的琼脂糖凝胶进行电泳, 检测酶切效果。
采用同源重组技术, 将回收纯化后的miR156A-SgRNA、miR156B-SgRNA和miR156C-SgRNA片段与经BsaI酶切线性化的载体pHDE-mcherry连接, 连接组装反应体系:50 ℃反应1 h, 热激法转化DH5α感受态细胞。
2.4.3 重组子转化后的菌液PCR鉴定将重组产物利用热激法转化大肠杆菌DH5α感受态细胞, 抗性培养12 h后, 挑单菌落培养3~4 h, 菌液PCR鉴定分析。PCR反应条件同2.4.1。
2.4.4 重组质粒pHDE-miR156提取及PCR鉴定选取阳性克隆菌液在含50 mg·mL-1的卡那霉素30 μL和30 mL液体LB培养基中, 37 ℃摇床过夜培养。提取质粒后进行质粒PCR鉴定, 1%浓度的琼脂糖凝胶电泳鉴定PCR产物。
2.4.5 重组质粒pHDE-miR156送样测序取经PCR鉴定为阳性的重组质粒pHDE-miR156送至上海生工生物工程股份有限公司广州分公司测序。
3 结果与分析 3.1 pHDE-mcherry载体构建分别以pHDE-mcherry质粒为模板, PCR扩增mcherry和35S启动子基因。其中mcherry约300 bp, 35S启动子约450 bp。将PCR产物电泳, 目的片段的PCR扩增结果(图 1)都出现目的片段条带, 其条带大小都与预期大小一致。将第一轮PCR产物割胶回收后作为模板进行重叠PCR, PCR产物电泳结果为一条约720 bp的条带(图 2), 与35S+Mch大小相符。
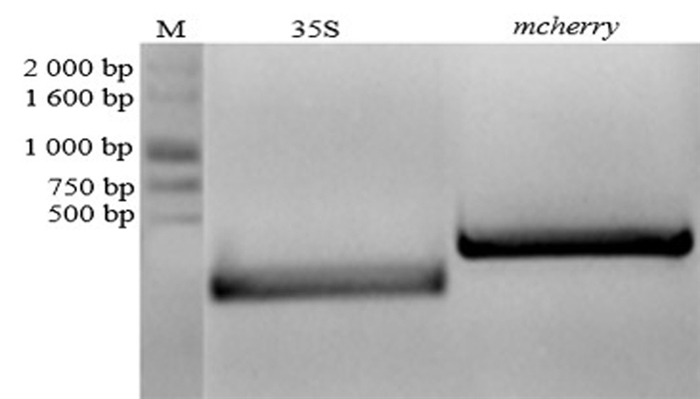
|
图 1 mcherry和35S片段的PCR结果 Fig. 1 PCR product of mcherry and 35S 注:M为DNA marker。 Note:M is DNA marker. |

|
图 2 35S+Mch的PCR结果 Fig. 2 PCR product of 35S+Mch 注:M为DNA marker。 Note:M is DNA marker. |
将扩增得到的目的片段35S+Mch, 与MfeⅠ酶切的pHDE质粒经重组酶同源重组后的转化感受态细胞, 涂布于卡那抗性的平板进行转化筛选, 待平板上出现菌落后随机挑取4个单菌落液体培养, 然后以培养的菌液为模板、35S-F和Mch-R为引物做菌液PCR检测, pHDE/Cas9载体线性化及重组转化结果(图 3)显示, E1号和E4号菌液PCR检测为阳性, 目的条带大小与预期一致。
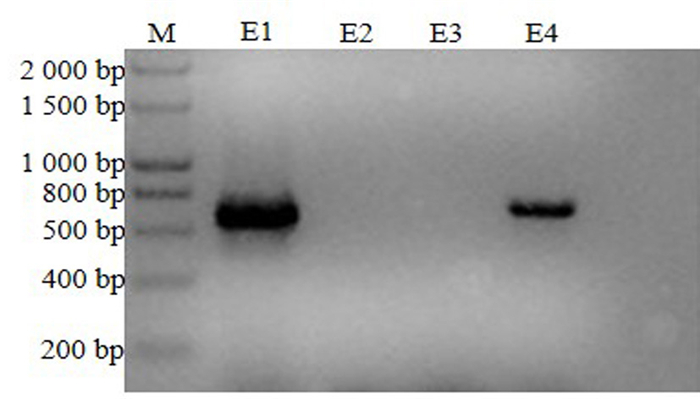
|
图 3 重组质粒pHDE-35S-mcherry菌液PCR检测结果 Fig. 3 Bacteria solution PCR product of recombinant plasmid pHDE-35S-mcherry M为DNA marker; E1~E3为重组质粒转化菌液; E4为阳性对照。 Note:M is DNA marker; E1-E3 are recombinant plasmid; E4 is positive control. |
重组质粒PCR鉴定结果(图 4)中均有清晰条带, 且条带大小在500~800 bp之间, 与目的片段35S+Mch条带(约720 bp)大小符合。重组质粒测序结果利用DNAStar软件进行序列分析, 测序峰图对应的序列无异常, 重组载体pHDE-mcherry的测序结果显示目的片段成功插入, 无一碱基发生突变, 与预期序列完全一致。
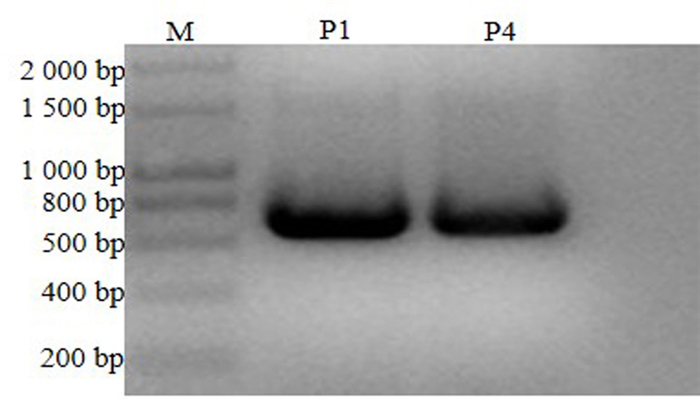
|
图 4 pHDE-35S-mcherry质粒PCR验证 Fig. 4 PCR product of recombinant plasmid pHDE-35S-mcherry 注:M为DNA marker; P1、P4为重组质粒PCR。 Note:M is DNA marker; P1, P4 are PCR product of recombinant plasmid. |
利用特异引物进行PCR扩增miR156A-gRNA、miR156B-gRNA和miR156C-gRNA, PCR扩增miR156-gRNA目的片段结果如图 5所示, 扩增的片段位于750~500 bp之间与预期条带位置大小(634 bp)一致, PCR扩增片段大小与预期相符。
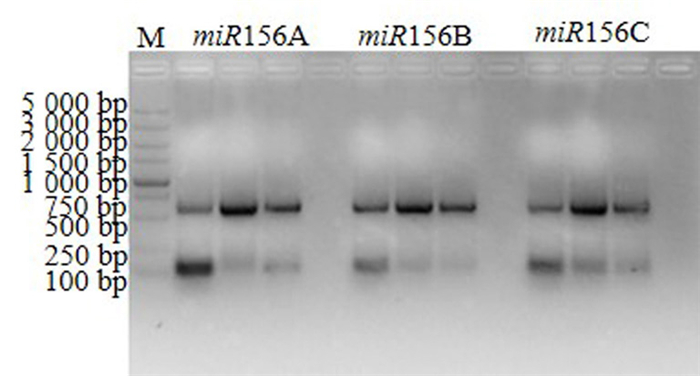
|
图 5 PCR扩增miR156-gRNA Fig. 5 PCR product of miR156-gRNA M为DNA marker。 Note:M is DNA marker. |
Bsa1酶切pHDE-mcherry后分别与miR156A-gRNA、miR156B-gRNA和miR156C-gRNA经同源重组酶连接组装成3种重组质粒, 转化感受态细胞后PCR鉴定结果见图 6, PCR扩增出预期的634 bp大小的条带, 阳性率100%。
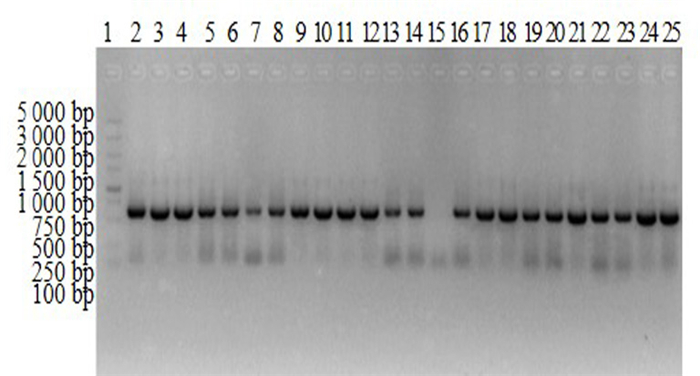
|
图 6 PCR扩增pHDE-mcherry-miR156 Fig. 6 PCR product of pHDE-mcherry-miR156 注:1为5 000 bp marker; 2~25为重组质粒。 Note:1 is 5 000 bp marker; 2-25 are recombinant plasmid. |
用碱裂解法提取上述菌液PCR结果为阳性的重组质粒, 进行质粒PCR。PCR电泳结果见图 7, 质粒PCR均能扩增出预期的634 bp大小相符的条带, 初步判定载体构建成功。
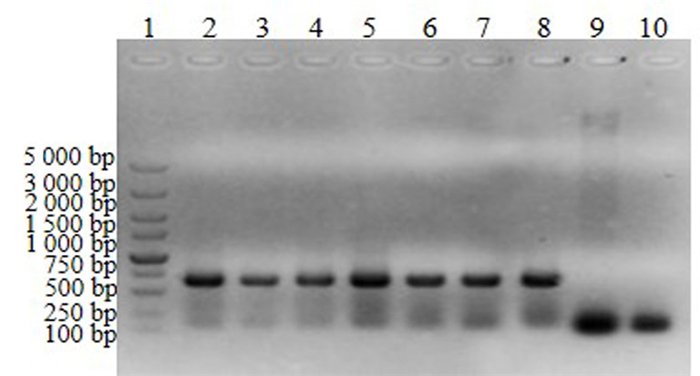
|
图 7 pHDE-mcherry-miR156质粒PCR结果 Fig. 7 PCR product of recombinant plasmid pHDE-mcherry-miR156 注:1为5 000 bp marker; 2~7为重组质粒; 8为阳性对照; 9~10为阴性对照。 Note:1 is 5 000 bp marker, 2-7 are recombinant plasmid; 8 is positive control; 9-10 are negative control. |
将重组质粒pHDE-miR156克隆提取的重组质粒进行电泳, 除含有少量RNA外, 无杂带出现, 3种重组质粒分别挑选一个阳性克隆送样测序。对重组质粒pHDE-mcherry-miR156A、pHDE-mcherry-miR156B和pHDE-mcherry-miR156C进行序列分析。测序峰图均显示设计的miR156有两靶点, 序列分析miR156 standard Seq与测序结果一致, 测序峰图对应的序列无异常, 表明重组质粒构建成功。
4 讨论与结论 4.1 CRISPR/Cas9技术在植物miRNA基因功能研究中的应用CRISPR/Cas9是锌指核酸内切酶(zinc finger nuclease, ZFN)、类转录激活因子效应物核酸酶(transcription activator like effector nucleases, TALEN)之后出现的第3代基因组定点编辑技术, 具备成本低廉, 操作简单, 效率高等优点。miRNAs是由20~24个碱基构成的具有保守结构域的非编码RNA。植物miRNA虽然具有高度保守的结构域, 但其对植物的生长发育各阶段具有重要的调控作用[11]。不同的miRNAs家族对植物生长发育具有不同的调控作用, 随着miRNAs研究的兴起, 越来越多的miRNAs家族在各种物种中被发现, 已有研究表明, miRNAs参与调控叶形态发育[12]、花器官的发育[13], 以及植物对非生物胁迫的响应[14]等。
开展植物的miRNA的功能研究可有效地改善植物的生长、发育进程。随着研究的深入, 越来越多的植物miRNA生物功能被验证, 如miR156与其靶基因SPLs所控制的营养生长阶段转变, 进而调节miR172, 然后miR172调节成花关键基因FT的表达。更多的研究表明, miR156与植物某些重要的性状调控有关, 同时在植物体胚发生中具有重要的调控机制[15]。已有研究表明, miR156通过切割其靶基因SPLs基因的mRNA或通过翻译抑制来调控SPLs表达, 对其生物学功能的研究已成为研究热点, 但关于miR156上游调控网络的研究尚处于起步阶段[16]。本课题组成功构建了miR156的CRISPR基因编辑载体, 为进一步研究巨桉miR156参与桉树体胚发生的分子机制奠定了一定的基础。目前, 尚未有利用CRISPR/Cas9研究桉树miRNA生物功能的报道。下阶段, 可将上述重组质粒转化农杆菌, 对巨桉外植体进行浸染, 验证转化后代中miR156的基因编辑情况, 对miR156的突变体后代进行相关研究, 以确定miR156在巨桉体胚发生过程中的功能。
4.2 可视化筛选的大片段编辑CRISPR体系的应用优势目前, CRISPR/Cas9系统在植物中的应用存在表达活性不高以及单位点基因编辑后代的酶切检测繁琐等问题。要想设计高效率的CRISPR/Cas9系统, 要考虑到该系统表达的时间、空间以及效率等几个方面[17-18]。一个miRNA序列设计两个Target靶序列, 可实现大片段突变, 减少了后续酶切鉴定突变后代的繁琐, 同时也提高了目标基因敲除的效率[19]。mcherry是一种来自于蘑菇珊瑚的红色荧光蛋白, 常用于标记和示踪某些分子和细胞组分。GAO et al[20]将在种皮中表达的At2S3启动子与mcherry融合在一起, 利用种皮可视筛选标记MCHERRY蛋白来进行筛选突变后代, 极大的提高了工作效率。通过35S启动红色荧光标记基因mcherry的表达, 可实现在植物转化早期中的可视化筛选, 提高基因编辑效率。
| [1] | CONG L, RAN F A, COX D, et al. Multiplex genome engineering using Crispr/Cas systems[J]. Science, 2013, 339(6 121): 819–823. |
| [2] | FENG Z Y, MAO Y F, XU N F, et al. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(12): 4 632–4637. DOI:10.1073/pnas.1400822111 |
| [3] | ZONG Y, WANG Y P, LI C, et al. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion[J]. Nature Biotechnology, 2017, 35(5): 438–440. DOI:10.1038/nbt.3811 |
| [4] | MAO Y F, BOTELLA J R, ZHU J K. Heritability of targeted gene modifications induced by plant-optimized CRISPR systems[J]. Cellular and Molecular Life Sciences, 2017, 76(4): 1 075–1093. |
| [5] | 刘丁源, 邱婷, 丁晓辉, 等. 快速构建多重sgRNA载体利用CRISPR/Cas9技术敲除拟南芥IAA2基因[J]. 遗传, 2016, 38(8): 756–764. |
| [6] | ZHANG C, MENG X H, WEI X L, et al. Highly efficient CRISPR mutagenesis by microhomology-mediated end joining in Aspergillus fumigatus[J]. Fungal Genetics and Biology, 2016, 86: 47–57. DOI:10.1016/j.fgb.2015.12.007 |
| [7] | OUYANG L J, LI L M. Effects of an inducible aiiA gene on disease resistance in Eucalyptus urophylla×Eucalyptus grandis[J]. Transgenic Research, 2016, 25(8): 441–452. |
| [8] | 杨民胜, 吴志化, 张维耀. 我国环北部湾地区发展桉树的思考[J]. 世界林业研究, 2016, 29(6): 64–69. |
| [9] | YAMAGUCHI A, WU M F, YANG L, et al. The microRNA-regulated SBP-box transcription factor SPL3 is a direct upstream activator of LEAFY, FRUITFULL, and APETALA1[J]. Developmental Cell, 2009, 17(2): 268–278. DOI:10.1016/j.devcel.2009.06.007 |
| [10] | CUI L G, SHAN J X, SHI M, et al. The miR156-SPL9-DFR pathway coordinates the relationship between development and abiotic stress tolerance in plants[J]. The Plant Journal, 2014, 80(6): 1 108–1117. DOI:10.1111/tpj.12712 |
| [11] | AXTELL M J, BARTEL D P. Antiquity of microRNAs and their targets in land plants[J]. The Plant Cell, 2005, 17(6): 1 658–1673. DOI:10.1105/tpc.105.032185 |
| [12] | MALLORY A C, REINHART B J, JONES-RHOADES M W, et al. MicroRNA control of PHABULOSA in leaf development:importance of pairing to the microRNA5, region[J]. The EMBO Journal, 2004, 23(16): 3 356–3364. DOI:10.1038/sj.emboj.7600340 |
| [13] | CHEN X M. A microRNA as a translational repressor of APETALA2 in Arabidopsis flower development[J]. Science, 2004, 303(5 666): 2022–2025. |
| [14] | SUNKAR R, ZHU J K. Novel and stress-regulated microRNAs and other small RNAs from Arabidopsis[J]. The Plant Cell, 2004, 16(8): 2 001–2019. DOI:10.1105/tpc.104.022830 |
| [15] | GUO C K, XU Y M, SHI A M, et al. Repression of miR156 by miR159 regulates the timing of the juvenile-to-adult transition in Arabidopsis[J]. The Plant Cell, 2017, 29(6): 1 293–1304. |
| [16] | LAUTER N, KAMPANI A, CARLSON S, et al. MicroRNA172 down-regulates glossy15 to promote vegetative phase change in maize[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(26): 9 412–9 417. DOI:10.1073/pnas.0503927102 |
| [17] | 段美艳, 李安宁, 赵志东, 等. 牛ATP5B基因启动子双荧光素酶报告基因载体构建与活性检测[J]. 西北农林科技大学学报(自然科学版), 2015, 43(8): 39–45. |
| [18] | 陈潇潇, 曹光球, 汪凤林, 等. 杉木纤维素合酶(ClCesA2)基因的克隆与表达分析[J]. 森林与环境学报, 2018, 38(1): 1–6. |
| [19] | ZHANG Z J, MAO Y F, HA S, et al. A multiplex CRISPR/Cas9 platform for fast and efficient editing of multiple genes in Arabidopsis[J]. Plant Cell Reports, 2016, 35(7): 1519–1533. DOI:10.1007/s00299-015-1900-z |
| [20] | GAO X H, CHEN J L, DAI X H, et al. An effective strategy for reliably isolating heritable and Cas9-free arabidopsis mutants generated by CRISPR/Cas9-mediated genome editing[J]. Plant Physiology, 2016, 171(3): 1794–1800. DOI:10.1104/pp.16.00663 |

 2018, Vol. 38
2018, Vol. 38
