文章信息
- 吕晓杰, 潘德灼, 李健, 陈世品, 陈伟
- LÜ Xiaojie, PAN Dezhuo, LI Jian, CHEN Shipin, CHEN Wei
- 油茶种仁成熟过程油脂合成代谢的转录组分析
- Transcriptomic analysis of lipid synthesis metabolism in Camellia oleifera seed kernel during its maturation period
- 森林与环境学报,2018, 38(2): 156-163.
- Journal of Forest and Environment,2018, 38(2): 156-163.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2018.02.005
-
文章历史
- 收稿日期: 2017-12-05
- 修回日期: 2018-02-01
2. 福建农林大学林学院, 福建 福州 350002
2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
油茶(Camellia oleifera Abel.)属山茶科(Theaceae)山茶属植物,是我国南方特有的木本油料树种[1]。油茶种子所压榨出的茶油是一种优质食用油,具有重要的保健功能,被誉为“东方橄榄油” [2]。随着人们生活水平的提高,对于优质食用油的需求越来越大,但是目前油茶面临的主要问题是油茶籽含油率不高,种仁含油率约40%,提高油茶籽的含油率是急需解决的问题[3]。由于油茶种子种仁的主要成分是油脂,因此,对其种仁的油脂合成代谢的研究对于提高油茶籽含油量具有重要意义。在油料作物中,参与油脂合成代谢的关键酶基因主要有乙酰辅酶A羧化酶基因(ACCase)、脂肪酸合酶基因(FAS)、脂肪酸脱饱和酶基因、二酰甘油乙酰转移酶基因(DGAT)等[4]。在拟南芥[5]、花生[6]、蓖麻[7]、麻疯树[8]、油桐[9]等油料作物已有研究中发现,参与油脂合成代谢的关键酶基因在油脂合成代谢中具有重要作用。
目前,与油茶相关的传统研究主要集中在茶油性状的分析及良种选育上[10-11]。近年来,对油茶生理代谢机制方面的研究有所进展。谭晓风等[12]首先以优良无性系油茶种仁为材料建立了油茶的cDNA文库。有研究发现油茶硬脂酰-酰基载体蛋白脱饱和酶基因(SAD)在进化上较为保守,与其他植物SAD的相似性较高,且比普通植物更有利于将饱和脂肪酸转化为油酸[13],油茶脂肪酸脱饱和酶基因2(FAD2)对于茶油脂肪酸组成具有关键调控作用[14],油茶种仁果糖-1,6-二磷酸醛缩酶基因4(CoFBA4)表达丰度与出油率呈显著正相关[15]。这些发现为研究油茶油脂代谢分子机制提供了一定的参考,但是油茶缺乏基因组信息,这对深入研究油茶基因的功能造成了一定困难。
转录组测序技术可以为研究油茶基因功能提供大量转录本信息,从中发掘重要基因进而分析功能。近年来,围绕油茶转录组的研究越来越多,江南等[16]利用RNA-Seq技术发现PLA2,FAD2,FAD3调控油茶种子发育过程中α-亚麻酸的合成。林萍等[17]对油茶种仁4个发育时期进行了转录组分析,并初步阐明了表达基因的种类和数量以及相关代谢通路。ZENG et al[18]通过转录组分析发现CoFBA和CoSAD的mRNA水平与油含量以及CoFAD2的基因表达水平与脂肪酸组成密切相关。但是针对油脂合成代谢的分子调控机制尚不明确,对福建省审定的油茶优良无性系品种“闽43”的转录组研究未见报道。为了进一步解析油茶油脂代谢调控机制,应用高通量转录组测序技术,根据代谢通路富集性分析表达差异的基因,对“闽43”油茶籽成熟过程中油脂代谢相关表达差异基因进行分析,探讨油脂合成代谢分子机制,研究结果可为阐明油茶油脂代谢分子机制、高含油量油茶分子育种工作提供理论依据。
1 材料与方法 1.1 材料准备与处理试验所用材料为福建省主要的栽培品种“闽43”油茶,为1985年筛选出,并在2008年通过福建省林木品种审定的优良无性系[19]。油茶果采集于福建省福州市闽侯桐口国有林场,北纬26°05′,东经119°17′,海拔186 m。霜降节气开始采集油茶果,采集时间分别为2015年10月24日(P1期)、11月7日(P2期)和11月21日(P3期)的3个时期,分别对应于“闽43”油茶果的成熟前期、成熟期和成熟后期。选择树龄为19 a、生长状态良好的油茶树9棵,每3棵树作为1个生物学重复,在每棵树的东南西北4个方位的中冠层分别采摘5粒形状大小相近、无病虫害的油茶果,每个生物学重复共计60粒油茶果。剥取油茶果的种仁,迅速置于液氮中冷冻后于-80 ℃冰箱储存,用于后续的转录组测序和RT-qPCR验证。新鲜种仁用于油茶含油量等生理指标的测定。
1.2 RNA提取与转录组测序将每个时期3次生物学重复的油茶种仁充分混匀于1个样品池,应用E.Z.N.A.® Plant RNA Kit提取试剂盒(Omega Bio-Tek公司,美国)提取每个样品池的总RNA。待总RNA质量检测合格后,构建cDNA文库。文库构建完成后,委托北京百迈克生物科技有限公司进行高通量转录组测序。
1.3 生物信息学分析 1.3.1 原始数据的处理应用Illumina Hiseq高通量测序平台对cDNA文库进行分析,产出大量Reads,即原始数据(raw data),其大部分Reads的质量达到或超过Q30。对原始数据进行过滤,去除带测序接头和引物序列的Reads获得的Reads即为纯净数据(clean data)。
1.3.2 序列的组装因油茶无参考基因组,应用Trinity软件对clean data进行组装[20]。首先将高质量的Reads打断为较短片段(K-mer),将这些小片段延伸成较长片段(contig),利用这些片段之间的重叠,得到片段集合(component),构建De Bruijin图,识别各个片段集合中的转录本序列,得到基因数据库(unigene)。
1.3.3 基因数据库功能注释和差异表达分析应用BLAST软件将基因数据库序列与蛋白质数据库NR(non-redundant)、KEGG (kyoto encyclopedia of genes and genomes)、GO(gene ontology)进行序列比对,得到unigene的注释信息[21-24]。应用FPKM(Fragments per kilobase of transcript per million mapped reads)方法计算unigene的表达丰度[25]。以FDR(false discovery Rate)<0.01且差异倍数(fold change)≥2作为筛选标准,获得3个时期表达差异的unigene。对差异表达的unigene进行KEGG和GO功能注释和分类。
1.4 差异基因表达验证应用MiniBEST Plant RNA Extraction Kit(TAKARA公司,日本)提取每个时期每个生物学重复样品的总RNA,参照TaKaRa Prime ScriptTMRT Reagent Kit with gDNA Eraser说明书合成cDNA。根据油茶转录组数据库应用Primer Premier 5.0软件设计引物,以GAPDH作为内参基因,所需的引物见表 1。参照TAKARASYBR Premix Ex TaqTM(TliRNaseH Plus)试剂盒说明书进行RT-qPCR分析,采用2-ΔΔCt法计算基因的差异倍数。
| 基因Unigene | 注释Description | 基因名称Gene name | 引物序列(5′-3′)Primer sequence |
| c135759.graph_c0 | glyceraldehyde 3-phosphate dehydrogenase | GAPDH | F:AGGTGGAAGAGCGGCCTCAT R:GGTCGCTTCTTTCTCTAGTC |
| c125388.graph_c0 | acetyl Co-enzyme a carboxyltransferase alpha subunit | ACCaseα-CT | F:CGCCCTACAAATCATCC R:TTTCCCGAGCCTTCTTC |
| c98815.graph_c0 | 3-ketoacyl-CoA synthase Ⅲ | KASⅢ | F:GCTGGGTCGCTAACATTC R:TCCCTACCTCCGCACTAA |
| c118582.graph_c0 | long chain acyl-CoA synthetase 2 | LACS2 | F:TGGACTCGCTCTGGGACTT R:CCACGGCATTTGAACC |
| c113232.graph_c0 | glycerol-3-phosphate 2-O-acyltransferase 4 | GPAT4 | F:GTCTCCTGCGTCACTTACA R:ACGGGCACAATCCTATC |
参考中华人民共和国国家标准GB/T 14488.1—2008《植物油料含油量测定》中的方法,对烘干的油茶种仁样品进行含油量的测定[26]。
2 结果与分析 2.1 测序数据统计经过测序质量控制,共得到24.00 Gb Clean Data,各样品Q30碱基百分比均不小于95.10%,GC含量均不小于47.28%,表明转录组测序的质量较好,可以用于后续分析研究。组装共得到123 157条Transcript和75 940条非冗余Unigene序列,对其进行长度统计分析(表 2),N50分别为1 363和1 119 bp,组装完整性较高,共有38 811条Unigene序列能够与公共数据库中的已知基因匹配,占所有非冗余基因的51.11%。
| 转录本长度 Length range/bp |
重叠群Contig | 转录本Transcript | 基因Unigene | |||||
| 数量Number | 占比Percent/% | 数量Number | 占比Percent/% | 数量Number | 占比Percent/% | |||
| 200~300 | 13 516 170 | 99.31 | 21 266 | 17.27 | 17 245 | 22.71 | ||
| 300~500 | 47 569 | 0.35 | 30 319 | 24.62 | 21 383 | 28.16 | ||
| 500~1 000 | 28 475 | 0.21 | 34 134 | 27.72 | 20 397 | 26.86 | ||
| 1 000~2 000 | 12 491 | 0.09 | 25 499 | 20.70 | 11748 | 15.47 | ||
| >2 000 | 5 086 | 0.04 | 11 939 | 9.69 | 5 167 | 6.80 | ||
| 合计Total number | 13 609 791 | 100 | 123 157 | 100 | 75 940 | 100 | ||
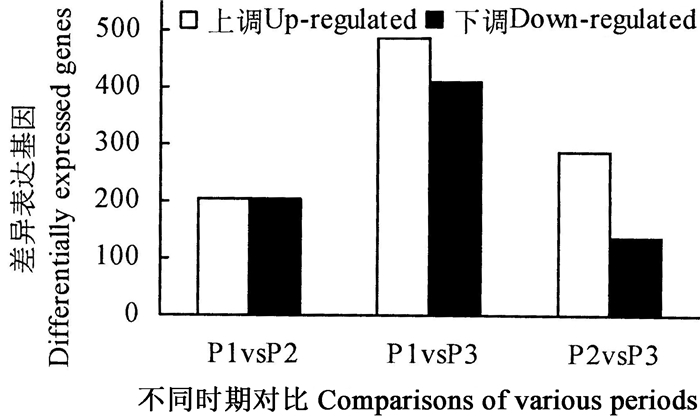
对P1、P2和P3的差异基因进行两两比较,由图 1显示,在P1 vs P2、P1 vs P3和P2 vs P3中分别有204、486和286条上调表达的基因,分别有203、407和137条下调表达的基因。P1到P3过程差异基因数目最多,说明成熟期前后,有较多差异基因表达。
|
图 1 油茶种仁3个发育时期差异表达基因 Fig. 1 Differentially expressed genes of C. oleifera seed kernels at 3 periods |
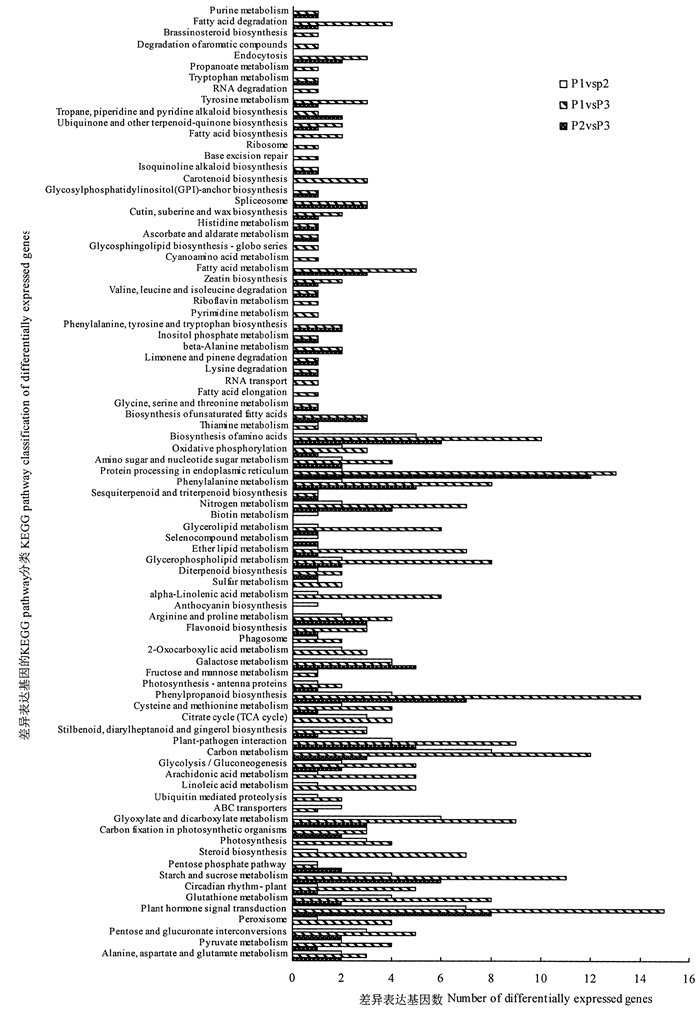
在KEGG数据库中,根据代谢通路分析,油茶种仁3个发育时期中,与脂肪酸和油脂代谢相关的途径中,P1和P2阶段过程中,差异基因有富集到甘油磷脂代谢途径;P1和P3阶段过程中,差异有富集到甘油磷脂代谢途径、脂肪酸代谢、脂肪酸合成、甘油酯代谢、不饱和脂肪酸、α-亚麻酸代谢、亚油酸代谢和花生四烯酸代谢;P2和P3阶段过程中,富集到甘油磷脂代谢途径、不饱和脂肪酸代谢、脂肪酸代谢(图 3)。Pathway富集性分析发现,与脂肪酸和甘油三酯代谢相关的4个基因乙酰辅酶A羧化酶羧基转移酶α亚基基因(ACCase α-CT)、β酮脂酰合酶基因(KASⅢ)、长链酰基辅酶A合酶基因2( LACS2)和甘油三磷酸酰基转移酶基因4(GPAT4)在3个时期均有较高表达。
|
图 2 油茶种仁转录组的差异表达基因的KEGG pathway分类 Fig. 2 KEGG pathway classification of differentially expressed genes of C. oleifera seed kernel |
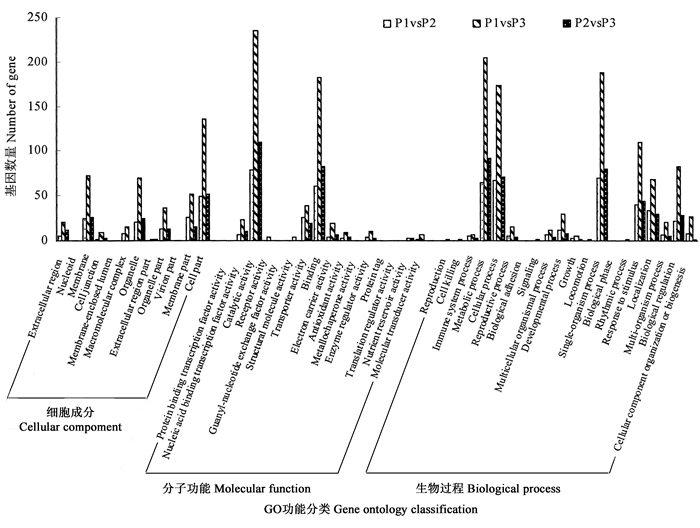
基于GO功能分类,油茶种仁3个发育时期的差异表达基因主要参与了20个生物过程,主要定位于12个细胞组分,主要涉及16个分子功能(图 3)。通过GO分类分析,发现富集到生物进程的差异基因最多,其中大部分的差异基因参与到了代谢过程和细胞过程。在P1到P2、P2到P3时期,差异基因主要参与了13个生物过程,定位于8个组分,涉及8个分子功能;P1到P3时期,差异基因参与18个生物过程,定位于9个细胞组分,涉及11个分子功能。其中,在细胞组分中,差异基因定位最多的是细胞部分,其次是细胞膜和细胞器;在分子功能中,差异基因涉及最多的是催化活性,其次是结合和转运体活性。
|
图 3 油茶种仁转录组的差异基因的GO功能分类 Fig. 3 Gene ontology classification of differentially expressed genes of C. oleifera seed kernel |
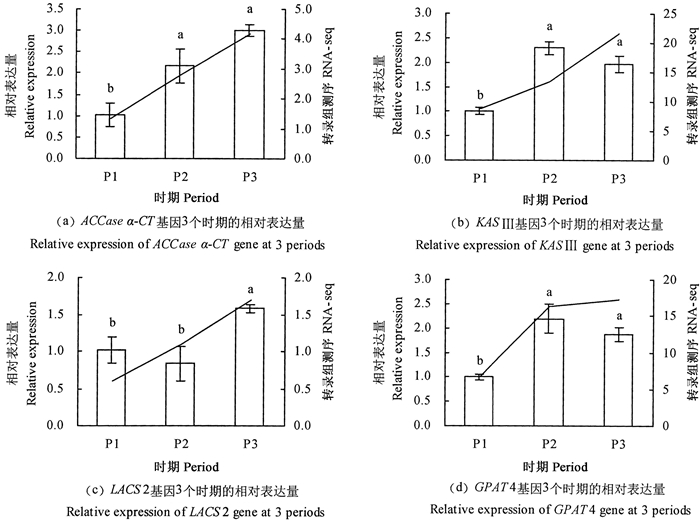
通过Pathway富集性分析和GO分类分析,选择4个显著差异的酶基因进行RT-qPCR验证(图 4),这4个基因的RT-qPCR的表达水平与转录组数据结果基本一致。随着种子的成熟,ACCase α-CT和KAS Ⅲ表达水平水平呈明显上升趋势。与P1相比较,P2的 LACS2表达水平未发生显著变化,到了P3时期,LACS2表达水平显著升高。相对于P1时期,P2和P3的 GPAT4表达水平均显著升高,但是P2和P3时期的 GPAT4表达水平未发现明显变化。
|
图 4 油茶种仁3个发育时期差异基因的表达水平 Fig. 4 Expression levels of differential gene from Camellia oleiferaseed kernels in 3 developing periods 注:直方柱上的不同字母表示不同处理间的显著差异(P< 0.05),直方图为Relative exprssion,直线为RNA-Seq。Note: Different letters indicate significant differences among the three treatments (P< 0.05), the histogram representsRelative expression, and the line represents RNA-seq. |
油茶树主要采收油茶籽榨取茶油,其中种仁是最主要的含油组织。通过测定油茶种仁的含油量,结果显示含油量从P1时期的35%上升到P3时期的47%(图 5),呈明显上升趋势,说明在油茶果成熟过程中,油脂保持一定水平的合成速率,并不断积累于油茶籽中。

|
图 5 油茶种仁不同时期含油率 Fig. 5 Oil content of C. oleifera seed kenel at different periods |
在油茶籽的3个发育时期,通过KEGG pathway发现参与油脂代谢的途径主要有甘油磷脂代谢途径、脂肪酸代谢、不饱和脂肪酸代谢、脂肪酸合成、甘油酯代谢、不饱和脂肪酸、α-亚麻酸代谢、亚油酸代谢和花生四烯酸代谢途径。在种子发育的3个时期,以上途径中富集到的差异基因参与了油脂代谢过程,并对油茶籽油脂合成代谢起调控作用。
植物油脂主要是以三酰甘油的形式储存,主要存在于植物的种子和果实中,三酰甘油由1分子甘油和3分子脂肪酸组成,有研究表明,油脂的积累受质体中脂肪酸含量的限制[27],所以脂肪酸合成是决定植物油脂含量的重要因素。ACCase酶包括同质型ACCase酶和异质型ACCase酶。异质型ACCase酶是催化脂肪酸合成反应的起始酶,是脂肪酸合成的限速酶,它催化乙酰辅酶A羧化为丙二酰辅酶A,丙二酰辅酶A继续在脂肪酸合酶的催化下进行连续的聚合反应生成脂肪酸。在黄豆发育的早期和中期, 油含量高的黄豆中ACCase酶活性明显高于油含量低的品种[28]。SELLWOOD et al[29]通过反义表达抑制油菜ACCase酶的活性获得转基因植株,转基因植株的成熟种子含油量显著低于野生型。油茶异质型ACCase的4个亚基(生物素羧化酶、生物素羧基载体蛋白、α-羧基转移酶和β-羧基转移酶)基因已被成功克隆,这4个亚基与花生、大豆的同源性较高[30],而且任何一个亚基的上调表达都会提高ACCase酶的活性,进而提高脂肪酸和油脂的合成。在油茶籽3个发育时期中,发现ACCase α-CT基因随着种子的成熟表达水平呈上升趋势,与此同时,含油量也不断增加。由此推断,在油茶种仁中,ACCase α-CT基因的上调表达可能促进脂肪酸的合成,从而提高油茶种仁含油量。
脂肪酸合酶催化的聚合反应中,以每次增加两个碳原子的形式完成碳链的延长,这种聚合反应是通过β酮脂酰合酶(KAS)催化,KAS酶有4种,分别为:KASⅠ、KASⅡ、KASⅢ和KASⅣ。KASⅢ是启动脂肪酸合成的关键酶,能够作用于酰基ACP,可以合成具有不同碳链结构的脂肪酸,因此KASⅢ可能是决定脂肪酸碳链结构的因子之一,是脂肪酸合成的限速酶之一[31]。有研究发现,在植物中KASⅢ影响脂肪酸碳链的长度,其酶活性直接影响脂肪酸的合成速率[32]。KASⅢ基因活性位点突变体不能在缺少长链脂肪酸的培养基上生长,这表明KASⅢ基因是脂肪酸合成所必需的[33]。在油茶籽3个发育时期,随着种仁的成熟,KASⅢ基因的表达水平不断增加,有利于脂肪酸的合成。
LACS可以将游离的脂肪酸合成为酰基辅酶A硫酯,此物质作为底物与甘油-3-磷酸参与到油脂的合成中。LACS在连接脂肪酸合成和油脂合成这两个代谢途径中起到了桥梁作用。目前在拟南芥的LACS家族有9个基因,其中LACS1和LACS9双突变时,种子的脂肪酸和油脂含量显著下降[34]。在水稻种子发育过程中,油脂含量变化和LACS活性呈显著正相关性[35]。同时,有研究发现AtLACS1、AtLACS2、AtLACS3、AtLACS4等基因的表达水平对于脂肪酸含量的提高有一定的作用[36];转AtLACS9基因的大豆含油率显著提高,这说明LACS9基因有助于油脂代谢的进行[37]。同样的,在油茶籽成熟过程,随着油茶籽含油量的增加,LACS2的表达水平也不断提高,这表明LACS2对油茶籽油脂的合成有一定的促进作用。
GPAT是三酰甘油合成的关键酶和起始酶,它催化甘油-3-磷酸sn-1位的酰化反应,形成溶血磷酯酸(LPA),后者再经过连续2次酰化反应形成三酰甘油。目前拟南芥GPAT家族有10个基因,分别为ATS1,AtGPAT1-AtGPAT9,在拟南芥种子中超表达经修饰的红花质体 GPAT基因可以显著提高种子含油量[38],AtGPAT9基因可能与种子储藏性油脂合成有关[39]。魏琦琦等[40]在油茶中首次克隆了两个GPAT基因,分别命名为CoATS1和CoGPAT6,并推测这两个基因的主要作用可能作用于膜脂。在本研究中,随着油茶籽的成熟,GPAT4基因的表达水平一直呈升高趋势,这可能促进了油脂的合成,有助于油含量的增加。
本文研究发现,油茶籽3个发育过程中,在KEGG pathway中发现与脂肪酸合成和油脂合成相关的4个关键基因的表达水平都随着成熟度的增加呈上升趋势,而随着油茶籽的成熟,种仁的含油量也一直在显著增加,说明这4个关键基因在种子油脂合成代谢中具有重要作用,促进了油脂的合成,进而提高了油茶籽油脂的含量。这为今后通过分子手段改造某些基因以达到提高油茶种仁含油量的目的起到了借鉴作用。但油脂代谢相关基因在木本植物中的具体作用机制以及这些基因如何调控脂质代谢和油的品质还有待于进一步的研究:这些研究结果为后续的油茶油脂合成代谢的分子调控机制研究提供了理论基础。
| [1] | 庄瑞林. 中国油茶[M]. 2版. 北京: 中国林业出版社, 2008: 1-13. |
| [2] | 周长富, 姚小华, 林萍, 等. 油茶种子发育过程组分含量动态研究[J]. 中国油料作物学报, 2013, 35(6): 680–685. DOI:10.7505/j.issn.1007-9084.2013.06.010 |
| [3] | 李文林, 黄凤洪, 王利宾. 油茶籽加工和综合利用研究进展[J]. 中国油脂, 2011, 36(11): 55–57. |
| [4] | 赵翠格, 刘頔, 李凤兰, 等. 植物种子油脂的生物合成及代谢基础研究进展[J]. 种子, 2010, 29(4): 56–62. |
| [5] | BAUD S, GUYON V, KRONENBERGER J, et al. Multifunctional acetyl-CoA carboxylase 1 is essential for very long chain fatty acid elongation and embryo development in Arabidopsis[J]. The Plant Journal, 2003, 33(1): 75–86. DOI:10.1046/j.1365-313X.2003.016010.x |
| [6] | PATEL M, JUNG S, MOORE K, et al. High-oleate peanut mutants result from a MITE insertion into the FAD2 gene[J]. Theoretical and Applied Genetics, 2004, 108(8): 1492–1502. DOI:10.1007/s00122-004-1590-3 |
| [7] | BROWN A P, KROON J T M, SWARBRECK D, et al. Tissue-specific whole transcriptome sequencing in castor, directed at understanding triacylglycerol lipid biosynthetic pathways[J]. PLoS One, 2012, 7(2): e30100. DOI:10.1371/journal.pone.0030100 |
| [8] | LIU H, WANG C P, KOMATSU S, et al. Proteomic analysis of the seed development in Jatropha curcas:from carbon flux to the lipid accumulation[J]. Journal of Proteomics, 2013, 91: 23–40. DOI:10.1016/j.jprot.2013.06.030 |
| [9] | 陈昊, 蒋桂雄, 龙洪旭, 等. 基于油桐种子3个不同发育时期转录组的油脂合成代谢途径分析[J]. 遗传, 2013, 35(12): 1403–1414. |
| [10] | 姜志娜, 谭晓风, 袁军, 等. 油茶果实和叶片中主要营养物质含量的变化规律[J]. 中南林业科技大学学报, 2012, 32(5): 42–45. |
| [11] | 彭邵锋, 陆佳, 陈永忠, 等. 油茶品种资源现状与良种筛选技术[J]. 经济林研究, 2012, 30(4): 174–179. |
| [12] | 谭晓风, 胡芳名, 谢禄山, 等. 油茶种子EST文库构建及主要表达基因的分析[J]. 林业科学, 2006, 42(1): 43–48. |
| [13] | 张党权, 谭晓风, 陈鸿鹏, 等. 油茶SAD基因的全长cDNA克隆及生物信息学分析[J]. 林业科学, 2008, 44(2): 155–159. DOI:10.11707/j.1001-7488.20080224 |
| [14] | 陈鸿鹏, 谭晓风, 谢耀坚, 等. 油茶FAD2基因的植物表达载体和RNA干扰载体构建及其功能分析[J]. 植物研究, 2015, 35(3): 347–354. DOI:10.7525/j.issn.1673-5102.2015.03.005 |
| [15] | 曾艳玲, 谭晓风, 蒋瑶, 等. 油茶果糖-1, 6-二磷酸醛缩酶基因(CoFBA4)的分子特征与表达分析[J]. 林业科学, 2013, 49(11): 164–170. |
| [16] | 江南, 谭晓风, 张琳, 等. 基于RNA-Seq的油茶种子α-亚麻酸代谢途径及相关基因分析[J]. 林业科学, 2014, 50(8): 68–75. |
| [17] | 林萍, 曹永庆, 姚小华, 等. 普通油茶种子4个发育时期的转录组分析[J]. 分子植物育种, 2011, 9(4): 498–505. |
| [18] | ZENG Y L, TAN X F, ZHANG L, et al. Identification and expression of fructose-1, 6-bisphosphate aldolase genes and their relations to oil content in developing seeds of tea oil tree (Camellia oleifera)[J]. PLoS One, 2014, 9(9): e107422. DOI:10.1371/journal.pone.0107422 |
| [19] | 谢国阳. 福建省油茶良种培育现状与对策[J]. 福建林业科技, 2012, 39(1): 153–155, 170. |
| [20] | GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29(7): 644–652. DOI:10.1038/nbt.1883 |
| [21] | ALTSCHUL S F, MADDEN T L, SCHÄFFER A A, et al. Gapped BLAST and PSI-BLAST:a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389–3402. DOI:10.1093/nar/25.17.3389 |
| [22] | 邓泱泱, 荔建琦, 吴松锋, 等. nr数据库分析及其本地化[J]. 计算机工程, 2006, 32(5): 71–73. |
| [23] | KANEHISA M, GOTO S, KAWASHIMA S, et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Research, 2004, 32(S1): D277–D280. |
| [24] | ASHBURNER M, BALL C A, BLAKE J A, et al. Gene ontology:tool for the unification of biology[J]. Nature Genetics, 2000, 25(1): 25–29. DOI:10.1038/75556 |
| [25] | TRAPNELL C, WILLIAMS B A, PERTEA G, et al. Transcript assembly and quantification by RNA-Seq reveals unannotated transcripts and isoform switching during cell differentiation[J]. Nature Biotechnology, 2010, 28(5): 511–515. DOI:10.1038/nbt.1621 |
| [26] | 国家粮食局. 植物油料含油量测定: GB/T 14488. 1-2008[S]. 北京: 中国标准出版社, 2008. |
| [27] | BAO X M, OHLROGGE J. Supply of fatty acid is one limiting factor in the accumulation of triacylglycerol in developing embryos[J]. Plant Physiology, 1999, 120(4): 1057–1062. DOI:10.1104/pp.120.4.1057 |
| [28] | GENGENBACH B G, SOMERS D A, WYSE D L, et al. Transgenic plants expressing maize acetyl COA carboxylase gene and method of altering oil content: US, US6222099[P]. 2001-04-24. |
| [29] | SELLWOOD C, SLABAS A R, RAWSTHORNE S. Effects of manipulating expression of acetyl-CoA carboxylase I in Brassica napus L. embryos[J]. Biochemical Society, 2000, 28(6): 598–600. DOI:10.1042/bst0280598 |
| [30] | 王保明. 油茶ACCase基因的克隆及功能研究[D]. 长沙: 中南林业科技大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10538-1012397883.htm |
| [31] | CHOI K H, HEATH R J, ROCK C O. β-Ketoacyl-acyl carrier protein synthase Ⅲ (FabH) is a determining factor in branched-chain fatty acid biosynthesis[J]. Journal of Bacteriology, 2000, 182(2): 365–370. DOI:10.1128/JB.182.2.365-370.2000 |
| [32] | DEHESH K, TAI H, EDWARDS P, et al. Overexpression of 3-ketoacyl-acyl-carrier protein synthase Ⅲs in plants reduces the rate of lipid synthesis[J]. Plant Physiology, 2001, 125(2): 1103–1114. DOI:10.1104/pp.125.2.1103 |
| [33] | LAI C Y, CRONAN J E. β-Ketoacyl-acyl carrier protein synthase Ⅲ (FabH) is essential for bacterial fatty acid synthesis[J]. Journal of Biological Chemistry, 2003, 278(51): 51494–51503. DOI:10.1074/jbc.M308638200 |
| [34] | CLOUGH S J, BENT A F. Floral dip:a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana[J]. The Plant Journal, 1998, 16(6): 735–743. DOI:10.1046/j.1365-313x.1998.00343.x |
| [35] | ICHIHARA K, KOBAYASHI N, SAITO K. Lipid synthesis and acyl-CoA synthetase in developing rice seeds[J]. Lipids, 2003, 38(8): 881–884. DOI:10.1007/s11745-003-1139-0 |
| [36] | PULSIFER I P, KLUGE S, ROWLAND O. Arabidopsis long-chain acyl-CoA synthetase 1(LACS1), LACS2, and LACS3 facilitate fatty acid uptake in yeast[J]. Plant Physiology and Biochemistry, 2012, 51: 31–39. DOI:10.1016/j.plaphy.2011.10.003 |
| [37] | 赵欢欢, 吴兴, 张锋, 等. 拟南芥AtLACS9基因的克隆及其植物表达载体构建[J]. 大豆科学, 2011, 30(2): 190–193. |
| [38] | JAIN R K, COFFEY M, LAI K, et al. Enhancement of seed oil content by expression of glycerol-3-phosphate acyltransferase genes[J]. Biochemical Society Transactions, 2000, 28(6): 958–961. DOI:10.1042/bst0280958 |
| [39] | GIDDA S K, SHOCKEY J M, ROTHSTEIN S J, et al. Arabidopsis thaliana GPAT8 and GPAT9 are localized to the ER and possess distinct ER retrieval signals:functional divergence of the dilysine ER retrieval motif in plant cells[J]. Plant Physiology and Biochemistry, 2009, 47(10): 867–879. DOI:10.1016/j.plaphy.2009.05.008 |
| [40] | 魏琦琦, 赵广, 何潇, 等. 油茶2个甘油-3-磷酸酰基转移酶基因的克隆与组织表达特异性分析[J]. 植物生理学报, 2016, 52(7): 1099–1109. |

 2018, Vol. 38
2018, Vol. 38


