文章信息
- 陈潇潇, 曹光球, 汪凤林, 罗红艳, 魏晓骁, 曹世江
- CHEN Xiaoxiao, CAO Guangqiu, WANG Fenglin, LUO Hongyan, WEI Xiaoxiao, CAO Shijiang
- 杉木纤维素合酶(ClCesA2)基因的克隆与表达分析
- Cloning and expression analysis of cellulose synthase ClCesA2 from Cunninghamia lanceolata
- 森林与环境学报,2018, 38(1): 1-6.
- Journal of Forest and Environment,2018, 38(1): 1-6.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2018.01.001
-
文章历史
- 收稿日期: 2017-04-14
- 修回日期: 2017-05-25
2. 国家林业局杉木工程技术研究中心, 福建 福州 350002
2. Chinese Fir Engineering Technology Research Center under State Forestry Administration, Fuzhou, Fujian 350002, China
纤维素是地球上重要的可再生资源,是植物细胞壁重要的骨架结构之一,是由D-葡萄糖通过β-1,4糖苷键连接而成的一种线性葡聚糖[1],在造纸、纺织、林业等诸多领域中有广泛的经济价值。高等植物纤维细胞发育过程复杂,受多种基因调控,纤维素合酶(CesA)是纤维形成的关键酶之一,主要催化纤维素的合成[2-5]。目前,已从40多种植物中克隆到1 400多个CesA基因的相关序列,在拟南芥[Arabidopsis thaliana (L.) Heynh.]中鉴定出10个CesA基因,毛果杨(Populus trichocarpa)中鉴定出18个CesA基因[6],绿竹[Dendrocalamopsis oldhami (Munro) Keng f.]中鉴定出8个CesA基因[7]。其中拟南芥的AtCesA1、AtCesA3、AtCesA6基因主要参与初生细胞壁合成,AtCesA4、AtCesA7、AtCesA8基因主要参与次生细胞壁合成[8-10]。在毛果杨中,PtCesA4、PtCesA7、PtCesA8、PtCesA17、PtCesA18基因构成了纤维素合酶复合体Ⅰ,主要参与细胞次生壁的合成,复合体Ⅱ由PtCesA3、PtCesA10、PtCesA11、PtCesA13、PtCesA15、PtCesA16基因构成,参与初生细胞壁和次生细胞壁的合成[11-13]。
杉木[Cunninghamia lanceolata (Lamb.) Hook.]是我国南方重要的造林树种,也是我国主要的用材林树种[14]。然而,我国杉木人工林的木质不紧密,易开裂和变形,在轮伐期短的林分中表现得尤为突出,极大程度地限制了杉木木材的用途。目前,国内学者开展一系列有关杉木木材细胞壁化学成分的研究,均是从化学的角度来探究细胞壁与其木材性质之间的关系,但对于遗传操作改良杉木的木材品质这一方法尚未涉及[15-17]。本课题组前期已从杉木中克隆出杉木纤维素合酶(ClCesA1)基因,并对该基因的结构以及功能做出了预测[18]。因此,对杉木纤维素合酶(ClCesA2)基因结构、功能以及其在不同组织中表达情况的研究和了解,对杉木材质的遗传改良具有重要意义。
1 材料与方法 1.1 材料试验材料为1年生杉木无性系061幼苗,由国家林业局杉木工程技术研究中心实验室提供。
1.2 杉木总RNA提取与cDNA合成总RNA的提取采用天根植物多糖多酚总RNA试剂盒进行;cDNA第1链的合成采用TIANScript cDNA第1链合成试剂盒进行。
1.3 ClCesA2基因的扩增与实时荧光定量聚合酶链式反应利用NCBI上的GenBank数据库已知的杉木DNA序列,设计特异引物及实时荧光定量聚合酶链式反应(polymerase chain reaction,PCR)引物,并递交上海生物工程技术有限公司合成,引物设计见表 1。PCR扩增反应条件如下(采用20 μL体系):cDNA 2 μL,引物ClCesA2-Fw(10 μM)1 μL,引物ClCesA2-Rv(10 μM)1 μL,10×KOD Buffer 2 μL,dNTP Mixture(2.5 mM)4 μL,DNA Polymerase KOD(2.5 U·μL-1)0.2 μL,ddH2O加至20 μL。95 ℃预变性3 min,94 ℃变性1 min,53 ℃退火3 min,72 ℃延伸3 min,共30个循环,最后72 ℃延伸10 min,4 ℃保存。扩增后产物经1%的琼脂糖凝胶电泳检测。利用实时荧光定量PCR仪器(LightCycler® Nano,Roche,瑞士)研究ClCesA2基因在不同组织的表达,qPCR扩增试剂为TransStart® Tip Green qPCR SuperMix,采用荧光染料为SYBR GreenⅠ,杉木EF1α基因作为内参基因。
| 引物名称 Primer name |
引物序列(5′-3′) Primer sequence |
| ClCesA2-Fw | ATGGAGGCCAACGCTGGC |
| ClCesA2-Rv | TCAGCAATTGACACCACATTGTTGTG |
| CesA2 Q Fw | GTCGCAGGATCTCATAACAGG |
| CesA2Q Rv | CACATTCATTGCAGGCTACA |
| EF1α-Fw | CAAAGAAGGGTGCCAATGA |
| EF1α-Rv | ACCAAACAACCGACCTACGA |
将PCR产物送至上海生物工程技术有限公司进行测序。测序结果用Vector NTI翻译成氨基酸序列在BLAST进行同源性比对,并使用MEGA6中Neighbor-Joining法构建系统进化树。用ProtParam tool在线软件预测ClCesA2蛋白的理化性质,ProtScale软件分析ClCesA2蛋白的疏水性,ProtFun分析预测ClCesA2蛋白的功能。用Scratch Protein Predictor软件对蛋白质序列进行2级结构预测,利用TMHMM软件进行蛋白质跨膜区预测。
2 结果与分析 2.1 总RNA质量检测提取的杉木叶片总RNA经1%琼脂糖凝胶电泳检测(图 1),18 S和28 S条带完整且清晰,28 S的亮度约为18 S的2倍,无拖尾现象,无DNA污染。超微量分光光度计检测A260/A280=1.91,说明其质量满足反转录及后续试验需求。
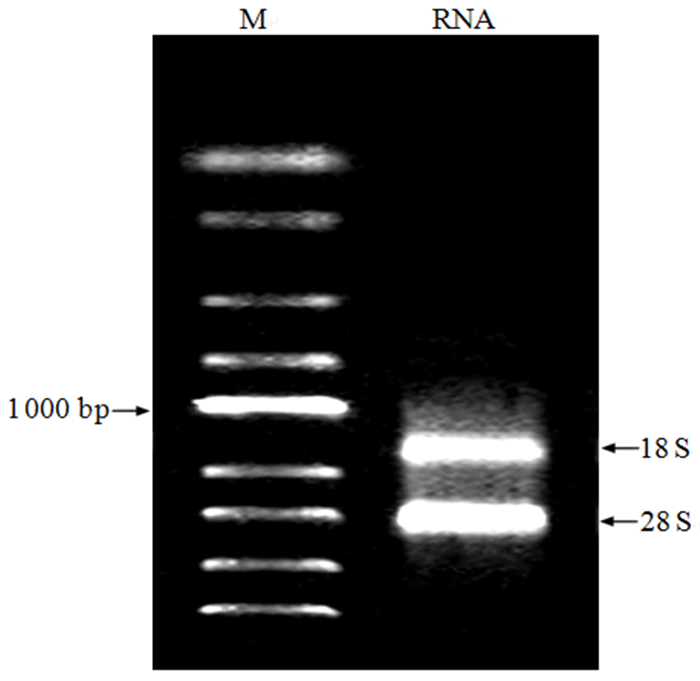
|
图 1 总RNA电泳图 Fig. 1 Electrophoretogram of total RNA |
以061杉木叶片提取的RNA反转录得到的cDNA为模板,通过设计的特异性引物进行PCR扩增,产物经1%琼脂糖凝胶电泳,得到长度约为3 200 bp的条带(图 2),目的片段进行测序后获得了长度为3 276 bp的序列, 经验证无误。
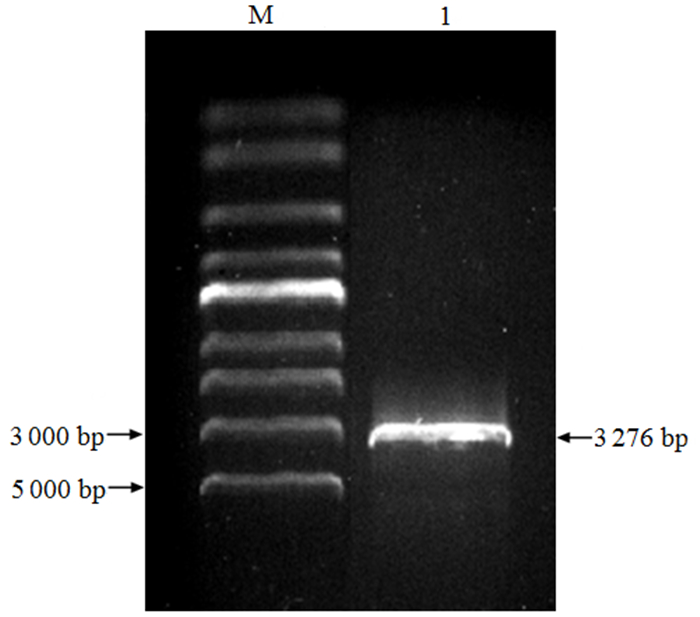
|
图 2 ClCesA2基因PCR扩增产物 Fig. 2 PCR amplification product of ClCesA2 gene |
在Pfam上对其机构域进行分析,在32~97氨基酸位点具有锌指结构CXXC(半胱氨酸氨-xx-半胱氨酸氨)。在787~890氨基酸位点具有保守的天冬氨酸残基,为糖基转移酶家族结构域Glyco-tranf-2-3(图 3),利用NCBI CDD在线工具对其鉴定在524~842氨基酸位点含有CesA-CelA-like结构域,因此该蛋白被确认为杉木CesA/Csl家族蛋白。运用MEGA 6构建分子进化树,将克隆到的基因翻译成的蛋白序列与拟南芥,水稻(Oryza sativa L.),颤杨(Populus tremuloides),欧美杂种山杨(Populus tremula L.×Populus tremuloides),毛白杨(Populus tomentosa Carr.),火炬松(Pinus taeda Linn.)的细胞CesA基因进行系统树分析。ClCesA2基因与细胞初生壁合成有关的基因AtCesA1,PtrCesA4聚在一起(图 4),推测ClCesA2基因可能与杉木细胞初生壁合成有关。

|
图 3 ClCesA2蛋白序列 Fig. 3 Protein sequence of ClCesA2 注:图中黄色区域为锌指结构;红色区域为跨膜结构区;红字区域为纤维素合酶底物结合结构。 Note: the yellow region indicates ring finger dimain, the red regin indicates transmembrane domain and the red words region indicates cellulose synthase substrate binding structure. |
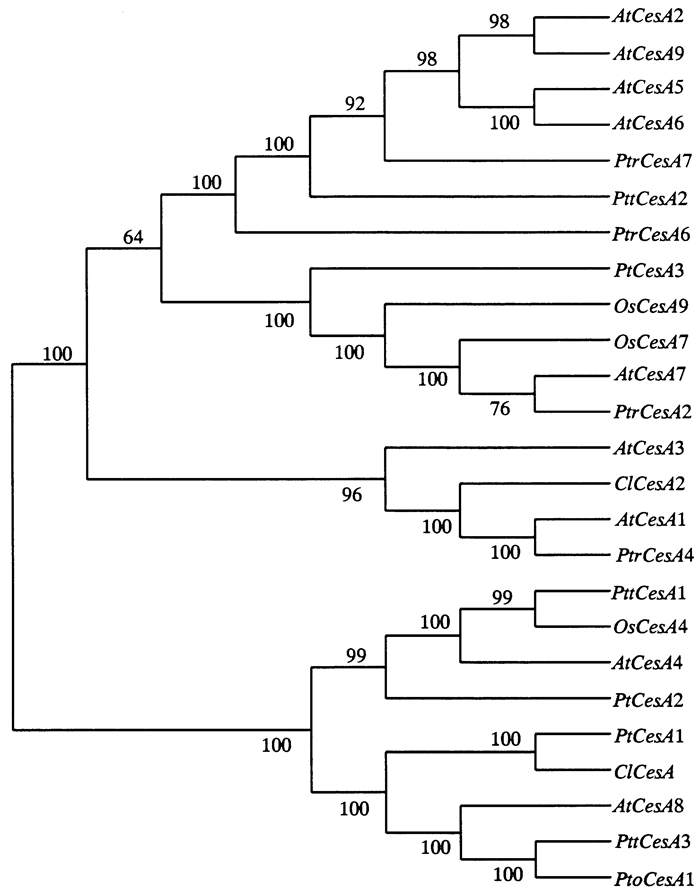
|
图 4 ClCesA2基因系统进化树 Fig. 4 Phylogenetic tree of ClCesA2 gene 注:分支上的数值表示Bootstrap验证中基于1 000次检验重复该节点的可信度。 Note: number on the branches represent the reliability percent of bootstrap test based on 1 000 replications. |
Protparam预测ClCesA2蛋白的理化性质,推测该蛋白相对分子质量为122 820.41 Da,等电点PI为6.65,理论推导半衰期为30 h,Leu含量最高,达8.6%,其次为Gly, 占7.5%,不稳定系数为39.21,属于稳定蛋白。经ProtScale软件分析可知, ClCesA2蛋白的疏水性最高, 分值为3.278,亲水性最高分为-3.544,整体而言,ClCesA2蛋白的整条多肽链亲水性氨基酸多于疏水性氨基酸,由此推测该蛋白属于亲水性蛋白(图 5)。利用ProtFun分析预测ClCesA2蛋白的功能,表明该蛋白具有生物合成相关的电压门控离子通道连接酶的作用。
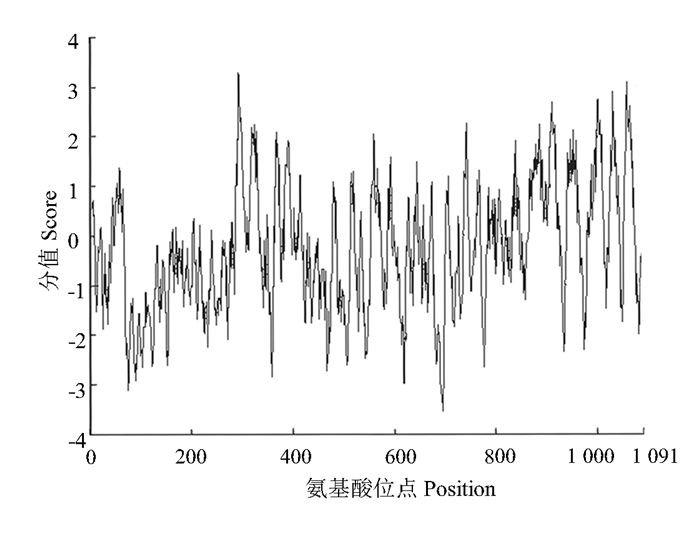
|
图 5 ClCesA2蛋白氨基酸序列的疏水性分析 Fig. 5 The hydrophobicity analysis of sequence of protein amino acid of ClCesA2 |
用PBIL LYON-GERLAND信息库对蛋白质序列进行2级结构预测,表明ClCesA2蛋白的2级结构中α-螺旋占98.81%,β-折叠占0.88%,无规则卷曲占0.31%,预计可形成二硫键13个,分别在1 087~1 091、590~602、403~432、39~42、770~809、81~84、882~888、647~648、670~676、61~69、58~66、575~674、675~847氨基酸位点,参与维持蛋白质构象的稳定。利用TMHMM软件进行蛋白质跨膜区预测,跨膜结构预测表明ClCesA2蛋白有8个跨膜区,分别是285~307、314~333、868~890、902~924、939~961、982~1 004、1 019~1 041、1 054~1 073氨基酸位点(图 6),最大跨膜区域为22个氨基酸残基,最小跨膜区域为19个氨基酸残基,平均跨膜区域为21个氨基酸残基。
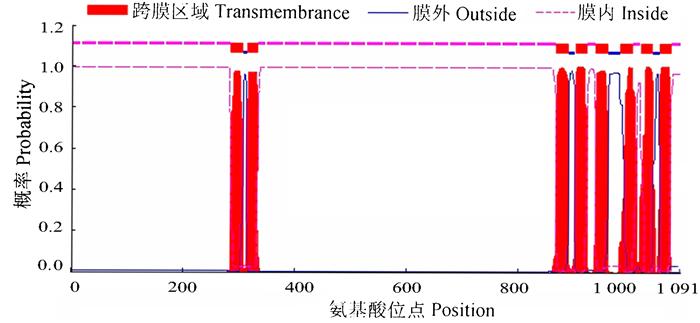
|
图 6 ClCesA2蛋白氨基酸序列的跨膜区预测 Fig. 6 Prediction of trasmembrane domains of protein amino acid of ClCesA2 |
在杉木根、茎、叶3种组织中, ClCesA2基因均有表达,表达量有显著差异(图 7)。其中,在茎中的表达量最高,大约为叶的20倍,根的表达量约为叶的10倍,叶的表达量最低。并且ClCesA2基因在杉木茎中的表达量较高,提示其在杉木韧皮纤维的合成过程中可能具有重要作用。
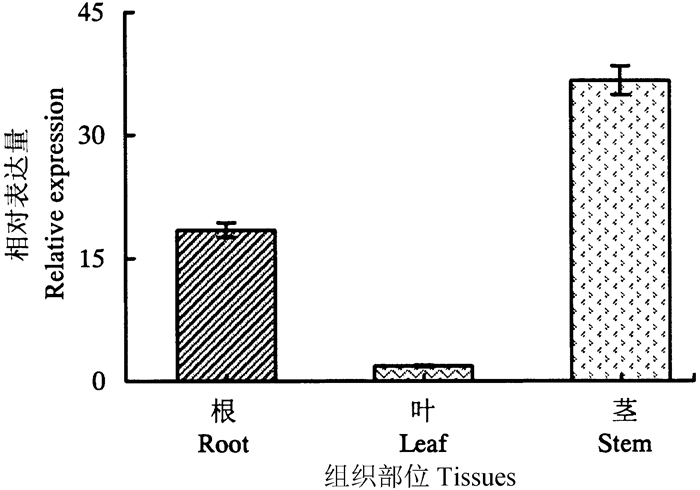
|
图 7 不同组织中ClCesA2基因相对表达量 Fig. 7 Relative expression of ClCesA2 gene in different tissues |
除植物外,一些细菌也可以合成纤维素。CesA基因最初的就是从醋酸杆菌中分离的[19-20]。随后先后从棉花、拟南芥、杨树等植物中克隆到了CesA基因,并进行了一系列的功能预测与验证。有关拟南芥中发现的10个CesA基因,已经先后从生化和遗传的角度证明了其在细胞壁合成中的功能。在欧洲颤杨中有证据表明ptrCesA6与茎的伸长生长有关,在杨树枝条生长的快速阶段表达量高[21]。水稻中也先后发现了12个CesA基因,有关CesA基因在水稻中功能的研究不仅限于纤维素合成的作用。水稻中的OsCesA4基因的过量表达会降低植株在幼苗期耐旱、耐高温、耐高盐性,对水稻的结实率也有一定影响[22]。但林木生物技术相对发展较慢,有关ClCesA基因在杉木中的其他方面的功能有待进一步研究与证实。
本课题组已发表一篇有关ClCesA1基因的文章[18]。ClCesA1基因先于本研究的ClCesA2基因克隆出来,且这两个基因均来自于杉木。生物信息学分析结果对比表明,ClCesA1基因的开放阅读框为2 955 bp,共编码984个氨基酸,ClCesA2基因开放阅读框全长3 276 bp,共编码1 091个氨基酸。ClCesA1基因和ClCesA2基因氨基酸相似性为62.4%,都有8个跨膜区域,最大跨膜区域同为22个氨基酸残基,但ClCesA2最小跨膜区域为19个氨基酸残基,ClCesA1基因最小跨膜区域为12个氨基酸残基。相对表达量分析结果对比显示,ClCesA1基因在根中表达量最高,ClCesA2基因在茎中表达量最高。根据系统进化树分析研究,该基因在功能上的预测与ClCesA1基因有所不同,ClCesA2基因可能参与杉木细胞初生壁的形成,而ClCesA1基因可能参与杉木细胞次生壁的形成。本课题组目前虽然已从杉木中克隆到两个CesA基因,但本课题组有关杉木的基因组的测序未完成,尚且不能确定杉木中是否还存在CesA家族的其他基因。
| [1] | KUMAR M, TURNER S. Plant cellulose synthesis:CesA proteins crossing kingdoms[J]. Phytochemistry, 2015, 112: 91–99. DOI:10.1016/j.phytochem.2014.07.009 |
| [2] | 刘长斌, 薛永常, 聂会忠. 高等植物纤维素合酶超家族[J]. 生命的化学, 2007, 27(6): 533–535. |
| [3] | 魏建华, 宋艳茹. 植物纤维素合酶基因研究进展[J]. 植物学通报, 2002, 19(6): 641–649. |
| [4] | JOSHI C P, MANSFIELD S D. The cellulose paradox-simple molecule, complex biosynthesis[J]. Current Opinion in Plant Biology, 2007, 10(3): 220–226. DOI:10.1016/j.pbi.2007.04.013 |
| [5] | 于莹, 郭文栋, 赵丽娟, 等. 亚麻纤维素合酶LuCesA8基因克隆与表达分析[J]. 东北农业大学学报, 2016, 47(8): 39–45, 54. |
| [6] | DJERBI S, LINDSKOG M, ARVESTAD L, et al. The genome sequence of black cottonwood(Populus trichocarpa)reveals 18 conserved cellulose synthase(CesA) genes[J]. Planta, 2005, 221(5): 739–746. DOI:10.1007/s00425-005-1498-4 |
| [7] | ATANASSOV H, PITTMAN J K, TURNER S R. Elucidating the mechanisms of assembly and subunit interaction of the cellulose synthase complex of Arabidopsis secondary cell walls[J]. Journal of Biological Chemistry, 2009, 284(6): 3833–3841. DOI:10.1074/jbc.M807456200 |
| [8] | 邓小波, 胡尚连, 曹颖, 等. 绿竹与毛竹纤维素合成酶(CesA)的生物信息学分析[J]. 福建林学院学报, 2011, 31(1): 84–90. |
| [9] | LI S D, LEI L, GU Y. Functional analysis of complexes with mixed primary and secondary cellulose synthases[J]. Plant Signaling & Behavior, 2013, 8(3): e23179. |
| [10] | CREUX N M, RANIK M, BERGER D K, et al. Comparative analysis of orthologous cellulose synthase promoters from Arabidopsis, Populus and Eucalyptus:evidence of conserved regulatory elements in angiosperms[J]. New Phytologist, 2008, 179(3): 722–737. DOI:10.1111/nph.2008.179.issue-3 |
| [11] | SONG D L, SHEN J H, LI L G. Characterization of cellulose synthase complexes in Populus xylem differentiation[J]. New Phytologist, 2010, 187(3): 777–790. DOI:10.1111/j.1469-8137.2010.03315.x |
| [12] | HOLLAND N, HOLLAND D, HELENTJARIS T, et al. A comparative analysis of the plant cellulose synthase(CesA) gene family[J]. Plant Physiology, 2000, 123(4): 1313–1323. DOI:10.1104/pp.123.4.1313 |
| [13] | 袁红梅, 郭文栋, 赵丽娟, 等. 亚麻纤维素合酶超基因家族的生物信息学及表达分析[J]. 中国农业科学, 2016, 49(23): 4656–4668. DOI:10.3864/j.issn.0578-1752.2016.23.018 |
| [14] | 贾治邦. 中国森林资源图集——第七次全国森林资源清查[M]. 北京: 中国林业出版社, 2009. |
| [15] | PFEFFER P E, GERASIMOWICZ W V, PIOTROWSKI E G, et al. Effect of paramagnetic iron on quantitation in carbon-13 cross polarization magic angle spinning nuclear magnetic resonance spectrometry of heterogeneous environmental matrixes[J]. Analytical Chemistry, 2002, 56(4): 734–741. |
| [16] | 任海青, 黄安民, 刘君良, 等. 杉木加工利用研究进展及建议[J]. 木材工业, 2006, 20(1): 25–27. |
| [17] | 陈美玲, 张双燕, 王传贵. 密粘褶菌生物性降解对杉木木材性质的影响[J]. 安徽农业大学学报, 2016, 43(3): 378–382. |
| [18] | 魏晓骁, 王士亚, 陈潇潇, 等. 一个新杉木纤维素合酶ClCesA基因的克隆与植物表达载体构建[J]. 福建农林大学学报(自然科学版), 2017, 46(1): 66–72. |
| [19] | SAXENAL I M, LIN F C, BROWN R M Jr. Cloning and sequencing of the cellulose synthase catalytic subunit gene of Acetobacter xylinum[J]. Plant Molecular Biology, 1990, 15(5): 673–683. DOI:10.1007/BF00016118 |
| [20] | SAXENA I M, BROWN R M Jr. Identification of a second cellulose synthase gene(acsAII)in Acetobacter xylinum[J]. Journal of Bacteriology, 1995, 177(18): 5276–5283. DOI:10.1128/jb.177.18.5276-5283.1995 |
| [21] | SAMUGA A, JOSHI C P. Cloning and characterization of cellulose synthase-like gene PtrCSLD2 from developing xylem of aspen trees[J]. Physiologia Plantarum, 2004, 120(4): 631–641. DOI:10.1111/ppl.2004.120.issue-4 |
| [22] | 刘丽娟. 水稻OsCesA4基因的纤维素合成与纤维性状调控功能研究[D]. 长沙: 湖南农业大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10537-1015604251.htm |

 2018, Vol. 38
2018, Vol. 38


