2. 农业农村部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306;
3. 上海海洋大学 食品质量安全检测实验室, 上海 201306;
4. 呼和浩特市农畜产品质量安全中心, 内蒙古 呼和浩特 010020;
5. 上海水产品加工及贮藏工程技术研究中心, 上海 201306
牡蛎(Ostreidae)作为一种近海养殖的海产品,产量位于中国海水养殖产品首位,2020年产量达542余万t[1]。中国沿海地区分布着种类多样的牡蛎种群。其中,商业化利用程度最高的是长牡蛎、福建牡蛎和香港牡蛎,而近江牡蛎和熊本牡蛎也有一定的养殖规模。其中长牡蛎是我国北方沿海主要养殖品种,福建牡蛎是闽浙地区主要养殖品种,香港牡蛎和近江牡蛎则是两广地区主要养殖品种[2]。因其营养价值高,生食味道鲜美,牡蛎成为主要的生食水产品之一,但受生活习性影响,牡蛎是一种善于富集微生物的滤食性动物。而在过去十几年间,海产品等一些食品中被发现存在大量广谱抗生素抗性细菌,其中许多具有可传播的耐药基因[3]。耐药基因的扩散和迁移主要通过水产品携带的微生物基因组可移动的遗传元件介导的水平基因转移或由原核生物群落介导的垂直基因转移实现的[4],同时环境中重金属也是影响细菌抗生素抗性的重要因子,有些细菌能够对重金属毒性产生耐受,并能够稳定遗传重金属耐受基因(heavy metal tolerance genes,HTGs),而这些遗传物质中存在同时编码抗生素耐药基因(antibiotic resistance genes,ARGs)的现象[5-6]。因此,重金属耐受性与抗生素抗性间存在的共选机制是一个值得关注的科学问题[7]。抗生素的过度使用和长期滥用造成了水产品及养殖环境(水体)中ARGs的形成与传播,以及种类和丰度的增加。环境中ARGs的残留或传播,使水产品在源头上受到污染,对人类健康产生了严重隐患。由于牡蛎特殊的生活习性,极容易在其体内发生抗性基因的转移,而在同一质粒和或转座子上通常发现编码金属和抗生素抗性的多个基因[8],从而产生协同抗性,我们有必要对牡蛎中抗生素与重金属抗性基因型分布情况及其相关性进行研究,以期对ARGs的来源、传播、扩散等机制进行研究,从源头上减少水产品中ARGs的出现。
1 材料与方法 1.1 材料与试剂 1.1.1 样本采集及处理地点及对象:2021年10月从上海市江阳水产市场对所售不同产地牡蛎进行取样。共计取样7份,根据产地分别将样品编号命名为:SDWH1、SDWH2、SDWH3(产地山东威海,图 1a);ZJTZ(产地浙江台州,图 1b);GDZJ1、GDZJ2(产地广东湛江,图 1c);GXBH(产地广西北海,图 1d)。
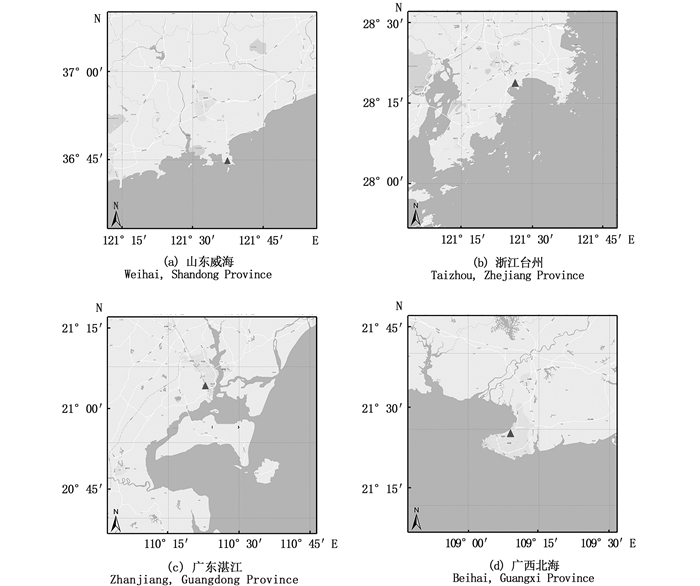
|
图 1 牡蛎样品产地信息 Fig. 1 Origin information of oyster samples |
根据不同牡蛎品种典型外观对采样牡蛎种类进行鉴别(表 1),山东样品为长牡蛎(又称太平洋牡蛎)、浙江样品为褶牡蛎,广东和广西样品均为近江牡蛎。
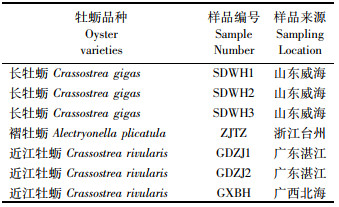
|
表 1 牡蛎样品信息表 Tab.1 Sample information of oysters |
样品采样与处理:每8个牡蛎记为1份样品。样品运输过程温度维持在0~5 ℃,抵达实验室后无法当天检测的样品保存在-80 ℃冰箱,并确保样品最多低温冷冻1次。镊子、手术刀、手术剪经高压灭菌后,将样品分离出肠道、腮和肌肉3部分(分别在各自对应的样品名称后面加入-I、-G和-M进行区分),7份样品共得到21份待测样品,分别装入无菌袋中待下一步处理。
1.1.2 试剂Tissue DNA Kit组织DNA提取试剂盒(美国Thermo Scientific公司);Taq DNA聚合酶,dNTP(大连TaKaRa公司)。
1.2 仪器与设备Advantage A10超纯水仪(德国Millipore公司);KingFisher Flex全自动核酸提取仪(美国Thermo Scientific公司);6235 PCR仪(德国Eppendorf公司);GelDocXR电泳及凝胶成像分析系统(美国Bio-Rad公司);Synergy 2酶标仪(美国BioTek公司)。
1.3 方法 1.3.1 牡蛎中微生物菌群多样性分析方法称样:选取牡蛎组织样品(提前解冻,每组2份, 每份10 mg,共14份,分别放至无菌1.5 mL离心管中)。
裂解:每个离心管加入200 μL Lysis Buffer和10 μL Pro. K,混合均匀;65 ℃金属浴15 min,至澄清,若不澄清可适当延长时间;向离心管中加入5 μL RNase A,混合均匀;室温放置10 min。
总DNA提取(自动化DNA提取仪):将裂解得到的全部溶液移入96深孔板(Sample板),向Sample板中加入200 μL Binding Buffer、300 μL无水乙醇、20 μL Magnetic Beads;另取96深孔板(Wash 1板)向对应位置加入Wash Buffer 800 μL;另取96深孔板(Wash 2板)向对应位置加入70% 乙醇800 μL;另取96深孔板(Wash 3板)向对应位置加入70% 乙醇800 μL;另取96 KF板(Elution板)加入Elution Buffer 100 μL。运行自动化DNA提取程序,结束后,移取Elution板中DNA至无菌1.5 mL离心管中-20 ℃保存。
用酶标仪测定DNA浓度,当浓度高于500 ng/μL后,在上海美吉生物医药科技有限公司生信云分析平台进行微生物菌群多样性分析。
1.3.2 重金属及抗生素耐药基因的PCR扩增方法选取牡蛎不同组织器官样品,按照1.3.1节中的方法获得宏基因组DNA。在25 μL的PCR反应体系下,用PCR进行六大类28种抗生素抗性基因(ARGs)和4种重金属耐受基因(HTGs)引物的扩增(表 2)。PCR扩增程序:95 ℃ 5 min;94 ℃ 1 min,55 ℃ 30 s,72 ℃ 1 min,共30个循环;72 ℃ 10 min。得到的PCR产物用琼脂糖凝胶电泳检测,并送至生工生物工程(上海)股份有限公司测序,经过Blast比对,确认其是否目的片段。重金属及抗生素抗性基因型对照信息见表 2。
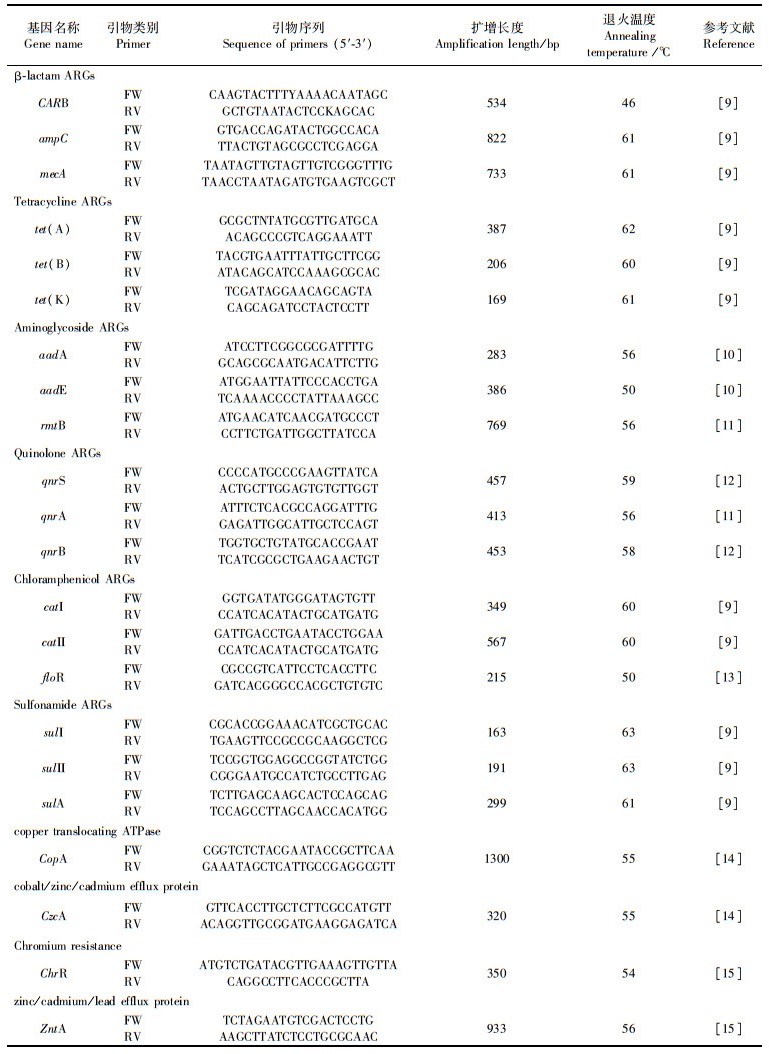
|
表 2 重金属及抗生素抗性基因信息表 Tab.2 Information table for resistance genes of heavy metals and antibiotics |
所有引物均由上海生工生物工程有限公司合成。
1.5 数据处理用SPSS 26.0进行统计分析,并使用Origin 2021对数据进行处理和制图。
2 结果与分析 2.1 牡蛎中微生物菌群多样性分析通过生信云分析平台对测序结果从种的水平进行分析(图 2)。以样本名为纵坐标,菌群物种在该样本中所占的比例为横坐标,柱颜色代表不同的菌群物种,柱长度代表该菌群物种所占比例大小。由图 2可知,含量占比前5位的细菌为不可培养的拟杆菌(uncultured Bacteroides)、未分类的芽孢杆菌(unclassified Bacillus cohn)、未分类的假单胞菌(unclassified Pseudomonas)、河流弧菌(Vibrio fluvialis)和嗜盐杆菌斯氏弓形菌(Arcobacter skirrowii),包含了2类腐败菌属(拟杆菌、假单胞菌)和1类致病菌属(弧菌)。拟杆菌是水产品加工过程中易感染的腐败菌,假单胞菌是冷藏水产品中的优势腐败菌,弧菌是新鲜水产品中的优势腐败菌以及致病菌[16]。

|
图 2 牡蛎中微生物菌群多样性分析 Fig. 2 Diversity analysis of microbial flora in oysters |
利用主成分分析(PCA)分析7份样本菌群组成差异(图 3)可以看出明显的因地理距离造成的显著菌群差异。其中ZJTZ(浙江台州)样品与其他样本菌群差异最为显著。同时从图 2中也可以看出,ZJTZ样品中优势菌群为芽孢杆菌,其他样品优势菌群则是多形拟杆菌。

|
图 3 不同产地牡蛎细菌多样性PCA分析 Fig. 3 PCA analysis of intestinal flora diversity of oysters from different habitats |
从ARGs的检出率来看:tet(A)、tet(B)、aadA、gryA、floR和sulⅡ的检出率为100.0%,sulⅠ的检出率为95.2%,catⅡ的检出率为90.5%。从ARGs的种类来看:氨基糖苷类ARGs检出率最高(84.1%),β-内酰胺类ARGs和喹诺酮类ARGs检出率最低(57.1%);具体为氨基糖苷类ARGs>氯霉素类ARGs>磺胺类ARGs>四环素类ARGs>β-内酰胺类ARGs/喹诺酮类ARGs。
从HTGs的检出率来看:CopA的检出率为71.4%,ChrR的检出率为23.8%,CzcA和ZntA未检出,HTGs的整体检出率为23.8%。
2.2.1 从牡蛎不同品种层面进行分析褶牡蛎和长牡蛎在抗性基因种类及丰度上差异不显著。在进行PCA分析后发现长牡蛎的抗性基因分布较为集中,表明抗性基因种类较为相似。而褶牡蛎的抗性基因分布较为分散,其中样品ZJTZ-I和SDWH1-G呈现明显离散。见图 4。
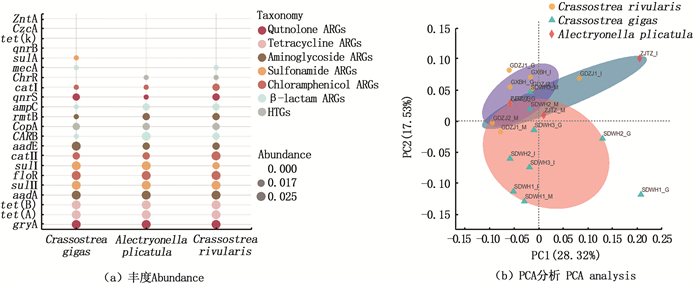
|
图 4 牡蛎不同品种抗性基因丰度及PCA分析 Fig. 4 Analysis of resistance genes abundance and PCA of different oyster varieties |
以牡蛎不同组织器官分析抗性基因:肌肉中检出率(65.6%)>腮中检出率(59.1%)>肠道中检出率(58.4%),但在抗性基因种类及丰度上差异不显著。肌肉中较高的检出率也提醒人们牡蛎的食用安全风险不容忽视。在进行PCA分析后发现肌肉中的抗性基因分布较为集中,表明抗性基因种类较为相似。而腮和肠道中的抗性基因分布较为分散,其中样品ZJTZ-I和SDWH1-G呈现明显离散。见图 5。
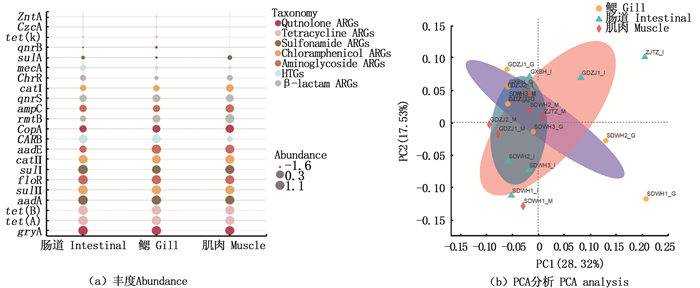
|
图 5 牡蛎不同组织器官抗性基因丰度及PCA分析 Fig. 5 Analysis of resistance genes abundance and PCA of different oyster tissues |
以不同样品来源分析抗性基因检出率:GXBH样品整体检出率最高(72.7%),SDWH1整体检出率最低(54.5%);具体为GXBH>GDZJ2>SDWH3>SDWH2/GDZJ1>ZJTZ>SDWH1。在抗性基因种类及丰度上各样品差异不显著。在进行PCA分析后发现同一样品可以进行明显的归一化分类,说明同一样品不同组织器官中抗性基因关联性较大。从离散程度来看,SDWH3和GDZJ2两个样品分布较为集中。见图 6。

|
图 6 牡蛎不同样品来源抗性基因丰度及PCA分析 Fig. 6 Analysis of resistance genes abundance and PCA of different oyster sample sources |
从不同抗性基因ARGs和HTGs的相关性分析(图 7)来看:CARB和qnrS相关性最高,ChrR则与sulA和mecA有较大相关性,CopA与各大类ARGs都有一定相关性,说明不同类型抗性基因有存在共用一个基因盒的可能性[17]。从对不同来源样品的相关性分析(图 7横坐标)来看,基本遵循了以不同产地样品进行归一化的分类,说明养殖环境可以对牡蛎中抗性基因的分布产生影响。牡蛎作为滩涂或者近海养殖的水产品,环境中排放的包括水产养殖环境中大量投放含重金属的促生长剂和杀菌剂、工业废水、生活废水等,这些环境因素对对牡蛎中抗性基因的分布产生的影响值得进一步研究。

|
图 7 牡蛎肠道、腮、肌肉中ARGs和HTGs Jaccard相关性分析 Fig. 7 Jaccard correlation analysis of of ARGs and HTGS in intestine, gill and muscle of oyster |
陈慧斌等[18]研究了4 ℃冷藏期间牡蛎鳃部菌群的动态变化,发现冷藏后期主要腐败优势菌群为假单胞菌属和气单胞菌属等。但目前对于水产品中肠道细菌种群特征的研究甚少,根据本文在牡蛎细菌多样性研究结果,拟杆菌、假单胞菌和弧菌3种菌在牡蛎中占比均很高,推断这些微生物是引起水产品腐败变质的主要污染源之一。新鲜牡蛎因其自身蛋白质含量较高,更有利于微生物的繁殖[19],进而影响牡蛎的K值、TVB-N值、质构和颜色等,因此微生物菌群可作为反映水产品新鲜度和腐败程度的一项重要指标[20]。同时,一些致病微生物会直接进入人体消化道导致一系列肠胃疾病[21]。因此,食用牡蛎前要注意流通运输过程中的冷藏保鲜,避免由有害微生物过量生长导致腐败引发的胃肠道疾病[22]。
在抗性基因多样性的分析中所有样本均检测到了大量的多重抗性基因,说明ARGs等抗性基因在水产品中广泛存在着,污染程度比较严重。研究人员在韩国水产品中检测出喹诺酮类抗性基因qnrS5、氯霉素抗性基因catA1和catB以及万古霉素抗性基因vanB[23-24]。除此之外,还在水产品中检测出了临床上医用抗生素相关的抗性基因[25]。随着养殖规模的扩大,养殖人员为了获得更高的经济利益,在水产养殖中便开始大规模的使用抗生素,引起水产环境中存在ARGs, 且种类多丰度高,而水产相关环境是水产品中ARGs的主要来源之一,所以对水产相关环境中ARGs的检测也是必不可少的。除此之外,水产养殖环境中抗生素污染不只是单一的。黄志坚等[26]通过PCR的方法分析了来自国内各个水产养殖环境中分离的139株细菌,发现这些细菌携带了超过4种ARGs,按丰度排序依次为氟氯霉素抗性基因(floR)、磺胺类抗性基因(sulⅠ、sulⅡ) 以及链霉素抗性基因(str),它们出现率均超过10%。此外甲氧苄啶抗性基因(dfrA20) 也有检出,检出率仅为2.88%,这表明水产养殖生物和养殖环境中存在不同程度的抗生素污染。
崔丁心等[27]对从牡蛎肌肉中分离出的25株细菌进行抗生素与重金属的交叉抗性研究,结果发现有13株出现了交叉抗性。微生物的基因组上天然存在某些ARGs[28],但种类较少,丰度较低,尚不构成水产品及相关环境中ARGs产生的主要原因。ARGs的获得和传递主要体现在生物体与环境的相互影响上。水产品的产地环境是水体,而环境中的ARGs会直接或间接的进入水体中,在水产品自身残留或传播,从而使水产品在源头上受到污染。此外,医疗农业上的抗生素可以直接影响环境中的原生菌,使其产生耐药性,并通过自然循环进入水体,造成水产品的污染。进入水产品中,携有ARGs的裸漏DNA可在其中长期存在,并且在相应的加工环节中依旧不会消失,而水产品中ARGs可通过食物链再次进入生物体内,开始又一次的恶性循环[29]。抗生素的过度使用和长期滥用污染了养殖水体,造成了水产品中ARGs的形成与传播、种类和丰度增加,对人类健康产生了严重隐患。但我国对于ARGs在水产品中来源、传播、扩散等研究并不详尽,并且没有引起足够重视。结合本文研究,发现牡蛎肌肉中虽然细菌多样性没有肠道等组织丰富,但耐药菌以及耐药基因含量却很高,提醒人们牡蛎的食用安全风险不容忽视。
本研究中牡蛎宏基因组中ARGs和HTGs呈现出对应性,说明重金属污染作为一种环境压力能够刺激菌株运行协同调控机制,通过协同调控ARGs的表达增强菌株的耐药性,重金属离子也可以协同调控ARGs降低菌株对抗生素的敏感度。环境中抗生素的存在会导致ARGs的产生,但有研究[30]表明,即使环境中不含抗生素,依然存在ARGs的产生和转移,这与重金属有着密切联系。这也可以解释细菌耐药表型与基因型大部分情况下不对应的现象。重金属污染作为一种环境压力能够刺激菌株运行协同调控机制,通过协同调控ARGs的表达增强菌株的耐药性;重金属离子也可以协同调控ARGs降低菌株对抗生素的敏感度[31]。LI等[32]通过Spearman相关分析和Mantel试验表明,重金属富集对微生物群落、ARGs和MRGs有显著影响。
总之,目前研究中抗生素的过度使用与耐药细菌的频繁检出密切相关,而重金属污染也影响着环境中抗性基因的选择。现如今对环境中抗生素的抗性研究主要集中在抗性基因的定性定量和一些相应抗性菌株的检测等,大多数的研究比较单一,缺乏整体性和系统性,而现实中,细菌主要生存在于各种因素相互影响相互作用的复杂生态环境中,例如多重抗生素污染,抗生素重金属复合污染等。所以,在以后的研究中除了更加深入研究抗生素ARGs在环境中传播扩散机制以外,也要注重对多重抗性、微生物的重金属和抗生素的抗性机制以及抗性基因组学的研究。
| [1] |
农业农村部渔业渔政管理局, 全国水产技术推广总站, 中国水产学会. 中国渔业统计年鉴-2021[M]. 北京: 中国农业出版社, 2021. Fishery Administration of the Ministry of Agriculture and Rural Areas, National Fisheries Technology Extension Center, China Society of Fisheries. 2021 China fisheries statistical yearbook[M]. Beijing: China Agricultural Press, 2021. |
| [2] |
国家贝类产业技术体系. 中国牡蛎产业发展报告[J]. 中国水产, 2021(6): 20-31. National Shellfish Industry Technology System. China oyster industry development report[J]. China Fisheries, 2021(6): 20-31. |
| [3] |
YE L, LU Z, LI X H, et al. Antibiotic-resistant bacteria associated with retail aquaculture products from Guangzhou, China[J]. Journal of Food Protection, 2013, 76(2): 295-301. DOI:10.4315/0362-028X.JFP-12-288 |
| [4] |
LEVY S B, MARSHALL B. Antibacterial resistance worldwide: causes, challenges and responses[J]. Nature Medicine, 2004, 10(12 S): S122-S129. |
| [5] |
ALLEN H K, DONATO J, WANG H H, et al. Call of the wild: antibiotic resistance genes in natural environments[J]. Nature Reviews Microbiology, 2010, 8(4): 251-259. DOI:10.1038/nrmicro2312 |
| [6] |
ZHU D, XIANG Q, YANG X R, et al. Trophic Transfer of antibiotic resistance genes in a soil detritus food chain[J]. Environmental Science & Technology, 2019, 53(13): 7770-7781. |
| [7] |
STEPANAUSKAS R, GLENN T C, JAGOE C H, et al. Coselection for microbial resistance to metals and antibiotics in freshwater microcosms[J]. Environmental Microbiology, 2006, 8(9): 1510-1514. DOI:10.1111/j.1462-2920.2006.01091.x |
| [8] |
JO S, SHIN C, SHIN Y, et al. Heavy metal and antibiotic co-resistance in Vibrio parahaemolyticus isolated from shellfish[J]. Marine Pollution Bulletin, 2020, 156: 111246. DOI:10.1016/j.marpolbul.2020.111246 |
| [9] |
JIANG L, HU X L, XU T, et al. Prevalence of antibiotic resistance genes and their relationship with antibiotics in the Huangpu River and the drinking water sources, Shanghai, China[J]. Science of the Total Environment, 2013, 458-460: 267-272. DOI:10.1016/j.scitotenv.2013.04.038 |
| [10] |
OUOBA L I I, LEI V, JENSEN L B. Resistance of potential probiotic lactic acid bacteria and bifidobacteria of African and European origin to antimicrobials: determination and transferability of the resistance genes to other bacteria[J]. International Journal of Food Microbiology, 2008, 121(2): 217-224. DOI:10.1016/j.ijfoodmicro.2007.11.018 |
| [11] |
HUANG S F, DAI W, SUN S, et al. Prevalence of plasmid-mediated quinolone resistance and aminoglycoside resistance determinants among carbapeneme non-susceptible Enterobacter cloacae[J]. PLoS One, 2012, 7(10): e47636. DOI:10.1371/journal.pone.0047636 |
| [12] |
XU J, XU Y, WANG H M, et al. Occurrence of antibiotics and antibiotic resistance genes in a sewage treatment plant and its effluent-receiving river[J]. Chemosphere, 2015, 119: 1379-1385. DOI:10.1016/j.chemosphere.2014.02.040 |
| [13] |
MAYNARD C, FAIRBROTHER J M, BEKAL S, et al. Antimicrobial resistance genes in enterotoxigenic Escherichia coli O149∶K91 isolates obtained over a 23-year period from pigs[J]. Antimicrobial Agents & Chemotherapy, 2003, 47(10): 3214-3221. |
| [14] |
BOUSKILL N J, BARNHART E P, GALLOWAY T S, et al. Quantification of changing Pseudomonas aeruginosa sodA, htpX and mt gene abundance in response to trace metal toxicity: a potential in situ biomarker of environmental health[J]. FEMS Microbiology Ecology, 2007, 60(2): 276-286. DOI:10.1111/j.1574-6941.2007.00296.x |
| [15] |
HE Y, JIN L L, SUN F J, et al. Antibiotic and heavy-metal resistance of Vibrio parahaemolyticus isolated from fresh shrimps in Shanghai fish markets, China[J]. Environmental Science and Pollution Research, 2016, 23(15): 15033-15040. DOI:10.1007/s11356-016-6614-4 |
| [16] |
蓝蔚青, 王蒙, 陈梦玲, 等. 水产品特定腐败菌群体感应及其植物源抑制剂研究进展[J]. 食品与机械, 2018, 34(9): 185-190. LAN W Q, WANG M, CHEN M L, et al. Research progress of specific spoilage organisms quorum sensing and plant-source inhibitors in aquatic products[J]. Food and Machinery, 2018, 34(9): 185-190. |
| [17] |
JIANG H, YU T, YANG Y T, et al. Co-occurrence of antibiotic and heavy metal resistance and sequence type diversity of Vibrio parahaemolyticus isolated from Penaeus vannamei at freshwater farms, seawater farms, and markets in Zhejiang province, China[J]. Frontiers in Microbiology, 2020, 11: 1294. DOI:10.3389/fmicb.2020.01294 |
| [18] |
陈慧斌, 刘智禹, 陈绍军, 等. 基于PCR-DGGE技术的冷藏牡蛎鳃部菌群分析[J]. 西南大学学报(自然科学版), 2013, 35(4): 151-156. CHEN H B, LIU Z Y, CHEN S J, et al. On characterization of gill associated bacterial flora in chilled stored oyster based on PCR-DGGE[J]. Journal of Southwest University (Natural Science Edition), 2013, 35(4): 151-156. |
| [19] |
付娇娇, 彭织云, 刘海泉, 等. 酸性电解水处理后南美白对虾贮藏过程中肠道微生物的多样性变化[J]. 食品工业科技, 2015, 36(4): 344-347, 356. FU J J, PENG Z Y, LIU H Q, et al. Changes of acidic electrolyzed water on intestinal microflora diversity of Penaeus vannawei during storage[J]. Science and Technology of Food Industry, 2015, 36(4): 344-347, 356. |
| [20] |
PRAPAIWONG N, WALLACE R K, ARIAS C R. Bacterial loads and microbial composition in high pressure treated oysters during storage[J]. International Journal of Food Microbiology, 2009, 131(2/3): 145-150. |
| [21] |
沈安梅. 上海市南汇区2007年感染性腹泻病监测分析[J]. 上海预防医学杂志, 2008, 20(12): 592-593. SHEN A M. Surveillance and analysis of infectious diarrhea in Nanhui District of Shanghai in 2007[J]. Shanghai Journal of Preventive Medicine, 2008, 20(12): 592-593. |
| [22] |
励建荣. 海水鱼类腐败机制及其保鲜技术研究进展[J]. 中国食品学报, 2018, 18(5): 1-12. LI J R. Research progress on spoilage mechanism and preservation technology of marine fish[J]. Journal of Chinese Institute of Food Science and Technology, 2018, 18(5): 1-12. |
| [23] |
HAN J E, KIM J H, CHERESCA C H JR, et al. First description of the qnrS-like (qnrS5) gene and analysis of quinolone resistance-determining regions in motile Aeromonas spp. from diseased fish and water[J]. Research in Microbiology, 2012, 163(1): 73-79. |
| [24] |
KIM M, KWON T H, JUNG S M, et al. Antibiotic resistance of bacteria isolated from the internal organs of edible snow crabs[J]. PLoS One, 2013, 8(8): e70887. |
| [25] |
娄阳, 张昭寰, 肖莉莉, 等. 食品源抗生素抗性基因的来源与分布状况研究进展[J]. 食品工业科技, 2015, 36(12): 368-374. LOU Y, ZHANG Z H, XIAO L L, et al. Research progress of source and distribution of antibiotic resistance genes in food[J]. Science and Technology of Food Industry, 2015, 36(12): 368-374. |
| [26] |
黄志坚, 陈旭凌, 路晓峰, 等. 水产养殖生物和养殖环境细菌鉴定及抗生素抗性基因检测[J]. 中山大学学报(自然科学版), 2012, 51(6): 92-96. HUANG Z J, CHEN X L, LU X F, et al. Identification and antibiotic resistance genes detection of bacteria in aquaculture organisms and aquatic environment[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2012, 51(6): 92-96. |
| [27] |
崔丁心, 陈玲, 陈慧, 等. 东寨港河口牡蛎中重金属-抗生素抗性细菌分析[J]. 海南师范大学学报(自然科学版), 2016, 29(3): 263-267, 273. CUI D X, CHEN L, CHEN H, et al. Analysis of resistant bacteria of antibiotic and heavy metal in oysters from Dongzhaigang estuary[J]. Journal of Hainan Normal University (Natural Science), 2016, 29(3): 263-267, 273. |
| [28] |
D'COSTA V M, KING C E, KALAN L, et al. Antibiotic resistance is ancient[J]. Nature, 2011, 477(7365): 457-461. |
| [29] |
DAANE L L, MOLINA J, SADOWSKY M J. Plasmid transfer between spatially separated donor and recipient bacteria in earthworm-containing soil microcosms[J]. Applied and Environmental Microbiology, 1997, 63(2): 679-686. |
| [30] |
SALYERS A A, AMÁBILE-CUEVAS C F. Why are antibiotic resistance genes so resistant to elimination?[J]. Antimicrobial Agents & Chemotherapy, 1997, 41(11): 2321-2325. |
| [31] |
BAKER-AUSTIN C, WRIGHT M S, STEPANAUSKAS R, et al. Co-selection of antibiotic and metal resistance[J]. Trends in Microbiology, 2006, 14(4): 176-182. |
| [32] |
LI Y Z, CHEN H Y, SONG L T, et al. Effects on microbiomes and resistomes and the source-specific ecological risks of heavy metals in the sediments of an urban river[J]. Journal of Hazardous Materials, 2021, 409: 124472. |
2. Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China;
3. Laboratory of Food Quality and Safety Testing, Shanghai Ocean University, Shanghai 201306, China;
4. Hohhot Agricultural and Animal Products Quality Safety Center, Hohhot 010020, Nei Mongol, China;
5. Shanghai Engineering Research Center of Aquatic Product Processing & Preservation, Shanghai 201306, China
 2022,
Vol. 31
2022,
Vol. 31


