2. 夏威夷太平洋大学 自然科学系, 檀香山 96813
20世纪90年代之前,人们普遍认为生物圈是指地表生物圈, 它分布在地表附近,范围大约为海平面上下垂直10 km,依靠太阳辐射获取能量。直到90年代初,奥地利科学家GOLD[1]在美国国家科学院院刊发表题为“深部热生物圈”的论文,指出除经典地表生物圈外,于地下约6~10 km的高温、高压的极端环境中,还存在另外一个生物圈,即“深部热生物圈”。研究[2]表明,深部生物圈中所含生物量非常大,约占地球总生物量的1/2~2/3。海洋深部生物圈(Sub-seafloor deep biosphere)是深部生物圈的组成部分,通常指栖息在海底深部(距海床表面1.5 m之下) 沉积物和洋壳中的生物群系[3-4],主要由微生物构成[5-6]。2012年的IODP 337航次,是目前为数不多的针对海底深部生物圈的大洋钻探航次。该航次在日本下北半岛C0020A站点进行了2 466 mbsf的钻井作业,取得了丰富的海底深部生物圈沉积物样品。地质学研究表明,C0020A在中新世早期是一片茂盛的海岸森林,在漫长的地质过程中随太平洋板块俯冲而逐渐下沉,形成富含有机质的弧前盆地并被海水吞没,最终形成了以砂岩、粉砂岩和炭质页岩为主的褐煤层(1.5~2.4 kmbsf)[7]。岩石学分析发现,含煤层以砂岩、粉砂岩和炭质页岩为主,有机成分以木屑煤为主,其中总有机碳(TOC)含量高达30%,并含有大量花粉和芽孢[8]。INAGAKI等[8]测定了煤层的含水量、颜色和镜质体反射率,结果表明煤层中的煤为成熟度较低的褐煤,呈生物扰动和压扁层理、透镜状层理或交错层理等沉积特征,表明煤层环境具有明显的陆相特征,为类似潮坪的近岸沉积环境[8]。此外,生物学研究表明,煤层中的微生物数量远高于紧邻的上层沉积物[8-9],且群落结构与陆地表层土壤环境中相似[9],以厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、变形菌门(Proteobacteria)、酸杆菌门(Acidobacteria)和拟杆菌门(Bacteroidetes)为主[10]。表明煤层中部分微生物可能为陆生来源,提示部分陆源微生物在进化过程中可能逐渐适应了煤层的营养和压力环境,并对深部生物圈中的营养要素循环起到重要作用。
1950年,PROOM等[11]从土壤样本中分离得到1株细菌,命名为Bacillus pantothenticus(泛酸芽孢杆菌)。1998年,HEYNDRICKX[12]通过扩增性rDNA限制性酶切片段分析(amplified rDNA restriction analysis, ARDRA),将泛酸芽孢杆菌与芽孢杆菌属的其他种区别开来,并划分为一个新属“Virgibacillus”(枝芽孢杆菌属)。研究发现,枝芽孢杆菌属在环境中分布十分广泛,从海洋到陆地[13-14],从动植物到人体肠道[15-16]均有发现。从目前已发表的有关数据来看,糖类是该属的主要碳源[10]。
FANG等[10]从IODP 337航次中获得深部煤层沉积物样品,并首次从中分离到枝芽孢杆菌属菌株,经鉴定命名为Virgibacillus pantothenticus(泛酸枝芽孢杆菌)19R1-5(19R)。有趣的是,19R与分离自土壤的同种菌株26T的16S rRNA基因序列相似度高达100%,DNA杂交同源性高达91.7%[10],表明19R可能来源于陆生环境。有研究表明高压环境明显增加微生物对能量及营养物质的需求[17],然而19R所处的煤层环境中惰性有机物含量较高,容易利用的有机物含量较低[8, 18]。那么,19R作为以糖类为主要碳源的微生物,是如何适应原位特殊的营养环境以及高压所造成的能量需求增加的呢?本研究对菌株19R和26T进行了基因组测序,首先通过建立全基因组系统发育树确定19R的来源,继而对二者代谢网络和转运蛋白进行比较,分析19R对糖类营养源的特殊利用能力。此外,本文也将对枝芽孢杆菌属的泛基因组构成进行分析,探讨19R能够在环境变迁中存活的分子基础。
1 材料和方法 1.1 实验材料菌株19R和26T分别购自日本微生物保藏中心(Japan Collection of Microorganisms,JCM)和中国海洋微生物菌种保藏管理中心(Marine Culture Collection of China,MCCC)。
1.2 菌株培养菌株活化后,挑取单菌落进行摇床扩繁(35 ℃、200 rpm/min),培养基为海洋肉汤2216E。培养至OD600值为1.2,取菌液50 mL,12 000 r/min,室温离心10 min,收集菌体。送上海美吉生物医药科技有限公司进行全基因组测序。其中26T采用第二代、第三代测序技术相结合的方法测基因组完成图,19R采用第二代测序技术测基因组扫描图。
1.3 生物信息学分析 1.3.1 ORF预测及基因组注释将两株菌的基因组序列分别上传至NCBI数据库并使用PGAP流程(https://www.ncbi.nlm.nih.gov/genome/annotation_prok/)进行ORF预测、tRNA和rRNA基因预测及蛋白序列注释。蛋白质信号肽由SignalP[19]在线预测(http://www.cbs.dtu.dk/services/SignalP/)。COG注释结果通过psi-blast[20]与COG数据库比对获得,参数设置:identities 50%, coverage 50%, e-value 1×10-5。
1.3.2 系统发育分析使用GTDB-tk[21]软件对细菌120个单拷贝基因进行预测和多序列比对,参数为--locapair、--maxiterae 100。提取并去除多序列比对结果中含有50%以上gap的碱基,使用Fasttree[22]建立最大似然性进化树,并利用iTOL[23]进行系统发育树的可视化。
1.3.3 代谢网络的重建使用KEGG BlastKOALA对基因组中所有蛋白序列进行在线注释(https://www.kegg.jp/blastkoala/)和代谢网络重建。
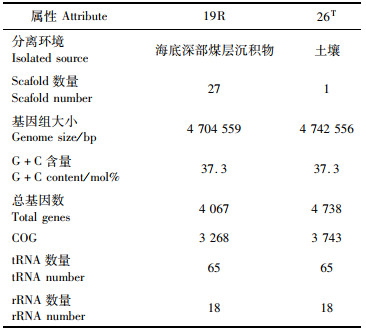
2 结果 2.1 19R和26T的基因组特征我们对26T采取二代、三代结合的方法,对19R采用二代的方法测序,经拼接获得26T的基因组完成图及19R的基因组草图, 基本信息如表 1所示。
|
表 1 19R和26T分离环境和基因组基本信息 Tab.1 19R and 26T isolated environmental and basic information of genomics |
19R的基因组草图为4 704 559 bp,包含27个Scafold,GC含量仅为37.3 mol%。经ORF预测,该基因组编码4 067个蛋白编码基因,其中有3 268个可被COG注释。碳水化合物的转运和代谢(COG-G)、转录(COG-K)以及氨基酸的转运和代谢(COG-E)在COG功能分类中均占绝大多数。26T的基因组由一个环状DNA构成,不含质粒,GC含量为37.3 mol%,基因组全长为4 742 556 bp,预测包含4 738个蛋白编码基因,其中3 743个可被COG注释,各COG分类的数目与19R基因组十分接近。此外,26T基因组编码了65个tRNA和18个rRNA。
值得注意的是,我们在26T中发现3种序列异质性的16S rRNA序列(拷贝数分别为4、1和1),相互间有若干单核苷酸多态性(SNP)的存在。鉴于二代测序读长较短,难以获得19R基因组的上述SNP,我们使用26T的各16S rRNA侧翼序列作为参考设计特异性引物(表 2),并结合16S rRNA通用引物对19R的16S rRNA基因序列进行巢式PCR扩增及测序。结果表明,二者对应的16S rRNA序列均完全一致,由此我们推测二者具有极高的亲缘关系。

|
表 2 19R的16S rRNA基因PCR所用引物 Tab.2 Primers for PCR of 16S rRNA gene in 19R |
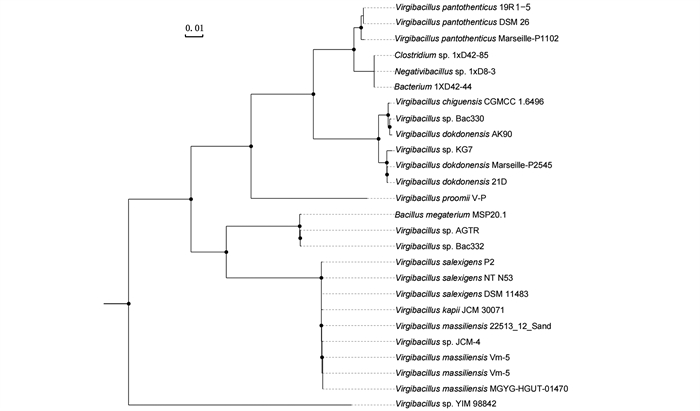
为研究19R的来源与演化,我们根据原核生物依据基因组分类数据库(GTDB)的120个保守蛋白序列,对NCBI RefSeq数据库中Firmicutes门全部基因组重新进行系统发育分析,确定了25个种属于枝芽孢杆菌属,其中包括4株分类错误的菌株(图 1)。重要的是,19R在系统发育树中与V. pantothenticus DSM 26, V. pantothenticus Marseille-P1102, Bacterium 1XD42-44, Clostridium sp. 1xD42-85和Negativibacillus sp. 1xD8-3等5株菌处于同一进化分支,而这5株菌均分离自陆地环境。表明19R极可能源自陆生环境,并在漫长的地壳运动中,与陆源有机质一起沉降、包埋,最终成为海洋沉积物中的微生物。
|
黑色的点表示bootstrap值在60~100。 The black dots indicate a bootstrap value from 60 to 100. 图 1 枝芽孢杆菌属的系统发育树 Fig. 1 Phylogenetic tree of Virgibacillus |
为研究19R对原位特殊营养环境的适应性机制,我们对19R和26T进行了代谢网络的重塑和比较。分析发现,二者均具有完整的糖酵解、三羧酸循环和磷酸戊糖途径,但D-山梨醇(D-sorbitol)代谢相关的6-磷酸山梨醇-2-脱氢酶仅存在于19R中(图 2),表明19R可能具有比26T更为广泛的糖类利用能力。

|
外圈椭圆表示物质运输系统;加粗文字和虚线分别表示19R特异性的底物和通路。 The outer circle ellipse represents the material transport systems; The bold characters and dotted lines indicate 19R-specific substrate and pathway. 图 2 根据KEGG预测的19R的代谢网络图 Fig. 2 Metabolic network of 19R predicted according to KEGG substrates and pathways, respectively |
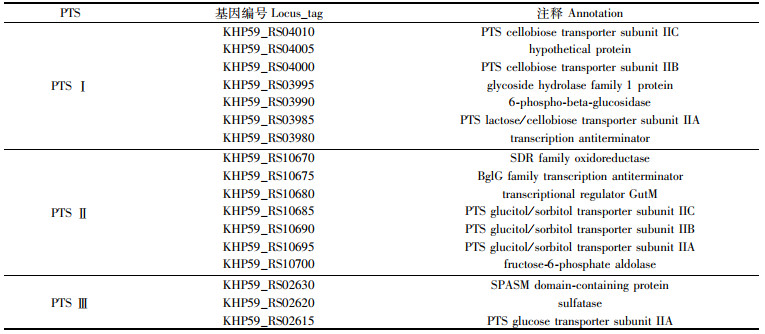
继而,我们对19R和26T的蛋白序列进行all-versus-all比对。结果显示,3 779个蛋白家族为二者共有,而有314个为19R独有,有352个为26T独有(BLASTp参数:identities 50%,coverage 50%,e-value 1×10-10)。对这些菌株特异性基因进行KEGG注释,我们发现19R除拥有26T的全部磷酸葡萄糖转移酶系统(PTS)以外,还拥有3个特殊的PTS,预测其糖类底物分别为lactose/cellobiose(乳糖/纤维二糖)、glucitol/sorbitol(山梨糖醇/山梨醇)和glucose(葡萄糖), 见表 3。该结果与FANG等[10]报道的19R具有更广泛的糖利用能力的结果相一致。
|
表 3 19R相对于26T特有的磷酸葡萄糖转移酶系统(PTS) Tab.3 Specific phosphoglucose transferase system (PTS) of 19R relative to 26T |
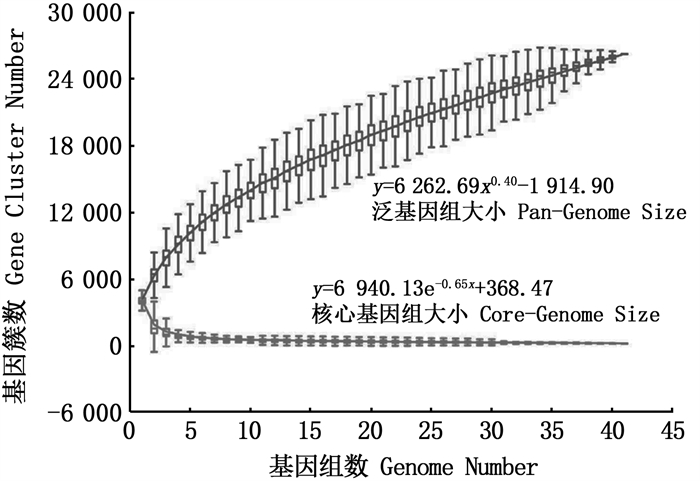
为研究19R对比于26T所特有的糖类转运基因的来源,我们对上述进化树中全部菌株的蛋白序列进行all-versus-all的比对,并使用OrthoMCL[24]软件进行聚类(BLASTp参数同上,mcl Inflation为1.4)。结果表明,枝芽孢杆菌属泛基因组(Pan-genome)共含有26 246个蛋白家族, 泛基因组大小与基因组数目拟合关系如图 3所示。值得注意的是,上述3个糖类转运蛋白所在的蛋白家族并非19R独有,这表明这些糖类转运相关基因为26T所丢失,而非19R所特异性获得。此外,在19R所在分支的6个近缘菌株中,仅19R同时拥有这3个转运体蛋白,说明保留较多的糖类转运基因可能是19R能够在环境变迁中生存的原因之一。
|
图 3 枝芽孢杆菌属泛基因组分析 Fig. 3 Pan-genomic analysis of the genus Virgibacillus |
关于日本下北半岛C0020A站位下的深部煤层微生物的来源,存在诸多假说[7-9, 16]。经过近十年的研究,科学家们从地质学和生物学的角度证明了该煤层微生物为陆生来源[7-9]。本研究首次从系统发育的角度,确定分离自该煤层的菌株19R为陆生来源,为上述观点提供了新的证据。本研究表明,表层生物圈与深部生物圈之间存在一定关联,表明深部微生物可能影响海洋乃至深部生物圈中的元素循环过程。
微生物对极端环境的适应性的机制是生态学的重要问题之一。STARNAWSKI等[25]通过PCR扩增测序和宏基因组测序技术对丹麦奥尔胡斯湾(Aarhus Bay)4个站点的沉积物进行了研究,发现原本在陆地环境中丰度不高的微生物群落随地质活动被埋藏到海底深层沉积物中并逐渐占据主导地位,但它们的基因组鲜有突变[25]。然而,该研究并未就水平基因转移在极端适应性方面的作用进行充分研究。在本研究中,我们发现在属水平上19R的3个的PTS并非该菌株所独有,这表明19R广泛的糖利用能力并非从沉积物环境中通过水平转移获得。然而,19R所在分支的6个近缘菌株中,仅19R同时拥有这3个PTS,说明极端环境造成的选择压力促使19R保留更广泛的糖类利用基因。值得说明的是,由于19R并未测得基因组完成图,我们只对PTS基因的类别进行统计,但无法确定其拷贝数目,故不能确定基因扩增是否在19R适应深部煤层环境的过程中起到作用。未来,获得19R的基因组完成图将有助于回答上述问题。
综上,本研究揭示了分离自海底深部生物圈的19R为陆生来源,随陆源有机质一起被埋藏至海底深部。19R保留的较广泛的糖类利用能力可能是其适应海底深部特殊营养环境的分子基础之一。
| [1] |
GOLD T. The deep, hot biosphere[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(13): 6045-6049. DOI:10.1073/pnas.89.13.6045 |
| [2] |
WHITMANWB, COLEMAN D C, WIEBE W J. Prokaryotes: the unseen majority[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583. DOI:10.1073/pnas.95.12.6578 |
| [3] |
D'HONDT S, JØRGENSEN B B, MILLER D J, et al. Distributions of microbial activities in deep subseafloor sediments[J]. Science, 2004, 306(5705): 2216-2221. DOI:10.1126/science.1101155 |
| [4] |
方家松, 张利. 探索深部生物圈[J]. 中国科学: 地球科学, 2011, 54(6): 750-759. FANG J S, ZHANG L. Exploring the deep biosphere[J]. Science China Earth Sciences, 2011, 54(2): 157-165. DOI:10.3969/j.issn.0253-2778.2011.02.010 |
| [5] |
D'HONDTS, RUTHERFORD S, SPIVACK A J. Metabolic activity of subsurface life in deep-sea sediments[J]. Science, 2002, 295(5562): 2067-2070. DOI:10.1126/science.1064878 |
| [6] |
董海良. 深地生物圈的最新研究进展以及发展趋势[J]. 科学通报, 2018, 63(36): 3883-3901. DONG H L. Recent developments and future directions of deep biosphere research[J]. Chinese Science Bulletin, 2018, 63(36): 3883-3901. |
| [7] |
JØRGENSEN B B. Microbial life in deep subseafloor coal beds[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(44): 11568-11570. DOI:10.1073/pnas.1716232114 |
| [8] |
INAGAKI F, HINRICHS K U, KUBO Y, et al. IODP expedition 337: deep coalbed biosphere off Shimokita-microbial processes and hydrocarbon system associated with deeply buried coalbed in the ocean[J]. Scientific Drilling, 2016, 21: 17-28. DOI:10.5194/sd-21-17-2016 |
| [9] |
INAGAKI F, HINRICHS K U, KUBO Y, et al. Exploring deep microbial life in coal-bearing sediment down to 2.5 km below the ocean floor[J]. Science, 2015, 349(6246): 420-424. DOI:10.1126/science.aaa6882 |
| [10] |
FANG J S, KATO C, RUNKO G M, et al. Predominance of viable spore-forming piezophilic bacteria in high-pressure enrichment cultures from 1.5 to 2.4 km-deep coal-bearing sediments below the ocean floor[J]. Frontiers in Microbiology, 2017, 8: 137. |
| [11] |
PROOM H, KNIGHT B C J G. Bacillus pantothenticus (n.sp.)[J]. Journal of General Microbiology, 1950, 4(3): 539-541. DOI:10.1099/00221287-4-3-539 |
| [12] |
HEYNDRICKX M. Virgibacillus: a new genus to accommodate Bacillus pantothenticus (Proom and Knight 1950). Emended description of Virgibacillus pantothenticus[J]. International Journal of Systematic Bacteriolog, 1998, 48(1): 99-106. DOI:10.1099/00207713-48-1-99 |
| [13] |
KÄMPFER P, ARUN A B, BUSSE H J, et al. Virgibacillus soli sp. nov., isolated from mountain soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(2): 275-280. DOI:10.1099/ijs.0.020974-0 |
| [14] |
YIN X J, YANG Y L, WANG S, et al. Virgibacillusoceani sp. nov.isolated from ocean sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(1): 159-164. |
| [15] |
KHELAIFIA S, CROCE O, LAGIER J C, et al. Noncontiguous finished genome sequence and description of Virgibacillus massiliensis sp. nov., a moderately halophilic bacterium isolated from human gut[J]. New Microbes and New Infections, 2015, 8: 78-88. DOI:10.1016/j.nmni.2015.09.012 |
| [16] |
TANASUPAWAT S, CHAMROENSAKSRI N, KUDO T, et al. Identification of moderately halophilic bacteria from Thai fermented fish (pla-ra) and proposal of Virgibacillus siamensis sp. nov.[J]. The Journal of General and Applied Microbiology, 2010, 56(5): 369-379. DOI:10.2323/jgam.56.369 |
| [17] |
SOUZA M O, CRECZYNSKI-PASA T B, SCOFANO H M, et al. High hydrostatic pressure perturbs the interactions between CF0F1 subunits and induces a dual effect on activity[J]. The International Journal of Biochemistry & Cell Biology, 2004, 36(5): 920-930. |
| [18] |
GLOMBITZA C, ADHIKARI R R, RIEDINGER N, et al. Microbial sulfate reduction potential in coal-bearing sediments down to 2.5 km below the seafloor off Shimokita Peninsula, Japan[J]. Frontiers in Microbiology, 2016, 7: 1576. |
| [19] |
ARMENTEROS J J A, TSIRIGOS K D, SØNDERBY C K, et al. SignalP 5.0 improves signal peptide predictions using deep neural networks[J]. Nature Biotechnology, 2019, 37(4): 420-423. DOI:10.1038/s41587-019-0036-z |
| [20] |
ALTSCHUL S F, MADDEN T L, SCHØFFER A A, et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [21] |
CHAUMEIL P A, MUSSIG A J, HUGENHOLTZ P, et al. GTDB-Tk: a toolkit to classify genomes with the genome taxonomy database[J]. Bioinformatics, 2020, 36(6): 1925-1927. |
| [22] |
PRICEMN, DEHAL P S, ARKIN A P. FastTree 2-approximately maximum-likelihood trees for large alignments[J]. PLoS One, 2010, 5(3): e9490. DOI:10.1371/journal.pone.0009490 |
| [23] |
LETUNIC I, BORK P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees[J]. Nucleic Acids Research, 2016, 44(W1): W242-W245. DOI:10.1093/nar/gkw290 |
| [24] |
LI L, STOECKERT JR C J, ROOS D S. OrthoMCL: identification of Orthologgroups for eukaryotic genomes[J]. Genome Research, 2003, 13(9): 2178-2189. DOI:10.1101/gr.1224503 |
| [25] |
STARNAWSKI P, BATAILLON T, ETTEMA T J G, et al. Microbial community assembly and evolution in subseafloor sediment[J]. Proceedings of the National Academy of Sciencesof the United States of America, 2017, 114(11): 2940-2945. DOI:10.1073/pnas.1614190114 |
2. Department of Natural Sciences, Pacific University of Hawaii, Honolulu 96813, USA
 2023,
Vol. 32
2023,
Vol. 32


