2. 上海深渊科学工程技术研究中心, 上海 201306;
3. 江苏省海涂研究中心, 江苏 南京 210036;
4. 青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室, 山东 青岛 266237;
5. 夏威夷太平洋大学自然科学系, 夏威夷 檀香山 96813
深渊是地球海洋的最深处,其海水深度超过6 000 m的海洋区域,涵盖了1%~2%的海底面积和45%的海洋垂直深度[1-4],是海洋生态系统的重要组成部分。虽然深渊具有高压、低温以及复杂的地质活动等极端环境特征[5],但近年来的研究[6]发现,深渊海沟沉积物具有远超普通深海环境的有机质含量和微生物碳周转率,是深海有机碳降解的“热点”区域。深渊微生物介导的有机碳的降解和转化是深海碳周转过程的重要环节,其对海洋原核生物群落结构和功能多样性的贡献不容忽视[6-7]。目前对深渊微生物群落的研究多是使用传统的分离培养方法[8-10]或者基于16S rRNA基因的高通量测序研究微生物群落多样性[11-22],而对海沟微生物的代谢潜力和功能特征的认识较少。已有的零星研究[23]显示,波多黎各海沟海水中的微生物群落具有丰富而独特的功能潜力。在马里亚纳海沟海水中,存在以烃类降解菌(如Oceanospirillales)为优势类群的微生物群落和大量编码与烃类降解有关酶的基因[24]。对马里亚纳海沟海水的另一项研究表明,深渊微生物主要营异养代谢,但也可能通过氧化CO来获取能量[25]。此外,对雅浦海沟的研究表明,海水(5 000 m、5 700 m和6 000 m)微生物中最主要的代谢方式是异养代谢(即降解碳水化合物、烃类化合物和芳香族化合物),但沉积物(4 435 m、4 568 m和6 578 m)中微生物的主要代谢方式可能是化能自养代谢[26],另外,海沟沉积物中的微生物还存在渗透胁迫、氧化应激、氧限制和磷酸盐限制等应激反应基因[26]。
国内外已经开展的关于深渊海沟中微生物功能代谢的研究多集中于海水微生物[23-30],对6 000 m以深的深渊沉积物中微生物的代谢潜力的研究很有限。本研究将运用宏基因组学和生物信息学相结合的方法,分析马里亚纳海沟“挑战者深渊”(深度达10 853 m)的表层沉积物中的微生物群落组成以及功能代谢潜力。研究结果将有助于揭示马里亚纳海沟深渊生物圈的微生物群落结构和代谢过程,提升对深渊海沟中生物地球化学循环和潜在微生物驱动机制的理解。
1 材料与方法 1.1 样品的采集本次样品是2016年由“张謇号”科学考察船在马里亚纳海沟MT(Mariana Trench)站位(11.403 7°N,142.363 0°E,水深为10 853 m)采集。沉积物样品通过依托于着陆器的箱式沉积物取样器收集样品后,使用长度30 cm、直径3.5 cm的无菌取样柱进行随机插管,随后用无菌橡胶塞进行封口保存。采集完成后迅速将样品置于-80 ℃进行保存,直至开展后续研究。
1.2 宏基因组DNA的提取和宏基因组测序首先将无菌取样柱中的沉积物样品按表层至深层的顺序进行切割,并称取10 g的0~2 cm层沉积物。本实验采用宏基因组DNA提取试剂盒(FastDNA® SPIN Kit for Soil,MP公司),将10 g沉积物样品置于试剂盒的样品管后,参照试剂盒说明书的提示与要求提取沉积物样品的宏基因组DNA,并加入50 μL的无菌水将DNA从过滤柱中进行洗脱。然后使用DNA纯化试剂盒(DNeasy®PowerClean® Pro Cleanup Kit,QIAGEN公司)对提取的宏基因组DNA进行纯化。使用1.2%的琼脂糖凝胶电泳和Invitrogen Qubit 3.0荧光计(Thermo Fisher Scientific)对纯化后的DNA样品进行质检,保证DNA的质量浓度高于10 ng/μL且DNA样品的电泳检测条带清晰无明显杂带。最后将满足建库标准的宏基因组DNA样品送至BGI公司,并使用BGIseq 500平台进行测序。
1.3 宏基因组数据分析本研究将测序后获得的原始数据进行质控(表 1),使用trimmomatic(v. 0.38)[31]将测序所得序列进行去接头和去除低质量碱基(参数为LEADING: 30;TRAILING: 30;CROP: 90;HEADCROP: 10;SLIDINGWINDOW: 4∶25;MINLEN: 50)。通过IDBA-UD(v. 1.1.3)[32]将干净序列(clean reads)进行组装、拼接为重叠群(contigs)(参数:kmer range 50~80, step 15)后,在拼接结果中筛选长度≥500 bp的重叠群作为最终的组装结果。基因预测使用prodigal(V2.6.3)[33]对拼接结果中的重叠群进行开放阅读框(Open Reading Frame,ORF)的预测(参数:-p meta)获得编码序列。基于KAIJU(http://kaiju.binf.ku.dk/)对宏基因组序列进行物种分类(参数:默认)[34],获得不同分类水平下的物种注释。编码序列通过BlastKOALA[35](https://www.kegg.jp/ghostkoala/)软件比对到京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)[35],参数默认;使用blastp[36](参数:-E 1e-5,其他:默认)将编码序列与蛋白相邻类的聚簇(clusters of orthologous groups,COG)[37]数据库(参数:-E 1e-5)进行比对;利用hmmer(v. 3.1b2)[38]工具(参数:-E 1e-5)将编码序列与碳水化合物活性酶数据库(carbohydrate-active enZYmes database,CAZy)[39]数据库进行比对,获得宏基因组序列的功能注释结果,并基于功能注释的结果计算基因的丰度信息。丰度计算方法:将对应到基因的序列数除以对应到到宏基因组的所有序列数与RNA的长度(reads per kilobase million,RPKM)[26],单位为reads/106 kb。

|
表 1 宏基因测序与组装信息 Tab.1 Metagenomic sequencing and assembly information |
本研究获得409 408 006条原始序列(reads),总碱基数为18.42 Gb,经质控处理后,获得了340 227 304个序列,优化碱基数为17.70 Gb,即干净序列占原始序列(raw reads)的96%以上。拼接组装后获得了403 297个重叠群,长度大于500 bp的重叠群有290 563个,GC含量为55.8%。经基因预测后,确定了238 154个开放阅读框序列(见表 1)。
基于KAIJU物种数据库,对本研究获得的宏基因组数据进行物种分类,共注释到49个门、90个纲、1 565个属,其中细菌序列占比85%,古菌序列占比14%,病毒序列占比0.2%。本研究注释到物种相对丰度大于1%的门有11个(图 1a),其主要物种包括变形菌门(Proteobacteria)、奇古菌门(Thaumarchaeota)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)和厚壁菌门(Firmicutes)等,分别占所有分类注释的42.44%、13.12%、7.92%、7.43%、6.55%和5.93%(图 1a)。在纲水平上,相对丰度超过1%的纲有11个(图 1b),其中变形菌门(Proteobacteria)的γ变形菌纲(Gammaproteobacteria)、α变形菌纲(Alphaproteobacteria)、放线菌纲(Actinomycetia)、浮霉菌纲(Planctomycetia)、β变形菌纲(Betaproteobacteria)和δ变形菌纲(Deltaproteobacteria)为海沟表层沉积物中的优势物种,分别占所有分类注释的15.62%、15.30%、6.42%、5.48%、5.19%和4.39%。本研究着重分析了相对丰度在前25名的属(图 1c),其中奇古菌门中的亚硝化侏儒菌属(Nitrosopumilus)在属水平上丰度最高,相对丰度高达9.32%;伍斯菌属(Woeseia)、链霉菌属(Streptomyces)和假单胞菌属(Pseudomonas)也为表层沉积物中的优势属,分别占所有分类注释的1.79%、1.12%和1.01%。
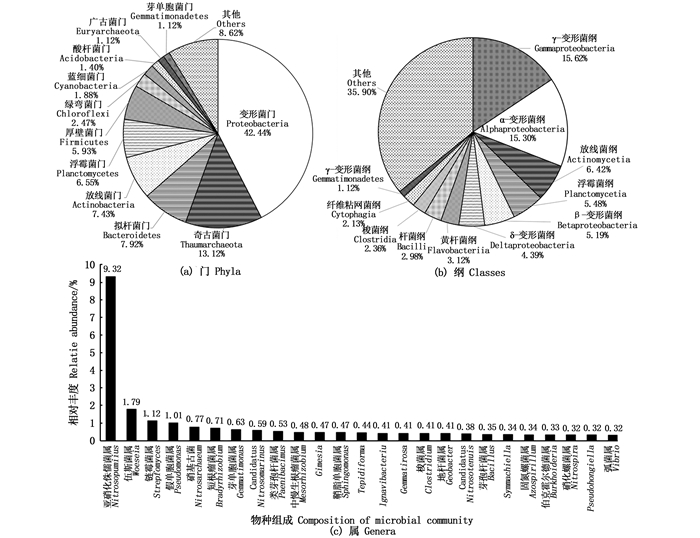
|
图 1 不同分类水平下沉积物中微生物的物种组成 Fig. 1 Composition of the sediment microbial communities at different classification levels |
基于COG数据库的功能注释结果显示(图 2),与代谢相关的功能基因中,氨基酸转运与代谢(8.53%)的基因相对丰度最高,能量生成和转换的基因相对丰度次之(7%)。此外,还注释到细胞壁/膜/包膜生物发生和翻译(6.38%)、核糖体结构与生物发生(5.85%)、辅酶转运和代谢(4.62%)、碳水化合物转运和代谢(4.49%)、脂肪转运和代谢(4.40%)、核苷酸转运和代谢(2.17%)、无机离子转运和代谢(3.80%)、次级产物的转运和代谢(3.07%)等功能基因。

|
纵坐标为COG数据库中不同功能的分类编号,框中的内容为编号对应的具体功能名称。横坐标为对应功能基因占所有功能基因的百分比。 Ordinate is the classification number of different functions in the COG database, and the contents in the box are the functions corresponding to the letters. The abscissa is the percentage of the corresponding functional genes in all the functional genes. 图 2 COG功能注释柱形图 Fig. 2 Bar diagram of COG function annotation |
基于KEGG和CAZy数据库进行功能注释,在所得的宏基因组序列中注释到与碳水化合物降解有关的各类酶,如糖苷水解酶(glycoside hydrolases,GHs)、碳水化合物酯酶(carbohydrate esterases,CEs)和多糖裂解酶(polysaccharide lyases,PLs),见图 3。此外,本研究还注释到与碳氢化合物降解(如芳香族化合物)有关的基因,例如编码降解邻苯二甲酸盐(Phthalate)有关酶的pht3、pht4、pht5、ligA和ligB(图 4)等基因,这些基因的相对丰度(以RPKM表示,单位:reads/106 kb)分别为126.78、27.74、98.65、13.30和22.46。
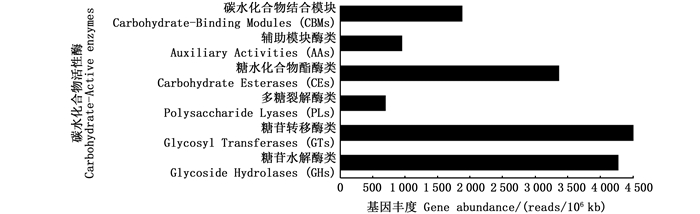
|
图 3 碳水化合物代谢相关酶的基因丰度(RPKM) Fig. 3 Gene abundances involved in the carbohydrates degradation (RPKM) |
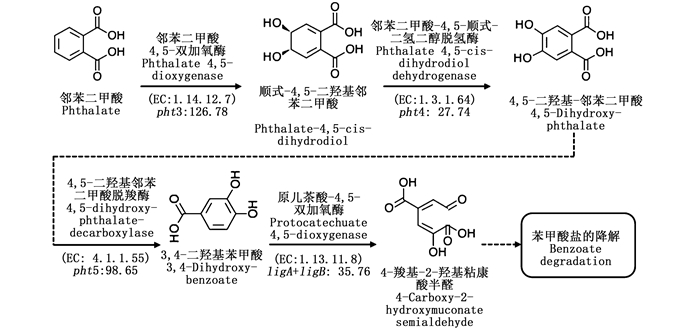
|
图中的反应箭头上方为降解邻苯二甲酸盐过程中所需酶的名称,下方为编码相应酶的功能基因和相对丰度(选用RPKM法),其单位为reads/106 kb。 The names of related enzymes of Phthalate degradation with their genes annotated are indicated over the reaction arrow in the diagram. The functional genes encoding the corresponding enzymes and relative abundance (expressed as RPKM) are below the reaction arrow, the unit of which is reads/106 kb. 图 4 降解邻苯二甲酸盐的代谢通路及关键功能基因的丰度(RPKM) Fig. 4 Gene abundances involved in the Phthalate degradation (RPKM) |
值得注意的是,本研究在深渊表层沉积物的微生物群落中发现了碳固定的代谢途径(图 5,表 2)。在所得的宏基因组序列中注释到完整的卡尔文循环(Calvin cycle)所需酶的所有编码基因:prk、rbc、pgk、gapA、gap、aldo、fba、fbp、glpX-SEBP、tkt和rpi(图 5)。此外,本研究还注释到3-羟基丙酸/4-羟基丁酸(3-hydroxypropionate/4-hydroxybutyrate,3HP/4HB)的固碳途径,其中的甲基丙二酰辅酶A变位酶(methylmalonyl-CoA mutase,mcmA)、酰基转移酶(acetyl-CoA C-acetyltransferase,atoB)等酶的编码基因表现出较高的相对丰度,RPKM值分别为638.20和538.57 reads/106 kb(表 2)。

|
在图中的反应箭头处显示了编码卡尔文循环过程中相关酶的基因名称及其基因丰度(选用RPKM法),其单位为reads/106 kb。 The name and abundance (RPKM) of the genes encoding the enzymes involved in the Calvin cycle are shown in the reaction arrow, the unit of which is reads/106 kb. 图 5 卡尔文循环的代谢过程 Fig. 5 Metabolic process of Calvin cycle |

|
表 2 3HP/4HB循环中注释到的相关酶 Tab.2 Related enzymes annotated in 3HP/4HB cycle |
研究发现,深渊沉积物中的微生物存在多个编码脱卤酶的基因,包括卤代烷烃脱卤酶(haloalkane dehalogenase)、卤代乙酸脱卤酶(haloacetate dehalogenase)和2-卤代酸脱卤酶(2-haloacid dehalogenase),编码这3种酶的基因相对丰度分别为179.29、29.88和154.3。(表 3)。深渊沉积物中的微生物还存在分别编码醛脱氢酶(Aldehyde dehydrogenase)和乙醇脱氢酶(Alcohol dehydrogenase)的ALDH、adhP和yiaY基因,其相对丰度分别为295.52、199.85和22.05 reads/106 kb,见表 3。
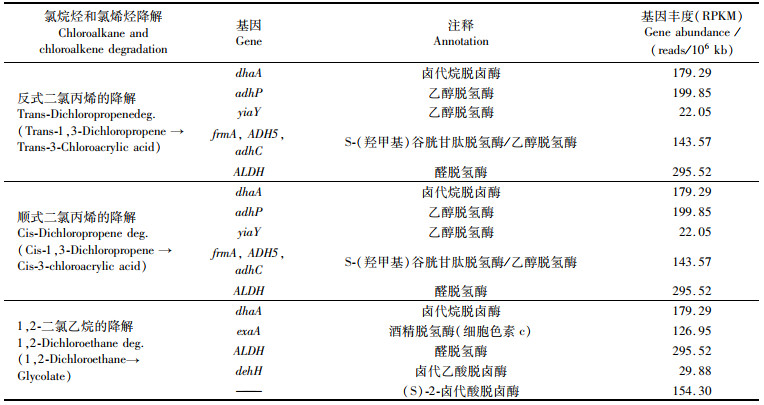
|
表 3 氯烷烃和氯烯烃的降解 Tab.3 Chloroalkane and chloroalkene degradation |
本研究在深渊表层沉积物中注释到了同化硝酸盐还原(assimilatory nitrogen reduction,ANRA)、异化硝酸盐还原(dissimilatory nitrogen reduction,DNRA)、反硝化(denitrification,DNF)和硝化等氮代谢过程中的关键基因(表 4)。结果表明,沉积物中的微生物存在编码ANRA(基因:nirA、narB和nasA)和DNRA(基因:nirB、nirD、narG、narH、napA、nrfA和nrfH等)过程中所需酶的全部基因(表 4)。此外,还注释到编码催化反硝化过程的关键酶——亚硝酸还原酶(nitrite reductase)的nirK、nirS基因(表 4)。值得注意的是,羟胺脱氢酶(hydroxylamine dehydrogenase)和氨单加氧酶(ammonia monooxygenase)是硝化过程的关键酶,编码这两种酶的基因分别为hao和amo,其相对丰度(以RPKM表示,单位:reads/106 kb)分别为48.10和112.92(表 4)。此外,在本研究的注释结果中还发现了亚硝酸盐氧化过程中的亚硝酸盐氧化还原酶(nitrite oxidoreductase),编码该酶的是nxr基因,其相对丰度为35.47 reads/106 kb。

|
表 4 与氮循环相关的功能基因的注释信息 Tab.4 Annotation of related functional genes in nitrogen cycle |
在本研究的宏基因组数据中注释到了同化硫酸盐还原(assimilatory sulfate reduction)、异化硫酸盐还原(dissimilatory sulfate reduction)和硫代硫酸盐氧化(thiosulfate oxidation)等与硫代谢有关的关键基因(表 5)。注释结果表明,沉积物中的微生物存在编码硫酸腺苷酰转移酶(sulfate adenylyltransferase)的sat/met3基因,其基因相对丰度(以RPKM表示)为239.54 reads/(106 kb)(表 5),如表 5所示,硫酸腺苷酰转移酶是异化硫酸盐还原过程中的第一个酶,也能够催化同化硫酸盐还原过程(表 5)。此外,宏基因组中还注释到编码核苷酰硫酸还原酶(adenylylsulfate reductase)的apr基因,该基因的相对丰度为114.37(表 5)。值得注意的是,沉积物中的微生物还存在多个涉及硫代硫酸盐氧化的基因,如soxA、soxB、soxC、soxD、soxX、soxY和soxZ(表 5)。
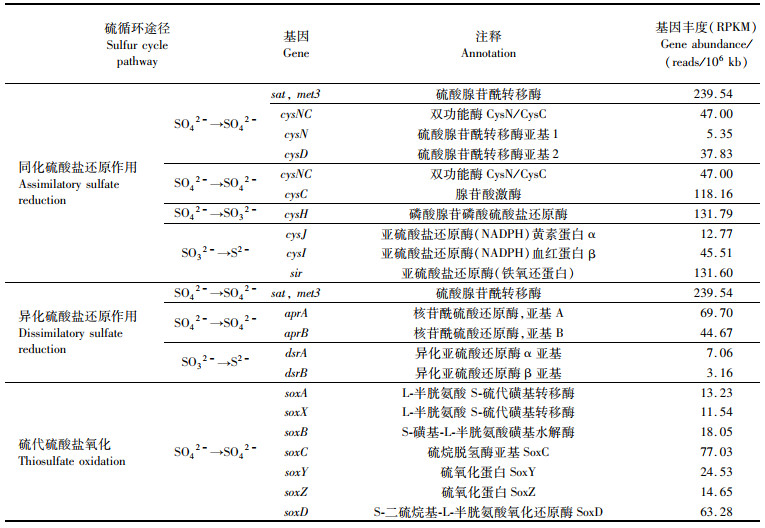
|
表 5 与硫循环相关的功能基因的注释信息 Tab.5 Annotation of related functional genes in sulfur cycle |
本研究基于KAIJU物种数据库,发现沉积物中的微生物群落存在高比例的变形菌门、拟杆菌门、放线菌门、浮霉菌门和厚壁菌门等异养微生物,其中变形菌门中的γ-、α-、β-和δ-变形菌纲是海沟表层沉积物中微生物群落的优势物种(图 1)。同时,通过对宏基因组的功能注释和分析发现,在本研究的深渊微生物群落中存在大量的编码与碳代谢有关酶的基因,如糖苷水解酶、碳水化合物酯酶和多糖裂解酶等(图 2,图 3)。这一结果与以往的研究一致,即深渊沉积物中的微生物总群落和潜在活性群落都以异养微生物和异养代谢过程为主[18-22]。这种微生物群落组成结构和功能代谢结构支持了在深渊海沟沉积物中所观察到的高水平的微生物氧消耗率和活跃的微生物碳周转过程[6-7]:一方面,海沟的“V”形构造使得上部有机颗粒物沿海沟侧坡向下输送[6],在重力的驱动下向海沟最深处移动[40-42],因此,与海沟侧坡以及普通深海平原相比,海沟底部具有更多的有机碳沉积,为深渊微生物的生存提供了大量的有机底物[40-42];而另一方面,深渊沉积物中的微生物在碳代谢相关酶的调控下,分解代谢这些有机碳。这些微生物的异养代谢过程对深渊沉积物中高效的碳周转率有重要作用,使得深渊沉积物中微生物的代谢对海洋生物地球化学循环过程具有重要影响。
值得一提的是,海沟中有机质具有显著的异质性和波动性[40-43]。通常状况下,活性有机物在沉降过程中会被上层海水中的微生物快速利用,而芳香族化合物[44]和卤代有机物[45]等结构复杂的化合物则通常会到达深海并在深海有机物库中富集。已有研究表明深海沉积物中存在编码降解芳香族化合物的厌氧氧化酶[46],且在雅浦海沟的沉积物中也发现了降解邻苯二甲酸盐等芳香族的基因[26]。本研究也表明马里亚纳海沟沉积物中的微生物存在编码降解邻苯二甲酸盐的代谢通路中所需酶的所有基因(pht3、pht4、pht5、ligA和ligB),见图 4,这些基因可以将邻苯二甲酸盐降解为4-羧基-2-羟基粘康酸半醛(4-carboxy-2-hydroxymuconate semialdehyde,HCMS)[44]。由于邻苯二甲酸盐是许多芳香族化合物降解过程中的常见中间体[47],因此,深渊微生物可能还可以降解结构更复杂的芳香族化合物。此外,虽然已有研究表明,深海沉积物中的微生物存在编码卤代烷烃脱卤酶和卤代乙酸脱卤酶的基因[48],但目前还尚未对深渊海沟沉积物中微生物的卤代有机物的降解潜力进行深入研究。本研究的结果显示深渊表层沉积物中的微生物宏基因组中存在高丰度的编码水解脱卤酶(如卤代烷烃脱卤酶、卤代乙酸脱卤酶和2-卤代酸脱卤酶)的基因,这3种酶能够利用羟基取代卤代有机物中的卤素分子[45],并介导多种卤代有机物(如1, 2-二氯乙烷、反式-二氯丙烯和顺式-二氯丙烯)的降解过程(表 3)。芳香族化合物和卤代有机物都是难降解的有机物[45]。当环境中的有机物贫乏时,深渊沉积物中的微生物可能通过这些重要活性酶降解周围环境中可用的难降解有机质,这对微生物的生存至关重要。
3.2 微生物的自养代谢生物硝化过程是地球氮循环的核心组成部分,而氨氧化过程(NH3→NO2-)是硝化过程的关键步骤[49]。氨氧化过程主要由氨氧化微生物(AOM)介导[49],氨氧化微生物分为氨氧化细菌(AOB)和氨氧化古菌(AOA),奇古菌门中的亚硝化侏儒菌属(Nitrosopumilus)是氨氧化古菌的典型微生物[50]。在本文所研究的深渊表层沉积物微生物群落中,亚硝化侏儒菌属(Nitrosopumilus)是相对丰度最高的微生物属(图 1)。进一步研究还发现深渊沉积物宏基因组中含有β变形菌门中的亚硝化球菌属(Nitrosococcus)、亚硝化单胞菌属(Nitrosomonas)和亚硝化螺菌属(Nitrosospira)[51]这3个已知的氨氧化细菌属,其相对丰度分别为0.21%、0.17%和0.08%。与这些潜在的氨氧化微生物物种信息相对应,在本研究所得的宏基因组数据中,目前所有已知的好氧氨氧化细菌和古菌的关键酶——氨单加氧酶(Ammonia monooxygenase,AMO)具有极高的相对丰度(RPKM值为112.92 reads/106 kb),见表 4。这些结果表明氨氧化过程可能是深渊表层沉积物中的重要代谢过程。
氨氧化过程会释放电子并生成能量,这些能量可以用于固定CO2、形成有机化合物和维持细胞生长等。不过,AOA和AOB在固碳方面存在重要差异,AOA通过3-羟基丙酸/4-羟基丁酸(3HP/4HB)途径固碳[52-53],而AOB则通过卡尔文循环(calvin cycle)进行固碳[54]。在马里亚纳海沟表层沉积物的宏基因组序列中,我们成功注释到3HP/4HB固碳途径所需的甲基丙二酰辅酶A变位酶(methylmalonyl-CoA mutase,mcmA)和酰基转移酶(acetyl-CoA C-acetyltransferase,atoB)等酶的编码基因(表 2),且这些基因在整个宏基因组中相对丰度较高,说明3HP/4HB途径的固碳过程在沉积物微生物中具有重要作用。同时,这一结果也与奇古菌门在深渊表层沉积物微生物群落组成中的高丰度结果相互对应。同时,深入分析还发现在深渊表层沉积物的宏基因组中存在着完整的卡尔文循环所需的所有基因(图 5),这一代谢通路的发现也与群落组成分析中所揭示的亚硝化球菌属(Nitrosococcus)、亚硝化单胞菌属(Nitrosomonas)和亚硝化螺菌属(Nitrosospira)等潜在的AOB类群相对应。另外,在宏基因组数据中还发现碳酸酐酶(carbonic anhydrase)的编码基因,其基因相对丰度(RPKM)为88.10 reads/106 kb,碳酸酐酶可以将海水或沉积物孔隙水中的HCO3-转化成CO2[55],为卡尔文循环提供底物。这些高丰度的自养固碳相关功能基因及潜在微生物种群的发现表明自养固碳可能是深渊表层沉积物中较为活跃的微生物过程,可以为深渊沉积物有机碳库提供活性较高的有机碳化合物。
3.3 高比例的新物种和其他代谢潜力在所得的宏基因组序列中存在大量的并未注释到物种的高质量序列(68.56%),可能是因为当前的物种数据库缺乏其参考序列[34],这意味着本研究中的微生物与当前物种数据库中已有生物的亲缘关系可能非常远,在深渊沉积物的微生物群落中或存在高比例的未知物种。这一可能性与此前对深渊物种多样性的研究报道[22]相符,共同支持了“地形隔离和极端的环境条件会导致深渊区新物种的产生”的假设[42, 56]。这些序列是否属于新物种依然有待进一步研究。此外,我们还发现大量的编码序列未在现有功能数据库中获得注释,这说明本研究中的微生物可能还存在其他潜在功能,也可能是因为宏基因组中不同物种有不同的密码子偏好性和物种特异的基因,因此需要适应性更广的预测算法对其进行预测[57]。此外,功能注释的结果仅代表该编码序列或许存在某些功能潜力,但是否真正行使这些功能还需要通过培养实验结合稳定同位素标记等多学科交叉手段进一步验证。
综上,在马里亚纳海沟表层沉积物的微生物群落中,数量众多的异养细菌(如γ-变形纲)是主导类群。此外,在所得的宏基因组序列中存在大量与各种碳水化合物、碳氢化合物和芳烃降解有关的基因,表明深渊微生物具有利用多种有机碳源的代谢潜力。本研究还表明马里亚纳海沟表层沉积物中的氨氧化古菌(奇古菌门)等潜在的化能自养微生物表现出较高的相对丰度,同时氨氧化、3HP/4HB、卡尔文循环等自养代谢途径在宏基因组中相对含量较高,说明自养固碳过程可能在深渊表层沉积物的碳周转中占据重要地位。研究结果证实了深渊微生物代谢的多样性,探究了海沟表层沉积物中的微生物组成及其代谢特征对深海的元素循环(如碳、氮和硫循环)的潜在影响,对探索深渊微生物生命过程及驱动深渊生物地球化学循环的内部机制具有重要意义。
| [1] |
MULDER T. Gravity processes and deposits on continental slope, rise and abyssal plains[M]//Developments in Sedimentology. Amsterdam: Elsevier, 2011, 63: 25-148.
|
| [2] |
SCHROPE M. Journey to the Bottom of the Sea[J]. Scientific American, 2014, 310(4): 60-69. DOI:10.1038/scientificamerican0414-60 |
| [3] |
LLOYD K G, MAY M K, KEVORKIAN R T, et al. Meta-analysis of quantification methods shows that Archaea and Bacteria have similar abundances in the subseafloor[J]. Applied and Environmental Microbiology, 2013, 79(24): 7790-7799. DOI:10.1128/AEM.02090-13 |
| [4] |
FANG J S, ZHANG L. Exploring the deep biosphere[J]. Science ChinaEarth Sciences, 2011, 54(2): 157-165. |
| [5] |
TAIRA K, KITAGAWA S, YAMASHIRO T, et al. Deep and bottom currents in the challenger deep, Mariana trench, measured with super-deep current meters[J]. Journal of Oceanography, 2004, 60(6): 919-926. DOI:10.1007/s10872-005-0001-y |
| [6] |
GLUD R N, WENZHÖFER F, MIDDELBOE M, et al. High rates of microbial carbon turnover in sediments in the deepest oceanic trench on Earth[J]. Nature Geoscience, 2013, 6(4): 284-288. DOI:10.1038/ngeo1773 |
| [7] |
LUO M, GLUD R N, PAN B B, et al. Benthic carbon mineralization in hadal trenches: insights from in situ determination of benthic oxygen consumption[J]. Geophysical Research Letters, 2018, 45(6): 2752-2760. DOI:10.1002/2017GL076232 |
| [8] |
YAYANOS A A, DIETZ A S, VAN BOXTEL R. Obligately barophilic bacterium from the Mariana trench[J]. Proceedings of the National Academy of Sciences of the United States of America, 1981, 78(8): 5212-5215. DOI:10.1073/pnas.78.8.5212 |
| [9] |
KATO C, LI L N, NOGI Y, et al. Extremely barophilic bacteria isolated from the Mariana Trench, Challenger Deep, at a depth of 11, 000 meters[J]. Applied and Environmental Microbiology, 1998, 64(4): 1510-1513. DOI:10.1128/AEM.64.4.1510-1513.1998 |
| [10] |
PATHOM-AREE W, STACH J E M, WARD A C, et al. Diversity of actinomycetes isolated from Challenger Deep sediment (10, 898 m) from the Mariana Trench[J]. Extremophiles, 2006, 10(3): 181-189. DOI:10.1007/s00792-005-0482-z |
| [11] |
ELOE E A, SHULSE C N, FADROSH D W, et al. Compositional differences in particle-associated and free-living microbial assemblages from an extreme deep-ocean environment[J]. Environmental Microbiology Reports, 2011, 3(4): 449-458. DOI:10.1111/j.1758-2229.2010.00223.x |
| [12] |
NUNOURA T, HIRAI M, YOSHIDA-TAKASHIMA Y, et al. Distribution and niche separation of planktonic microbial communities in the water columns from the surface to the Hadal waters of the Japan trench under the Eutrophic ocean[J]. Frontiers in Microbiology, 2016, 7: 1261. |
| [13] |
NUNOURA T, TAKAKI Y, HIRAI M, et al. Hadal biosphere: Insight into the microbial ecosystem in the deepest ocean on Earth[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(11): E1230-E1236. |
| [14] |
TARN J, PEOPLES LM, HARDY K, et al. Identification of free-living and particle-associated microbial communities present in Hadal regions of the Mariana trench[J]. Frontiers in Microbiology, 2016, 7: 665. |
| [15] |
LIU R L, WANG L, LIU Q F, et al. Depth-resolved distribution of particle-attached and free-living bacterial communities in the water column of the New Britain Trench[J]. Frontiers in Microbiology, 2018, 9: 625. DOI:10.3389/fmicb.2018.00625 |
| [16] |
PEOPLES L M, DONALDSON S, OSUNTOKUN O, et al. Vertically distinct microbial communities in the Mariana and Kermadec trenches[J]. PLoS One, 2018, 13(4): e0195102. DOI:10.1371/journal.pone.0195102 |
| [17] |
WANG Y, GAO Z M, LI J, et al. Hadal water sampling by in situ microbial filtration and fixation (ISMIFF) apparatus[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2019, 144: 132-137. DOI:10.1016/j.dsr.2019.01.009 |
| [18] |
NUNOURA T, NISHIZAWA M, HIRAI M, et al. Microbial diversity in sediments from the bottom of the challenger deep, the Mariana Trench[J]. Microbes and Environments, 2018, 33(2): 186-194. DOI:10.1264/jsme2.ME17194 |
| [19] |
HIRAOKA S, HIRAI M, MATSUI Y, et al. Microbial community and geochemical analyses of trans-trench sediments for understanding the roles of hadal environments[J]. The ISME Journal, 2020, 14(3): 740-756. DOI:10.1038/s41396-019-0564-z |
| [20] |
CUI G J, LI J, GAO Z M, et al. Spatial variations of microbial communities in abyssal and hadal sediments across the Challenger Deep[J]. PeerJ, 2019, 7: e6961. DOI:10.7717/peerj.6961 |
| [21] |
PEOPLES L M, GRAMMATOPOULOU E, POMBROL M, et al. Microbial community diversity within sediments from two geographically separated Hadal Trenches[J]. Frontiers in Microbiology, 2019, 10: 347. DOI:10.3389/fmicb.2019.00347 |
| [22] |
LIU R L, WANG Z X, WANG L, et al. Bulk and active sediment prokaryotic communities in the Mariana and Mussau trenches[J]. Frontiers in Microbiology, 2020, 11: 1521. DOI:10.3389/fmicb.2020.01521 |
| [23] |
ELOE E A, FADROSH D W, NOVOTNY M, et al. Going deeper: metagenome of a hadopelagic microbial community[J]. PLoS One, 2011, 6(5): e20388. DOI:10.1371/journal.pone.0020388 |
| [24] |
LIU J W, ZHENG Y F, LIN H Y, et al. Proliferation of hydrocarbon-degrading microbes at the bottom of the Mariana Trench[J]. Microbiome, 2019, 7(1): 47. DOI:10.1186/s40168-019-0652-3 |
| [25] |
GAO Z M, HUANG J M, CUI G J, et al. In situ meta-omic insights into the community compositions and ecological roles of hadal microbes in the Mariana Trench[J]. Environmental Microbiology, 2019, 21(11): 4092-4108. DOI:10.1111/1462-2920.14759 |
| [26] |
ZHANG X X, XU W, LIU Y, et al. Metagenomics reveals microbial diversity and metabolic potentials of seawater and surface sediment from a Hadal biosphere at the Yap Trench[J]. Frontiers in Microbiology, 2018, 9: 2402. DOI:10.3389/fmicb.2018.02402 |
| [27] |
LEÓN-ZAYAS R, PEOPLES L, BIDDLE J F, et al. The metabolic potential of the single cell genomes obtained from the Challenger Deep, Mariana Trench within the candidate superphylum Parcubacteria (OD1)[J]. Environmental Microbiology, 2017, 19(7): 2769-2784. DOI:10.1111/1462-2920.13789 |
| [28] |
LEÓN-ZAYAS R, NOVOTNY M, PODELL S, et al. Single cells within the Puerto Rico trench suggest Hadal adaptation of microbial lineages[J]. Applied and Environmental Microbiology, 2015, 81(24): 8265-8276. DOI:10.1128/AEM.01659-15 |
| [29] |
WEI Z F, LI W L, HUANG J M, et al. Metagenomic studies of SAR202 bacteria at the full-ocean depth in the Mariana Trench[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2020, 165: 103396. DOI:10.1016/j.dsr.2020.103396 |
| [30] |
HUANG J M, WANG Y. Genomic differences within the phylum Marinimicrobia: From waters to sediments in the Mariana Trench[J]. Marine Genomics, 2020, 50: 100699. DOI:10.1016/j.margen.2019.100699 |
| [31] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [32] |
PENG Y, LEUNG H C M, YIU S M, et al. IDBA-UD: a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [33] |
HYATT D, CHEN G L, LOCASCIO P F, et al. Prodigal: prokaryotic gene recognition and translation initiation site identification[J]. BMC Bioinformatics, 2010, 11(1): 119. DOI:10.1186/1471-2105-11-119 |
| [34] |
MENZEL P, NG K L, KROGH A. Fast and sensitive taxonomic classification for metagenomics with Kaiju[J]. Nature Communications, 2016, 7: 11257. DOI:10.1038/ncomms11257 |
| [35] |
KANEHISA M, SATO Y, MORISHIMA K. BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 |
| [36] |
CAMACHO C, COULOURIS G, AVAGYAN V, et al. BLAST+: architecture and applications[J]. BMC Bioinformatics, 2009, 10: 421. DOI:10.1186/1471-2105-10-421 |
| [37] |
YIN Y B, MAO X Z, YANG J C, et al. dbCAN: a web resource for automated carbohydrate-active enzyme annotation[J]. Nucleic Acids Research, 2012, 40(W1): W445-W451. DOI:10.1093/nar/gks479 |
| [38] |
EDDY S R. A new generation of homology search tools based on probabilistic inference[J]. Genome Informatics, 2009, 23(1): 205-211. |
| [39] |
TATUSOV R L, FEDOROVA N D, JACKSON J D, et al. The COG database: an updated version includes eukaryotes[J]. BMC Bioinformatics, 2003, 4: 41. DOI:10.1186/1471-2105-4-41 |
| [40] |
JAMIESON A. The Hadal zone: life in the deepest oceans[M]. Cambridge: Cambridge University Press, 2015: 217-238.
|
| [41] |
ICHINO M C, CLARK M R, DRAZEN J C, et al. The distribution of benthic biomass in hadal trenches: A modelling approach to investigate the effect of vertical and lateral organic matter transport to the seafloor[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2015, 100: 21-33. DOI:10.1016/j.dsr.2015.01.010 |
| [42] |
LIU R L, WANG L, WEI Y L, et al. The hadal biosphere: Recent insights and new directions[J]. DeepSea Research Part Ⅱ: Topical Studies in Oceanography, 2018, 155: 11-18. DOI:10.1016/j.dsr2.2017.04.015 |
| [43] |
IKEHARA K, USAMI K, KANAMATSU T. Repeated occurrence of surface-sediment remobilization along the landward slope of the Japan Trench by great earthquakes[J]. Earth, Planets and Space, 2020, 72: 114. DOI:10.1186/s40623-020-01241-y |
| [44] |
FATAYER S, COPPOLA A I, SCHULZ F, et al. Direct visualization of individual aromatic compound structures in low molecular weight marine dissolved organic carbon[J]. Geophysical Research Letters, 2018, 45(11): 5590-5598. DOI:10.1029/2018GL077457 |
| [45] |
ANG T F, MAIANGWA J, SALLEH A B, et al. Dehalogenases: from improved performance to potential microbial dehalogenation applications[J]. Molecules, 2018, 23(5): 1100. DOI:10.3390/molecules23051100 |
| [46] |
DONG X Y, GREENING C, RATTRAY J E, et al. Metabolic potential of uncultured bacteria and archaea associated with petroleum seepage in deep-sea sediments[J]. Nature Communications, 2019, 10: 1816. DOI:10.1038/s41467-019-09747-0 |
| [47] |
刘芃岩, 冯关涛, 刘金巍, 等. 邻苯二甲酸酯的光降解研究[J]. 环境科学学报, 2009, 29(5): 1049-1055. LIU P Y, FENG G T, LIU J W, et al. Photochemical degradation of phthalates: effects of UV and hydrogen peroxide[J]. Acta Scientiae Circumstantiae, 2009, 29(5): 1049-1055. DOI:10.3321/j.issn:0253-2468.2009.05.024 |
| [48] |
VUILLEMIN A, KERRIGAN Z, D'Hondt S, et al. Exploring the abundance, metabolic potential and gene expression of subseafloor Chloroflexi in million-year-old oxic and anoxic abyssal clay[J]. FEMS Microbiology Ecology, 2020, 96(12): fiaa223. DOI:10.1093/femsec/fiaa223 |
| [49] |
徐建宇, 毛艳萍. 从典型硝化细菌到全程氨氧化微生物: 发现及研究进展[J]. 微生物学通报, 2019, 46(4): 879-890. XU J Y, MAO Y P. From canonical nitrite oxidizing bacteria to complete ammonia oxidizer: discovery and advances[J]. Microbiology China, 2019, 46(4): 879-890. |
| [50] |
洪义国, 何翔, 吴佳鹏, 等. 海洋氨氧化古菌及其驱动的碳氮生物地球化学循环过程研究进展[J]. 中国科学院大学学报, 2020, 37(4): 433-441. HONG Y G, HE X, WU J P, et al. Carbon-nitrogen biogeochemical cycle processes driven by ammonia-oxidizing archaea in marine environment[J]. Journal of University of Chinese Academy of Sciences, 2020, 37(4): 433-441. |
| [51] |
GAO J F, FAN X Y, PAN K L, et al. Diversity, abundance and activity of ammonia-oxidizing microorganisms in fine particulate matter[J]. Scientific Reports, 2016, 6: 38785. DOI:10.1038/srep38785 |
| [52] |
WALKER C B, DE LA TORRE J R, KLOTZ M G, et al. Nitrosopumilusmaritimus genome reveals unique mechanisms for nitrification and autotrophy in globally distributed marine crenarchaea[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(19): 8818-8823. DOI:10.1073/pnas.0913533107 |
| [53] |
TOURNA M, STIEGLMEIER M, SPANG A, et al. Nitrososphaera viennensis, an ammonia oxidizing archaeon from soil[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(20): 8420-8425. DOI:10.1073/pnas.1013488108 |
| [54] |
于少兰, 乔延路, 韩彦琼, 等. 好氧氨氧化微生物系统发育及生理生态学差异[J]. 微生物学通报, 2015, 42(12): 2457-2465. YU S L, QIAO Y L, HAN Y Q, et al. Differences between ammonia-oxidizing microorganisms in phylogeny and physiological ecology[J]. Microbiology China, 2015, 42(12): 2457-2465. |
| [55] |
VERHAMME D T, PROSSER J I, NICOL G W. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms[J]. The ISME Journal, 2011, 5(6): 1067-1071. DOI:10.1038/ismej.2010.191 |
| [56] |
JAMIESON A J, FUJII T, MAYOR D J, et al. Hadal trenches: the ecology of the deepest places on Earth[J]. Trends in Ecology &Evolution, 2010, 25(3): 190-197. |
| [57] |
王慧丽, 郭安源. 环境微生物宏基因组学数据库利用[J]. 生物技术通报, 2015, 31(11): 78-88. WANG H L, GUO A Y. An introduction to metagenome databases of environmental microbiology[J]. Biotechnology Bulletin, 2015, 31(11): 78-88. |
2. Shanghai Engineering Research Center of Hadal Science and Technology, Shanghai 201306, China;
3. Tidal Flat Research Center of Jiangsu Province, Nanjing 210036, Jiangsu, China;
4. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, Shandong, China;
5. Department of Natural Sciences, Hawaii Pacific University, Honolulu HI 96813, U. S. A
 2022,
Vol. 31
2022,
Vol. 31


