2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306;
3. 上海海洋大学 农业农村部淡水水产种质资源重点实验室, 上海 201306
斑马鱼(Danio rerio)早期胚胎透明[1],其循环系统与包括人类在内的其他脊椎动物发育相似[2],因而斑马鱼是研究心血管遗传学和基因功能的优势模型[3]。斑马鱼心脏起源于侧板中胚层两侧的心脏原基[4],然后延伸形成心管(heart tube)[5]。胚胎在受精后24小时(24 hours post fertilization,hpf)形成一个没有瓣膜(leaflets)的原始线状心管结构[6]。在36~48 hpf时的心脏环化过程中,心脏腔体开始形成[7],这一阶段对内外因素非常敏感[8]。接着在100 hpf后,心脏在房室管(AV canal,AVC)形成一个瓣膜[9]。心脏环化是必要且高度保守的形态发生过程,将脊椎动物心脏管的不同区域置于它们最终的位置。此外,在鸟类和哺乳动物中,心脏神经嵴细胞(cardiac neural crest cells,cNCCs)参与心脏流出道(outflow tract,OFT)的形成,并产生OFT远端周围的平滑肌细胞[10]。
心脏发育是一个复杂而周密的过程,并且受到遗传因素的严格调控,包括诸多与形态发生相关的基因。其中,Hox转录因子在脊椎动物早期发育过程中起着重要作用[11]。Hox基因编码一类包含同源结构域的转录因子家族,几乎在所有真核细胞中都存在[12]。其中,Hox基因在影响神经嵴细胞分化能力方面起着重要作用[13]。通过影响神经嵴细胞的分化,几种Hox蛋白和共结合因子被认为对咽弓动脉和心脏的发育具有重要意义[11]。Hox基因的表达谱具有时空共线性的特点,即位于基因组上3′端的基因表达区域靠近躯体的头部,且表达时间比5′端的基因早[14]。心脏作为躯体前部的器官,也可能受到前端Hox基因的影响。MAKKI等[15]在小鼠中发现,Hoxa1缺失会导致晚期胚胎心脏流出道和大动脉畸形。斑马鱼hoxb5b的基因敲降会导致心脏房室分化异常,心房数量增加,心房变大[16]。hoxb4a基因仅在造血功能方面有少量研究[17],与心脏发育的关系未见报道。
为了研究hoxb4a在斑马鱼胚胎发育中的作用,本文首先使用基因沉默技术对hoxb4a进行敲降,发现hoxb4a在3 dpf时出现心包腔肿大、心脏环化异常的表型,进而利用qPCR检测心脏相关基因在hoxb4a沉默后的表达量变化。qPCR结果显示,敲降hoxb4a后,第一心域、第二心域和心脏神经嵴细胞的部分关键基因均出现显著性表达量下调,为深入发掘hoxb4a在心脏发育中的调控机理提供参考。
1 材料与方法 1.1 实验材料 1.1.1 斑马鱼鱼系的来源与养殖所用野生型斑马鱼均为AB品系。转基因野生型斑马鱼Tg(myl7 -GFP)和Tg(kdrl-GFP)为北京大学张博教授惠赠。本实验对斑马鱼的养殖和科研技术处理,均按照上海海洋大学动物伦理相关规定进行(IACUC SHOU-DW-2021-042)。
斑马鱼养殖在循环水系统中,循环水经过紫外消毒和曝气处理,水温28.5 ℃,pH为7.0~8.0。斑马鱼产卵在配鱼缸中进行,胚胎收集后养殖在纯净水中,于28.5 ℃恒温箱中培养,并保持每天换水。一般在受精后第3天开始破膜,到第5天投喂草履虫,第20天开始少量投喂卤虫,持续喂养至第30天,可将幼鱼放到循环水系统中养殖。雄鱼和雌鱼交配后产生的Ⅰ细胞期受精卵用于显微注射。
1.1.2 质粒与载体来源pXT7质粒由北京大学张博教授惠赠,过表达载体hoxb4a CDS-pXT7由本实验室克隆构建。
1.1.3 仪器和试剂本实验所用仪器包括PCR仪(Bio-rad,美国,C1000 Touch)、荧光定量PCR仪(Roche,德国,LightCycler480Ⅱ)、正置荧光显微镜(ZEISS,德国,Axio Imager2)。
实验所用试剂主要包括体外转录试剂盒(Life Technologies,美国)、FastStartTM通用SYBR Green预混液(Roche,德国)、NBT/BCIP即用型显色液(Roche,德国)。
1.2 实验方法 1.2.1 整体胚胎原位杂交在斑马鱼基因编码区(CDS)跨内含子的区域设计探针引物,在下游引物前加上T7启动子。以野生型斑马鱼的cDNA为模板PCR,PCR产物纯化之后准备进行体外转录,合成整体胚胎原位杂交所需的探针(表 1)。

|
表 1 探针引物序列 Tab.1 Sequence of probe primers |
第一步将斑马鱼胚胎固定。若要收集超过48 hpf的胚胎,需要在24 hpf时加入PTU(1-苯基-2-硫脲)抑制斑马鱼生长黑色素。收集胚胎到1.5 mL离心管中,尽量吸去离心管中的养殖水,然后加入1 mL 4%多聚甲醛,4 ℃摇床过夜。第2天用1× PBST清洗2次,每次5 min,然后用体积分数为30%、50%、70%、100%的乙醇依次进行脱水。用体积分数为70%、50%、30%的乙醇依次复水,然后在PBST中洗2次,每次5 min。使用10 μg/mL蛋白酶K(Roche,德国)处理20 min后,置于不加探针的杂交液中,放入65 ℃杂交炉中孵育4~5 h,然后加入含有1 ng/μL探针的杂交液,在杂交炉中杂交过夜。杂交配制5%羔羊血清和2 mg/mL BSA的封闭液,加入离心管中在室温下对胚胎进行封闭3~4 h。封闭结束后,按体积比1∶ 5 000的比例加入抗地高辛的抗体,在4 ℃的摇床过夜。用PBST室温清洗6次,每次15 min。Staining Buffer洗5 min。然后开始准备显色,每个样品中加入1 mL的NBT/BCIP,铝箔纸包裹以避光显色,其阳性信号呈蓝色。信号强度、角度等数据通过Image J软件测量。测量标准:以流出道中点作为起点,以房室间隔中点作为终点,两者之间距离作为心室长度。以房室间隔中点作为起点,以流入道中点作为终点,经过两点组成的直线与心室线段的夹角作为心脏环化倾角。
1.2.2 基因敲降从GeneTools公司订购hoxb4a基因的反义核酸,将其溶解于RNase-free的水中使其终浓度为1 mmol/L。所用的morpholino(MO)序列为hoxb4a:5′-AGCTGCACTTACCGATGTTTACGTG-3′。其中,hoxb4a MO所用注射质量浓度为8 ng/胚胎,每枚胚胎注射量为1 nL。
1.2.3 基因过表达首先克隆hoxb4a的编码区(CDS),并连接到斑马鱼中可表达的pXT7质粒载体中,测序正确后,在37 ℃摇菌扩大培养。使用快速小提质粒试剂盒提取基因过表达质粒,并使用紫外分光光度计测得质粒的质量浓度。使用Xba Ⅰ酶将过表达质粒线性化以便用于转录。纯合后的线性质粒片段通过Mmessage mMACHINE T7 Ultra试剂盒体外转录合成hoxb4a的mRNA,整个过程保证RNase-free。hoxb4a mRNA所用注射质量浓度为20 pg/胚胎,每枚胚胎注射量为1 nL。
1.2.4 qPCR检测基因表达量变化采用Roche®480进行qPCR检测基因表达量变化。相对表达量采用2-ΔΔCt,利用GraphPad 6.0进行数据作图和显著性差异分析。

|
表 2 qPCR引物序列 Tab.2 Sequence of qPCR primers |
胚胎用2%的甲基纤维素固定,利用Zeiss Axio Imager M2正置荧光显微镜成像。目镜倍数:1×,物镜放大倍数:10×。
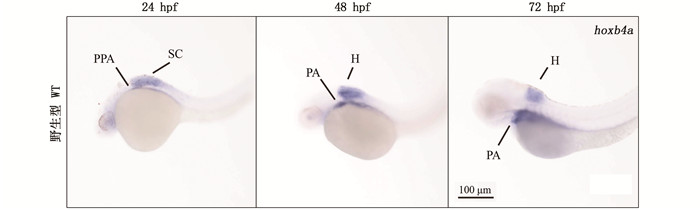
2 结果 2.1 斑马鱼胚胎发育过程hoxb4a基因的表达利用原位杂交技术,对hoxb4a在野生型胚胎中多个时期进行表达定位。实验发现24 hpf时,hoxb4a表达在后脑、脊髓、神经管位置。48 hpf和72 hpf时,hoxb4a的表达均集中在后脑和鳃弓(图 1)。从48 hpf到72 hpf可以观察到: hoxb4a在鳃弓的表达范围扩大、表达量增强;在后脑的表达范围缩小、表达量降低。
|
PPA.后鳃弓;PA.鳃弓;SC.脊髓;H.后脑。 PPA.posterior pharyngeal arch; PA. pharyngeal arch; SC. spinal cord; H. hindbrain. 图 1 斑马鱼hoxb4a基因在野生型胚胎24 hpf、48 hpf和72 hpf时的原位杂交表达图 Fig. 1 in situ hybridization expression map of hoxb4a in wild-type embryos at 24 hpf, 48 hpf and 72 hpf |
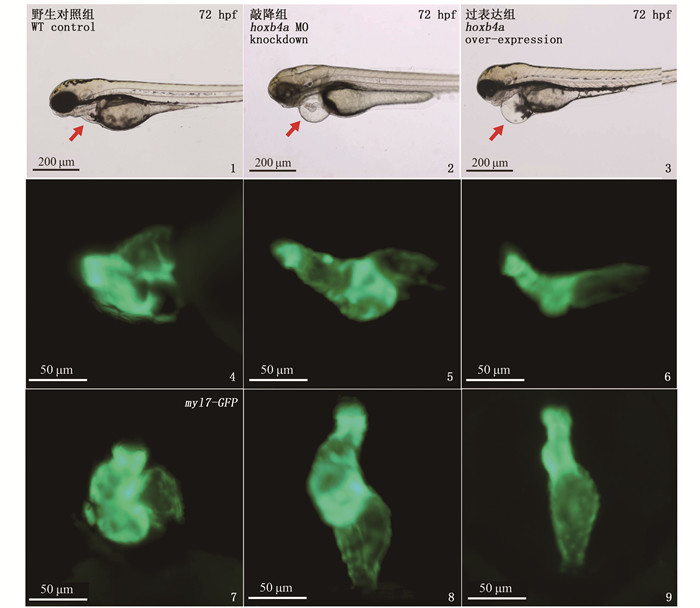
敲降hoxb4a基因后,72 hpf光镜下观察斑马鱼胚胎的心包腔开始肿大(图版Ⅰ)。后续使用myl7 (心肌细胞标记的探针)对3 dpf的斑马鱼胚胎进行原位杂交,发现敲降hoxb4a后,心脏无法正常环化,且心房和心室的腔室缩小(图 2)。hoxb4a基因mRNA过表达的斑马鱼胚胎,在72 hpf心脏环化异常,心房和心室紧缩的表型见图版Ⅰ。
|
1-3. 3 dpf时,野生型对照组、敲降组(注射了8 ng morpholino)、过表达组(注射了20 pg hoxb4a mRNA)胚胎光镜图(侧面观,头在左), 红色箭头指示心脏部位。4-6. 在转基因斑马鱼系Tg (myl7 : GFP)(心肌细胞被GFP标记)背景下,野生型对照组、敲降组(注射了8 ng morpholino)、过表达组(注射了20 pg hoxb4a mRNA)胚胎在3 dpf时的荧光光镜图片(侧面观,头在左)。7-9. 在转基因斑马鱼系Tg (myl7 : GFP)背景下,野生型对照组、敲降组(注射了8 ng morpholino)、过表达组(注射了20 pg hoxb4a mRNA)胚胎在3 dpf时期的荧光光镜图片(腹面观,头在上)。 1-3. At 3 dpf, the light microscopic images of wild-type control group, knockdown group (injected with 8 ng morpholino) and overexpression group (injected with 20 pg hoxb4a mRNA) embryos (side view with head on the left), with red arrows indicating the heart site. 4-6. In the background of transgenic zebrafish line TG (myl7 : GFP) (myocardial cells labeled with GFP), the fluorescence microscope images of wild-type control group, knockdown group (injected with 8 ng morpholino) and overexpression group (injected with 20 pg hoxb4a mRNA) embryos at 3 dpf (side view, head on the left). 7-9. In the background of transgenic zebrafish line Tg (myl7 : GFP), fluorescent microscope images of wild-type control group, knockdown group (injected 8 ng morpholino) and overexpression group (injected 20 pg hoxb4a mRNA) embryos at 3 dpf (ventral view with head on top). 图版 Ⅰ 斑马鱼hoxb4a异常表达会导致心脏发育畸形 Plate Ⅰ Zebrafish hoxb4a misexpression leading to abnormal cardiac development |
|
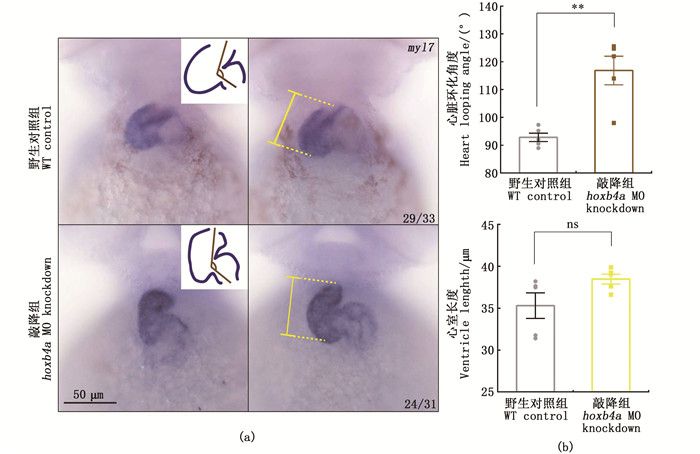
(a)72 hpf时,野生型对照组、敲降组(注射了8 ng morpholino实验组)胚胎原位杂交表达图(腹面观,头在上),棕色线段指示在环化角度。(b)野生型对照组、注射了8 ng hoxb4a morpholino实验组心脏环化角度、心室长度差异,黄色线段指示心室长度。*.P < 0.05;**.P < 0.01。 (a) At 72hpf, in situ hybridization expression map of wild-type control group, knockdown group (injected 8 ng morpholino) embryos (ventral view with head on top), The brown line segment indicates the angle of looping. (b) There were significant differences in looping angle and ventricular length between wild-type control group and experimental group. The yellow line segment indicates the length of the ventricle. *. P < 0.05; **. P < 0.01。 图 2 心肌标记基因原位杂交观察hoxb4a morphant表型 Fig. 2 in situ hybridization of myocardial marker gene to observe the phenotype of hoxb4a morphant |
整体胚胎原位杂交检测显示,72 hpf时,野生型对照组的心室心房环化角度为89°~96°,而敲降斑马鱼hoxb4a基因后,心室心房环化倾角均为99°~126°,总体明显增大(P < 0.05),但心室长度未表现明显异常(P>0.05)。见图 2b。
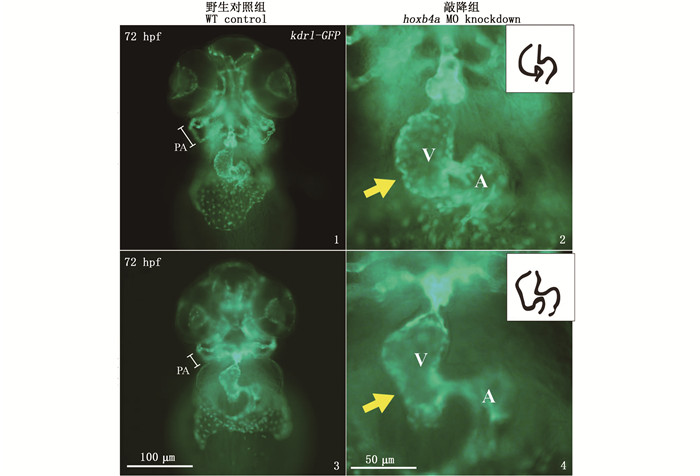
2.4 hoxb4a基因敲降导致鳃弓和心内膜异常为了进一步分析hoxb4a基因沉默后的心脏表型,利用血管内皮及心内膜特异性标记基因kdrl的转基因鱼系对morphant进行对比观察。结果显示:72 hpf时,相比于野生型对照组,注射了8 ng morpholino的hoxb4a morphant胚胎出现鳃弓缢缩、心内膜外缘形态畸形(图版Ⅱ)。
|
1-2. 3 dpf时,在转基因斑马鱼系Tg (kdrl : GFP)背景下,野生型对照组、敲降组(注射了8 ng morpholino)胚胎的荧光光镜图片(腹面观,头在上), 白色箭头指示鳃弓部位。3-4. 3 dpf时,在转基因斑马鱼系Tg (kdrl : GFP)背景下,野生型对照组、敲降组(注射了8 ng morpholino)胚胎的荧光光镜图片(腹面观,头在上), 黄色箭头指示心内膜部位。PA. 鳃弓; V. 心室;A. 心房。 1-2. At 3dpf, under the background of transgenic zebrafish line Tg (kdrl : GFP), the fluorescent microscope images of wild-type control group and knockdown group (injected 8 ng morpholino) embryos (ventral view with head on the top), the white arrow indicates the pharyngeal arch. 3-4. At 3dpf, under the background of transgenic zebrafish line Tg (kdrl : GFP), the fluorescent microscope images of wild-type control group and knockdown group (injected 8 ng morpholino) embryos (ventral view with head on the top), the yellow arrow indicates the endocardium. PA. pharyngeal arch; V. ventricle; A. atrium. 图版 Ⅱ 斑马鱼hoxb4a基因沉默会导致鳃弓和心内膜畸形 Plate Ⅱ Silencing of hoxb4a in zebrafish leading to pharyngeal arch and endocardial malformation |
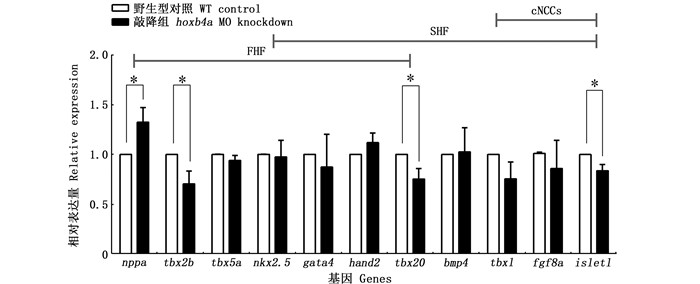
利用qPCR探究hoxb4a调控的心脏相关下游基因,发现腔室分化关键基因nppa上调(P < 0.05),房室间隔形成关键基因tbx2b下调(P < 0.05),腔室形成激活因子tbx20下调(P < 0.05),神经嵴关键基因islet1下调(P < 0.05),见图 3。
|
*为有显著性差异,P < 0.05,3组独立生物学重复实验(n=3)。 * There was significant difference (P < 0.05), three groups of independent biological repeated experiments(n=3). 图 3 心脏相关基因在hoxb4a morphant中的表达量变化 Fig. 3 Expression of heart-related genes in hoxb4a morphant |
心脏环化是个复杂的过程。在斑马鱼中抑制心脏神经嵴细胞会导致心脏环化受损[18]。小鼠中,pSHF(posterior SHF)的部分细胞决定了心脏的环化方式[19],其SHF衍生物的损失会导致心脏环化失败[20]。综上所述,FHF、SHF、cNCCs均参与心脏环化。本文中利用基因敲降与过表达实验,均证实hoxb4a的异常表达会导致心脏环化异常,并且hoxb4a可能以基因剂量依赖性方式[21]调节心脏发育[22]。结合qPCR结果显示,当斑马鱼hoxb4a沉默时,各心脏谱系中部分基因均受到影响,其中,tbx20、tbx2b显著下调,nppa明显上调。在小鼠中,GAVRILOV等[23]发现Tbx20突变可导致心脏环化失败、房室分化异常。2002年,HABETS等[24]报道了在人类和大鼠的心脏发育过程中,NPPA基因的表达是心脏腔室形成的首要标志之一,且小鼠中Tbx2可抑制Nppa活性。因此,猜测斑马鱼中hoxb4a基因沉默后的tbx2b表达下调是引起nppa上调的原因。总体上,tbx20、tbx2b和nppa的表达量变化可能是导致心脏环化异常的因素之一。
3.2 hoxb4a参与心脏发育的方式2015年,CAVANAUGH等以神经嵴转基因斑马鱼进行谱系追踪,发现神经嵴在两个不同的发育阶段均参与了心脏的发生。神经嵴细胞通过第1和第2鳃弓(pharyngeal arches,PA)迁移进入心管参与心肌的发生。直到50 hpf时,第二波神经嵴细胞开始沿鳃弓迁移至心脏并参与心脏形态发生,而hox基因在神经嵴细胞分化迁移规范上起着重要作用[25]。本研究显示,hoxb4a在48 hpf至72 hpf内在鳃弓处存在持续高表达的现象(图 1)。通过转基因鱼系结果,hoxb4a基因敲降后会导致鳃弓异常表型(图版Ⅱ)。同时,根据qPCR数据(图 3),作为心脏神经嵴细胞的标记基因,islet1的表达量在hoxb4a沉默后显著下调。综上所述,猜测hoxb4a可能通过影响神经嵴细胞参与斑马鱼心脏发育。
本文通过基因沉默和mRNA基因过表达技术,高效快速地在斑马鱼F0探索了hoxb4a的功能,并未改变其基因型,发现hoxb4a基因的敲低或过表达均会在斑马鱼胚胎发育至3 dpf时产生心脏环化失败的异常表型,表明hoxb4a基因在调控心脏早期发育中发挥作用。本研究进一步通过原位杂交和qPCR技术对hoxb4a基因敲低胚胎进行分析,发现hoxb4a可能通过影响心脏神经嵴基因islet1参与心脏调控,进而干预腔室分化基因nppa和心脏环化相关基因tbx20、tbx2b的表达,最终导致了心脏发育的畸形。本研究为进一步通过CRISPR技术敲除hoxb4a研究其在心脏发育中的作用奠定了良好的基础,为挖掘hoxb4a基因调控心脏发育的机制及加深对心脏调控网络的认识都具有重要意义。
| [1] |
STAINIER D Y, LEE R K, FISHMAN M C. Cardiovascular development in the zebrafish. I. Myocardial fate map and heart tube formation[J]. Development, 1993, 119(1): 31-40. DOI:10.1242/dev.119.1.31 |
| [2] |
HOVE J R, KÖSTER R W, FOROUHAR A S, et al. Intracardiac fluid forces are an essential epigenetic factor for embryonic cardiogenesis[J]. Nature, 2003, 421(6919): 172-177. DOI:10.1038/nature01282 |
| [3] |
BARK D L JR, JOHNSON B, GARRITY D, et al. Valveless pumping mechanics of the embryonic heart during cardiac looping: pressure and flow through micro-PIV[J]. Journal of Biomechanics, 2017, 50: 50-55. DOI:10.1016/j.jbiomech.2016.11.036 |
| [4] |
PRUMMEL K D, NIEUWENHUIZE S, MOSIMANN C. The lateral plate mesoderm[J]. Development, 2020, 147(12): dev175059. DOI:10.1242/dev.175059 |
| [5] |
GLICKMAN N S, YELON D. Cardiac development in zebrafish : coordination of form and function[J]. Seminars in Cell & Developmental Biology, 2002, 13(6): 507-513. |
| [6] |
FOROUHAR A S, LIEBLING M, HICKERSON A, et al. The embryonic vertebrate heart tube is a dynamic suction pump[J]. Science, 2006, 312(5774): 751-753. DOI:10.1126/science.1123775 |
| [7] |
BAKKERS J. Zebrafish as a model to study cardiac development and human cardiac disease[J]. Cardiovascular Research, 2011, 91(2): 279-288. DOI:10.1093/cvr/cvr098 |
| [8] |
JOHNSON B, BARK D JR, VAN HERCK I, et al. Altered mechanical state in the embryonic heart results in time-dependent decreases in cardiac function[J]. Biomechanics and Modeling in Mechanobiology, 2015, 14(6): 1379-1389. DOI:10.1007/s10237-015-0681-1 |
| [9] |
VERMOT J, FOROUHAR A S, LIEBLING M, et al. Reversing blood flows act through klf2a to ensure normal valvulogenesis in the developing heart[J]. PLoS Biology, 2009, 7(11): e1000246. DOI:10.1371/journal.pbio.1000246 |
| [10] |
JIANG X, ROWITCH D H, SORIANO P, et al. Fate of the mammalian cardiac neural crest[J]. Development, 2000, 127(8): 1607-1616. DOI:10.1242/dev.127.8.1607 |
| [11] |
MALLO M, ALONSO C R. The regulation of Hox gene expression during animal development[J]. Development, 2013, 140(19): 3951-3963. DOI:10.1242/dev.068346 |
| [12] |
AMORES A, FORCE A, YAN Y L, et al. Zebrafish hox clusters and vertebrate genome evolution[J]. Science, 1998, 282(5394): 1711-1714. DOI:10.1126/science.282.5394.1711 |
| [13] |
ROUX M, LAFOREST B, EUDES N, et al. Hoxa1 and Hoxb1 are required for pharyngeal arch artery development[J]. Mechanisms of Development, 2017, 143: 1-8. DOI:10.1016/j.mod.2016.11.006 |
| [14] |
PEARSON J C, LEMONS D, MCGINNIS W. Modulating Hox gene functions during animal body patterning[J]. Nature Reviews Genetics, 2005, 6(12): 893-904. DOI:10.1038/nrg1726 |
| [15] |
MAKKI N, CAPECCHI M R. Cardiovascular defects in a mouse model of HOXA1 syndrome[J]. Human Molecular Genetics, 2012, 21(1): 26-31. DOI:10.1093/hmg/ddr434 |
| [16] |
WAXMAN J S, KEEGAN B R, ROBERTS R W, et al. Hoxb5b acts downstream of retinoic acid signaling in the forelimb field to restrict heart field potential in zebrafish[J]. Developmental Cell, 2008, 15(6): 923-934. DOI:10.1016/j.devcel.2008.09.009 |
| [17] |
KROSL J, AUSTIN P, BESLU N, et al. In vitro expansion of hematopoietic stem cells by recombinant TAT-HOXB4 protein[J]. Nature Medicine, 2003, 9(11): 1428-1432. DOI:10.1038/nm951 |
| [18] |
BERNDT C, POSCHMANN G, STüHLER K, et al. Zebrafish heart development is regulated via glutaredoxin 2 dependent migration and survival of neural crest cells[J]. Redox Biology, 2014, 2: 673-678. DOI:10.1016/j.redox.2014.04.012 |
| [19] |
DOMíNGUEZ J N, MEILHAC S M, BLAND Y S, et al. Asymmetric fate of the posterior part of the second heart field results in unexpected left/right contributions to both poles of the heart[J]. Circulation Research, 2012, 111(10): 1323-1335. DOI:10.1161/CIRCRESAHA.112.271247 |
| [20] |
HINITS Y, PAN L Y, WALKER C, et al. Zebrafish Mef2ca and Mef2cb are essential for both first and second heart field cardiomyocyte differentiation[J]. Developmental Biology, 2012, 369(2): 199-210. DOI:10.1016/j.ydbio.2012.06.019 |
| [21] |
MCFADDEN D G, BARBOSA A C, RICHARDSON J A, et al. The Hand1 and Hand2 transcription factors regulate expansion of the embryonic cardiac ventricles in a gene dosage-dependent manner[J]. Development, 2005, 132(1): 189-201. DOI:10.1242/dev.01562 |
| [22] |
COLLINS E M, THOMPSON A. HOX genes in normal, engineered and malignant hematopoiesis[J]. The International Journal of Developmental Biology, 2018, 62(11/12): 847-856. |
| [23] |
GAVRILOV S, HARVEY R P, PAPAIOANNOU V E. Lack of genetic interaction between Tbx20 and Tbx3 in early mouse heart development[J]. PLoS One, 2013, 8(7): e70149. DOI:10.1371/journal.pone.0070149 |
| [24] |
HABETS P E M H, MOORMAN A F M, CLOUT D E W, et al. Cooperative action of Tbx2 and Nkx2.5 inhibits ANF expression in the atrioventricular canal: implications for cardiac chamber formation[J]. Genes & Development, 2002, 16(10): 1234-1246. |
| [25] |
CAVANANGH A M, HUANG J, CHEN J N. Two developmentally distinct populations of neural crest cells contribute to the zebrafish heart[J]. Developmental Biology, 2015, 404(2): 103-112. DOI:10.1016/j.ydbio.2015.06.002 |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. Key Laboratory of Freshwater Aquatic Genetic Resources, Ministry of Agriculture and Rural Affairs, Shanghai Ocean University, Shanghai 201306, China
 2022,
Vol. 31
2022,
Vol. 31


