2. 广州海洋地质调查局自然资源部海洋矿产资源重点实验室, 广东 广州 510760;
3. 中国地质大学 地质过程与矿产资源国家重点实验室, 湖北 武汉 430074;
4. 青岛海洋科学与技术国家实验室海洋生物与生物技术实验室, 山东 青岛 266237;
5. 夏威夷太平洋大学自然科学系, 檀香山 96813
海洋表层浮游植物通过光合作用产生的有机物主要以颗粒有机物(particulate organic matter, POM)的形式输送到深海[1-2]。下沉颗粒持续为深海微生物供应有机碳和其他营养物质[3-4],微生物的迁移与承载其转运的POM在微生物学、化学和酶学上构成一个连续整体[5]。在颗粒物沉降过程中,POM聚集物优先被颗粒附着的附生菌(particle-attached mode, PAM)所定殖并分泌胞外酶[6-7]分解POM,同时释放溶解有机物(dissolved organic matter, DOM)[5, 8-10]。释放的DOM在下沉颗粒后方形成富碳富营养的羽流,从而促进周围水体中自由菌(free-living microorganism, FLM)的代谢作用[5, 11-12]。因此,FANG等[5]提出了PDPMC模型,揭示“POM-DOM-嗜高压细菌连续一体化机制”。
海洋异养微生物是参与高分子有机聚合体分解和降解的关键角色,主导了水体中有机碳通量的变化[13]。研究[14-15]表明,从海洋表面到海底,随着深度的增加微生物的丰度和特征均展现不同的特点,这主要与环境因素(压力、温度、溶解氧氧浓度等)变化相关。微生物群落的垂直分层近年来已被广泛报道[16-19],研究发现静水压力对微生物群落组成、生理和代谢有着显著影响。然而,以往的研究主要集中在对于原位海水的微生物群落组成的探索,仅能揭示特定深度的微生物群落组成,而在POM分解和降解过程中,微生物群落在颗粒下沉过程中,有机物的化学组成的变化与其对静水压力的响应机制尚不清楚。
本研究利用13C标记的硅藻碎屑作为POM加入表层海水,并以逐渐加压的培养方式模拟颗粒在海洋中下沉的过程,重点探讨了微生物相互作用和群落演替以及静水压力对POM分解和降解的影响。主要目的是验证颗粒物下沉过程中静水压力和营养资源的变化驱动PAM和FLM的演替的假说,即随着深度增加,静水压力增加而营养资源减少过程中微生物的群落演替与功能变化规律。
1 材料与方法 1.1 采样及基本参数样品为张謇号2019年航次于马里亚纳海沟的M站位(11°19.79′N, 142°11.96′E),通过盐-温-深CTD传感器(型号sea bird SBE 911)共采集250 L的50米浅表层海水进行微生物群落分析。180 L海水首先通过孔径10 μm过滤器(规格142 mm,Merck Millipore Ltd.)进行预过滤以去除大颗粒和浮游动物,再通过3.0和0.22 μm孔径聚碳酸酯滤膜依次过滤,分别收集附生菌和自由菌[20],测定原位海水(in-situ water,ISW)微生物群落组成。剩余的70 L海水用于DNA-SIP标记高压培养实验样品采集和与培养实验,所有实验均在船上进行。
1.2 模拟颗粒沉降高压培养实验硅藻培养参考LIU等[21]于2020年发表的培养方法,在实验室中获得了13C-标记和12C-对照的Thalassiosiraweissflogii CCMP 1051T硅藻碎屑。硅藻碎屑在正式培养前使用液氮进行研磨, 并在121 ℃高温灭菌20 min后烘干,储存在-20 ℃冰箱中。本次试验所使用的13C-标记和12C-对照的硅藻碎屑中13C原子标记度百分比分别为79.2%和1.59%。
试验设计为模拟颗粒物从表层(0.1 MPa)下沉到深海(60 MPa)的过程,培养试验在常压(0.1 MPa)和高压(10、30和60 MPa)4种压力条件下进行,每个压力组6个培养体系,包括13C标记组和12C对照组,每组各3个平行。为保证表层海水样品中微生物群落结构无明显变化,CTD采集海水样品后即刻配制培养体系并现场进行不同压力条件下的培养。培养体系为密封培养袋(Kapak,美国)中加入200 mL 50 m海水,0.06 g 13C或12C硅藻碎屑,和67 mL于培养前充氧4 h的Fluorinert(FC-40, 3MTM Corp.)[22]。使用高压培养釜进行压力培养,以水为介质增加压力,以模拟海洋中不同深度的压力,将高压釜置于4 ℃冷库中。4个深度组同时开始培养,在微生物生长的稳定期,0.1 MPa的6个样品首先结束培养,取出样品过滤分离PAM与FLM进行后续分析; 其他3组加压至10 MPa(1 000 m),此时维持1 000 m深度海水的温度和压力进行平行培养,当微生物生长达到稳定期后,1 000 m深度组的6个样品终止试验,取样进行后续分析。剩下2个深度组依此类推继续加压培养,直到最后1个深度组加压到60 MPa(6 000 m)。
1.3 POC和DOC的化学分析在整体培养过程中,从开始至结束平均在相应的5个时间点取混合后培养液20 mL于安瓿瓶中以测定培养体系的POC与DOC的浓度变化。通过0.7 μm的GF/F玻璃纤维滤膜分别获得滤膜(即POC)与滤液(即DOC),POC、DOC的浓度测定分别采取TOC-LCPH总有机碳分析仪(日本岛津公司)和TOC-V总有机碳分析仪(日本岛津公司)。POC降解率计算公式[23]如下:
 (1)
(1)
式中:Ct为t小时POC浓度; C0为POC初始浓度; t为培养时间, h;k为POC的降解速率。
1.4 DNA提取及PCR扩增使用Fast DNA Spin Kit for soil (MP Bio,USA)分别提取附生菌和自由菌的总DNA。16S rRNA基因测序以V4区为目标区域进行引物设计[24],在515F(5′-GTGCCAGCMGCCGCGGTAA-3′)前端和806R(5′-GGACTACHVGGGTWTCTAAT-3′)后端分别连接长度为6~8 bp的Barcode序列标签以此区分不同样品; 扩增片段大小约为291 bp。PCR体系为50 μL,主要包括:Premix TaqTM混合染料25 μL, 10 μmol/L前端后端引物各1 μL, DNA 1 μL, 无菌ddH2O 22 μL。PCR方法: 94 ℃ 3 min,94 ℃ 45 s,50 ℃ 60 s,72 ℃ 90 s, 72 ℃ 10 min,共35个循环。
1.5 超高速离心梯度分馏及实时荧光定量PCR密度梯度离心过程参照贾仲君[25]2011年发表的DNA-SIP超离心梯度分馏法。采用实时荧光定量PCR (qPCR)检测DNA样品的第3至14层的拷贝数,qPCR所用引物为细菌通用引物对338F(5′-CCTACGGGAGGCAGCAG-3′),518R(5′-ATTACCGCGGCTGCTGG-3′)[26]。采取绝对定量PCR的标准化方法:使用标准质粒制备6个浓度梯度(4.3×10~4.3×106)的标准稀释曲线。qPCR体系为20 μL,主要包括:Go Taq qPCR Master Mix酶10 μL,2 μmol/L前端后端引物各2 μL, DNA模板4 μL,无菌ddH2O 2 μL。qPCR步骤使用仪器7 500 Real Time PCR System Amplifier (ABI,USA),qPCR扩增程序:初始变性(95 ℃,10 min); 40次循环变性(95 ℃,15 s; 60 ℃,1 min); 95 ℃退火15 s,60 ℃退火1 min; 95 ℃延伸30 s, 60 ℃延伸15 s。本次细菌16S rDNA基因的扩增效率均大于99%,R2值小于0.99。PAM与FLM的细胞丰度是根据每个细菌细胞平均含有4.08个16S rRNA基因拷贝数计算的[27-28]。
1.6 IlluminaMiSeq测序及数据分析平台上按照上海美吉生物制药技术有限公司的标准协议进行配对测序。利用UCHIME对获得的原始序列进行鉴定和去除。使用UPARSE (7.0.1090版本,http://www.drive5.com/uparse/)对操作分类单元(OTUs)进行聚类,相似性截止率为97%。每个OTU的分类均在Silva 16S rRNA数据库(SSU123)中进行,置信阈值为70%。本次数据分析过程筛选至少在一个样本中相对丰度≥1%的OTU进行进一步分析。采用MOTHUR (V(1.30.2版本,https://www.mothur.org/wiki/Download_mothur)计算Chao1指数、Shannon指数和Simpson指数等多样性指数。使用VENNY 2.1[29]制作Venn图,展现不同培养条件下与ISW中2种不同组分间OTU分布的变化。基于物种组成在OTU水平上构建Bray-Curtis距离矩阵来表示Beta多样性,采用QIIME (1.9.1版本,http://qiime.org/install/index.html)进行多维尺度NMDS分析。采用ANOSIM相似性分析用于检验组间和组内差异。
1.7 网络构建与分析在基于随机矩阵理论(RMT)的分子生态网络分析平台(MENAP,http://129.15.40.240/mena.cgi),利用OTU表和16S rRNA基因测序结果的相应reads构建共现生态网络[30-31]。基于Pearson相关系数计算所有网络拓扑特征的相关系数,并将所有网络可视化数据导出并在Cytoscape(版本3.8.2)进行可视化绘图。通过随机网络优化计算[32]对模块内连通性(Zi)和模块间连通性(Pi)2个参数进行计算和比较,确定网络节点拓扑属性的分类:模块集线器(module hubs,Zi≥2.5且Pi < 0.62);网络集线器(network hubs,Zi≥2.5且Pi≤0.62);连接器(connectors,Zi < 2.5且Pi≥0.62);周边外设(peripherals,Zi < 2.5且Pi < 0.62)[33]。
2 结果 2.1 POC和DOC浓度变化在不同静水压力下,微生物对POM的分解和降解导致POC和DOC浓度随时间变化。除0.1 MPa时POC浓度在12 h先降低后增加外,其他压力下POC浓度均随时间降低。DOC浓度变化规律比较明显,总体上随时间呈增加趋势,在60 MPa时,DOC积累尤为明显(图 1)。总体而言,POC浓度随时间和压力呈下降趋势,而DOC浓度呈上升趋势。
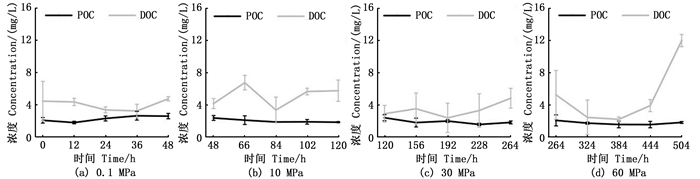
|
图 1 培养过程POC和DOC浓度变化 Fig. 1 Variations of POC and DOC concentrations during the incubation |
原位海水中附生菌的细菌丰度为2.96×105 cells/mL,自由菌的细菌丰度则为2.9×107 cells/mL[图 2(a)]。在0.1 MPa条件下培养过后,附生菌的细菌丰度增加2个数量级,之后附生菌和自由菌的细胞丰度均随着压力的增加而降低。同样与原位条件相比,培养后的物种丰富度(Chao1指数)大幅下降,之后随着压力的增加物种丰富度保持相对稳定[图 2(b)]。附生菌和自由菌组合的Alpha多样性(Shannon指数)和物种均匀度(Simpson even指数)在不同压力下也有所不同:在原位海水中和60 MPa压力下,附生菌的Alpha多样性明显高于自由菌,而在其他3个压力(0.1、10和30 MPa)下,则展现出相反的趋势[图 2(c)]; 在除60 MPa外的所有压力下,自由菌的物种均匀度都高于附生菌[图 2(d)]。
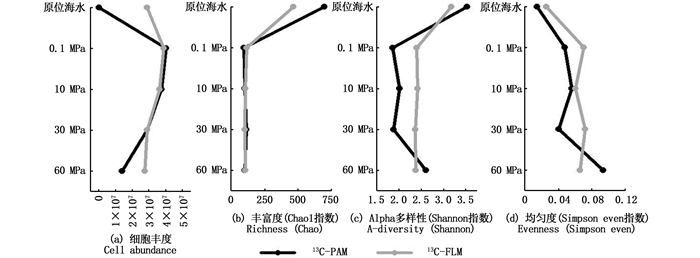
|
图 2 培养前后附生菌和自由菌组分的细胞丰度与多样性指数 Fig. 2 Cell abundance and diversity index of PAM and FLM before and after incubation |
通过超高速离心可成功分离13C标记的DNA,细菌的基因最大拷贝数出现在第5~7层,即“重”DNA馏分。将活性OTU定义为13C-重DNA馏分中OTU丰度与12C-对照中重层对应层相应OTU丰度相减后,相对丰度≥1%的OTU[21]。本次共鉴定出19个活性OTUs,主要隶属于Gammaproteobacteria(14个OTUs)、Alphaproteobacteria(2个OTUs),以及Bacteroidia(3个OTUs)。不同压力下的活性类群在附生菌或自由菌组分之间不均匀分布,根据图 3中呈现的分布模式,主要有以下几个类群。

|
系统发育树用最大似然法构建并使用MUSCLE进行多序列比对,利用bootstrap分析(500)评估了树的稳健性,近似百分比值均显示在节点上。Bar =每100个核苷酸有5个取代。偏好附生营生与自由营生的OTU分别用红色和黑色文本表示。右侧热图中的方框显示了在0.1、10、30和60 MPa下,活性(上方)和总体系(下方)中该物种的相对丰度。 Phylogenetic relationship was inferred by the maximum likelihood method. Multiple sequence alignments were performed with MUSCLE. The robustness of the tree was evaluated using bootstrap analysis (500). Percentage values are indicated at the nodes. Bar = 5 substitutions per 100 nucleotides. PAM and FLM are indicated in red and black text on the left side respectively.Boxes on the right show the relative abundance of active (upper) and total (lower) microorganisms at 0.1, 10, 30, and 60 MPa. 图 3 DNA-SIP筛选后活性微生物的系统发育树与相对丰度 Fig. 3 Phylogeny and relative abundance of active microorganisms detected by DNA-SIP |
首先,一些类群在总体系中或在活性筛选后均在不同压力下明显呈现为优势物种,这些大多为γ -Proteobacteria群,主要有OTU333(Vibrio)、OTU370(Alteromonas)、OTU210(Pseudoalteromonas)和OTU369(Pseudoalteromonas)。其次,有一些类群在总培养体系和活性筛选后均表现出对于附生或自由营生的单一偏好,如OTU129(Lesingera)、OTU158 (Neptunomonas)、OTU548(Oleibacter)、OTU338(Vibrio pomeroyi)、OTU215(Methylophaga)、OTU211(Leeuwenhoekiella)以及OTU 220(Mesonia); 而有两个物种则在筛选后呈现不一样的生活方式:OTU239 (Pseudomonas)和OTU313(Aliiroseovarius),即随着压力的增加,微生物改变了生活策略。最后,几个在低压下相对丰度极低的物种在高压下展现了较高的活性,包括OTU297(Cobetia)与OTU326(Shewanella),推测这些微生物在深海有机质再矿化过程中发挥了更重要的作用。
2.4 微生物生活方式的变化通过计算对数比值比(OR)来确定微生物对于生活方式(附生菌或自由菌)的偏好,比值比参考LI等[34]所述计算方法,OR>0则为PAM偏好,OR<0为FLM偏好,OR≈0即为在两种生活方式无明显偏好。如图 4所示,检测到的活跃PAM菌群仅由Gammaproteobacteria(Cobetia、Shewanella、Alteromonas和Pseudoalteromonas)组成,而FLM菌群则由更多样的谱系组成,其中包括Gamma-、Alphaproteobacteria和Bacteroidia(Tenacibaculum、Alcanivorax、Marinobacter、Oleibacter、Vibrio、Neptunomonas、Methylophaga、Leeuwenhoekiella、Leisingera、Aliiroseovarius)以及未分类的Favobacteriaceae。这一结果表明,在本研究检测的所有压力下微生物的生态位分配都发生了改变。仅在一种生活方式下呈现偏好的类群大多是非活性OTU,如PAM偏好的OTU329(Halobacteriovorax,非活性)和OTU297(Cobetia,活性),FLM偏好的OTU176(Marinomonas,非活性)、OTU220(未分类的Flavobacteriaceae,活性)和OTU132(Olleya,非活性)。
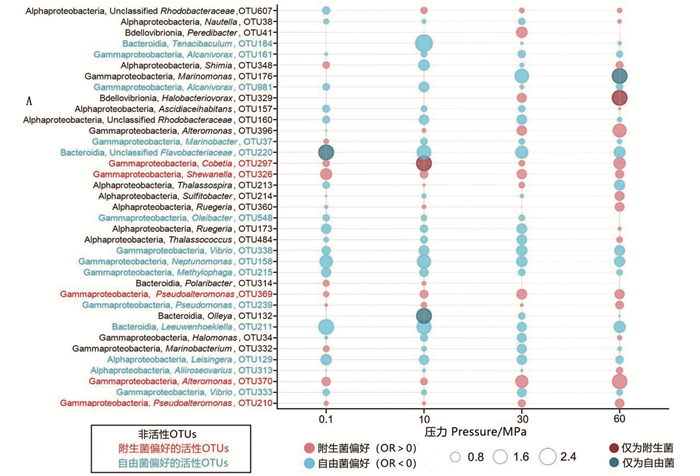
|
不同压力下总微生物群落中相对丰度≥1%的OTUs的对数比值比气泡图,其中红色气泡表示具有正比值比的OTUs,即偏好颗粒附生方式。蓝色气泡代表负比值比的OTUs,即偏好自由营生的生活方式。暗红色和深蓝色气泡分别代表仅存在于PAM和FLM组分中的OTUs。 Odds ratio for each of the OTUs with relatively abundance (≥1%) in the total microbial communities at different pressures. Red bubbles represent OTUs with a positive odds ratio, i.e., preference for the PA lifestyle. Blue bubbles represent OTUs with a negative odds ratio, i.e., preference for FL lifestyle. Scale is shown at the bottom. Dark red and dark blue bubbles represent OTUs that existed only in the PAM and FLM fraction, respectively. 图 4 微生物对于生活方式(附生菌或自由菌)的偏好 Fig. 4 Microorganism's preference for lifestyle (PAM or FLM) |
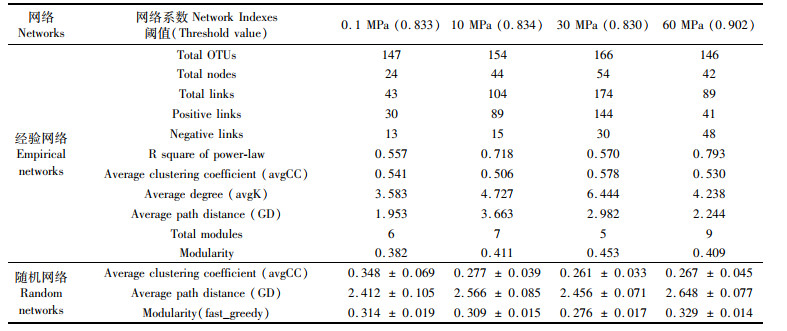
网络图呈现了OTUs(节点)代表的不同微生物种群之间的生态相互作用(连接线),本次分析所得的4个微生物共生网络分别对应的4个压力下的模拟沉降培养试验结果[图 5(a)]。网络的大小(即节点的数量)从0.1 MPa的24个提高到10 MPa的44个以及30 MPa的54个,随后在60 MPa减少为42个。网络复杂度(network complexity)呈现出相同的变化趋势,从0.1 MPa到30 MPa增加,然后在60 MPa时下降,表现为高平均连通性(avgK)和较小的平均路径距离(GD)。网络的拓扑性质表明,所构建的均为无标度(scale-free)网络(R2 = 0.557~0.793),即网络的连通性遵循幂律,网络中少数节点与其他节点之间有较多的连接,且大部分节点之间仅有较少连接[30-31]。比较经验网络和随机网络的平均聚类系数(avgCC)也可得出构建的网络具有小世界特征(表 1)。
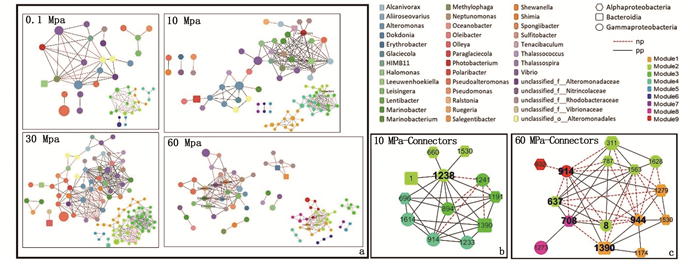
|
共现网络揭示了(a)4个压力(0.1、10、30和60 MPa)下不同OTUs(节点)之间的微生物相互作用; 在10 MPa(b)和60 MPa(c)的网络中发现了连接器(Connectors)。节点颜色代表不同的OTU,节点大小与节点的平均相对丰度成正比。黑色实线和红色虚线分别代表正相关和负相关。PP-正相关; NP-负相关。 The co-occurrence networks reveal microbial interactions between different OTUs (species) at 0.1, 10, 30, and 60 MPa. Connectors were identified in the 10 (b) and 60 MPa (c) networks. More information about the connectors is shown in Table A2 and Table A3. The node color represents different OTUs, and the node size is proportional to its average relative abundance. The black solid lines and red dash lines represent positive and negative links respectively. NP, negative links; PP, positive links. 图 5 共现网络与子网络 Fig. 5 The co-occurrence networks and subnetworks |
|
表 1 共现网络的拓扑性质参数 Tab.1 The topological properties parameters of the co-occurrence networks |
网络集线器、模块集线器和连接器是网络构成中的关键节点。在本次构建的网络中没有观察到网络集线器或模块集线器,在10 MPa网络的模块2中发现一个连接器OTU1238(Photobacterium),它与网络中其他节点的相关性大都为正相关[图 5(b)]。60 MPa网络更碎片化,共计算得出6个连接器,分别是OTU944(Marinobacterium)、OTU1390 (Leeuwenhoekiella)、OTU8(Alteromonas)、OTU637(Shimia)、OTU708(Ruegeria)和OTU914(Vibrio)[图 5(c)]。
3 讨论 3.1 海洋表层微生物对深海代谢的作用表层海洋中产生的颗粒物构成了碎屑集合体[35],其丰富的碳含量和营养物质吸引了大量的异养细菌定殖降解[11, 36]。因此,碎屑颗粒下沉过程中将有机物和微生物从海洋表层带到深海,促进了深海的物质与能量代谢。以前的研究[37-39]已经表明在下降的颗粒上附着有大量的异养微生物,其细胞丰度比周围水体高一个数量级。有假说[5, 12, 40]认为,颗粒有机物在水体的下沉过程中,附生菌可分泌胞外酶分解POM并产生DOM。本次试验结果表明,POC的浓度随着时间和压力的增加而降低,同时DOC的浓度随着压力的增加而增加,且在最高压力60 MPa下观测到最高的DOC浓度(图 1)。这表明在模拟沉降培养过程中微生物对POM的分解效率高,且DOM的产生和积累效率高。本次结果同样验证了这个假设:附生菌作为典型的富营养生物[41],在分解POC的过程中产生了大量的DOC,远高于其可利用的数量,因在下降的粒子后面会形成一个DOC羽流[5, 11-12]。本次研究结果同样表明,PAM对POM降解与FLM分解利用DOM的之间存在耦合关系[6, 42]。高压下DOC浓度的增加也可能与深海微生物生理和代谢能力的变化有关,在高压下PAM和FLM对DOC的利用效率降低,从而导致DOC的累积。前人研究[42-43]表明,深海微生物的代谢能力及对有机物的降解能力往往受到高压的抑制。本次研究同样发现POM降解速率随着压力增长而降低,这些结果与前人[21, 43-45]的发现一致,同样研究结果显示表层海水中的微生物为深海提供了重要的代谢基础。
3.2 不同压力下附生菌和自由菌在颗粒降解中的作用研究结果显示,Gammaproteobacteria类群中Alteromonas、Pseudoalteromonas、Vibrio和Marinobacter,普遍存在于附生菌和自由菌中并积极参与了POM降解(图 3),其中Alteromonas和Pseudoalteromonas更倾向于颗粒附着的生活方式(图 4),这与过去在地中海[46-47]、南海[48]、波多黎各海沟[20, 34]、新不列颠海沟[49]和太平洋深部[50]的研究结果一致。与附生菌相比,自由菌的群落组成更加多样化(图 2)。过去有假说[5, 8]认为,附生菌是海洋中的富营养生物和机会主义者,由于水体中下沉颗粒含有丰富的碳和营养资源,因此其更倾向于颗粒附着的生活方式。事实上,本次研究结果显示在颗粒下沉过程中活跃的微生物发生了生活方式上的演替,且在深海中微生物的生活方式在系统发育和代谢上并不是保守的,这些结论与SALAZAR等[2]的观点形成了鲜明对比。曾经有研究者[5, 8, 51]提到海洋中的颗粒物富含碳和营养物质并极大地支持微生物代谢,本次研究结果很好佐证了这一观点,特别是在深海高压下,颗粒附着的生活方式是首选的策略,随着静水压力的增加,大多数微生物从自由营生方式转向颗粒附着方式(图 4)。此外,许多微生物以双重生活方式对颗粒有机物进行分解降解,在高压下,自由菌的活性比例明显增加(图 4)。深海微生物群落演替不仅受到营养物质可利用性的驱动,同时也受到静水压力的影响。在深海中,POM分解产生的有机质在压力作用下质量和数量减少,变得更加难降解[52],因此在深海碳循环中,能够降解难降解有机物的微生物应占主导地位并发挥更重要的作用[5]。在活性筛选结果中,FLM的相对丰度远高于PAM(图 4),可以预测自由菌群落在POM降解过程中发挥着更为重要的作用,且微生物的生活方式、获取营养策略与其在海洋中的生态功能并不严格相关。
3.3 不同压力下微生物网络相互作用和群落稳定性生态系统的功能是通过复杂的微生物群落网络中的物种相互作用来实现的[30, 53]。本次研究使用基于RMT的网络分析方法来说明网络结构和关联交互作用[30]。通过对无标度、小世界、模块化和层次化等一般网络拓扑特征的分析,可以反映水体中颗粒物下沉时,微生物网络的相互作用及其对环境变化(即静水压力的变化)的响应。拓扑性质参数显示,4种微生物群落的整体网络结构明显不同。从表 1可以看出,随着压力的增加,群落的连通性(avgK)、网络模块性和聚类系数(avgCC)的增加说明群落的微生物网络在压力下变得更加脆弱。60MPa压力下的微生物群落网络与其他3种压力下的差异尤为明显,上述指标均有所下降,说明社区网络的弹性和稳定性有所提高。60 MPa下的微生物网络主要是负相关,代表偏害共栖、捕食和竞争等关系[54-56]。然而在深海的高压环境下,偏害共栖和捕食行为不太可能发生,因而物种竞争很可能主导了深海中表层来源微生物之间的相互作用,主要呈现为争夺下沉颗粒附带的有限资源。此外,60MPa压力下的网络比较分散,共有9个模块(图 5),而其他3种压力下的网络模块数较少。网络模块可以被认为是生态位或微生物功能单元[57],代表一组功能类群,它们之间具有高度紧密联系,但与其他模块的联系很少[30]。在海洋中,随着水体剖面上越来越多的分解颗粒对碳和营养物质的可用性的降低,微生物网络在高压下呈现碎片化,形成了更多的子网络,以增加群落的弹性和稳定性[58-59]。这一结果表明,微生物在群落网络水平上适应了深海环境普遍存在的高压和寡营养特征。
| [1] |
AZAM F, MALFATTI F. Microbial structuring of marine ecosystems[J]. Nature Reviews Microbiology, 2007, 5(10): 782-791. DOI:10.1038/nrmicro1747 |
| [2] |
SALAZAR G, CORNEJO-CASTILLOFM, BORRULL E, et al. Particle-association lifestyle is a phylogenetically conserved trait in bathypelagic prokaryotes[J]. Molecular Ecology, 2015, 24(22): 5692-5706. DOI:10.1111/mec.13419 |
| [3] |
MESTRE M, RUIZ-GONZÁLEZC, LOGARES R, et al. Sinking particles promote vertical connectivity in the ocean microbiome[J]. Proceedings of the National Academy of Sciencesof the United States of America, 2018, 115(29): E6799-E6807. |
| [4] |
SANDERS R, MORRIS P J, POULTON A J, et al. Does a ballast effect occur in the surface ocean?[J]. Geophysical Research Letters, 2010, 37(8): L08602. |
| [5] |
FANG J S, LI Z, LI J T, et al. The POM-DOM piezophilic microorganism continuum(PDPMC): The role of piezophilic microorganisms in the global ocean carbon cycle[J]. Science China Earth Sciences, 2015, 58(1): 106-115. DOI:10.1007/s11430-014-4985-2 |
| [6] |
SMITH DC, SIMON M, ALLDREDGE AL, et al. Intense hydrolytic enzyme activity on marine aggregates and implications for rapid particle dissolution[J]. Nature, 1992, 359(6391): 139-142. DOI:10.1038/359139a0 |
| [7] |
LIU Q F, FANG J S, LI J T, et al. Depth-resolved variations of cultivable bacteria and their extracellular enzymes in the water column of the new Britain trench[J]. Frontiers in Microbiology, 2018, 9: 135. DOI:10.3389/fmicb.2018.00135 |
| [8] |
AZAM F, LONG R A. Sea snow microcosms[J]. Nature, 2001, 414(6863): 495-498. DOI:10.1038/35107174 |
| [9] |
ARISTEGUI J, GASOL J M, DUARTE C M, et al. Microbial oceanography of the dark ocean's pelagic realm[J]. Limnology and Oceanography, 2009, 54(5): 1501-1529. DOI:10.4319/lo.2009.54.5.1501 |
| [10] |
DUCKLOW H W, CARLSON C A. Oceanic bacterial production[M]//MARSHALL K C. Advances in Microbial Ecology. Boston, MA: Springer, 1992, 12: 113-181.
|
| [11] |
KIØBOE T, JACKSON G A. Marine snow, organic solute plumes, and optimal chemosensory behavior of bacteria[J]. Limnology and Oceanography, 2001, 46(6): 1309-1318. DOI:10.4319/lo.2001.46.6.1309 |
| [12] |
SIMON M, GROSSART H P, SCHWEITZER B, et al. Microbial ecology of organic aggregates in aquatic ecosystems[J]. Aquatic Microbial Ecology, 2002, 28(2): 175-211. |
| [13] |
MESTRE M, BORRULL E, SALA M M, et al. Patterns of bacterial diversity in the marine planktonic particulate matter continuum[J]. The ISME Journal, 2017, 11(4): 999-1010. DOI:10.1038/ismej.2016.166 |
| [14] |
MATÍN-CUADRADOAB, LÓPEZ-GARCÍAP, ALBA J C, et al. Metagenomics of the deep mediterranean, a warm bathypelagic habitat[J]. PLoS One, 2007, 2(9): e914. DOI:10.1371/journal.pone.0000914 |
| [15] |
MÉVEL G, VERNET M, GOUTX M, et al. Seasonal to hour variation scales in abundance and production of total and particle-attached bacteria in the open NW Mediterranean Sea (0-1000 m)[J]. Biogeosciences, 2008, 5(6): 1573-1586. DOI:10.5194/bg-5-1573-2008 |
| [16] |
DELONG E F, PRESTON C M, MINCER T, et al. Community genomics among stratified microbial assemblages in the ocean's interior[J]. Science, 2006, 311(5760): 496-503. DOI:10.1126/science.1120250 |
| [17] |
RÖSEL S, GROSSARTH P. Contrasting dynamics in activity and community composition of free-living and particle-associated bacteria in spring[J]. Aquatic Microbial Ecology, 2012, 66(2): 169-181. DOI:10.3354/ame01568 |
| [18] |
NUNOURA T, TAKAKI Y, HIRAI M, et al. Hadal biosphere: insight into the microbial ecosystem in the deepest ocean on Earth[J]. Proceedings of the National Academy of Sciencesof the United States of America, 2015, 112(11): E1230-E1236. |
| [19] |
NUNOURA T, HIRAI M, YOSHIDA-TAKASHIMAY, et al. Distribution and niche separation of planktonic microbial communities in the water columns from the surface to the hadal waters of the Japan Trench under the eutrophic ocean[J]. Frontiers in Microbiology, 2016, 7: 1261. |
| [20] |
ELOE E A, SHULSE C N, FADROSH D W, et al. Compositional differences in particle-associated and free-living microbial assemblages from an extreme deep-ocean environment[J]. Environmental Microbiology Reports, 2011, 3(4): 449-458. DOI:10.1111/j.1758-2229.2010.00223.x |
| [21] |
LIU Y, FANG J S, JIA Z J, et al. DNA stable-isotope probing reveals potential key players for microbial decomposition and degradation of diatom-derived marine particulate matter[J]. Microbiology Open, 2020, 9(5): e1013. |
| [22] |
FANG J S, UHLE M, BILLMARK K, et al. Fractionation of carbon isotopes in biosynthesis of fatty acids by a piezophilic bacterium Moritella japonica strain DSK1[J]. Geochimicaet Cosmochimica Acta, 2006, 70(7): 1753-1760. DOI:10.1016/j.gca.2005.12.011 |
| [23] |
SUROY M, PANAGIOTOPOULOS C, BOUTORHJ, et al. Degradation of diatom carbohydrates: a case study with N-and Si-stressed Thalassiosiraweissflogii[J]. Journal of Experimental Marine Biology and Ecology, 2015, 470: 1-11. DOI:10.1016/j.jembe.2015.04.018 |
| [24] |
LIU J, HUA Z S, CHEN L X, et al. Correlating microbial diversity patterns with geochemistry in an extreme and heterogeneous environment of mine tailings[J]. Applied and Environmental Microbiology, 2014, 80(12): 3677-3686. DOI:10.1128/AEM.00294-14 |
| [25] |
贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用[J]. 微生物学报, 2011, 51(12): 1585-1594. JIA Z J. Principle and application of DNA-based stable isotope probing-A review[J]. Acta Microbiologica Sinica, 2011, 51(12): 1585-1594. |
| [26] |
THIJS S, DE BEECK M O, BECKERS B, et al. Comparative evaluation of four bacteria-specific primer pairs for 16S rRNAgene surveys[J]. Frontiers in Microbiology, 2017, 8: 494. |
| [27] |
LEE Z M P, BUSSEMAⅢ C, SCHMIDT T M. rrnDB: documenting the number of rRNA and tRNA genes in bacteria and archaea[J]. Nucleic Acids Research, 2009, 37(S1): D489-D493. |
| [28] |
LI J T, WEI B B, WANG J N, et al. Variation in abundance and community structure of particle-attached and free-living bacteria in the South China Sea[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2015, 122: 64-73. DOI:10.1016/j.dsr2.2015.07.006 |
| [29] |
OLIVEROS J C. VENN Y. An interactive tool for comparing lists with Venn Diagrams[EB/OL]. 2007. https://bioinfogp.cnb.csic.es/tools/venny/index.html.
|
| [30] |
ZHOU J Z, DENG Y, LUO F, et al. Functional molecular ecological networks[J]. Microbiology, 2010, 1(4): e00169-10. |
| [31] |
DENG Y, JIANG Y H, YANG Y F, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13: 113. DOI:10.1186/1471-2105-13-113 |
| [32] |
GUIMERà R, AMARAL L A N. Functional cartography of complex metabolic networks[J]. Nature, 2005, 433(7028): 895-900. DOI:10.1038/nature03288 |
| [33] |
TU Q C, YAN Q Y, DENG Y, et al. Biogeographic patterns of microbial co-occurrence ecological networks in six American forests[J]. Soil Biology and Biochemistry, 2020, 148: 107897. DOI:10.1016/j.soilbio.2020.107897 |
| [34] |
LI J T, GU L Y, BAI S J, et al. Characterization of particle-associated and free-living bacterial and archaeal communities along the water columns of the South China Sea[J]. Biogeosciences, 2021, 18(1): 113-133. DOI:10.5194/bg-18-113-2021 |
| [35] |
BIDDANDA B A, POMEROY L R. Microbial aggregation and degradation of phytoplankton-derived detritus in seawater. I. Microbial succession[J]. Marine Ecology Progress Series, 1988, 42: 79-88. DOI:10.3354/meps042079 |
| [36] |
ALLDREDGE A L, COHEN Y. Can microscalechemical patches persist in the sea? Microelectrode study of marine snow, fecal pellets[J]. Science, 1987, 235(4789): 689-691. DOI:10.1126/science.235.4789.689 |
| [37] |
REINTHALER T, AKEN H V, VETH C, et al. Prokaryotic respiration and production in the meso-and bathypelagic realm of the eastern and western north Atlantic basin[J]. Limnology and Oceanography, 2006, 51(3): 1262-1273. DOI:10.4319/lo.2006.51.3.1262 |
| [38] |
SOGIN M L, MORRISON H G, HUBER J A, et al. Microbial diversity in the deep sea and the underexplored "rare biosphere"[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(32): 12115-12120. DOI:10.1073/pnas.0605127103 |
| [39] |
KELLOGG C T E, DEMING J W. Comparison of free-living, suspended particle, and aggregate-associated bacterial and archaealcommunities in the Laptev Sea[J]. Aquatic Microbial Ecology, 2009, 57(1): 1-18. DOI:10.1007/s00248-008-9465-9 |
| [40] |
CHO B C, AZAM F. Major role of bacteria in biogeochemical fluxes in the ocean's interior[J]. Nature, 1988, 332(6163): 441-443. DOI:10.1038/332441a0 |
| [41] |
LAURO F M, BARTLETT D H. Prokaryotic lifestyles in deep sea habitats[J]. Extremophiles, 2008, 12(1): 15-25. DOI:10.1007/s00792-006-0059-5 |
| [42] |
TURKEY C M. The effect of pressure on leucine and thymidine incorporation by free-living bacteria and by bacteria attached to sinking oceanic particles[J]. Deep Sea Research Part I: Oceanographic Research Papers, 1993, 40(11/12): 2193-2206. |
| [43] |
TAMBURINI C, BOUTRIF M, GAREL M, et al. Prokaryotic responses to hydrostatic pressure in the ocean: a review[J]. Environmental Microbiology, 2013, 15(5): 1262-1274. DOI:10.1111/1462-2920.12084 |
| [44] |
TAMBURINI C, GARCIN J, GREGORI G J, et al. Pressure effects on surface mediterranean prokaryotes and biogenic silica dissolution during a diatom sinking experiment[J]. Aquatic Microbial Ecology, 2006, 43(3): 267-276. |
| [45] |
TAMBURINI C, GOUTX M, GUIGUE C, et al. Effects of hydrostatic pressure on microbial alteration of sinking fecal pellets[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2009, 56(18): 1533-1546. DOI:10.1016/j.dsr2.2008.12.035 |
| [46] |
ACINAS SG, ANTON J, RODRIGUEZ-VALERA F. Diversity of free- living and attached bacteria in offshore western Mediterranean waters as depicted by analysis of genes encoding 16S rRNA[J]. Applied and Environmental Microbiology, 1999, 65(2): 514-522. DOI:10.1128/AEM.65.2.514-522.1999 |
| [47] |
CRESPO B G, POMMIER T, FERNÁNDEZ-GÓMEZ B, et al. Taxonomic composition of the particle-attached and free-living bacterial assemblages in the northwest Mediterranean sea analyzed by pyrosequencing of the 16s rRNA[J]. Microbiologyopen, 2013, 2(4): 541-552. DOI:10.1002/mbo3.92 |
| [48] |
DANG H Y, LOVELL C R. Bacterial primary colonization and early succession on surfaces in marine waters as determined by amplified rRNA gene restriction analysis and sequence analysis of 16S rRNAgenes[J]. Applied and Environmental Microbiology, 2000, 66(2): 467-475. DOI:10.1128/AEM.66.2.467-475.2000 |
| [49] |
LIU R L, WANG L, LIU Q F, et al. Depth-resolved distribution of particle-attached and free-living bacterial communities in the water column of the New Britain Trench[J]. Frontiers in Microbiology, 2018, 9: 625. DOI:10.3389/fmicb.2018.00625 |
| [50] |
BOEUF D, EDWARDS B R, EPPLEY J M, et al. Biological composition and microbial dynamics of sinking particulate organic matter at abyssal depths in the oligotrophic open ocean[J]. Proceedings of the National Academy of Sciences, 2019, 116(24): 11824-11832. DOI:10.1073/pnas.1903080116 |
| [51] |
ALLDREDGE A L, GOTSCHALK C C. The relative contribution of marine snow of different origins to biological processes in coastal waters[J]. Continental Shelf Research, 1990, 10(1): 41-58. DOI:10.1016/0278-4343(90)90034-J |
| [52] |
JIAO N Z, HERNDL G J, HANSELL D A, et al. Microbial production of recalcitrant dissolved organic matter: Long-term carbon storage in the global ocean[J]. Nature Reviews Microbiology, 2010, 8(8): 593-599. DOI:10.1038/nrmicro2386 |
| [53] |
MONTOYA J M, PIMM S L, SOLÉR V. Ecological networks and their fragility[J]. Nature, 2006, 442(7100): 259-264. DOI:10.1038/nature04927 |
| [54] |
BRENNER K, YOU L C, ARNOLD F H. Engineering microbial consortia: a new frontier in synthetic biology[J]. Trends in Biotechnology, 2008, 26(9): 483-489. DOI:10.1016/j.tibtech.2008.05.004 |
| [55] |
FAUST K, RAES J. Microbial interactions: from networks to models[J]. Nature Reviews Microbiology, 2012, 10(8): 538-550. DOI:10.1038/nrmicro2832 |
| [56] |
SHI S, NUCCIO E E, SHI Z J, et al. The interconnected rhizosphere: High network complexity dominates rhizosphere assemblages[J]. Ecology Letters, 2016, 19(8): 926-936. DOI:10.1111/ele.12630 |
| [57] |
LUO F, ZHONG J X, YANG Y F, et al. Application of random matrix theory to biological networks[J]. Physics Letters A, 2006, 357(6): 420-423. DOI:10.1016/j.physleta.2006.04.076 |
| [58] |
KRAUSE A E, FRANK K A, MASON D M, et al. Compartments revealed in food-web structure[J]. Nature, 2003, 426(6964): 282-285. DOI:10.1038/nature02115 |
| [59] |
WU L W, YANG Y F, CHEN S, et al. Long-term successional dynamics of microbial association networks in anaerobic digestion processes[J]. Water Research, 2016, 104: 1-10. DOI:10.1016/j.watres.2016.07.072 |
2. Key Laboratory of Marine Mineral Resources, Ministry of Natural Resources, Guangzhou Marine Geological Survey, Guangzhou 510760, Guangdong, China;
3. State Key Laboratory of Geological Processes and Mineral Resources, Department of Earth Sciences, China University of Geosciences, Wuhan 430074, Hubei, China;
4. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, Shandong, China;
5. Department of Natural Science, Hawaii Pacific University, Honolulu 96813, HI, United States
 2022,
Vol. 31
2022,
Vol. 31


