2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室,上海 201306
光唇鱼属(Acrossocheilus Oshima, 1919)隶属鲤形目(Cypriniformes)鲤科(Cyprinidae)鲃亚科(Barbinae),我国主要分布于长江及其以南水系,包括我国台湾省和海南岛,少数种类见于越南和老挝,是一群生活于急流环境中的中小型淡水鱼类[1-4]。Fishbase数据库(https://www.fishbase.cn/search.php)显示光唇鱼属曾有47个种和亚种,去除同物异名,目前仍认为有26个有效种,其中20个种和亚种分布在中国。该属物种传统形态分类主要将体色和条纹、下唇结构和背鳍末根不分支鳍条的粗细及后缘锯齿的有无等作为主要鉴别特征,但由于其种类多,同一物种可能因栖息环境的不同导致种群间出现形态差异。另外,个体不同发育阶段形态差异及雌雄两性异形的存在等导致光唇鱼属物种间性状交叉颇多,物种鉴定非常困难,目前仍没有可被广范认可的形态分类体系。
我国东南部的浙江、福建和台湾境内独立河系众多,生态环境多样,因此该区域光唇鱼属种类也极为丰富,共记录有8种[1, 4-5]。但是该区域这些物种的有效性和地理分布问题至今仍然存在争议,如: 伍献文等[1]和单乡红等[4]认为台湾有光唇鱼A. fasciatus(Steindachner,1892)、台湾光唇鱼A. formosanus(Regan,1908)和厚唇光唇鱼A. labiatus(Regan,1908)3种,光唇鱼和厚唇光唇鱼还分布于浙江和福建等地,台湾光唇鱼在浙江也有分布。SUNG等[6]采用同工酶技术,对台湾各水系光唇鱼属鱼类进行分析,最终将其合并为同一种即台湾光唇鱼A.paradoxus(Gunther,1868),并认为是台湾特有种[7-8]。YUAN等[8]以线粒体控制区序列的系统发育研究,指出大陆东南部水系广泛分布的厚唇光唇鱼和光唇鱼应为同一种,而分布于闽江的厚唇光唇鱼和光唇鱼与武夷光唇鱼A.wuyiensis(Wu & Chen, 1981)为同物异名,武夷光唇鱼为其有效种名。此后亦有基于线粒体cyt b基因的研究认为台湾光唇鱼也分布在大陆闽江水系,并提出台湾光唇鱼起源于大陆,经过两次冰河时期的3次拓殖事件到达台湾[9]。侯晓静[10]采用多基因分子标记的大陆光唇鱼属亲缘地理研究结果与YUAN等[8]对大陆光唇鱼的分类结果相一致。HOU等[11]基于线粒体基因组的光唇鱼系统发育研究也表明台湾光唇鱼与武夷光唇鱼亲缘关系最近。因此,现今基本认为台湾只有1种光唇鱼即台湾光唇鱼,与闽江的武夷光唇鱼为近源种,但二者是否为彼此独立的物种仍然存在疑问。
DNA条形码(DNA Barcoding)是利用DNA序列对物种进行鉴定的分子生物学方法,由于它不过于依赖保存样本的完整性,不受生物体不同发育阶段影响,在相似种鉴别中有更高的准确性,被广泛应用于物种鉴定和遗传多样性研究[12-13]。COⅠ基因因具备进化速率适宜,易于扩增等优点被认为是理想的DNA条形码[14]。研究表明,COⅠ基因在鱼类物种间有较高的物种鉴定可靠性,可以很好地区分约98%的海洋鱼类和93%的淡水鱼类[12, 15-16]。本研究借助COⅠ基因作为遗传标记,分析台湾光唇鱼与分布于闽江武夷光唇鱼物种有效性,为光唇鱼属的分类研究等提供依据。
1 材料与方法 1.1 样本采集所用标本采集于2011年5月—2020年7月。依据单乡红等[4]进行分类鉴定,然后按照YUAN等[8]和侯晓静[10]研究结果,选择物种典型性样本进行分析,将分布于浙江各水系原定名为厚唇光唇鱼和光唇鱼视为光唇鱼,将分布于闽江的视为武夷光唇鱼。共有6种光唇鱼属鱼类9个地理种群共76尾样本,另外选取东方墨头鱼(Garra orientalis)、纹唇鱼(Osteochilus salsburyi)和台湾白甲鱼(Onychostoma barbatulum)3种4尾样本作为外类群用于系统发育分析。标本经鉴定打好标签并取下肌肉后,置于95%乙醇中固定,所有标本保存于上海海洋大学鱼类标本室(表 1)。

|
表 1 光唇鱼属的样本信息、21个单倍型的分布频率及群体的遗传多样性 Tab.1 Sample information, distribution frequency of 21 haplotypes, haplotype diversity (h) and nucleotide diversity (π) of Acrossocheilus |
采用生工生物工程公司(上海)动物基因组DNA提取试剂盒提取DNA。利用NCBI Primer-BLAST设计引物(表 2)。PCR扩增体系为25 μL包含: Taq PCR Master Mix(BBI)12.5 μL; ddH2O 9.5 μL; 上下游引物各1.0 μL(浓度10 μmol/L);DNA模板1.0 μL。反应程序:94 ℃预变性4 min,扩增35个循环(94 ℃变性30 s,53 ℃退火45 s,72 ℃延伸1 min),最后72 ℃延伸10 min,4 ℃保存。送至上海生工生物工程公司进行测序,由于COⅠ基因全长较长,为保证测序准确性,设计中间引物加测了中间部分的序列。

|
表 2 COⅠ扩增引物 Tab.2 Amplification primers of COⅠ sequences |
用DNAstar中的SeqMen[17]读取测序峰图,拼接并进行人工校正。用MEGA 7.0[18]对序列进行比对并分析变异情况,并用Kimura 2-parameter模型计算种间和种内遗传距离。用DnaSP 5.1[19]软件统计单倍型数,计算单倍型多样性Haplotypes diversity(h),核苷酸多样性Nucleotide diversity(π)。
1.4 系统发育关系分析与物种界定采用最大似然法(maximum likelihood,ML)、最大简约法(maximum parsimony, MP)和贝叶斯法(Bayesian inference,BI)构建分子系统发育树。利用jModeltest 2.0[20]以AIC(Akaike information criterion)为标准选出最佳模型(TIM3+I)。使用PhyML 3.0[21]构建最大似然(ML)树,并通过1 000次重复自举测试评估统计置信度。
采用Mrbayes 3.2.6 [22]进行贝叶斯推理(BI)分析,以随机树为起始树,替代模型参数Nst设置为6,采用马尔科夫链的蒙特卡洛(Markov Chain Monte Carlo, MCMC) 方法, 同时运行4条马尔科夫链(1条冷链及3条热链)运算1 000万代,每1 000次取样1次,去掉运算开始25%的不可信老化树, 直到链的收敛分离频率平均标准方差小于0.01停止运算。将得到的一致树用FigTree 1.4.0查看并编辑。
使用PAUP 4.0[23]对数据进行最大简约法(MP)系统发育分析,采用启发式搜索(heuristic search),树二等分再连接(tree-bisection-reconnection, TBR)的分支交换法。逐步加入算法(stepwise addition tree), random-addition sequences设置为10次。转换(transition, Ti)和颠换(transversion, Tv)设置为相同的权重,分支置信度以1 000次自引导法(bootstrap analysis, BP)进行评估。
以ASAP (assemble species by automatic partitioning)法进行物种界定[24],将光唇鱼属各单倍型序列输入ASAP网站(https://bioinfo.mnhn.fr/abi/public/asap/asapweb.html)运行物种界定,采用Jukes-Cantor(JC69)模型进行分析。
2 结果 2.1 COⅠ基因序列特征经比对以及手工校正,得到COⅠ基因全长序列1 551 bp,共编码517个氨基酸。其中:保守位点(conserved sites)1 298个,占83.69%;可变位点(variable sites)253个,占16.31%。简约信息位点(parsimony-informative sites)247个,占15.92%;单突变位点(singleton sites)6个,占0.38%。碱基组成:A=27.0%、T=28.9%、C=26.7% 和G=17.4%, A+T的含量较高为55.9%,明显高于G+C的含量(44.1%)。核苷酸碱基组成表现出较为明显的碱基偏倚性。
2.2 单倍型分布情况及遗传多样性6种76尾光唇鱼样本共检测到21种单倍型,各群体中单倍型个数为2~6个。平均单倍型多样性(h)为0.915 8±0.017 8,平均核苷酸多样性(π)为0.053 03±0.004 08(表 1),如此高的核苷酸多样性水平是由于物种间分化较大所致。除光唇鱼因来自两个水系且样本较少外,其余各种的单倍型多样性指数皆处于0.6左右的中等水平;核苷酸多样性(π)在0.001左右,呈现低核苷酸多样性水平,其中温州光唇鱼和武夷光唇鱼的核苷酸多样性最低,分别为0.000 77和0.000 82。
2.3 系统发育关系以东方墨头鱼、纹唇鱼和台湾白甲鱼作为外类群,采用MP、ML和BI等3种方法分别构建系统发育树,其结果显示3种树的拓扑结构基本一致(图 1)。6种光唇鱼除武夷光唇鱼外皆各自形成了单系群,其中半刺光唇鱼处于树的基部,台湾光唇鱼和武夷光唇鱼聚为单系群,具有极高的支持率(100/100/1),它们与光唇鱼、温州光唇鱼和克氏光唇鱼组成的单系群互为姐妹群。
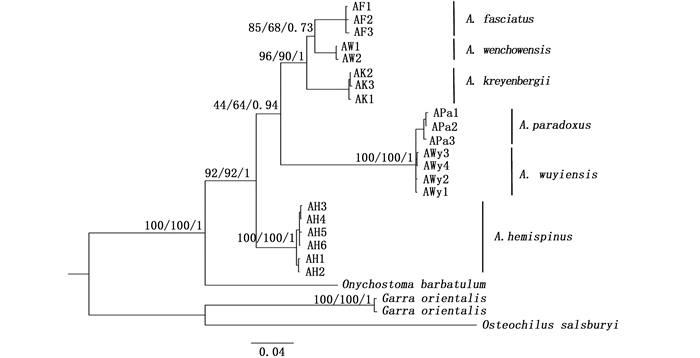
|
分支长度取自BI树。节点上的数字依次为MP/ML/BI分析的自展支持率(bootstrap value, BV)和后验概率值(posterior probability, PP)。 Branch length is taken from the BI analyses. The numbers on the nodes are the bootstrap values and posterior probabilities from MP/ML/BI analyses. 图 1 基于COⅠ基因序列采用最大简约法(MP)、最大似然(ML)和贝叶斯法(BI)3种方法建立的系统发育树 Fig. 1 Phylogenetic trees derived from Maximum parsimony (MP), Maximum likelihood (ML) and Bayesian (BI)methods showing the phylogeney of Acrossocheilus based on complete mitochondrial CO Ⅰ sequences |
种间和种内平均K 2-P遗传距离如表 3所示。种内遗传距离为0.077%~0.302%,种间遗传距离为0.932%~10.826%。如果从种间遗传距离来看,仅台湾光唇鱼与武夷光唇鱼的遗传距离小于2%,为0.932%,其次为温州光唇鱼与光唇鱼、克氏光唇鱼的遗传距离为4%~5%,其余各种间的遗传距离均在10%以上水平。
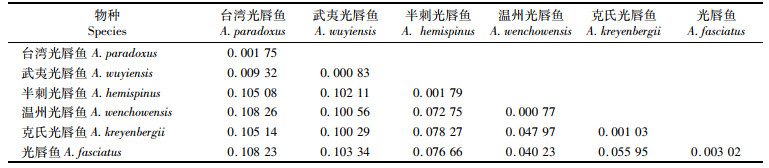
|
表 3 光唇鱼属群体内、群体间K 2-P遗传距离 Tab.3 Average K 2-P distance between and within nine groups of Acrossocheilus populations |
ASAP物种界定分析结果显示(图 2),除外类群3种以外,光唇鱼属分5群为最佳,包括:第1群为半刺光唇鱼、第2群为光唇鱼、第3群为温州光唇鱼、第4群为克氏光唇鱼、第5群为台湾光唇鱼和武夷光唇鱼, ASAP分数最低的为最佳分群。
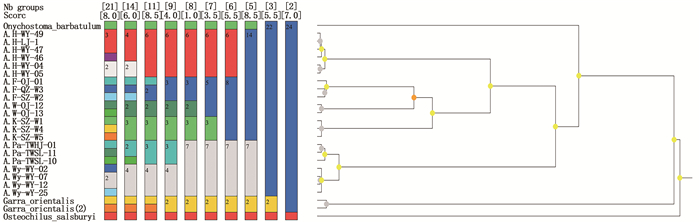
|
图 2 ASAP物种界定法的分析结果 Fig. 2 Result based on assemble species by automatic partitioning method (ASAP) |
HEBERT等[25]提出的基于COⅠ基因序列鉴别物种的关键是种间遗传距离必须大于种内遗传距离, 而且距离差异大于10倍。并且HEBERT[26]对动物界11个动物门13 320个物种进行研究发现物种内的遗传距离绝大多数小于1%,很少有大于2%。本研究结果显示,台湾的台湾光唇鱼与闽江的武夷光唇鱼构成单系群,其遗传距离仅为0.932%,远远小于2%,而这两种与本研究中同属其他物种之间的遗传距离均超过了10%。尽管这两个物种间的遗传距离未完全达到群体内遗传距离台湾光唇鱼(0.175%)和武夷光唇鱼(0.083%)的10倍以上,但以ASAP物种界定法分析结果也将台湾光唇鱼和武夷光唇鱼归为同一物种。JU等[9]研究结果显示,台湾和闽江的台湾光唇鱼种群并没有各自形成单系群,而是形成4个单系群,闽江种群(应为武夷光唇鱼)为一单系群与台湾北部种群形成的单系群为姐妹群关系,台湾其余种群组成的两个单系群为姐妹群关系,表明闽江种群与台湾北部地区的种群间亲缘关系更近,甚至近于台湾岛北部与南部种群间的亲缘关系。通过检视标本形态特征来看,二者体色和横条纹一致,背鳍鳍膜均为黑色,下唇结构因个体大小和性别差异区别也不稳定,另外武夷光唇鱼背鳍末根不分枝、鳍条后缘均有细锯齿,15尾台湾光唇鱼标本中背鳍末根不分枝鳍条后缘光滑和有细锯齿的类型皆有(6尾Vs 9尾)。因此,没有明显和稳定的形态特征作为二者的鉴别性状。综上分析,闽江分布的武夷光唇鱼A.wuyiensis(Wu & Chen,1981)与台湾光唇鱼A.paradoxus(Gunther,1868)应为同一物种,武夷光唇鱼为台湾光唇鱼的同物异名。
尽管本研究的系统发育关系分析结果显示台湾与闽江的台湾光唇鱼种群共同构成了单系群,但YUAN等[8]的结果支持闽江和台湾的台湾光唇鱼种群各自为单系群,JU等[9]的研究结果也支持闽江种群为一单独谱系。因而对于该物种,至少应该分为闽江和台湾两个进化显著单位(evolutionary significant unit, ESU)分别进行保护。研究结果表明,闽江和台湾的台湾光唇鱼种群遗传多样性均处于较低水平,闽江种群核苷酸多样性相比台湾种群水平更低,因此闽江种群更应加强保护。
| [1] |
伍献文, 曹文宣, 易伯鲁, 等. 中国鲤科鱼类志[M]. 上海: 上海科学技术出版社, 1982. WU X W, CAO W X, YI B L, et al. The cyprinid fishes of China[M]. Shanghai: Shanghai Scientific & Technical Publishers, 1982. |
| [2] |
郑慈英. 珠江鱼类志[M]. 北京: 科学出版社, 1989. ZHENG C Y. The fishes of pearl river[M]. Beijing: Science Press, 1989. |
| [3] |
朱元鼎, 伍汉霖, 金鑫渡, 等. 福建鱼类志[M]. 福州: 福建科学技术出版社, 1985. ZHU Y D, WU H L, JIN X D, et al. The fishes of Fujian[M]. Fuzhou: Science and Technology of Fujian Press, 1985. |
| [4] |
单乡红, 林人端, 乐佩琦, 等. 中国动物志, 硬骨鱼纲, 鲤形目(下卷)[M]. 北京: 科学出版社, 2000. SHAN X H, LIN R D, LE P Q, et al. Fauna sinica, Barbinae, Cypriniformes, Osteichthyes[M]. Beijing: Science Press, 2000. |
| [5] |
吴秀鸿, 陈焕新, 曹兴源, 等. 福建光唇鱼属一新种[J]. 武夷科学, 1981, 1: 126-128. WU X H, CHEN H X, CAO X Y, et al. A new species of fish of the genus Acrossocheilus from Fujian[J]. Wuyi Science Journal, 1981, 1: 126-128. |
| [6] |
SUNG W S, LEE S C, YU M J. Taxonomic status of the fishes of Acrossocheilus formosanus and A. labiatus (Cyprinidae: Barbinae) from Taiwan based on isozyme electrophoresis[J]. Bulletin of the Institute of Zoology Academia Sinica, 1993, 32(2): 127-139. |
| [7] |
SHEN S C. Fishes of Taiwan[M]. Taipei, China: Department of Zoology, Taiwan University, 1993.
|
| [8] |
YUAN L Y, LIU X X, ZHANG E. Mitochondrial phylogeny of Chinese barred species of the cyprinid genus Acrossocheilus Oshima, 1919 (Teleostei: Cypriniformes) and its taxonomic implications[J]. Zootaxa, 2015, 4059(1): 151-168. DOI:10.11646/zootaxa.4059.1.8 |
| [9] |
JU Y M, HSU K C, YANG J Q, et al. Mitochondrial diversity and phylogeography of Acrossocheilus paradoxus (Teleostei: Cyprinidae)[J]. Mitochondrial DNA Part A, 2018, 29(8): 1194-1202. DOI:10.1080/24701394.2018.1431227 |
| [10] |
侯晓静. 中国光唇鱼属亲缘地理研究[D]. 上海: 上海海洋大学, 2019. HOU X J. Phylogeography of genus Acrossocheilus (Teleostei: Cyprinidea) in China[D]. Shanghai: Shanghai Ocean University, 2019. |
| [11] |
HOU X J, LIN H D, TANG W Q, et al. Complete mitochondrial genome of the freshwater fish Acrossocheilus longipinnis (Teleostei: Cyprinidae): genome characterization and phylogenetic analysis[J]. Biologia, 2020, 75(11): 1871-1880. DOI:10.2478/s11756-020-00440-y |
| [12] |
WARD R D. FISH-BOL, a case study for DNA barcodes[M]//KRESS W J, ERICKSON D L. DNA Barcodes: Methods and Protocols. Totowa: Humana Press, 2012: 423-439.
|
| [13] |
席晓晴, 鲍宝龙, 章守宇. DNA条形码在鱼类胃含物种类鉴定中的应用[J]. 上海海洋大学学报, 2015, 24(2): 203-210. XI X Q, BAO B L, ZHANG S Y. Application of DNA barcoding in species analysis of fish stomach content[J]. Journal of Shanghai Ocean University, 2015, 24(2): 203-210. |
| [14] |
HANNER R, BECKER S, IVANOVA N V, et al. FISH-BOL and seafood identification: Geographically dispersed case studies reveal systemic market substitution across Canada[J]. Mitochondrial DNA, 2011, 22(s1): 106-122. |
| [15] |
陈月华, 何培民, 杨金权. 基于DNA条形码的如东海域浒苔附着鱼卵的物种鉴定[J]. 上海海洋大学学报, 2018, 27(1): 1-7. CHEN Y H, HE P M, YANG J Q. Molecular identification of fish eggs in Enteromorpha of Rudong sea area based on DNA barcode[J]. Journal of Shanghai Ocean University, 2018, 27(1): 1-7. |
| [16] |
LAKRA W S, SINGH M, GOSWAMI M, et al. DNA barcoding Indian freshwater fishes[J]. Mitochondrial DNAPart A, 2016, 27(6): 4510-4517. DOI:10.3109/19401736.2015.1101540 |
| [17] |
SWINDELL S R, PLASTERER T N, SEQMA N. Contig assembly[J]. Methods in Molecular Biology, 1997, 70: 75-89. |
| [18] |
KUMAR S, STECHER G, TAMURA K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [19] |
LIBRADO P, ROZAS J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. |
| [20] |
DARRIBAD, TABOADA G L, DOALLO R, et al. jModelTest 2: more models, new heuristics and parallel computing[J]. Nature Methods, 2012, 9(8): 772. |
| [21] |
GUINDON S, DUFAYARD J F, LEFORT V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0[J]. Systematic Biology, 2010, 59(3): 307-321. |
| [22] |
RONQUIST F, HUELSENBECK J P. MrBayes 3: Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 2003, 19(12): 1572-1574. |
| [23] |
SWOFFORD D L. PAUP phylogenetic analysis using parsimony (*and Other Methods). Version 4.0b10[M]. Sunderland: Sinauer Associates, 2002.
|
| [24] |
PUILLANDRE N, BROUILLET S, ACHAZ G. ASAP: assemble species by automatic partitioning[J]. Molecular Ecology Resources, 2021, 21(2): 609-620. |
| [25] |
HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(1512): 313-321. |
| [26] |
HEBERT P D N, RATNASINGHAM S, WAARD J R D. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society of London B: Biological Sciences, 2003, 270(s): 96-99. |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China
 2022,
Vol. 31
2022,
Vol. 31


