白洋淀流域位于海河流域大清河中部,由大小9条呈扇形河网汇流于白洋淀,在第三纪晚期经历了兴起、扩张与收缩的演变过程,在第四纪形成最初环境[1]。拒马河为白洋淀西北部的支流,发源于河北省涞源县西北太行山麓,自西向东流至张坊分为南北二支,北拒马河下游为白沟河,与南拒马河(现今为干涸状态)在白沟镇汇合后注入白洋淀。沙河位于白洋淀西部,发源于山西省灵丘县太白山碾盘岭北麓,沿地势向东南方向流入王快水库,后通过沙河干渠注入白洋淀。第四纪冰期和间冰期的往复振荡,造成气候的冷暖波动,白洋淀流域经历了周期性海进海退[1-2],使得该地区鱼类群落结构和种群历史动态受到了强烈的影响。白洋淀流域为南方类型和北方类型的过渡地带,生物多样性较丰富,是中华多刺鱼(Pungitius sinensis)的最南分布区,多鳞白甲鱼(Onychostoma macrolepi)的最北分布区,北方须鳅(Barbatula nuda)的最东南分布区。在气候变化的背景下,人类活动的影响加剧了白洋淀流域部分河流干涸消亡、物种消失的现象,例如,南拒马河干涸消亡,拒马河和沙河曾分布的花斑副沙鳅(Parabotia fasciata)、武昌副沙鳅(Parabotia banarescui)、漓江副沙鳅(Parabotia lijiangensis)、东方薄鳅(Leptobotia orierntalis)、黄线薄鳅(Leptobotia flavolineata)、多鳞产颌鱼(Varicorhinus macrolepis)和北京鳊(Parabramis pekinensis)[3-4]均已在白洋淀流域消失。遗传多样性的研究对物种资源评定及种群保护有重要意义[5],是评价物种演化潜力及环境生存能力的一个重要指标[6-7]。因此,开展白洋淀流域种群遗传多样性研究对白洋淀流域种质资源保护和利用提供基础数据,为白洋淀流域保护区域的划分提供依据。
宽鳍鱲(Zacco platypus)隶属于鲤形目(Cypriniform)鲤科(Cyprinidae),广泛分布于我国东部沿海各溪流中[8]。目前,国内关于宽鳍鱲的研究[9-10]显示宽鳍鱲可能存在隐形种。以秦岭为界,淮河以北宽鳍鱲种群具有较近的亲缘关系[11-12]。对宽鳍鱲遗传多样性的研究[9-13]主要集中于南方地区,例如:梁晓旭等[14]基于Cytb基因对广东境内9条水系宽鳍鱲的遗传多样性研究显示,广西中西部地区为宽鳍鱲种群的扩散中心;ZHENG等[13]基于Cytb基因对黄山地区、四川、湖南、广西以及北京宽鳍鱲种群遗传多样性研究显示,黄山地区宽鳍鱲种群较其他地区具有较大的遗传分化;刘慧芬等[15]基于Cytb基因对河南境内的长江、淮河、黄河以及海河宽鳍鱲种群遗传多样性研究显示,长江水系宽鳍鱲种群遗传分化较大。2018—2019年对白洋淀流域的“一淀、两库和四条河流”,即白洋淀区、王快水库、西大洋水库及入淀或库区河流,沙河、拒马河、府河和孝义河等水域渔获物的调查发现,宽鳍鱲仅在沙河和拒马河有分布,且为优势物种之一。本文以Cytb基因及COI基因为分子遗传标记,对沙河和拒马河宽鳍鱲遗传多样性及种群结构进行分析,探究种群动态变化的主要历史原因,为白洋淀流域宽鳍鱲种群保护提供理论基础。
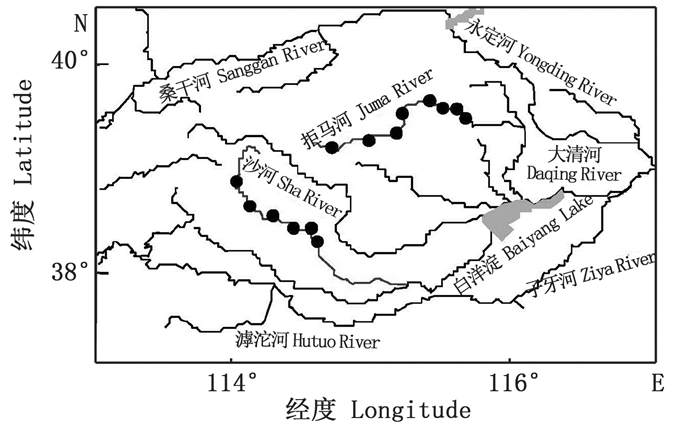
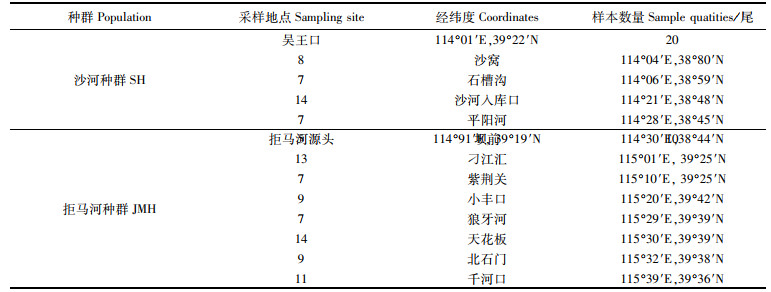
1 材料与方法 1.1 试验材料样本于2018年10月—2019年11月采自白洋淀流域沙河及拒马河,共计141尾,见图 1和表 1。采集工具主要为地笼和丝网。样本采集后,根据形态特征及分子数据进行鉴定,新鲜样本用95%乙醇固定,置于-20 ℃冰箱中保存备用,保存于河北大学水生生物学博物馆。
|
图 1 采样点示意图 Fig. 1 Map showing the collection sites |
|
表 1 样本采集信息 Tab.1 Sampling information |
取每尾样本左侧胸鳍剪碎,使用组织基因组提取试剂盒DP304(北京天根生化科技有限公司)进行DNA提取。Cytb基因扩增和测序引物为L14724(GACTTGAAAAACCACCGTTG) 和H15915(CTCCGATCTCCGGATTACAAGAC)[16];COI基因扩增和测序引物为F1(TCA- ACCAACCACAAAGACATTGGCAC)和R1(TAGACTTCTGGGTGGCCAAAGAATCA)[17]。Cytb的PCR反应条件:95 ℃预变性3 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸40 s,共30个循环;最后72 ℃延伸10 min,4 ℃停止反应;COI 基因退火温度为51 ℃。其他扩增条件与Cytb基因相同。PCR产物用1%琼脂糖凝胶电泳检测,条带清晰明亮产物送往通用生物系统(安徽)有限公司完成序列测定。
1.3 数据处理和分析将DNA序列用BioEdit 7.0.9 [18]软件进行比对和拼接,利用MEGA 5.05[19]软件分析序列碱基组成、变异情况,计算转换/颠换比率等相关系数,基于Kimura-2参数模型计算地理种群间的遗传距离。利用DNAsp 5.1[20]软件进行遗传多样性相关分析,计算种群间核苷酸歧义度Dxy,并分析核苷酸多样性(π)及单倍型多样性(h)。使用Arlequin 3.5[21]软件计算种群分化系数(FST)并对种群进行中性检验及错配分析,采用贝叶斯天际线(Bayesian skyline plot, BSP)检测有效种群的变动。运用BEAST 2.4.8软件[22],以Cytb基因每百万年1%的进化速率作为参考,基于贝叶斯溯组天际线模型(Bayesian coalescent skyline),采用马尔科夫链的蒙特卡洛算法(Monte Carlo Markov chain,MCMC)运行1×108代,结果通过Tracer 1.7[23]中的Bayesian skyline重建功能进行读取。以jModelTest 2.1.7软件[24]检测到最适合的进化模型为HKY+F+G4模型。使用MrBayes 3.1.1[25]和IQtree程序[26]分别基于贝叶斯法(BI)和最大似然法(ML)构建单倍型系统发育树,BI分析运用MrBayes 3.1.1程序,4条马尔科夫链同时运行1×106代,每100代对系统树进行抽样,最终构建一致树,通过后验概率评估每个节点。ML分析采用Phylosuit软件中的IQtree程序,并通过具有1 000个重复的bootstrap分析评估每个节点的可信度。选取鲤科马口鱼属马口鱼(Opsariichthys bidens,Cytb序列号AY646649及COI序列号MF122595)作为外来群构建系统发育树。利用PoPart 1.7[27]软件,采用中接法(Median-joining)构建单倍型网络图。利用Structure2.3.4[28]软件分析种群遗传结构。参考鲤科鱼类线粒体Cytb每百万年0.76%~1.00%的进化速率[29-30],利用公式(1)估算各类群间分歧时间[31]。
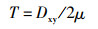 (1)
(1)
式中:Dxy为核苷酸歧义度,由DNAsp软件计算得出; μ为平均进化速率; T代表分歧时间。根据FST值计算种群间的基因流,计算公式为
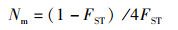 (2)
(2)
式中:Nm指地理种群之间的基因流; FST为地理种群之间的遗传分化系数。
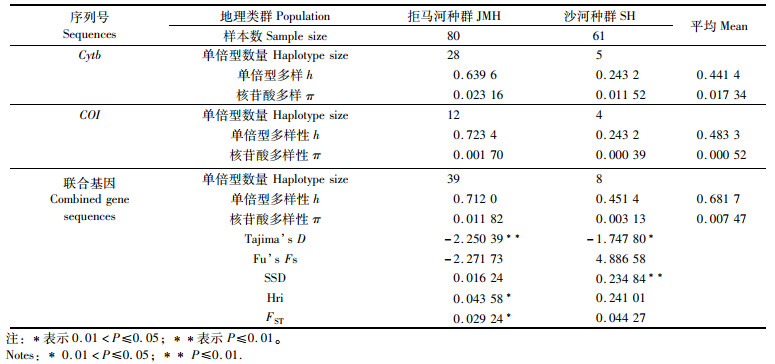
2 结果 2.1 序列特征和遗传多样性分析来自沙河及拒马河141条线粒体Cytb序列对比后总长度为1 110 bp:保守位点1 066个,约占序列总长度的96.04%;简约信息位点17个,约占序列总长度的1.53%;变异位点(多态性位点)44个,约占序列总长度的3.96%;碱基组成T为31.16%,C为26.51%,A为26.19%,G为16.15%;141个个体中共确认单倍型31个,单倍型多样性(h)平均值为0.441 4,其中沙河种群为0.243 2,拒马河种群为0.639 6,核苷酸多样性(π)平均值为0.017 34,其中沙河种群为0.011 52,拒马河种群为0.023 16(表 2)。141条COI序列对比后总长度为643 bp:保守位点632个,约占序列总长度的98.29%;简约信息位点8个,约占序列总长度的1.24%;变异位点11个,约占序列总长度的1.71%;碱基组成T为29.91%,C为26.54%,A为24.57%,G为18.98%;141个个体中共确认单倍型15个,单倍型多样性(h)平均值为0.483 3,其中沙河种群为0.243 2,拒马河种群为0.723 4,核苷酸多样性(π)平均值为0.017 34,其中沙河种群为0.000 39,拒马河种群为0.001 70。与Cytb相比,COI序列相对更为保守,具有较少的变异位点。
|
表 2 宽鳍鱲线粒体基因遗传多样性、中性检验与错配分析 Tab.2 Genetic diversity, neutrality tests and mismatch distribution based on mtDNA of Zacco platypus |
141条序列联合后总长度为1 753 bp,共确认单倍型有45个,种群间共享单倍型有2个(Hap_2和Hap_17),见表 2。保守位点1 698个,约占序列总长度的96.86%;简约信息位点25个,约占序列总长度的1.42%;变异位点55个,约占序列总长度的3.14%;碱基组成T为30.67%,C为26.53%,A为25.61%,G为17.19%;单倍型多样性(h)平均值为0.681 7,其中沙河种群为0.451 4,拒马河种群为0.712 0,核苷酸多样性(π)平均值为0.007 47,其中沙河种群为0.003 13,拒马河种群为0.011 82。其中,拒马河种群较沙河种群具有相对高的单倍型多样性和核苷酸多样。
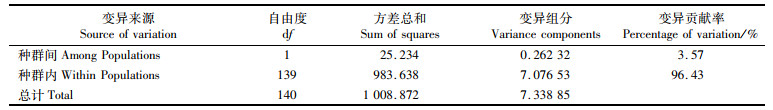
基于联合基因AMOVA分析显示,遗传变异主要来源于种群内,占遗传总变异的96.43%,高于种群间变异(3.57%),见表 3。种群间的遗传分化系数(FST)为0.035 74(P < 0.01),种群间基因流Nm为6.745,遗传距离为0.003,地理种群之间存在基因交流现象(图 2)。
|
表 3 基于Cytb和COI 基因联合计算种群内和种群间变异的分子方差分析 Tab.3 Analysis of molecular variance (AMOVA) among populations and within populations based on combined Cytb and COI gene sequences |
|
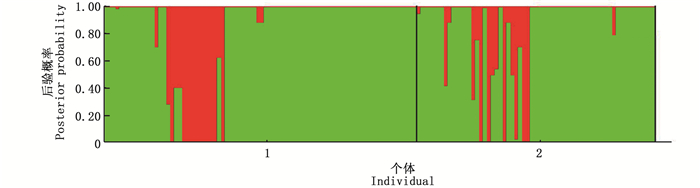
红色与绿色代表不同种源地种群中的个体属于不同祖先种群的可能性(K=2);1.拒马河种群;2.沙河种群。 Red and green represent the possibility that samples from different provenance populations belong to different ancestral populations(K=2);1.JMH population; 2.SH population. 图 2 宽鳍鱲种群遗传结构图 Fig. 2 Genetic structure of the population of Zacco platypus |
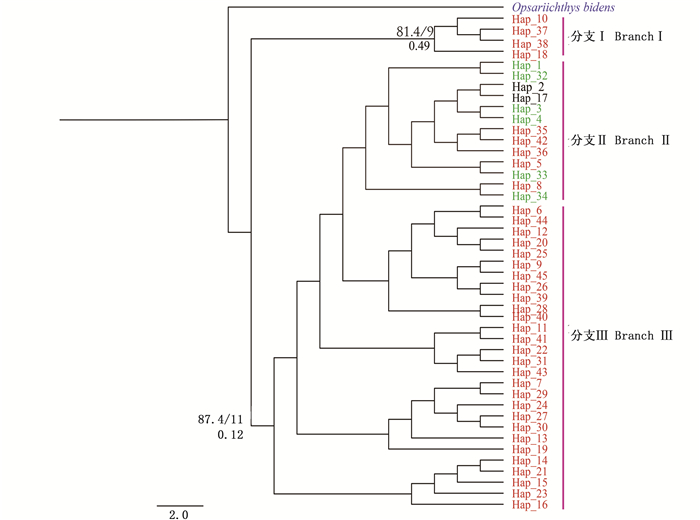
基于联合基因,以BI法和ML法分别构建单倍型系统发育树(图 3),BI树和ML树拓扑结构大体一致。拓扑结构可分为3大分支,分支Ⅰ和分支Ⅲ均为拒马河种群单倍型,分支Ⅱ包含沙河种群单倍型与拒马河种群单倍型以及种群间共享单倍型hap_2和hap_17。以Cytb基因作为参考,根据核苷酸歧义度Dxy估算分支分歧时间:拒马河种群及沙河种群分歧时间约为0.089百万年前~0.067百万年前。单倍型网格图(图 4)显示: 拒马河种群独有单倍型与缺失单倍型连线较多,呈网状结构,局部为辐射状结构;沙河种群以共享单倍型hap_2为中心呈现辐射状结构。
|
红色.拒马河种群单倍型;绿色.沙河种群单倍型;黑色.共享单倍型;蓝色.外类群;节点上方为BI后验概率,下方为ML支持率。 Red.haplotypes of Juma River; Green.haplotypes of Sha River; Black.median vectors; Blue.Outgroup; Posterior probability for Bayesian inferences above branch and bootstrap support for ML below the branch. 图 3 基于Cytb和COI基因联合构建的宽鳍鱲贝叶斯(BI)单倍型系统发育树 Fig. 3 Haplotype Bayesian tree (BI) based on combined Cytb and COI gene sequences of Zacco platypus |

|
红色.拒马河种群(JMH);绿色.沙河种群(SH);黑色.缺失单倍型。 Red. haplotypes of Juma River; Green.haplotypes of Sha River; Black.median vectors. 图 4 基于Cytb基因和COI基因联合分析的单倍型网络图 Fig. 4 Haplotype network based on combined Cytb and COI gene sequences |
中性检验显示,拒马河种群Tajima’s D呈显著负值(P < 0.05),Fu’s Fs为不显著负值(表 2),表明不符合中性进化假说;错配分析中Hri统计显著(P < 0.05),表明拒绝种群扩张假说;错配分布图显示为锯齿状下降曲线(图 5a),说明拒马河种群未出现种群扩张现象,但贝叶斯天际线点图(图 5b)显示拒马河种群在0.004百万年前经历了种群扩张,后趋于稳定。沙河种群Tajima’s D为显著负值(P < 0.05),Fu’s Fs为正值(表 2),表明未偏离假设,符合中性进化假说;错配分析显示SSD值统计显著(P < 0.05),错配分布图(图 5c)为平滑下降曲线,表明拒绝种群扩张假说;贝叶斯天际线点图(图 5d)显示沙河种群有效种群较稳定,即沙河种群未经历过种群扩张。白洋淀流域宽鳍鱲有效种群呈先扩张后趋于稳定状态(图 5e)。
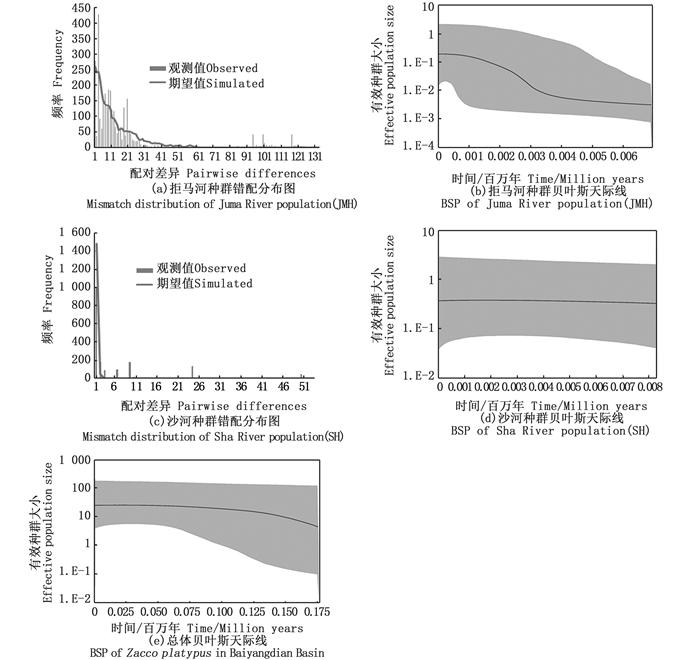
|
图 5 宽鳍鱲不同种群错配分布图及贝叶斯天际线点图 Fig. 5 Mismatch distribution and BSP (Bayesian Skyline Plots) of different populations of Zacco platypus |
种群遗传多样性能够反映生物对环境变迁与自然选择的效应。种群遗传多样性越高,其演化潜力越大,适应生存环境能力越强,种群遗传多样性越低,承受的生存压力越大[6]。刘慧芬等[15]基于COI基因对河南省的海河、黄河、淮河和长江水系宽鳍鱲种群遗传结构研究显示,海河种群的单倍型多样性和核苷酸多样性(h=0.333 33, π=0.000 53)低于长江、淮河和黄河等水系。曹安娜[12]基于Cytb和COI基因对河南的海河、北京的滦河、辽宁的辽河以及安徽和浙江等地的宽鳍鱲遗传多样性研究显示,海河种群的单倍型多样性和核苷酸多样性(h=0.627 0, π=0.001 03和h=0.480 0, π=0.000 31)低于辽河和浙江等地。本文基于Cytb基因和COI基因对白洋淀流域沙河及拒马河宽鳍鱲种群遗传多样性的研究,结果与以上研究相似,显示出较低的遗传多样性,平均单倍型多样性分别为0.441 4和0.483 3,平均核苷酸多样性分别为0.017 34和0.000 52。沙河种群(h=0.451 4,π=0.003 13)较拒马河种群(h=0.712 0,π=0.011 82)呈现更低的单倍型多样性与核苷酸多样性。一般认为低的单倍型多样性和低的核苷酸多样性表示种群近期经历过奠基者效应或瓶颈效应[32]。白洋淀流域位于第四纪冰期活动频繁地带,经历了6次海进海退[2],该地区种群历史动态和群落结构受到了强烈影响,承受了较大的生存压力,种群遗传结构通常为低的单倍型多样性和低的核苷酸多样性。白洋淀流域由于受人类活动影响,鱼类优势种由大型化、对环境要求较高的鱼类,转变为小型化、适应能力强的鱼类[33],此现象在流域内仍然存在,因此建议采取禁止捕捞及污染物定期处理等措施,加强流域环境的管理,有效保护生物栖息环境,尽量减少流域内鱼类资源的丧失。
3.2 遗传分化种群的遗传结构变异分析能够评估种群对环境变化的适应能力以及生存和演化的潜力[34]。拒马河种群与沙河种群的变异来源主要是种群内变异(96.43%),种群间的遗传分化系数FST小于0.05(FST=0.035 74,P < 0.01),表明这2个种群之间没有遗传分化[35];结合系统发育树和单倍型网格图,沙河种群与拒马河种群存在共享单倍型,种群之间基因流较大(Nm>1),在相当程度上阻碍了种群间的分化[36],沙河种群与拒马河种群存在基因渐渗现象。通常分布较广的物种在没有地理屏障的情况下,地理距离越远其遗传分化程度越大[37]。宽鳍鱲分布极为广泛,在我国东部各大水系中均有分布。刘慧芬等[15]研究显示宽鳍鱲海河种群与其地理距离较远的长江种群遗传分化较大,而与其他地理距离较近的黄河种群遗传分化很小。曹安娜[12]基于COI基因的研究也显示地理距离较近的辽河种群、滦河种群和海河种群遗传分化小,而与地理距离较远的浙江和安徽等地的种群遗传分化大。这在一定程度上与地质历史事件相吻合,在地质史上,古黄河、古辽河以及两河中间水系,如海河水系和滦河水系曾为同一水系[38],宽鳍鱲在第四纪冰期期间曾在淮河以北辽河以南等水系进行迁徙,种群间有基因交流。
3.3 种群历史动态分析当种群经历过扩张事件时,中性检验Tajima’s D和Fu’s Fs的值呈显著负值(P < 0.01)[39],核苷酸错配分布图为单峰泊松分布,错配分析Hri和SSD检验均不显著(P>0.05)[36]。实际上,种群历史动态远比模型分析的结果更加复杂,且不同模型分析的结果可能不一致。本研究中,拒马河种群核苷酸分布图未呈单峰泊松分布,为锯齿状下降曲线;中性检验Fu’s Fs呈不显著负值,Tajima’s D呈显著负值(P < 0.01),有学者认为Fu’s Fs检验对种群扩张敏感[40],因此判断拒马河种群近期未经历过种群扩张。但贝叶斯天际线图显示,拒马河种群在0.004百万年前经历了种群扩张,结合单倍型网络图,拒马河种群局部呈多个星状辐射,表示拒马河种群在历史上曾发生局部扩张事件,种群扩张事件发生在末次冰期结束时间(0.075百万年前~0.01百万年前)。沙河种群保持稳定状态,较拒马河种群呈现低单倍型多样性与低核苷酸多样性,结合单倍型网络图,推测沙河种群曾经历过奠基者效应,在末次冰期结束时,冰川融化使得河流水位上升,不同河流之间有了临时联系,部分拒马河种群个体迁移至沙河形成沙河种群。由于沙河种群进化历史短,积累的遗传变异较拒马河种群少,因此沙河种群单倍型多样性与核苷酸多样性均低于拒马河种群。参考鲤科鱼类Cytb基因进化速率,根据Dxy估算了拒马河种群及沙河种群分歧时间大约为晚更新世0.089百万年前~0.067百万年前,处于第四次间冰期和末次冰期期间。
| [1] |
常利伟. 白洋淀湖群的演变研究[D]. 长春: 东北师范大学, 2014. CHANG L W. The evolution of Baiyangdian Lake group research[D]. Changchun: Northeast Normal University, 2014. |
| [2] |
姚政权, 石学法. 渤海湾沿岸第四纪海侵研究进展[J]. 海洋地质前沿, 2015, 31(2): 9-16, 70. YAO Z Q, SHI X F. A review of quaternary transgression researches along the Bohai Bay[J]. Marine Geology Frontiers, 2015, 31(2): 9-16, 70. |
| [3] |
李国良. 关于河北省淡水鱼类区系的探讨[J]. 动物学杂志, 1986, 21(4): 4-9, 12. LI G L. Discussion on freshwater fish fauna in Hebei province[J]. Chinese Journal of Zoology, 1986, 21(4): 4-9, 12. |
| [4] |
赵连有. 河北省鳅科鱼类的检索[J]. 河北师范大学学报(自然科学版), 1991(4): 96-97. ZHAO L Y. Retrieval of loach fishes in Hebei province[J]. Journal of Hebei Normal University (Natural Science Edition), 1991(4): 96-97. |
| [5] |
LOHMAN D J, PEGGIE D, PIERCE N E, et al. Phylogeography and genetic diversity of a widespread old world butterfly, Lampides boeticus (Lepidoptera: Lycaenidae)[J]. BMC Evolutionary Biology, 2008, 8: 301. DOI:10.1186/1471-2148-8-301 |
| [6] |
韩增超. 黑颈长尾雉圈养种群的遗传多样性分析[D]. 桂林: 广西师范大学, 2012. HAN Z C. Analysis of the genetic diversity in Syrmaticus humiae in artificially reared[D]. Guilin: Guangxi Normal University, 2012. |
| [7] |
MILOT E, GIBBS H L, HOBSON K A. Phylogeography and genetic structure of northern populations of the yellow warbler (Dendroica petechia)[J]. Molecular Ecology, 2000, 9(6): 667-681. DOI:10.1046/j.1365-294x.2000.00897.x |
| [8] |
陈宜瑜. 中国动物志, 硬骨鱼纲, 鲤形目(中卷)[M]. 北京: 科学出版社, 1998. CHEN Y Y. Fauna sinica, Osteichthyes, Cypriniformes (c)[M]. Beijing: Science Press, 1998. |
| [9] |
PERDICES A, CUNHA C, COELHO M M. Phylogenetic structure of Zacco platypus (Teleostei, Cyprinidae) populations on the upper and middle Chang Jiang (=Yangtze) drainage inferred from cytochrome b sequences[J]. Molecular Phylogenetics and Evolution, 2004, 31(1): 192-203. DOI:10.1016/j.ympev.2003.07.001 |
| [10] |
WANG X, LIU F, YU D, et al. Mitochondrial divergence suggests unexpected high species diversity in the opsariichthine fishes (Teleostei: Cyprinidae) and the revalidation of Opsariichthys macrolepis[J]. Ecology and Evolution, 2019, 9(5): 2664-2677. DOI:10.1002/ece3.4933 |
| [11] |
邢迎春. 中国宽鳍鱲Zacco platypus (Cypriniformes: Cyprinidae)分子系统学, 物种分化及其生物学特性的研究[D]. 武汉: 华中师范大学, 2007. XING Y C. Study on molecular systematics, speciation and biological characteristics of Zacco platypus (Cypriniformes: Cyprinidae) in China[D]. Wuhan: Central China Normal University, 2007. |
| [12] |
曹安娜. 中国不同地区宽鳍鱲的系统地理学及甬江种群的生物学研究[D]. 广州: 中山大学, 2018. CAO A N. Phylogeographies of Zacco platypus populations from different places in China and the biological study of Yongjiang population[D]. Guangzhou: Sun Yat-Sen University, 2018. |
| [13] |
ZHENG X, ZHOU T Q, WAN T, et al. Huangshan population of Chinese Zacco platypus (Teleostei, Cyprinidae) harbors diverse matrilines and high genetic diversity[J]. Zoological Research, 2016, 37(2): 103-109. |
| [14] |
梁晓旭, 庆宁, 杨柯林, 等. 广东地区宽鳍鱲种群遗传变异和亲缘地理[J]. 水生生物学报, 2010, 34(4): 806-814. LIANG X X, QING N, YANG K L, et al. Population genetic variation and phylogeography of Zacco platypus in Guangdong region[J]. Acta Hydrobiologica Sinica, 2010, 34(4): 806-814. |
| [15] |
刘慧芬, 张超, 王静, 等. 河南境内四水系宽鳍鱲野生群体的遗传多样性[J]. 中国水产科学, 2018, 25(2): 269-277. LIU H F, ZHANG C, WANG J, et al. Genetic diversity of wild Zacco platypus populations among four river systems in Henan province analyzed by mitochondrial CO I gene[J]. Journal of Fishery Sciences of China, 2018, 25(2): 269-277. |
| [16] |
XIAO W H, ZHANG Y P, LIU H Z. Molecular systematics of Xenocyprinae (Teleostei: Cyprinidae): taxonomy, biogeography, and coevolution of a special group restricted in East Asia[J]. Molecular Phylogenetics and Evolution, 2001, 18(2): 163-173. DOI:10.1006/mpev.2000.0879 |
| [17] |
WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia's fish species[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1847-1857. DOI:10.1098/rstb.2005.1716 |
| [18] |
HUSE S M, HUBER J A, MORRISON H G, et al. Accuracy and quality of massively parallel DNA pyrosequencing[J]. Genome Biology, 2007, 8(7): R143. DOI:10.1186/gb-2007-8-7-r143 |
| [19] |
TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [20] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [21] |
EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0): an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2007, 1: 47-50. |
| [22] |
BOUCKAERT R, HELED J, K? HNERT D, et al. BEAST 2:a software platform for Bayesian evolutionary analysis[J]. PLoS Computational Biology, 2014, 10(4): e1003537. DOI:10.1371/journal.pcbi.1003537 |
| [23] |
RAMBAUT A, DRUMMOND A. Tracer v1.4[J]. Encyclopedia of Atmospheric Sciences, 2007, 141(3567): 2297-2305. |
| [24] |
GUINDON S, GASCUEL O. A simple, fast, and accurate algorithm to estimate large phylogenies by maximum likelihood[J]. Systematic Biology, 2003, 52(5): 696-704. DOI:10.1080/10635150390235520 |
| [25] |
HUELSENBECK J P, RONQUIST F. MRBAYES: Bayesian inference of phylogenetic trees[J]. Bioinformatics, 2001, 17(8): 754-755. DOI:10.1093/bioinformatics/17.8.754 |
| [26] |
NGUYEN L T, SCHMIDT H A, VON HAESELER A, et al. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J]. Molecular Biology and Evolution, 2015, 32(1): 268-274. DOI:10.1093/molbev/msu300 |
| [27] |
LEIGH J W, BRYANT D, NAKAGAWA S. POPART: full-feature software for haplotype network construction[J]. Methods in Ecology and Evolution, 2015, 6(9): 1110-1116. DOI:10.1111/2041-210X.12410 |
| [28] |
PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959. DOI:10.1093/genetics/155.2.945 |
| [29] |
PERDICES A, VASIL'EVA E, VASIL'EV V. From Asia to Europe across Siberia: phylogeography of the Siberian spined loach (Teleostei, Cobitidae)[J]. Zoologica Scripta, 2015, 44(1): 29-40. DOI:10.1111/zsc.12085 |
| [30] |
DURAND J D, TSIGENOPOULOS C S, ÜNLÜ E, et al. Phylogeny and biogeography of the family Cyprinidae in the middle east inferred from cytochrome b DNA-evolutionary significance of this region[J]. Molecular Phylogenetics and Evolution, 2002, 22(1): 91-100. DOI:10.1006/mpev.2001.1040 |
| [31] |
张艳萍, 杜岩岩, 娄忠玉, 等. 甘肃省几种裂腹鱼类系统发育关系探讨[J]. 西北师范大学学报(自然科学版), 2013, 49(5): 91-96, 102. ZHANG Y P, DU Y Y, LOU Z Y, et al. Molecular phylogeny of schizothoracinae fishes in Gansu Province based on mitochondrial cytochrome b gene sequences[J]. Journal of Northwest Normal University (Natural Science), 2013, 49(5): 91-96, 102. DOI:10.3969/j.issn.1001-988X.2013.05.019 |
| [32] |
司从利. 珠江水系鳜鱼的遗传多样性研究[D]. 广州: 暨南大学, 2012. SI C L. Study on genetic diversity of Siniperca chuatsi sensu lato in the Pearl River[D]. Guangzhou: Jinan University, 2012. |
| [33] |
夏雪岭, 翟雷田. 几种在白洋淀野生状态下绝迹的经济鱼类及原因[J]. 河北渔业, 2014(5): 63-64. XIA X L, ZHAI L T. Several economic fishes extinct in the wild state of Baiyangdian Basin and the reasons[J]. Hebei Fisheries, 2014(5): 67-68. |
| [34] |
ARBOGAST B S. Phylogeography: the history and formation of species[J]. American Zoologist, 2001, 41(1): 134-135. DOI:10.1093/icb/41.1.134 |
| [35] |
冷欣, 王中生, 安树青, 等. 岛屿特有种全缘冬青遗传多样性的ISSR分析[J]. 生物多样性, 2005, 13(6): 546-554. LENG X, WANG Z S, AN S Q, et al. ISSR analysis of genetic diversity of Ilex integra, an insular endemic plant[J]. Biodiversity Science, 2005, 13(6): 546-554. DOI:10.3321/j.issn:1005-0094.2005.06.008 |
| [36] |
WRIGHT S. The genetical structure of populations[J]. Annals of Eugenics, 1951, 15(4): 323-354. |
| [37] |
王晓云, 徐旭, 程波翔, 等. 西伯利亚蓼EST-SSR引物开发和国内资源的遗传背景分析[J]. 中国实验方剂学杂志, 2018, 24(5): 43-49. WANG X Y, XU X, CHENG B X, et al. Development of EST-SSR Markers and genetic background analysis for Polygonum sibiricum in China[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2018, 24(5): 43-49. |
| [38] |
解玉浩. 辽河的鱼类区系[J]. 鱼类学论文集, 1981, 2: 111-120. XIE Y H. The ichthyofauna of the Liao River[J]. Transactions of the Chinese Ichthyological Society, 1981, 2: 111-120. |
| [39] |
张争世, 胡冰洁, 叶祥益, 等. 基于mtDNA Cyt b序列分析齐口裂腹鱼群体遗传多样性[J]. 水生生物学报, 2017, 41(3): 609-616. ZHANG Z S, HU B J, YE X Y, et al. Genetic diversity of the prenant's schizothoracin (Schizothorax prenanti) based on partial mtDNA Cyt b sequences[J]. Acta Hydrobiologica Sinica, 2017, 41(3): 609-616. |
| [40] |
FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915-925. DOI:10.1093/genetics/147.2.915 |
 2021,
Vol. 30
2021,
Vol. 30


