2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306
尼罗罗非鱼(Oreochromis niloticus)原产于非洲,属于鲈形目(Perciformes)丽鱼科(Cichlids)口孵非鲫属(Oreochromis)[1]。罗非鱼是变温水生动物,持续性高温会造成发病率升高、养殖经济效益降低等危害,对我国罗非鱼养殖业带来不良影响[2]。生物的抗逆性受多个信号通路调控,需要多基因共同参与表达[3]。近年来,有较多的研究从转录水平上分析生物对环境胁迫的响应机制。如:BILYK等[4]通过RNA-Seq探究博氏南冰(Pagothenia borchgrevinki)响应温度胁迫的分子机制,并首次推测鱼类应对温度胁迫时基因的转录会受性别影响;HUANG等[5]进行虹鳟(Oncorhynchus mykiss)的头肾组织在热应激条件下转录组表达谱的比较分析;HU等[6]通过转录组测序分析低温驯化的斑马鱼(Danio rerio)和尼罗罗非鱼响应低温环境的遗传网络调控机制。因此,探究不同性别尼罗罗非鱼响应持续性高温处理的适应机制,不仅可获得热适应的基因调控网络,而且对培育出适应全球气候变暖条件下生长的鱼类品种来说非常重要。
有研究报道TUNEL染色检测发现:在8 ℃下尼罗罗非鱼鳃组织存在特异性凋亡信号[6]。为探究尼罗罗非鱼对高温环境响应的适应机制,取罗非鱼背部肌肉、大脑和鳃进行TUNEL染色来分析细胞凋亡情况,并对高温下凋亡信号最强的背部肌肉进行转录组测序分析[7],探讨差异基因的表达情况,初步阐明不同性别的尼罗罗非鱼对高温环境的响应机制。为建立热应激条件下不同性别尼罗罗非鱼的代谢途径,构建信号转导通路、发掘耐温靶基因提供理论依据。
1 材料与方法 1.1 样品采集实验材料来自于广西水产科学研究院国家级罗非鱼良种场,是以尼罗罗非鱼为亲本繁育10 d的苗种,将苗种随机分为2组,每组各50尾鱼苗,高温实验组养殖于36 ℃(自28 ℃开始梯度升温,1 ℃/2 h,直至36 ℃),对照组平行养殖于28 ℃。养殖70 d后,通过尼罗罗非鱼性别连锁分子标记鉴定性别,将雌鱼和雄鱼分开[8],并按下列方式命名:实验组70 d-36 ℃-雌鱼(L70 d 36 CM)、实验组70 d-36 ℃-雄鱼(L70d36XM)、对照组70 d-28 ℃-雌鱼(L70d28CM)和对照组70 d-28 ℃-雄鱼(L70d28XM)。
1.2 方法 1.2.1 形态学统计监测了实验组(36 ℃)和对照组(28 ℃)的雌鱼和雄鱼生长发育情况,在处理70 d时采用游标卡尺测量其体长(精确到0.01 mm),用毛巾轻轻将鱼体表面水分擦干,采用电子天平称重(精确到0.01 mg),用GraphPad软件对其体长和体质量变化进行统计分析。
1.2.2 TUNEL染色取实验组和对照组处理70 d的罗非鱼脑、鳃和背部肌肉进行组织包埋,制作石蜡切片,随后取切片进行TUNEL染色,具体操作步骤参考Vazyme公司的TUNEL FITC Apoptosis Detection kit,将处理好的样品使用Laser confocal显微镜进行拍照分析。
1.2.3 RNA提取每个处理组分别取5个罗非鱼背部肌肉样本,用Invitrogen公司的TRIzol® Reagent试剂盒提取样品总RNA,将5个RNA样品等浓度混合后用于文库构建。抽提RNA的纯度均满足OD值(260/280)为1.8~2.2,且含量大于3 μg,浓度大于200 ng/μL。
1.2.4 RNA测序和质量控制RNA样品质检合格后可进行制备测序文库,用Illumina Hiseq ×Ten进行测序。对原始数据进行过滤,去除低质量的碱基,得到有效数据。
1.2.5 差异表达基因GO和KEGG富集分析转录组序列,通过计算FPKM值来确定基因的表达量,采用DESeq[9]软件包进行基因差异表达分析,筛选阈值为Fold Change≥2,FDR < 0.05。利用GO[10]、KEGG[11]等数据库对差异表达基因进行功能注释。为了确定与高温应激相关的功能类别,利用DAVID(https://david.ncifcrf.gov/)在GO数据库中通过计算差异表达基因参与生物学过程、细胞组分、分子功能的超几何分布,进行功能分类注释和富集分析。同时,利用KEEG数据库获得Unigenes的代谢通路注释信息。以上分析均以P < 0.05作为统计学显著性的阈值。
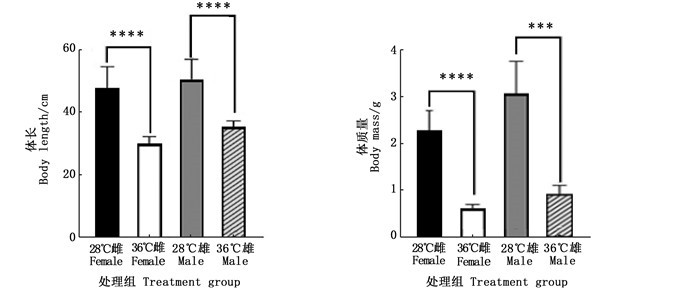
2 结果 2.1 形态学统计监测了实验组和对照组雌鱼和雄鱼在处理70 d的生长发育状况,记录其体质量和体长,利用GraphPad软件进行统计分析和作图。由图 1可以看出,在高温处理70 d之后,实验组的生长发育速度显著迟缓于对照组(P < 0.001),均表现为雄鱼生长发育比雌鱼快。
|
***表示两个温度处理组差异性极显著(P < 0.001);****表示两个温度处理组差异性极显著(P < 0.000 1)。 *** indicates there was extremely significant difference between the two different temperature treatment groups (P < 0.001); **** indicates there was extremely signiticant difference between the two different temperature treatment groups(P < 0.000 1). 图 1 不同温度雌鱼和雄鱼生长指标 Fig. 1 Growth index of females and males at different temperatures |
取实验组和对照组处理70 d的罗非鱼的背部肌肉、大脑和鳃进行TUNEL染色来分析细胞凋亡情况。通过TUNEL染色法发现,在罗非鱼的肌肉组织中有高水平的TUNEL标记,凋亡信号十分强烈(图 2),在其他组织中信号较弱。推测肌肉组织可能与罗非鱼对环境温度升高的响应有关系。

|
图 2 罗非鱼组织TUNEL凋亡检测 Fig. 2 Munofluorescent detection of the apoptosis by TUNEL assay in tissues of tilapia |
经过测序质量控制,各样品Q30碱基百分比均不小于91.59%,GC含量在49%左右。比对效率是RNA-seq数据利用率的直接体现。各样品的Mapped Reads与尼罗罗非鱼参考基因组的比对效率均为85.75%~89.81%,Uniq Mapped Reads的比对效率均为81.50%~85.55%,可以进行后续的分析与研究。见表 1。

|
表 1 与参考基因组的序列比对结果 Tab.1 Mapping information with reference genome |
在GO、KEGG、COG、KOG、NR和eggNOG 5个数据库中,L70d28CM和L70d36CM实验组共注释到3 321个基因,L70d28XM和L70d36XM实验组共注释到4 529个基因。通过比较雌鱼高温处理组与对照组以及雄鱼高温处理组与对照组在70 d时的基因表达量变化。L70d28CM与L70d36CM进行比较,共鉴定出3 405个差异表达基因,其中上调基因1 966个,下调基因1 439个;L70d28XM与L70d36XM进行比较,共鉴定出4 645个差异表达基因,其中上调基因3 460个,下调基因1 185个,均表现为在36 ℃时上调基因数量多于下调基因。
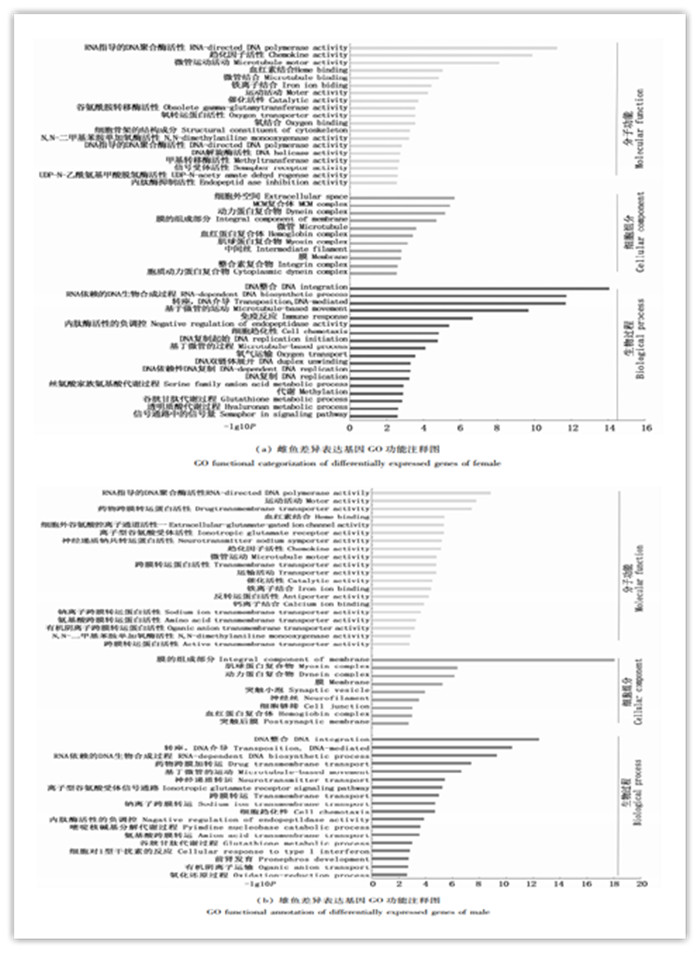
2.5 GO功能分析和KEGG聚类分析为确定与高温适应相关的功能类别,根据差异基因分析的结果对差异基因进行GO功能注释分析。在L70d28CM和L70d36CM实验组中,生物过程部分主要以DNA整合、DNA介导的转座、基于微管的运动、免疫反应和细胞趋化性的功能为主;细胞组分中主要以MCM复合体、动力蛋白复合体和微管的组成成分为主;分子功能主要以趋化因子活性、催化活性、血红素结合和药物跨膜转运蛋白活性为主;在L70d28XM和L70d36XM实验组中,生物过程部分主要以DNA整合、DNA介导的转座、药物转膜运输、神经递质转运和钠离子跨膜运输;细胞组分中主要以肌球蛋白复合体、动力蛋白复合体和膜的组成成分为主;分子功能主要以血红素结合、催化活性、跨膜运输和转运活性为主(图 3)。
|
图 3 显著差异表达基因的GO功能注释 Fig. 3 GO functional annotation of the significantly differentially expressed genes |
利用KEGG数据库对筛选到的、不同生长温度的差异表达基因进行聚类分析后发现,罗非鱼在高温响应过程中,不仅与环境信息处理有关,还与鱼的生长发育、新陈代谢通路有关。L70d28CM和L70d36CM两个雌性组的差异表达基因主要聚类在细胞过程和代谢过程,其中嘌呤代谢富集的差异基因数最多,细胞周期、嘧啶代谢和DNA复制通路富集的差异基因数较多;L70d28XM和L70d36XM两个雄性组的差异表达基因主要集中在代谢过程和组织系统过程,包括药物代谢-细胞色素P450、心肌细胞的肾上腺素信号传导、心肌收缩和紧密连接等通路上,其中紧密连接和心肌细胞的肾上腺素信号传导富集的差异基因数最多(图 4)。

|
圆圈代表一个KEGG通路,纵坐标为通路名称,横坐标为富集因子,圆圈颜色代表P,颜色越红代表显著富集性越可靠,圆圈越大代表富集的基因数目越多。 Circle represents one KEGG pathway, ordinate represents the name of the pathway, and the abscissa represents the enrichment factor, the color of the circle represents P. The darker the red color, the more reliable the significant enrichment, and the larger the circle, the greater the number of enriched genes. 图 4 差异表达基因KEGG通路 Fig. 4 KEGG pathway of differentially expressed genes |
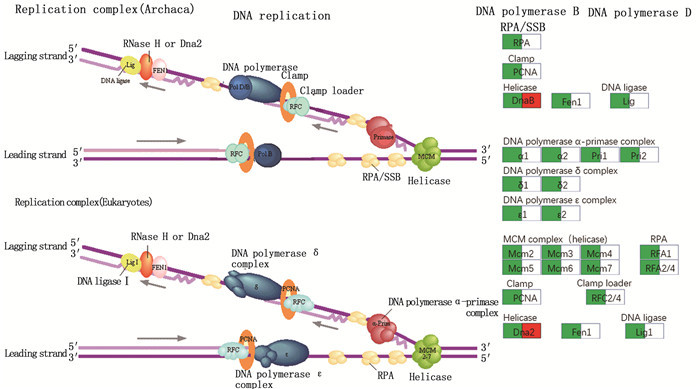
KEGG通路分析显示,不同性别罗非鱼在受到持续性高温处理后,通过不同的代谢途径来调节生命活动以维持机体正常生长。在雌鱼中,随着高温应激反应的持续,DNA复制这条通路的下调基因也随着增多,主要集中在Mcm蛋白家族、增殖细胞核抗原(PCNA)、DNA聚合酶复合体、复制因子C(RFC)等。长期的高温环境可能会导致复制叉的减慢或停滞,DNA复制过程受到抑制,进而抑制了细胞的增殖和分化;但该通路在雄鱼中富集不明显,表明在雄鱼中,这一过程受高温环境的影响不大。
3 讨论温度与鱼类生存息息相关,热应激是影响鱼体生长发育最严重的环境胁迫[12]。热应激会抑制DNA复制,导致复制叉的减慢或停滞[13],也会影响蛋白质、脂质、碳水化合物等代谢调节[14]。广温性鱼类适应温度变化的策略有调节细胞周期、改变细胞膜脂质成分、激活免疫应答反应基因、调节酶的催化活性和催化效率等[15]。在目前全球气候变暖的背景下, 揭示其热应激胁迫的分子机制,培育出耐高温的新品种显得尤为迫切。在环境因子胁迫下,大多数胁迫相关基因的转录水平会上升,而生长发育相关基因的转录水平有所下降,这是由转录因子与靶基因启动子的顺式作用元件的特异性结合引起的[16]。
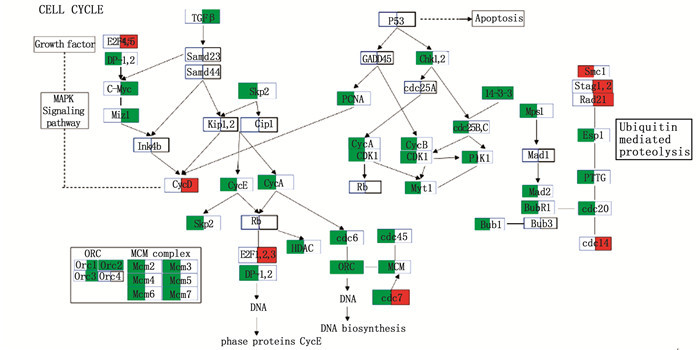
使用RNA-Seq分析的方法,对高温处理组和对照组的不同性别尼罗罗非鱼肌肉组织进行差异基因的表达谱分析,结果表明了两种性别具有各自特有的抗性机制:在雌鱼中显著富集的途径是嘌呤代谢、嘧啶代谢、细胞周期和DNA复制,其中嘌呤代谢和嘧啶代谢相关基因发生上调,而DNA复制通路(图 5)和细胞周期通路(图 6)相关基因如PCNA、Mcm、cdc7等发生显著性下调,与BILYK等[4]的研究结果细胞周期通路中Mcm下调一致,表明温度升高会导致细胞增殖和蛋白质生物合成普遍减慢;而在雄鱼中显著富集在心肌收缩、心肌细胞的肾上腺素信号转导、碳代谢和药物代谢等通路上,相关基因cdc7、p38MAPK和E2F等发生上调。本研究通过转录组分析获得的差异表达基因,其功能、代谢途径及相关生理特性的具体机制还需要进一步试验验证。
|
方框左边为雌鱼,右边为雄鱼。红色为上调基因,绿色为下调基因。 Left of table represents female differentially expressed genes, right of table represents male. Green represents the down-regulated genes, red represents the up-regulated genes. 图 5 DNA复制通路 Fig. 5 DNA replication pathway |
|
方框左边为雌鱼,右边为雄鱼。红色为上调基因,绿色为下调基因。 Left of table represents female differentially expressed genes, right of table represents male. Green represents the down-regulated genes, red represents the up-regulated genes. 图 6 细胞周期通路 Fig. 6 Cell cycle pathway |
综上所述,不同性别的罗非鱼响应高温环境的适应机制不同:雌鱼主要表现在细胞增殖和代谢系统调节;雄鱼则主要表现在代谢系统和组织系统调节。不同的KEGG途径为我们提供了可能导致鱼类暴露于持续高温发生的不可逆组织损伤的触发因素,从转录水平的调控机制给不同性别的鱼类适应高温环境的研究提供新思路。
| [1] |
陈蓝荪. 世界罗非鱼捕捞和养殖的动态特征研究[J]. 上海水产大学学报, 2006, 15(4): 477-482. CHEN L S. Study of dynamic characteristics of world tilapia capture and aquaculture industry[J]. Journal of Shanghai Fisheries University, 2006, 15(4): 477-482. |
| [2] |
罗祖娟, 林勇, 杨慧赞, 等. 高温对罗非鱼生长发育及病害的影响[J]. 广西畜牧兽医, 2013, 29(1): 48-50. LUO Z J, LIN Y, YANG H Z, et al. Effect of high temperature on growth and development and disease of tilapia[J]. Guangxi Animal Husbandry and Veterinary Medicine, 2013, 29(1): 48-50. DOI:10.3969/j.issn.1002-5235.2013.01.026 |
| [3] |
崔文晓, 马爱军, 黄智慧, 等. 大菱鲆(Scophthalmus maximus) PRL基因、Na+-K+-ATPase α1基因对盐度胁迫的响应[J]. 渔业科学进展, 2017, 38(6): 32-40. CUI W X, MA A J, HUANG Z H, et al. Effects of salinity stress on the mRNA expression of PRL and Na+-K+-ATPase α1 of turbot (Scophthalmus maximus)[J]. Progress in Fishery Sciences, 2017, 38(6): 32-40. |
| [4] |
BILYK K T, CHENG C H C. RNA-seq analyses of cellular responses to elevated body temperature in the high Antarctic cryopelagic nototheniid fish Pagothenia borchgrevinki[J]. Marine Genomics, 2014, 18: 163-171. DOI:10.1016/j.margen.2014.06.006 |
| [5] |
HUANG J Q, LI Y J, LIU Z, et al. Transcriptomic responses to heat stress in rainbow trout Oncorhynchus mykiss head kidney[J]. Fish & Shellfish Immunology, 2018, 82: 32-40. |
| [6] |
HU P, LIU M L, LIU Y M, et al. Transcriptome comparison reveals a genetic network regulating the lower temperature limit in fish[J]. Scientific Reports, 2016, 6: 28952. DOI:10.1038/srep28952 |
| [7] |
SOMERO G N, GIESE A C, WOHLSCHLAG D E. Cold adaptation of the Antarctic fish Trematomus bernacchii[J]. Comparative Biochemistry and Physiology, 1968, 26(1): 223-233. DOI:10.1016/0010-406X(68)90327-7 |
| [8] |
SUN Y L, JIANG D N, ZENG S, et al. Screening and characterization of sex-linked DNA markers and marker-assisted selection in the Nile tilapia (Oreochromis niloticus)[J]. Aquaculture, 2014, 433: 19-27. DOI:10.1016/j.aquaculture.2014.05.035 |
| [9] |
ANDERS S, HUBER W. Differential expression analysis for sequence count data[J]. Genome Biology, 2010, 11(10): R106. DOI:10.1186/gb-2010-11-10-r106 |
| [10] |
YOUNG M D, WAKEFIELD M J, SMYTH G K, et al. Gene ontology analysis for RNA-seq:accounting for selection bias[J]. Genome Biology, 2010, 11(2): R14. DOI:10.1186/gb-2010-11-2-r14 |
| [11] |
KANEHISA M, ARAKI M, GOTO S, et al. KEGG for linking genomes to life and the environment[J]. Nucleic Acids Research, 2007, 36(s1): D480-D484. |
| [12] |
周朝伟, 胡续雯, 雷骆, 等. 热应激对银鲫生化指标和HSP70 mRNA表达的影响[J]. 渔业科学进展, 2018, 39(6): 65-71. ZHOU C W, HU X W, LEI L, et al. Effects of heat stress on biochemical indices and HSP70 mRNA expression in gibel carp (Carassius auratus gibelio)[J]. Progress in Fishery Sciences, 2018, 39(6): 65-71. |
| [13] |
VELICHKO A K, PETROVA N V, RAZIN S V, et al. Mechanism of heat stress-induced cellular senescence elucidates the exclusive vulnerability of early S-phase cells to mild genotoxic stress[J]. Nucleic Acids Research, 2015, 43(13): 6309-6320. DOI:10.1093/nar/gkv573 |
| [14] |
SLIMEN I B, NAJAR T, GHRAM A, et al. Heat stress effects on livestock:molecular, cellular and metabolic aspects, a review[J]. Journal of Animal Physiology and Animal Nutrition, 2016, 100(3): 401-412. DOI:10.1111/jpn.12379 |
| [15] |
JOHNSTON I A, GOLDSPINK G. Thermodynamic activation parameters of fish myofibrillar ATPase enzyme and evolutionary adaptations to temperature[J]. Nature, 1975, 257(5527): 620-622. DOI:10.1038/257620a0 |
| [16] |
赵丽娟, 易小娅, 曾幼玲. 植物逆境相关C2H2型锌指蛋白的研究进展[J]. 分子植物育种, 2016, 14(3): 578-585. ZHAO L J, YI X Y, ZENG Y L. Research progress on plant stress related C2H2 type zinc finger protein[J]. Molecular Plant Breeding, 2016, 14(3): 578-585. |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
 2021,
Vol. 30
2021,
Vol. 30


