2. 上海海洋大学 上海市水产养殖工程技术研究中心, 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306
三角帆蚌(Hyriopsis cumingii)是我国淡水珍珠行业的主要育珠品种之一,在人工养殖的淡水蚌中,三角帆蚌的育珠量占80%以上[1]。三角帆蚌所产珍珠珠质细腻光滑、色泽鲜艳、形状较圆[2-3],但是三角帆蚌的育珠能力与性别相关,雄性三角帆蚌培育的珍珠在总质量、直径、色泽等方面均优于雌性三角帆蚌所育珍珠[4]。目前,三角帆蚌性别决定和性别分化基因尚未发现,性别决定机制还不清楚,所以研究相关基因以实现单性化养殖至关重要。
KLHL10是大型BTB(Broad-complex, Tramtrack, and Bric-a-brac)kelch蛋白家族的成员,又称为BBK(BTB、BACK和Kelch)蛋白家族。KLHL10基因的特征是氨基末端的BTB/POZ结构域和羧基末端的6个kelch重复序列[5-6]。在哺乳动物中,其对受精、雄性生殖器形态发生、雄性生殖腺发育、精细胞的发展至关重要[7]。小鼠中,KLHL10蛋白在生长和成熟的精子细胞中表达,且KLHL10一个等位基因的突变会导致雄性小鼠不育,因为基因突变使精子形成被破坏,精子成熟不同步,晚期精子数量减少[8-10]。在软体动物方面,已证明KLHL10基因与雄性玛雅章鱼(Octopus maya)的生殖能力有关[11]。但是KLHL10基因的功能在其他贝类尚未报道。
RNAi近年来普遍应用于基因功能探究和信号转导通路等方面[12-13]。目前已广泛应用于贝类基因功能的研究[14-16],但是不同干扰链的干扰效率存在很大的差异[17]。本研究初次克隆了三角帆蚌KLHL10基因,通过荧光定量技术(qRT- PCR)检测了其在多个组织及不同年龄性腺组织中的表达量,同时利用干扰技术对KLHL10在性腺中的功能进行初探,为探索KLHL10基因在三角帆蚌性别分化和决定中的功能和作用做准备。
1 材料与方法 1.1 实验材料1~8月龄和12、24、36月龄健康三角帆蚌取自浙江省金华市武义伟明水产有限公司养殖基地,带回实验室后取样,取24月龄三角帆蚌的性腺、鳃、闭壳肌、斧足、外套膜、肝胰腺,及1~8月龄、12月龄和36月龄三角帆蚌的性腺组织,立即保存在液氮中,-80 ℃冰箱保存备用,每组3个重复。
实验所用引物序列合成及测序均由生工生物技术(上海)有限公司完成,见表 1。
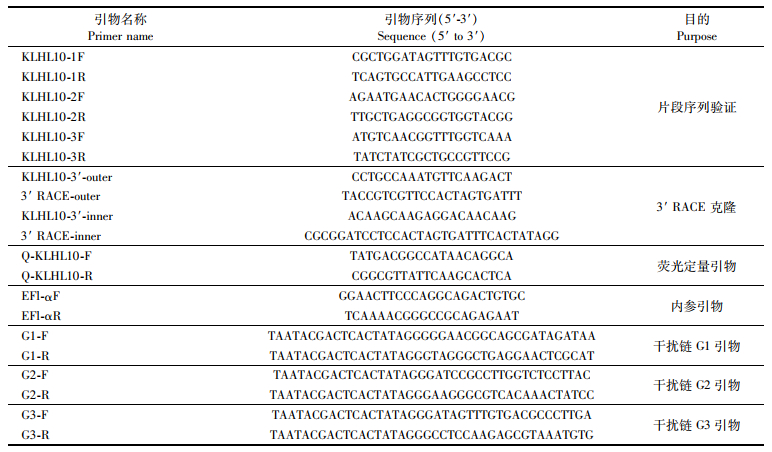
|
表 1 引物名称及序列 Tab.1 Primer names and sequences |
利用TRIZOL法提取24月龄三角帆蚌各组织的RNA,用NanoDrop 2000c (Thermo Scientific,US)检查RNA的纯度,再通过1.0%琼脂糖凝胶电泳检查RNA的完整性,根据3′-Full RACE Core Set with PrimeScriptTM RTase试剂盒反转成cDNA第一条链作为克隆3′的模板。根据已知序列设计KLHL10基因3′ RACE引物KLHL10-3′-outer和KLHL10-3′-inner(表 1),进行巢式PCR反应。以RACE cDNA第一链作为模板,用KLHL10-3′-outer引物和3′ RACE-outer引物进行第1轮PCR,然后以第1轮PCR产物作为模板,用KLHL10-3′-inner引物和3′ RACE-inner引物进行第2轮PCR。PCR产物经1.0%琼脂糖凝胶电泳检测,切胶回收,连接到pMD®19-T载体中,转化至感受态大肠杆菌DH5α,蓝白斑筛选后挑选白色菌株进行摇菌、送测。
1.3 KLHL10基因生物信息学分析通过NCBI的ORF Finder程序(https://www.ncbi.nlm.nih.gov/orffinder/)获得氨基酸序列的开放阅读框预测;通过BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)将所得到的基因序列与同源物种进行核苷酸和氨基酸序列相似性分析;通过SignalP 4.1 Server程序(http://www.cbs.dtu.dk/services/SignalP/)预测信号肽、通过TMHMM Server v2.0程序(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构;通过NetPhos 3.1程序(http://www.cbs.dtu.dk/services/NetPhos/)查找磷酸化位点。通过ProtParam程序(https://web.expasy.org/protparam/)预测蛋白质的物理化学参数;SMART程序(http://smart.embl-heidelberg.de/)预测蛋白质的二级结构、SWISS-MODEL程序(https://swissmodel.expasy.org/)预测三级结构;DNAMAN软件将克隆的cDNA全长和氨基酸序列进行整理分析;Bioedit软件进行多重序列比对;MEGA 7.0软件进行系统进化树的构建,Bootstrap重复1 000次计算各物种间的置信值。
1.4 荧光定量PCR分析将上述所取组织利用TRIZOL法提取RNA,并按照PrimeScriptTM RT Reagent Kit with gDNA Eraser试剂盒(TaKaRa, 日本)反转,将3个平行等量混合,稀释5倍,以此作为实时荧光定量PCR(qRT-PCR)的模板,反应体系如下:5×gDNA Eraser Buffer 5 μL,gDNA Eraser 1 μL,总RNA适量(500 ng),RNase Free dH2O补足至10 μL,42 ℃反应2 min;在上述反应液中加入PrimeScript RT Enzyme Mix Ⅰ 1 μL,RT Primer Mix 4 μL,5×PrimeScript Buffer 4 μL,RNase Free dH2O 1 μL,37 ℃ 15 min,85 ℃ 5 s,4 ℃保存,使用前稀释5倍。根据KLHL10基因的ORF区设计荧光定量的引物,以EF1-α作为内参,反应体系(20 μL):2×TB Green Premix Ex Taq(TaKaRa) 10 μL,引物各0.6 μL,cDNA模板1.6 μL,ddH2O 7.2 μL,每个样品3个重复。用2-ΔΔCT法计算相对表达量,用SPSS 18.0软件进行差异显著分析,用SigmaPlot12.3作图。
1.5 RNA干扰根据三角帆蚌KLHL10基因序列用Prime Prime 5设计3对引物,长200~500 bp,在每对正反引物前加上T7序列(表 1),发送至生工生物(上海)工程有限公司合成。dsRNA合成步骤:(1)通过普通PCR扩增所需探针片段,将产物切胶回收并进行纯化,测定其浓度,浓度符合要求者将作为后续的体外转录反应模板。(2)体外转录体系为模板RNA适量(共1 μg),5×T7 Transcription 4 μL,10 mmol/L NTP Mix 8 μL,T7 Transcription E-Mix 2 μL, RNase Free dH2O补足至20 μL,PCR仪37 ℃孵育2 h。(3)去除DNA,退火形成双链RNA,并去除单链RNA。取相同浓度的RNA混匀,置于PCR仪中70 ℃、10 min,随后室温放置20 min,然后每20 μL体系中加入1 μL RNase Solution和1 μL DNase Ⅰ,PCR仪37 ℃孵育30 min。(4)纯化双链RNA。上述液体中加入1/10体积的3 mol/L醋酸钠和相同体积的异丙醇,混匀,冰浴5 min,高速离心10 min;去上清后加入0.5 mL的70%乙醇洗涤;去除乙醇,室温风干15 min,将纯化后的dsRNA用无酶水溶解,稀释至一定浓度后,-80 ℃冰箱保存。将3条干扰链分别命名为G1、G2、G3。
干扰实验所用12月龄三角帆蚌在实验室暂养7 d后开始注射,分为4组,空白对照组、干扰组1(G1)、干扰组2(G2)和干扰组3(G3),每组10只,分别置于箱体中。将3条干扰链均稀释为120 ng/μL,实验组每只蚌注射100 μL干扰链,注射部位为闭壳肌,7 d后进行性腺组织取样,并提取RNA。将反转后的cDNA进行荧光定量。每组3只三角帆蚌RNA进行混样。
2 结果 2.1 KLHL10基因cDNA全长和氨基酸序列分析本实验克隆获得了三角帆蚌KLHL10基因cDNA全长,为2 361 bp(登录号:MT257263),包括93 bp的5′非编码区(5′-UTR)、447 bp的3′非编码区(3′-UTR)和1 821 bp的ORF区,编码606个氨基酸(图 1)。评估该蛋白质分子质量约为69.3 ku,理论等电点(PI)为5.20,不具备信号肽和跨膜结构,磷酸化位点分析发现17个丝氨酸(S)磷酸化位点,20个苏氨酸(T)磷酸化位点,11个酪氨酸(Y)磷酸化位点。利用SMART进行结构域分析发现KLHL10基因具有BTB/POZ结构域(37~134位氨基酸)、BACK结构域(139~242位氨基酸)和6个kelch(290~572位氨基酸)重复序列(图 2a)。利用SWISS-MODEL预测了KLHL10基因的蛋白三级结构(图 2b),QMEAN为-3.56,说明该蛋白与模板蛋白匹配度较高。其中α螺旋占27%,β折叠占32%,无规则卷曲为12%。

|
小写字母表示3′-UTR和5′-UTR。起始密码子和终止密码子由方框标出。预测蛋白的BTB/POZ结构域、BACK结构域和kelch重复序列分别由阴影、下划线及波浪线标出; 加尾信号由粗线标出。 Lowercase letters indicate 3′-UTR and 5′-UTR. The boxes represent the start and stop codons respectively. The shaded and underlined portion represented the BTB/POZ and BACK domains respectively, the kelch repeat motifs are indicated by wave lines; Tail signal is marked by thick line. 图 1 KLHL10 cDNA完整序列及编码的氨基酸序列 Fig. 1 Full cDNA length and encoded amino acid sequence of KLHL10 gene |

|
图 2 结构预测 Fig. 2 Predicted structures |
利用NCBI数据库将三角帆蚌的KLHL10氨基酸序列与其他物种的KLHL10氨基酸序列比对显示,三角帆蚌KLHL10与智人(Homo sapiens)、小鼠(Mus musculus)的同源性分别为55.57%和55.41%,与美洲牡蛎(Crassostrea virginica)、长牡蛎(Crassostrea gigas)、虾夷扇贝(Mizuhopecten yessoensis)的同源性分别为75.00%、75.00%和69.98%。通过BioEdit进行多序列比对,发现三角帆蚌KLHL10基因与其他9个物种的结构域相似度较高(图 3),表明KLHL10基因在进化过程中相对保守。

|
图 3 三角帆蚌与其他物种KLHL10氨基酸序列比对 Fig. 3 Alignment analysis of KLHL10 amino acid between H. cumingii and others |
采用MEGA 7.0软件,以邻接法(neighbor-joining,NJ)构建系统进化树,进化树分为两大支,脊椎动物为一支,无脊椎动物为一支。其中,无脊椎动物中三角帆蚌与其他贝类聚为一支,果蝇单独为一支,三角帆蚌的KLHL10基因与美洲牡蛎和长牡蛎亲缘关系较近(图 4)。
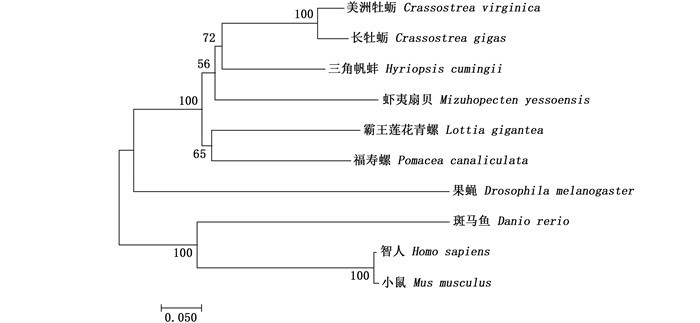
|
基于MEGA 7.0使用NJ法构建,节点上的数字表示重复1 000次的Bootstrap检验置信值。 The tree was constructed by MEGA 7.0 using the neighbour-joining method with 1 000 Bootstrap repeats. 图 4 三角帆蚌与其他物种KLHL10蛋白质进化树 Fig. 4 Phylogenetic analysis of KLHL10 protein between H. cumingii and others |
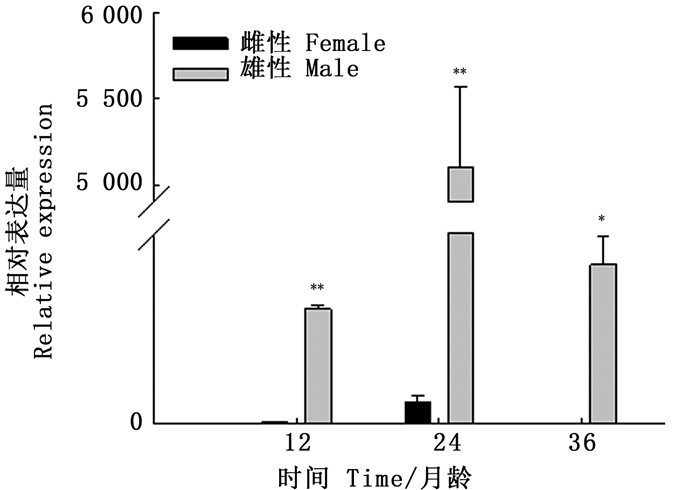
以EF1-α为内参,通过实时荧光定量PCR检测KLHL10基因在24月龄三角帆蚌性腺、鳃、闭壳肌、外套膜、斧足和肝胰腺6个组织,1~8月龄性腺以及1、2、3龄雌雄性腺中的表达情况。结果显示,KLHL10基因在雌雄6个组织中均有表达,在精巢中的表达量最高,且显著高于其他组织(P < 0.01),见图 5;幼龄时期的结果显示,KLHL10基因在三角帆蚌6月龄时表达量最高(图 6);在12、24、36月龄雌雄性腺中,KLHL10基因在精巢中的表达量均高于同龄卵巢中的表达量,且24月龄时的表达量最高(图 7)。
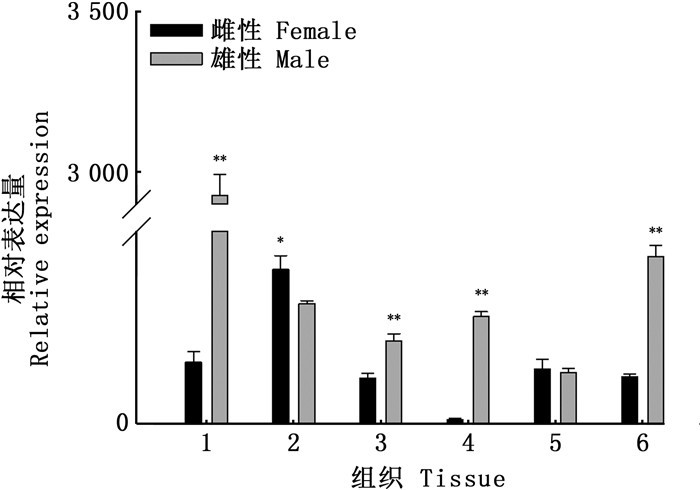
|
1.性腺;2.鳃;3.外套膜;4.斧足;5.闭壳肌;6.肝胰腺;*表示雌雄间存在显著性差异(*. P < 0.05;**. P < 0.01)。 1.Gonad; 2.Gill; 3.Mantle; 4.Foot; 5.Adductor; 6.Hepatopancreas; Significant differences are indicated by asterisks (*. P < 0.05;**. P < 0.01). 图 5 KLHL10基因在雌雄各组织中的表达 Fig. 5 Expression of KLHL10 in various tissues of female and male adults |
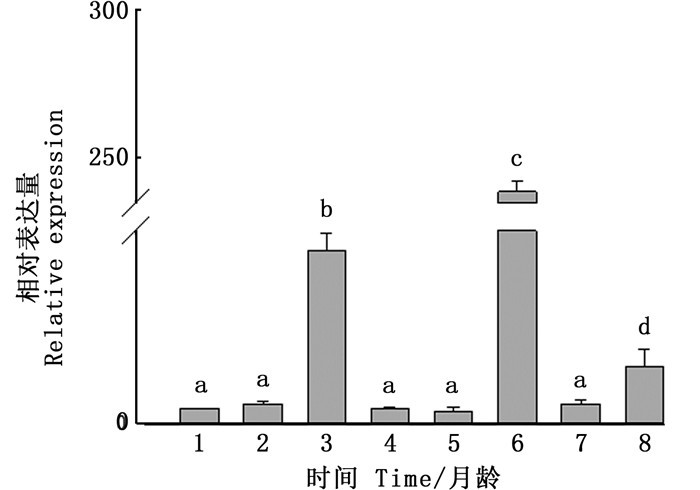
|
不同字母(a, b, c, d)表示性腺不同发育时期表达存在显著性差异(P < 0.05),标有相同字母表示组间无显著性差异。 Different letters (a, b, c, d) indicate significant difference in expression of gonads at different developmental stages(P < 0.05), identical letters indicate no significant difference between groups. 图 6 KLHL10基因在早期(1~8月龄)性腺组织中的表达 Fig. 6 Expression of KLHL10 in gonads at early stage |
|
*表示雌雄间存在显著性差异(*. P < 0.05;**. P < 0.01)。 Significant differences are indicated by asterisks (*. P < 0.05;**. P < 0.01). 图 7 KLHL10基因在12、24、36月龄雌雄性腺中的相对表达 Fig. 7 Relative expression of KLHL10 in female and male gonads at 12-, 24-, and 36-month-old |
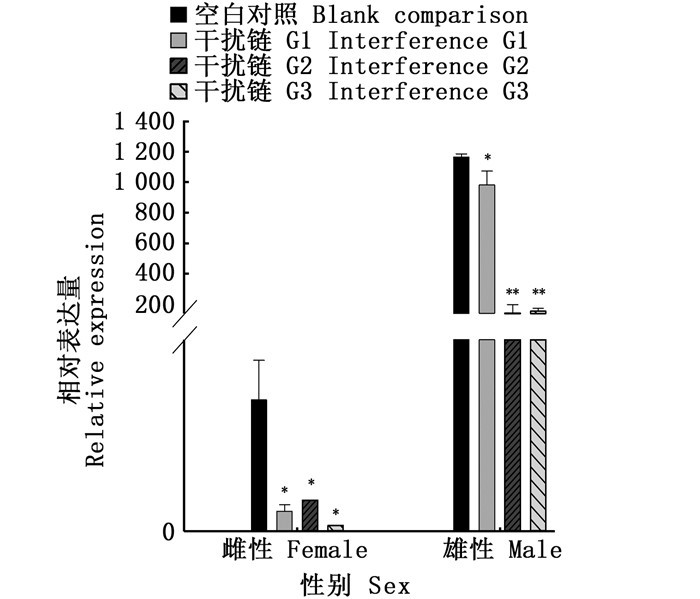
qRT-PCR检测干扰后三角帆蚌性腺组织中KLHL10基因mRNA的表达量,结果(图 8)显示本实验合成的3条干扰链均起到了一定的干扰作用。SPSS分析结果显示:在雌性三角帆蚌中,G1、G2和G3干扰后KLHL10基因mRNA的表达量与空白组差异显著(P < 0.05);在雄性三角帆蚌中,G1干扰后KLHL10基因的表达量与空白组差异显著(P < 0.05),G2和G3与空白组的比较结果为差异极显著(P < 0.01);3条干扰链对雌性三角帆蚌的干扰率分别为84.6%、76.2%和95.8%,对雄性三角帆蚌的干扰率分别为15.7%、87.7%和86.7%。
|
*表示与对照组存在显著性差异(*. P < 0.05;**. P < 0.01)。 Significant differences are indicated by asterisks (*. P < 0.05;**. P < 0.01). 图 8 RNA干扰后KLHL10基因在雌雄性腺组织中的相对表达 Fig. 8 Relative expression of KLHL10 in female and male gonad tissues after RNA interference |
本实验首次克隆了三角帆蚌KLHL10基因的cDNA全长序列,包含1个长为606个氨基酸的ORF区。蛋白质结构预测显示其含有BTB蛋白家族特有的BTB/POZ结构域和kelch重复序列,多序列比对结果表明三角帆蚌KLHL10基因的氨基酸序列与其他物种相似度较高,特别是与美洲牡蛎等软体动物相似度更高,证明KLHL10基因在进化过程中高度保守,也暗示KLHL10基因在软体动物中的功能可能与脊椎动物相似。
BBK蛋白家族是BTB蛋白家族的一大成员,首先在果蝇中发现,含有BTB/POZ结构域,参与多种生物学过程,例如迁移,细胞骨架排列,细胞形态调节,蛋白质泛素化和基因表达[18-21]。目前关于KLHL10基因的研究并不多,大多数是关于其BTB/POZ结构域的研究,功能性研究较少[22-25]。文献[26]表明,KLHL10基因与精子息息相关,在人类中,KLHL10基因是生殖细胞特异性基因,男性不育往往伴随着KLHL10基因的错误和剪切突变。同时,KLHL10基因在小鼠的精子延长分化过程中也具有重要作用[27]。在斑点乌鳢中也已证明KLHL10基因是精子发育相关基因[28]。在果蝇中也发现KLHL10蛋白通过与Cullin3泛素化作用激活精子发育所需的半胱天冬酶[29]。
本实验利用qRT-PCR检测KLHL10基因在三角帆蚌成熟个体中的表达情况,结果显示其在性腺、鳃、闭壳肌、斧足、外套膜和肝胰腺中均有表达,除了在精巢中特异性高表达外,在其他组织中均是微量表达。而12、24、36月龄时期,KLHL10基因在精巢中的表达量先升后降,其中12月龄三角帆蚌性腺处于增殖期,24月龄时期性腺处于成熟生长阶段,此时期性腺组织由原始性腺细胞开始转化为原始生殖细胞,36月龄时期性腺发育完全成熟,具有大量成熟可排放的生殖细胞[30]。KLHL10基因在12月龄到24月龄精巢中表达量升高,表明KLHL10基因可能参与三角帆蚌性腺的增殖发育,同时KLHL10基因在12、24、36月龄精巢中高表达,推测KLHL10基因是雄性相关基因,并参与精巢和生殖细胞发育。同时,幼龄阶段KLHL10基因的表达结果显示,其在6月龄时期高表达,而三角帆蚌在5月龄开始性别分化[31],由此猜测KLHL10基因除了参与精巢发育外,还参与三角帆蚌性别分化。由于KLHL10基因相关功能研究文献较少,此次实验主要基于基因表达分析,其在三角帆蚌中的功能值得进一步深究。
通过RNA干扰发现不同的干扰链对同1个基因的干扰效果有所不同。本实验中,3条干扰链G1、G2和G3对雌性三角帆蚌的干扰率分别为84.6%、76.2%和95.8%,对雄性三角帆蚌的干扰率分别为15.7%、87.7%和86.7%。其中,干扰链G3在雌雄性腺中的干扰率均在80%以上,表明干扰链G3对三角帆蚌KLHL10基因具有明显的干扰效果,此链将用于后续研究KLHL10基因在三角帆蚌性腺发育中的作用。本实验为进一步研究三角帆蚌的性别分化和性别决定相关基因及机制提供一定参考价值。
| [1] |
汪桂玲, 白志毅, 刘晓军, 等. 三角帆蚌种质资源研究进展[J]. 水产学报, 2014, 38(9): 1618-1627. WANG G L, BAI Z Y, LIU X J, et al. Research progress on germplasm resources of Hyriopsis cumingii[J]. Journal of Fisheries of China, 2014, 38(9): 1618-1627. |
| [2] |
姜琦, 白志毅, 孙朝虎. 三角帆蚌所育不同颜色珍珠及其相关组织金属元素种类和含量差异分析[J]. 上海海洋大学学报, 2019, 28(6): 882-889. JIANG Q, BAI Z Y, SUN C H. Analysis of metallic element types and contents in pearls of different colors and their related tissues in Hyriopsis cumingii[J]. Journal of Shanghai Ocean University, 2019, 28(6): 882-889. |
| [3] |
ZHAO Y C, BAI Z Y, FU L L, et al. Comparison of growth and pearl production in males and females of the freshwater mussel, Hyriopsis cumingii, in China[J]. Aquaculture International, 2013, 21(6): 1301-1310. DOI:10.1007/s10499-013-9632-y |
| [4] |
李西雷, 李卿青, 朱庭耀, 等. 添加类胡萝卜素对三角帆蚌总类胡萝卜素含量及贝壳珍珠质颜色的影响[J]. 上海海洋大学学报, 2018, 27(4): 477-486. LI X L, LI Q Q, ZHU T Y, et al. Effect of dietary carotenoid on total carotenoids content and shell color in Hyriopsis cumingii[J]. Journal of Shanghai Ocean University, 2018, 27(4): 477-486. |
| [5] |
YANG Z F, LI Q, MA X, et al. Mutation analysis in Chinese families with autosomal dominant hereditary cataracts[J]. Current Eye Research, 2015, 40(12): 1225-1231. DOI:10.3109/02713683.2014.997885 |
| [6] |
ADAMS J, KELSO R, COOLEY L. The kelch repeat superfamily of proteins:propellers of cell function[J]. Trends in Cell Biology, 2000, 10(1): 17-24. DOI:10.1016/S0962-8924(99)01673-6 |
| [7] |
STOGIOS P J, DOWNS G S, JAUHAL J J S, et al. Sequence and structural analysis of BTB domain proteins[J]. Genome Biology, 2005, 6(10): R82. DOI:10.1186/gb-2005-6-10-r82 |
| [8] |
ROBINSON D N, COOLEY L. Drosophila kelch is an oligomeric ring canal actin organizer[J]. Journal of Cell Biology, 1997, 138(4): 799-810. DOI:10.1083/jcb.138.4.799 |
| [9] |
赵丽. MIJ大鼠Klhl10、Insl3、Acr、Zmynd15基因的克隆与序列分析[D].石家庄: 河北医科大学, 2015. ZHAO L. Cloning and analysis of Klhl10, Insl3, Acr and Zmynd15genes in MIJ rats[D]. Shijiazhuang: Hebei Medical University, 2015. |
| [10] |
YAN W, MA L, BURNS K H, et al. Haploinsufficiency of kelch-like protein homolog 10 causes infertility in male mice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(20): 7793-7798. DOI:10.1073/pnas.0308025101 |
| [11] |
LÓPEZ-GALINDO L, JUÓREZ O E, LARIOS-SORIANO E, et al. Transcriptomic analysis reveals insights on male infertility in Octopus maya under chronic thermal stress[J]. Frontiers in Physiology, 2019, 9: 1920. DOI:10.3389/fphys.2018.01920 |
| [12] |
BOSHER J M, LABOUESSE M. RNA interference:genetic wand and genetic watchdog[J]. Nature Cell Biology, 2000, 2(2): E31-E36. DOI:10.1038/35000102 |
| [13] |
谢东昌.斑节对虾Akirin基因的分子克隆及免疫功能研究[D].上海: 上海海洋大学, 2019. XIE D C. Molecular cloning and immunological characterization of Akirin in black tiger shrimp (Penaeus monodon)[D]. Shanghai: Shanghai Ocean University, 2019. |
| [14] |
FABIOUX C, CORPOREAU C, QUILLIEN V, et al. In vivo RNA interference in oyster-vasa silencing inhibits germ cell development[J]. The FEBS Journal, 2009, 276(9): 2566-2573. DOI:10.1111/j.1742-4658.2009.06982.x |
| [15] |
王芹, 汪桂玲, 陈亚, 等. RNA干扰沉默三角帆蚌HcCA3基因对贝类矿化作用的影响[J]. 基因组学与应用生物学, 2018, 37(2): 741-747. WANG Q, WANG G L, CHEN Y, et al. Effect of silencing HcCA3 gene in Hyriopsis cumingii by RNA interference on mineralization of shellfish[J]. Genomics and Applied Biology, 2018, 37(2): 741-747. |
| [16] |
JIN C, REN H Y, PU J W, et al. Identification of nacre matrix protein genes hic14 and hic19 and their roles in crystal growth and pearl formation in the mussel Hyriopsis cumingii[J]. Biotechnology and Applied Biochemistry, 2019, 66(4): 545-554. DOI:10.1002/bab.1752 |
| [17] |
BARRETO F S, SCHOVILLE S D, BURTON R S. Reverse genetics in the tide pool:knock-down of target gene expression via RNA interference in the copepod Tigriopus californicus[J]. Molecular Ecology Resources, 2015, 15(4): 868-879. DOI:10.1111/1755-0998.12359 |
| [18] |
HARRISON S D, TRAVERS A A. The tramtrack gene encodes a Drosophila finger protein that interacts with the ftz transcriptional regulatory region and shows a novel embryonic expression pattern[J]. The EMBO Journal, 1990, 9(1): 207-216. DOI:10.1002/j.1460-2075.1990.tb08097.x |
| [19] |
GODT D, COUDERC J L, CRAMTON S E, et al. Pattern formation in the limbs of Drosophila:bric à brac is expressed in both a gradient and a wave-like pattern and is required for specification and proper segmentation of the tarsus[J]. Development, 1993, 119(3): 799-812. DOI:10.1242/dev.119.3.799 |
| [20] |
COUDERC J L, GODT D, ZOLLMAN S, et al. The bric à brac locus consists of two paralogous genes encoding BTB/POZ domain proteins and acts as a homeotic and morphogenetic regulator of imaginal development in Drosophila[J]. Development, 2002, 129(10): 2419-2433. DOI:10.1242/dev.129.10.2419 |
| [21] |
LVHRIG S, KOLB S, MELLIES N, et al. The novel BTB-kelch protein, KBTBD8, is located in the Golgi apparatus and translocates to the spindle apparatus during mitosis[J]. Cell Division, 2013, 8(1): 3. DOI:10.1186/1747-1028-8-3 |
| [22] |
WANG S H, ZHENG H L, ESAKI Y, et al. Cullin3 is a KLHL10-interacting protein preferentially expressed during late spermiogenesis[J]. Biology of Reproduction, 2006, 74(1): 102-108. DOI:10.1095/biolreprod.105.045484 |
| [23] |
PINTARD L, WILLEMS A, PETER M. Cullin-based ubiquitin ligases:Cul3-BTB complexes join the family[J]. The EMBO Journal, 2004, 23(8): 1681-1687. DOI:10.1038/sj.emboj.7600186 |
| [24] |
PALLETT M A, REN H W, ZHANG R Y, et al. Vaccinia virus BBK E3 ligase adaptor A55 targets importin-dependent NF-κB activation and inhibits CD8+ T-Cell memory[J]. Journal of Virology, 2019, 93(10): e00051-19. |
| [25] |
STOGIOS P J, PRIVE G G. The BACK domain in BTB-kelch proteins[J]. Trends in Biochemical Sciences, 2004, 29(12): 634-637. DOI:10.1016/j.tibs.2004.10.003 |
| [26] |
YATSENKO A N, ROY A, CHEN R H, et al. Non-invasive genetic diagnosis of male infertility using spermatozoal RNA:KLHL10 mutations in oligozoospermic patients impair homodimerization[J]. Human Molecular Genetics, 2006, 15(23): 3411-3419. DOI:10.1093/hmg/ddl417 |
| [27] |
HONDA S, MIKI Y, MIYAMOTO Y, et al. Oocyte-specific gene Oog1 suppresses the expression of spermatogenesis-specific genes in oocytes[J]. The Journal of Reproduction and Development, 2018, 64(4): 297-301. DOI:10.1262/jrd.2018-024 |
| [28] |
ROY A, BASAK R, RAI U. De novo sequencing and comparative analysis of testicular transcriptome from different reproductive phases in freshwater spotted snakehead Channa punctatus[J]. PLoS One, 2017, 12(3): e0173178. DOI:10.1371/journal.pone.0173178 |
| [29] |
ARAMA E, BADER M, RIECKHOF E G, et al. A ubiquitin ligase complex regulates caspase activation during sperm differentiation in Drosophila[J]. PLoS Biology, 2007, 5(10): e251. DOI:10.1371/journal.pbio.0050251 |
| [30] |
潘彬斌.池养三角帆蚌性腺发育组织学研究[D].上海: 上海海洋大学, 2010. PAN B B. Histological study on the gonad development of Hyriposis cumingii cultured in the pond[D]. Shanghai: Shanghai Ocean University, 2010. |
| [31] |
薛婷.淡水珍珠蚌DUI发生及性腺发育研究[D].上海: 上海海洋大学, 2016. XUE T. Study on DUI occurring and gonad development of freshwater pearl mussels[D]. Shanghai: Shanghai Ocean University, 2016. |
2. Shanghai Engineering Research Center of Aquaculture, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
 2021,
Vol. 30
2021,
Vol. 30


