2. 上海海洋大学 水产动物遗传育种中心上海市协同创新中心, 上海 201306
中国拥有大量的盐碱水资源,面积约为6.03×1012 m2[1],但开发利用难度大,需要适宜的水产养殖品种[2]。盐碱水具有盐度高、pH高、碱度高、离子成分复杂等特点[3],其中碱度是指水中能与酸发生中和反应的物质总量,包括HCO3-、CO32-等弱酸根, OH-等强碱以及NH3等弱碱性物质,而天然水体中主要以碳酸盐碱度为主[4]。通常,适当的碱度有利于水生生物生存,但过高的碳酸盐碱度对水生生物具有较强的毒害作用[5-6]。碱度毒性会影响血液中pH缓冲体系,会损伤鳃、黏膜等离子交换组织[7]。尼罗罗非鱼(Oreochromis niloticus)是一种广盐性鱼类。本实验室在开展耐盐碱罗非鱼新品种选育的过程中也试图研究其适应盐碱环境的机制[8]。在高碳酸盐碱度胁迫下,血氨浓度上升,罗非鱼会启动尿素与谷氨酰胺代谢途径共同参与调节血氨浓度[9]。碱度胁迫会影响尼罗罗非鱼鳃离子细胞数量和形态[10]。碳酸酐酶(carbonic anhydrase, CA)家族中的CAⅡ、CAⅣ、溶质载体家族蛋白(solute carrier family, SLC)中的SLC4A4、SLC26A6参与碱度胁迫下离子转运、渗透压平衡调节[11]。
microRNA(miRNAs)是一类内源性、短链非编码小RNA[12]。miRNAs主要通过与靶基因的3′非翻译区(3′UTR)的碱基不完全配对,抑制或降解靶基因的表达[13]。研究表明miRNA广泛参与动物[14-15]和植物[16]适应环境的过程。在植物中的研究表明miRNA在适应盐度[17]、碱度[18]、温度[19]和湿度[20]的变化中发挥重要作用。在尼罗罗非鱼的研究中发现:miR-204通过靶向血管内皮生长因子(vascular endothelial growth factor, VEGF)响应低氧胁迫[21];miR-30c和miR-206分别通过靶向热休克蛋白(heat Shock Proteins 70, HSP70)和胰岛素样生长因子(insulin-like growth factor-1, IGF-1)来应对渗透压变化[22-23];miR-21通过靶向血管内皮生长因子VEGFB和VEGFC来参与适应碱度胁迫[24]。本实验室已通过高通量测序技术发现碳酸盐碱度胁迫后罗非鱼鳃组织中67个差异表达显著miRNAs,并结合实时荧光定量PCR对7个miRNA(miR-122、miR-192、miR-194a、miR-24a、miR-30d、miR-143和miR-155)进行了初步研究,但尚未对其作用和靶基因进行深入研究。
选取miR-192深入研究其在尼罗罗非鱼应答碱度胁迫中的作用,首先采用实时荧光定量PCR检测在急性碱度胁迫尼罗罗非鱼后,96 h内鳃组织中miR-192的表达变化。利用生物信息软件预测miR-192的靶基因,并对靶基因进行KEGG通路分析。在众多可能的靶基因中筛选出溶质转运蛋白(solute carriers16A7, SLC16A7)基因进行实时荧光定量PCR、双荧光素酶报告实验以及活体体内注射miR-192抑制剂实验进一步验证靶向关系。本研究为探明miRNAs调控尼罗罗非鱼应答碱度胁迫的分子调控机制提供实验基础。
1 材料与方法 1.1 材料实验用尼罗罗非鱼取样于上海海洋大学罗非鱼种质资源试验站,体质量为(110±10) g,暂养于室内控温循环水族箱内,每天定时投喂2次,投喂量约为鱼体质量的3%。待完全适应新环境后,挑选规格均匀、体质健康的个体开展碱度胁迫实验。本研究经上海海洋大学动物实验伦理委员会批准。
1.2 碳酸盐碱度胁迫实验设计实验容器为60 cm×45 cm×40 cm的玻璃水槽,实验用碱水提前配制,在曝气完全的淡水中加入所需NaHCO3 (分析纯),稳定48 h后使用。实验前1 d停止喂食,实验开始时将鱼从过渡淡水中直接移至碱度组中进行急性胁迫,每组投放10尾鱼,设置3个重复。于胁迫后0、6、12、24、48、72和96 h采样,胁迫过程中没有死亡现象,每个重复组取1尾鱼。
根据预实验结果,设置淡水组FW和碱度组AW6(6 g/L NaHCO3),实验期间应适当换水保持碱度。配置完成后,用滴定法对水体碱度进行测量,FW组碱度为2.01 mmol/L,AW6组碱度为68.60 mmol/L。
取胁迫后0、6、12、24、48和96 h的鳃组织进行实时荧光定量,检测miR-192和SLC16A7的表达量。取0 h其他组织用于miR-192的组织表达谱分析。
1.3 总RNA提取及cDNA合成利用Trizol法提取尼罗罗非鱼的鳃组织总RNA,提取步骤严格按照TRNzol-A+ Reagent (TIANGEN)说明书操作,以1.5%的琼脂糖凝胶电泳检测RNA完整性,并测定质量浓度及纯度,稀释至1 ng/μL保存于-80 ℃冰箱备用。提取0 h尼罗罗非鱼的鳃、脑、心、肌肉、肝、肾、肠组织的总RNA,用于组织表达谱检测。
所提取的总RNA经1.5%的琼脂糖凝胶电泳检测RNA无降解条带和污染条带,核酸浓度检测仪测定的A260/A280为1.8~2.0,可用于后续实验。
使用Prime Script® RT Reagent Kit with gDNA Eraser(TaKaRa)试剂盒合成SLC16A7的cDNA第一链,反应体系第一步,去除RNA中残存的DNA(10 μL):5 × gDNA Eraser Buffer 2.0 μL,gDNA Eraser 1.0 μL,RNA 1.0 μL,RNase Free dH2O 6.0 μL。第二步,合成cDNA第一链(20 μL):经第一步处理的RNA 10.0 μL,PrimeScript RT Enzyme MixⅠ 1.0 μL,RT引物1.0 μL,5×PrimeScript Buffer 4.0 μL,RNase Free dH2O 4.0 μL。
合成miR-192的cDNA第一链,第2步中用miR-192 RT引物替代RT引物,其余同SLC16A7 cDNA第一链的合成。
制备的cDNA保存在4 ℃冰箱,用于后续实验。
1.4 引物设计与筛选根据测序所得尼罗罗非鱼miR-192序列,采用生工生物工程股份有限公司提供的茎环序列,使用Primer 5设计茎环引物,选取U6 snRNA作为内参基因。
选取β-actin作为内参基因,对SLC16A7设计引物。引物通过软件Primer 5设计,由上海生工生物工程股份有限公司合成。选择3′UTR区域用于双荧光素实验中的引物设计,通过普通PCR反应筛选验证,引物信息详见表 1。

|
表 1 引物序列信息 Tab.1 Primer sequence information |
使用TB GreenTM Premix Ex TaqTM (Tli RNaseH Plus)试剂盒进行荧光定量PCR,反应体系如下(20 μL):TB GreenTM Premix Ex TaqTM (Tli RNaseH Plus)(2×) 10.0 μL,PCR正向引物(10 μmol/L) 0.5 μL,PCR反向目录(10 μmol/L) 0.5 μL,cDNA 2.0 μL,无RNase酶水7.0 μL。反应程序:95 ℃ 2 min预变性; 95 ℃ 15 s变性; 58.5 ℃ 1 min退火延伸,反应进行35个循环。
数据分析采用CFX Manager软件进行。基线值、阈值和阈循环值由软件自动设定,每个反应设置3个平行组。每批反应均设阴性对照,确保反应体系未受污染,采取相对定量2-ΔΔCT[25]法计算表达量。
1.6 双荧光素酶报告载体构建与鉴定构建pmirGLO-SLC16A7-3′-UTR双荧光素酶报告载体:根据Ensembl上的基因序列(ENSONIG00000016934)通过RT-PCR方法扩增SLC16A7基因3′UTR片段。使用1.5%琼脂糖凝胶电泳分析PCR反应产物,并对产物进行纯化。用DraⅠ和XbaⅠ对纯化的PCR产物和载体进行双酶切,然后T4酶连接(连接时PCR产物与荧光素酶报告载体的摩尔数之比为10:1,连接体系为10 μL)。将连接产物导入DH5α感受态细胞进行阳性克隆验证,双酶切及电泳检测正确后,送公司测序鉴定。鉴定正确的重组载体命名为pmirGLO-SLC16A7-3′-UTR。
瞬时转染HEK-293T细胞:采用24孔板进行培养,接种密度为5 ×104。使用含10%胎牛血清(FBS)的DMEM培养基置于CO2培养箱中培养,在37 ℃、5% CO2条件下培养24 h至细胞密度达70%~85%时开始转染。转染分为3组:重组载体pmirGLO-SLC16A7-3′-UTR(对照组)、重组载体pmirGLO-SLC16A7-3′-UTR + miR-192 mimic(处理组)和重组载体pmirGLO-SLC16A7-3′-UTR + miR-192 mimic NC(处理NC组)。转染时每组细胞设6个平行,先吸去原培养基,取25 μL OPTI-MEM培养基稀释1 μL LipofectamineTM 2 000并充分混匀(转染稀释液),取25 μL OPTI-MEM培养基稀释1 μg miR-192 mimics或者mimic NC并充分混匀(处理稀释液),将上述处理稀释液缓慢滴入转染稀释液中,轻轻混匀后于室温孵育5 min。加入转染脂质体复合物至细胞中,转染6 h后更换新鲜OPTI-MEM培养基。在37 ℃,5% CO2条件下孵育细胞2 d后分析转染细胞的荧光值。
1.7 双荧光素酶报告基因系统检测使用Dual-Luciferase® Reporter Assay System(Promega)试剂盒,在Promega GLOMAX上检测荧光信号。将5×PLB稀释成1×PLB备用,预混好LARⅡ和Stop & Glo Reagent反应液。弃去旧培养基,用PBS清洗2遍,每孔加入100 μL 1×PLB裂解细胞,置于摇床上15 min。将50 μL LARⅡ加入1.5 mL EP管中,取10 μL细胞裂解液加入EP管,置于仪器检测信号;加入50 μL Stop & Glo Reagent,再次置入仪器,检测信号。根据测得的萤火虫荧光素酶活性值F(firely luciferase)及海肾荧光素酶活性值R(renilla luciferase),计算得出相对荧光素酶活性ΔCT=F/R。
1.8 罗非鱼体内注射miRNA抑制剂实验罗非鱼分别注射生理盐水(对照组)和miR-192抑制剂(处理组)。提前使用RNase-free无菌水稀释miR-192抑制剂,每尾每次注射剂量为500 μL的miR-192抑制剂稀释液(200 μmol/L),对照组使用500 μL 0.65%的生理盐水。采用胸腔注射,每天2次,注射3 d,注射时间固定。距离第1次注射96 h后,取肌肉、鳃、肾和肝组织进行测量。
1.9 统计学处理实验数据采用SPSS 17.0软件进行统计分析。分析结果采用平均值±标准差(Mean±SD)表示,当P < 0.05时表示差异具有统计学意义。
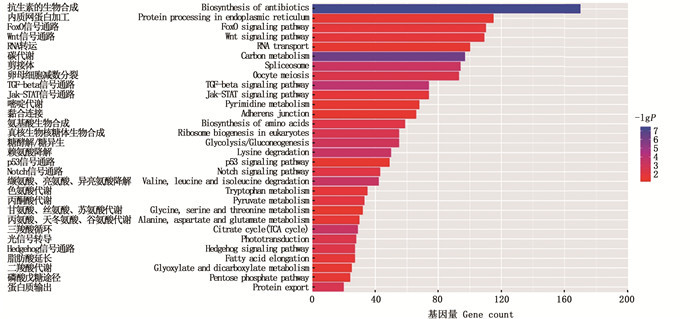
2 结果 2.1 miR-192的生物信息学分析和靶基因预测使用miRanda和RNAhybrid对miR-192的靶基因进行预测,并对预测的结果进行KEGG通路富集分析(P < 0.05),见图 1。结果显示,预测的靶基因在损伤修复相关通路(biosynthesis of antibiotics、protein processing in endoplasmic reticulum)和碳代谢通路(carbon metabolism)上显著富集,也与p53、Wnt、FoxO等信号通路相关联。在靶基因预测结果中,SLC16A7的靶向结合位点被两个软件同时预测位于3′UTR的区域(图 2),推测其被miR-192直接靶向调节的可能性较大,因此选择SLC16A7作为后续验证靶基因。
|
图 1 预测靶基因的KEGG通路富集分析图 Fig. 1 Enrichment analysis map of KEGG pathway for predicting target genes |

|
图 2 对靶基因SLC16A7的预测结果 Fig. 2 Prediction of the target gene SLC16A7 |
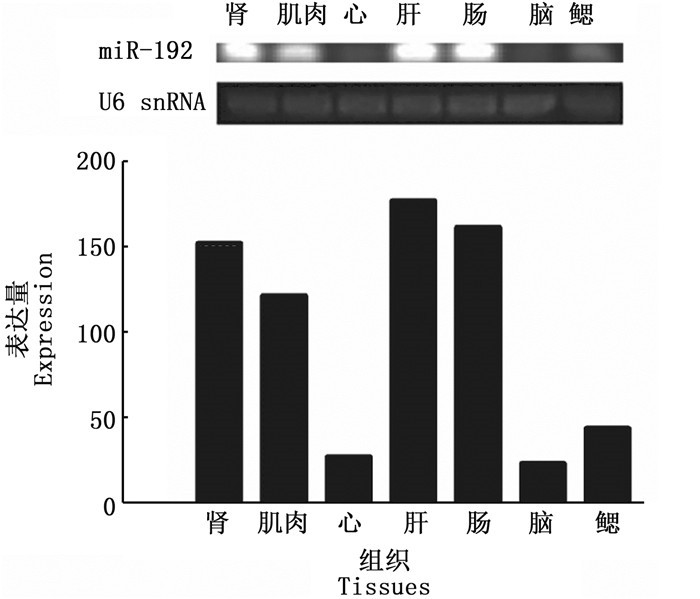
取0 h各个组织的cDNA进行PCR扩增,对扩增产物进行凝胶电泳检测,检测其组织表达谱。由图 3可知,miR-192在肾、肌肉、心、肝、肠、脑和鳃中都表达,其中在肾、肝和肠中表达水平较高。使用Image J对凝胶电泳条带的亮度进行定量,见图 3。
|
图 3 miR-192的组织表达谱 Fig. 3 Tissue expression of miR-192 |
miR-192和SLC16A7在碱度胁迫下鳃组织中的表达如图 4所示。在AW中,miR-192表达显著下降,SLC16A7的表达显著上升。

|
柱状图上方“*”代表差异显著(P < 0.05)。 "*"above the histogram shows significant difference (P < 0.05). 图 4 miR-192和SLC16A7在鳃组织96 h内的表达 Fig. 4 Expression of miR-192 and SLC16A7 in gill within 96 h |
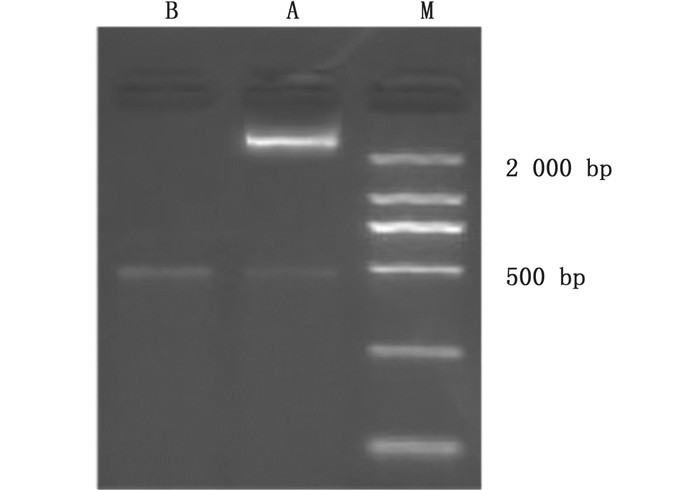
根据miR-192在SLC16A7-3′UTR的靶向位点设计引物,PCR产物经琼脂糖凝胶电泳显示为一条长度约500 bp的条带,与实际长度(508 bp)相符,测序结果证实该片断基因序列完全正确。pmirGLO载体经过酶切线性化,插入目的基因片段,制备pmirGLO-SLC16A7-3′-UTR双荧光素酶重组载体。构建好的载体经过双酶切鉴定,结果显示酶切后得到的片段与预期片段大小相符。见图 5。
|
A.pmirGLO-SLC16A7-3′-UTR双荧光素酶报告载体;B.PCR产物;M.Marker。 A. Dual luciferase reporter of pmirGLO-SLC16A7-3′-UTR; B. Production of PCR; M. Marker. 图 5 琼脂糖凝胶电泳结果 Fig. 5 Result of agarose gel electrophoresis |
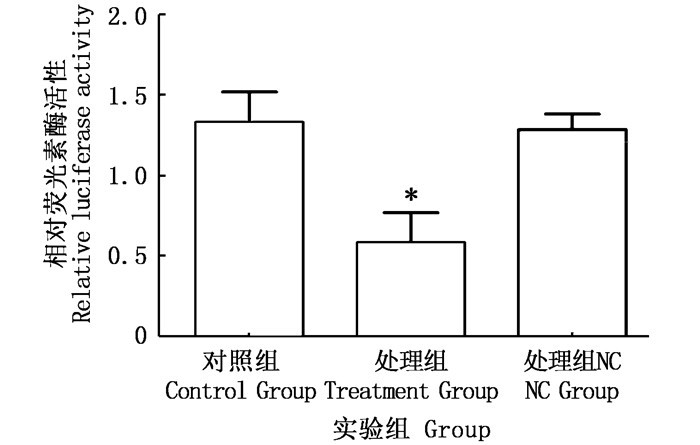
处理组、对照组和处理NC组均检测出荧光信号,但处理组的荧光素酶活性显著低于对照组和处理NC组。miR-192 mimics显著降低了pmirGLO-SLC16A7-3′-UTR双荧光素酶报告载体的荧光素酶表达。见图 6。
|
柱状图上方“*”代表差异显著(P < 0.05)。 "*" above the histogram shows significant difference (P < 0.05). 图 6 双荧光素酶活性检测 Fig. 6 Detection of dual luciferase activity |
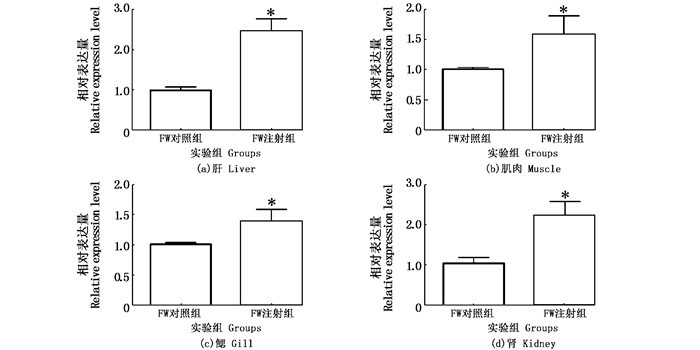
与注射生理盐水(对照组)相比miR-192抑制剂(处理组)中肝、鳃、肾、肌肉中miR-192表达均受到抑制,表达水平下降(P < 0.05),见图 7。SLC16A7的表达均显著上升(P < 0.05),见图 8。

|
柱状图上方“*”代表差异显著(P < 0.05)。 "*" above the histogram shows significant difference (P < 0.05). 图 7 体内注射miRNA抑制剂实验中miR-192的表达 Fig. 7 Expression of miR-192 in vivo injection of miRNA antagomir |
|
柱状图上方“*”代表差异显著(P < 0.05)。 "*" above the histogram shows significant difference (P < 0.05). 图 8 体内注射miRNA抑制剂实验中SLC16A7的表达 Fig. 8 Expression of SLC16A7 in vivo injection of miRNA antagomir |
研究表明,miR-192是一种高度保守的miRNA,在生物生长和疾病发生的过程中发挥重要作用。例如:在人肝癌细胞中,miR-192的表达显著下降,其靶向的SLC39A6参与癌细胞转移过程[26];在人结直肠癌细胞中,miR-192通过靶向C-X-C基序趋化因子配体2基因(C-X-C chemokines 2, CXCL2)抑制癌细胞迁移[27];在水稻中,miR-192负调控ATP结合蛋白(ATP-binding cassette transporter, ABC)基因参与对金属镉胁迫的应答过程[28]。miR-192在尼罗罗非鱼的肾、肝、胃肠、肌肉和鳃组织中均有表达,而碱度胁迫下在鳃组织中的表达随时间显著下调。对miR-192预测靶基因的KEGG通路富集分析发现,miR-192的靶基因与多个重要的信号传导(foxO signaling pathway、wnt signaling pathway等)、氨基酸代谢(lysine degradation等)、抗生素的生物合成(biosynthesis of antibiotics)、内质网中的蛋白质加工(protein processing in endoplasmic reticulum)等通路相关联。上述通路都在罗非鱼适宜碱度胁迫过程中起到重要作用[8],如参与逆境响应、生物损伤修复等。综上说明miR-192参与了罗非鱼应答及适应碱度胁迫的过程,也暗示了其可能的作用。
本研究筛选了靶基因SLC16A7并进行深入研究。通过实时荧光定量技术发现在碱度胁迫下miR-192显著下调,同时SLC16A7的表达显著上调。双荧光素酶报告实验发现,miR-192会与SLC16A7的3′UTR区域结合,降低相应荧光表达。体内注射miR-192抑制剂后,罗非鱼多个组织中miR-192的表达显著下调,同时SLC16A7显著上调。结果说明,SLC16A7是miR-192直接调控的靶基因,即miR-192通过与SLC16A7的mRNA 3′UTR区域结合,对其进行转录后水平调控。
溶质转运蛋白16家族(solute carriers family 16, SLC16)是细胞膜(含胞内膜)上最重要的膜转运蛋白家族之一,它参与了细胞间的物质运输和能量传递等重要生理活动[29],其中SLC16A1、SLC16A3、SLC16A7和SLC16A8在人体内的主要作用是转运乳酸盐、丙酮酸和酮体[30]。例如,当氧气供应不足、糖酵解受到刺激时,细胞内产生大量乳酸,SLC16A1和SLC16A3会参与酸碱调控并将乳酸代谢出细胞[31]。在高盐碱度环境下,生物体内pH会发生变动,耗氧率升高[32],细胞内可能会聚集乳酸代谢产物[33]。推测SLC16A7可能通过调节乳酸代谢和细胞内pH而参与罗非鱼适应碱度胁迫的过程。
| [1] |
刘永新, 方辉, 来琦芳, 等. 我国盐碱水渔业现状与发展对策[J]. 中国工程科学, 2016, 18(3): 74-78. LIU Y X, FANG H, LAI Q F, et al. The current state and development strategy for China's saline-alkaline fisheries[J]. Engineering Science, 2016, 18(3): 74-78. DOI:10.3969/j.issn.1009-1742.2016.03.013 |
| [2] |
雷衍之, 董双林, 沈成钢. 碳酸盐碱度对鱼类毒性作用的研究[J]. 水产学报, 1985, 9(2): 171-183. LEI Y Z, DONG S L, SHEN C G. Study on the toxicity of carbonate-alkaline to fishes[J]. Journal of Fisheries of China, 1985, 9(2): 171-183. |
| [3] |
章征忠, 张兆琪, 董双林, 等. pH、盐度、碱度对淡水养殖种类影响的研究进展[J]. 中国水产科学, 1999, 6(4): 95-98. ZHANG Z Z, ZHANG Z Q, DONG S L, et al. Advance in research on the effects of pH, salinity and alkalinity on freshwater cultured species[J]. Journal of Fishery Sciences of China, 1999, 6(4): 95-98. DOI:10.3321/j.issn:1005-8737.1999.04.022 |
| [4] |
王萍, 刘济源, 么宗利, 等. 水生动物盐碱适应生理学研究进展[J]. 长江大学学报(自然科学版), 2015, 12(15): 44-47. WANG P, LIU J Y, YAO Z L, et al. Research advances in saline-alkali adaptation physiology of aquatic animals[J]. Journal of Yangtze University (Natural Science Edition), 2015, 12(15): 44-47. |
| [5] |
么宗利, 应成琦, 周凯, 等. 碳酸盐碱度胁迫下凡纳滨对虾基因的差异表达[J]. 中国水产科学, 2012, 19(1): 1-12. YAO Z L, YING C Q, ZHOU K, et al. Gene expression profiles of Litopenaeus vannamei in response to carbonate alkalinity stress[J]. Journal of Fishery Sciences of China, 2012, 19(1): 1-12. |
| [6] |
YAO Z L, LAI Q F, ZHOU K, et al. Developmental biology of medaka fish (Oryzias latipes) exposed to alkalinity stress[J]. Journal of Applied Ichthyology, 2010, 26(3): 397-402. DOI:10.1111/j.1439-0426.2009.01360.x |
| [7] |
孙龙仪, 王云祥, 李秀梅. 碱度毒性和防治措施的探讨[J]. 天津水产, 1997(1): 34-35. SUN L Y, WANG Y X, LI X M. Discussion on alkalinity toxicity and control measures[J]. Tianjin Fisheries, 1997(1): 34-35. |
| [8] |
ZHAO Y, ZHANG C S, ZHOU H T, et al. Transcriptome changes for Nile tilapia (Oreochromis niloticus) in response to alkalinity stress[J]. Comparative Biochemistry and Physiology Part D:Genomics and Proteomics, 2020, 33: 100651. DOI:10.1016/j.cbd.2019.100651 |
| [9] |
涂翰卿.碳酸盐碱度胁迫下尼罗罗非鱼氨代谢途径与Rh蛋白氨转运作用[D].上海: 上海海洋大学, 2018. TU H Q. Ammonia metabolism pathway and ammonia transport of Rh proteins of Oreochromis niloticus under the stress of carbonate alkalinity[D]. Shanghai: Shanghai Ocean University, 2018. |
| [10] |
王燕, 赵金良, 吴俊伟, 等. 碱度胁迫对尼罗罗非鱼鳃离子细胞形态以及鳃、肾和肠中HCO3-转运因子的影响[J]. 动物学杂志, 2016, 51(6): 1027-1037. WANG Y, ZHAO J L, WU J W, et al. Effects of alkalinity on morphology of Gill Ionocytes and HCO3- transporters in gill, kidney and intestine of Nile tilapia (Oreochromis niloticus)[J]. Chinese Journal of Zoology, 2016, 51(6): 1027-1037. |
| [11] |
王燕, 赵金良, 赵岩, 等. 碱度对尼罗罗非鱼血清渗透压、离子浓度及离子转运酶活力的影响[J]. 生态科学, 2017, 36(4): 12-20. WANG Y, ZHAO J L, ZHAO Y, et al. Influence of alkalinity on serum osmolality, ion concentration and ion transport enzymes activity of Oreochromis niloticus[J]. Ecological Science, 2017, 36(4): 12-20. |
| [12] |
LAU N C, LIM L P, WEINSTEIN E G, et al. An abundant class of tiny RNAs with probable regulatory roles in Caenorhabditis elegans[J]. Science, 2001, 294(5543): 858-862. DOI:10.1126/science.1065062 |
| [13] |
REINHART B J, WEINSTEIN E G, RHOADES M W, et al. MicroRNAs in plants[J]. Genes & Development, 2002, 16(13): 1616-1626. |
| [14] |
HER G M, HSU C C, HONG J R, et al. Overexpression of gankyrin induces liver steatosis in zebrafish (Danio rerio)[J]. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids, 2011, 1811(9): 536-548. |
| [15] |
MENNIGEN J A, PANSERAT S, LARQUIER M, et al. Postprandial regulation of hepatic microRNAs predicted to target the insulin pathway in rainbow trout[J]. PLoS One, 2012, 7(6): e38604. DOI:10.1371/journal.pone.0038604 |
| [16] |
王维, 张玉娟, 陈洁, 等. 植物逆境胁迫相关miRNA研究进展[J]. 生物技术通报, 2015, 31(1): 1-10. WANG W, ZHANG Y J, CHEN J, et al. Research progress of MicroRNAs in plant stress responses[J]. Biotechnology Bulletin, 2015, 31(1): 1-10. |
| [17] |
ZHAO B T, GE L F, LIANG R Q, et al. Members of miR-169 family are induced by high salinity and transiently inhibit the NF-YA transcription factor[J]. BMC Molecular Biology, 2009, 10(1): 29. DOI:10.1186/1471-2199-10-29 |
| [18] |
LI H Y, DONG Y Y, YIN H L, et al. Characterization of the stress associated microRNAs in Glycine max by deep sequencing[J]. BMC Plant Biology, 2011, 11(1): 170. DOI:10.1186/1471-2229-11-170 |
| [19] |
ZHOU X F, WANG G D, SUTOH K, et al. Identification of cold-inducible microRNAs in plants by transcriptome analysis[J]. Biochimica et Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2008, 1779(11): 780-788. DOI:10.1016/j.bbagrm.2008.04.005 |
| [20] |
ZHAO B T, LIANG R Q, GE L F, et al. Identification of drought-induced microRNAs in rice[J]. Biochemical and Biophysical Research Communications, 2007, 354(2): 585-590. DOI:10.1016/j.bbrc.2007.01.022 |
| [21] |
ZHAO Y, ZHU C D, YAN B, et al. miRNA-directed regulation of VEGF in tilapia under hypoxia condition[J]. Biochemical and Biophysical Research Communications, 2014, 454(1): 183-188. DOI:10.1016/j.bbrc.2014.10.068 |
| [22] |
YAN B, GUO J T, ZHAO L H, et al. MiR-30c:a novel regulator of salt tolerance in tilapia[J]. Biochemical and Biophysical Research Communications, 2012, 425(2): 315-320. DOI:10.1016/j.bbrc.2012.07.088 |
| [23] |
YAN B, ZHU C D, GUO J T, et al. MiR-206 regulates the growth of the teleost tilapia (Oreochromis niloticus) through the modulation of IGF-1 gene expression[J]. Journal of Experimental Biology, 2013, 216(7): 1265-1269. |
| [24] |
ZHAO Y, WU J W, WANG Y, et al. Role of miR-21 in alkalinity stress tolerance in tilapia[J]. Biochemical and Biophysical Research Communications, 2016, 471(1): 26-33. DOI:10.1016/j.bbrc.2016.02.007 |
| [25] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [26] |
LIAN J W, JING Y, DONG Q Z, et al. miR-192, a prognostic indicator, targets the SLC39A6/SNAIL pathway to reduce tumor metastasis in human hepatocellular carcinoma[J]. Oncotarget, 2016, 7(3): 2672-2683. DOI:10.18632/oncotarget.6603 |
| [27] |
郑敏. miR-192靶向CXCL2基因对结直肠癌细胞侵袭迁移能力的抑制作用[D].唐山: 华北理工大学, 2017. ZHENG M. miR-192 targets C-X-C motif chemokine ligand 2 and inhibits invasion and migration in human colorectal cancer[D]. Tangshan: North China University of Science and Technology, 2017. |
| [28] |
刘海丽. miR192、miR166及其靶基因HD-Zip对水稻镉胁迫应答的功能研究[D].杭州: 中国计量学院, 2013. LIU H L. Functional analysis on cadmium response of miR192、mirR166 and it's target HD-Zip in rice (Oryza sativa)[D]. Hangzhou: China Jiliang University, 2013. |
| [29] |
MITCHELL P. Translocations through natural membranes[M]. NORD F F. Advances in Enzymology and Related Areas of Molecular Biology. New York: John Wiley & Sons, Inc, 1967: 33-87.
|
| [30] |
王月, 徐冰红, 刘虎, 等. 溶质转运蛋白超家族的功能及结构研究进展[J]. 现代生物医学进展, 2017, 17(24): 4775-4783, 4793. WANG Y, XU B H, LIU H, et al. Research progress on the function and structure of solute carrier superfamily transporters[J]. Progress in Modern Biomedicine, 2017, 17(24): 4775-4783, 4793. |
| [31] |
HALESTRAP A P. The SLC16 gene family-Structure, role and regulation in health and disease[J]. Molecular Aspects of Medicine, 2013, 34(2/3): 337-349. |
| [32] |
ZHANG P D, ZHANG X M, LI J, et al. The effects of body weight, temperature, salinity, pH, light intensity and feeding condition on lethal DO levels of whiteleg shrimp, Litopenaeus vannamei (Boone, 1931)[J]. Aquaculture, 2006, 256(1/4): 579-587. |
| [33] |
张桂芝.单羧酸转运蛋白MCT1基因反义表达重组载体转染人肺腺癌细胞及其生物学效应的研究[D].重庆: 第三军医大学, 2001. ZHANG G Z. Study on Monocarboxylate transporter-1 gene anti-sense reconstructed vector transfection into lung adenocarcinoma A549 cells and its biological affects[D]. Chongqing: Third Military Medical University, 2001. |
2. Shanghai Collaborative Innovation for Aquatic Animal Genetics and Breeding, Shanghai Ocean University, Shanghai 201306, China
 2021,
Vol. 30
2021,
Vol. 30


