2. 农业农村部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
诺如病毒(norovirus)属于杯状病毒科(Caliciviridae)诺如病毒属(Norovirus), 目前,诺如病毒可分为10个基因群(GⅠ-GⅩ), 其中GⅡ型是引起人类急性肠胃炎的主要病因,包括至少27种不同的基因型[1-2]。2006—2016年,中国地区有超过132起(8 133例)诺如病毒疫情的暴发,其中82%的案例都是由GⅡ型诺如病毒引起的,造成了严重的医疗负担和经济损失[3]。牡蛎是诺如病毒在环境中流行和传播的重要载体[4]。诺如病毒相关的临床疫情通常与消费受污染的牡蛎有关[5-6]。大量研究证实,超过80%的人诺如病毒基因型存在于牡蛎中[7]。因此,牡蛎中诺如病毒的快速、准确的检测将有利于相关环境的监测和疾病的防控。
巢式RT-PCR又称为嵌套式RT-PCR, 是在RT-PCR基础上发展而来的,使用2对引物对目的片段进行扩增,具有灵敏度高,特异性强等优点,在人[8]和水产品[9]病原生物的检测上已得到广泛的应用。自2002年以来,KAGEYAMA等[5]、KOJIMA等[10]和NISHIDA等[11]针对诺如病毒衣壳蛋白基因(VP1)设计了2对引物(COG2F/G2SKR, G2SKF/G2SKR), 被广泛用于诺如病毒的分型与检测。然而,近年来越来越多的研究发现在检测牡蛎中的诺如病毒时,该方法经常出现大小不一的非特异性PCR产物,而后续的测序结果证实这些序列多源于牡蛎本身或其肠道细菌的基因[12], 表明现有的巢式PCR方法在应用于牡蛎中诺如病毒的检测时容易受到样品本底的影响,特异性差,造成假阳性等结果。诺如病毒基因组极易变异且基因重组频繁发生[2], 因此,现有的PCR引物因基因型覆盖度不够还会出现假阴性等情况[12-13]。
针对上述问题,本研究基于现有公共数据库(NCBI GenBank)中所有的GⅡ型诺如病毒的序列,重新设计了针对GⅡ型诺如病毒的通用型巢式RT-PCR引物并通过牡蛎样品的实际检测对其特异性、灵敏度和稳定性等3个方面进行了系统地评估,并与旧引物进行了对比[12], 为用于牡蛎中GⅡ型诺如病毒的检测提供一种可靠的技术手段。
1 材料与方法 1.1 主要试剂及仪器 1.1.1 实验试剂动物组织总RNA提取试剂盒购自上海捷瑞生物工程有限公司;RNA稳定和储存溶液RNAlater、病毒RNA提取试剂盒购自上海翊瑞生物科技有限公司;一步法反转录PCR试剂盒、PCR Master Mix购自宝生物工程(大连)有限公司;100 bp DNA Ladder购自天根生化科技(北京)有限公司和上海奈彻生物科技有限公司。
1.1.2 实验样品及预处理实验所用太平洋牡蛎(Crassostrea gigas)购自上海市芦潮港海鲜市场(长10~12 cm、宽6~8 cm)。解剖牡蛎的消化腺组织后分装成50~100 mg每管,保存在RNA保护剂中并冻于-80 ℃冰箱。中国疾病预防控制中心(以下简称中国CDC)提供的诺如病毒GⅡ型阳性粪便样品(GⅡ.3, GⅡ.4, GⅡ.6, GⅡ.12, GⅡ.6[P7], GⅡ.2[P16]和GⅡ.17)经实验室测序验证后,保存于-80 ℃冰箱。
1.1.3 实验仪器AC2-4S1-CN生物安全柜、ACB-4A1超净工作台购自新加坡艺思高科技有限公司(ESCO); FastPre-24 MP组织均质器购自MP Biomedicals(美国); 5331 PCR仪、5382000074恒温混匀金属浴、5702R台式低温离心机购自艾本德中国有限公司(Eppendorf); PowerPac HC电泳仪、ChemiDoc XRS+ 凝胶成像系统和凝胶成像分析仪购自伯乐生命医学产品(上海)有限公司(Bio-Rad)。
1.2 实验方法 1.2.1 引物设计与合成根据GUO等[12]的研究经验,建立了1个用于引物设计的本地数据库,其中包含了从NCBI(https://www.ncbi.nlm.nih.gov)下载的在线能得到的所有GⅡ型诺如病毒的序列,用诺如病毒基因分型工具(https://www.rivm.nl/mpf/typingtool)对其进行基因分型,并按照基因型进行分组。利用生物信息学软件Geneious Prime进行序列比对分析,基于保守区域的序列设计出针对GⅡ型诺如病毒的通用型引物,并通过NCBI上nr库的Primer Blast工具评估新引物的特异性。所设计GⅡ型诺如病毒的巢式RT-PCR通用型新引物及旧引物合成于上海生工生物有限公司,序列如表 1所示。

|
表 1 巢式RT-PCR的引物组合 Tab.1 Primer combination of nested RT-PCR |
牡蛎中总RNA的提取:取1管牡蛎消化腺组织用MP组织均质器打成匀浆,按照动物组织总RNA提取试剂盒说明书提取牡蛎消化腺的总RNA, 直接作为RT-PCR的模板使用。
粪便中病毒RNA的提取:按照病毒RNA提取试剂盒说明书提取粪便中的病毒RNA, 直接作为RT-PCR的模板使用。
1.2.3 旧巢式RT-PCR反应旧巢式RT-PCR法(COG2F/G2SKR, G2SKF/G2SKR, 以下简称G2SKF/G2SKR)分为RT-PCR和第2轮PCR。将所提取的实际样品的RNA作为模板进行RT-PCR反应,RT-PCR反应体系共25 μL, 含2×1 Buffer 12.5 μL, COG2F 1 μL, G2SKR 1 μL, Enzyme Mix 1 μL, ddH2O 8.5 μL, 模板1 μL。
反应程序:50 ℃反转录30 min, 94 ℃预变性2 min, 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸30 s, 30个循环。
将第1轮产物稀释10倍作为模板进行第2轮PCR反应,第2轮PCR反应体系共25 μL, 含PCR Mix 12.5 μL, G2SKF 1 μL, G2SKR 1 μL, ddH2O 9.5 μL, 模板1 μL。
反应程序:94 ℃预变性4 min, 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸30 s, 35个循环,72 ℃延伸7 min。两轮PCR扩增结束后用2%的琼脂糖凝胶电泳检测PCR产物。
1.2.4 新巢式RT-PCR反应新巢式RT-PCR法(COG2F/NG2OR, NG2OF/NG2OR, 以下简称NG2OF/NG2OR)分为RT-PCR和第2轮PCR。将所提取的实际样品的RNA作为模板进行RT-PCR反应,RT-PCR反应体系共25 μL, 含2×1 Buffer 12.5μL, COG2F 1 μL, NG2OR 1 μL, Enzyme Mix 1 μL, ddH2O 8.5 μL, 模板1 μL。
反应程序:50 ℃反转录30 min, 94 ℃预变性2 min, 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸30 s, 30个循环。
将第1轮产物稀释10倍作为模板进行第2轮PCR反应,第2轮PCR反应体系共25 μL, 含PCR Mix 12.5 μL, NG2OF 1 μL, NG2OR 1 μL, ddH2O 9.5 μL, 模板1 μL。
反应程序:94 ℃预变性4 min, 94 ℃变性30 s, 55 ℃退火30 s, 72 ℃延伸30 s, 35个循环,72 ℃延伸7 min。两轮PCR扩增结束后用2%的琼脂糖凝胶电泳检测PCR产物。
1.2.5 覆盖度评估采用由中国CDC提供的GⅡ型诺如病毒阳性粪便样品(GⅡ.3, GⅡ.4, GⅡ.6, GⅡ.12, GⅡ.6[P7], GⅡ.2[P16]和GⅡ.17), 分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)按照1.2.3节和1.2.4节中反应体系及条件进行巢式RT-PCR扩增和电泳检测。
1.2.6 特异性评估采用由实验室前期得到的有非特异性扩增现象的牡蛎样品(背景对照)和GⅠ型诺如病毒阳性的牡蛎样品(背景对照和阴性对照), 分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)按照1.2.3节和1.2.4节中反应体系及条件进行巢式RT-PCR扩增和电泳检测。
1.2.7 灵敏度评估采用随机选取的3个GⅡ型诺如病毒阳性且已知浓度的牡蛎RNA(分别为9.32×103、2.64×104、5.79×104拷贝/μL)和它们的10倍梯度稀释液作为模板[14], 分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)按照1.2.3节和1.2.4节中反应体系及条件进行巢式RT-PCR扩增和电泳检测。
1.2.8 实际样品检测采用92只新鲜采集的牡蛎样品,分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)按照1.2.3节和1.2.4节中反应体系及条件进行巢式RT-PCR扩增和电泳检测。
2 结果 2.1 引物设计本地数据库中包含了在线下载的共29 784条(截至2017年12月31日)GⅡ诺如病毒序列。小于1 000 nt (nucleotide)的序列通常位于P区(protruding domain), 保守性差,不适合用于引物设计,因而被筛除。因此,共得到6 419条长度为1 000~7 627 nt的序列,将其进行基因分型,得到44条GⅡ.1, 241条GⅡ.2, 359条GⅡ.3, 3 561条GⅡ.4, 31条GⅡ.5, 104条GⅡ.6, 147条GⅡ.7, 8条GⅡ.8, 5条GⅡ.9, 10条GⅡ.10, 8条GⅡ.11, 227条GⅡ. 12, 76条GⅡ.13, 186条GⅡ.14, 6条GⅡ.15, 352条GⅡ.16, 796条GⅡ.17, 8条GⅡ.18, 8条GⅡ.19, 12条GⅡ.20, 186条GⅡ.21, 39条GⅡ.22和5条GⅡ.23序列。用生物信息学软件Geneious Prime对6 419条序列进行多重比对(multiple sequences alignment)分析,发现GⅡ型诺如病毒的核酸序列变异性过高,无法找到合适的保守区域对23种基因型进行全部覆盖。鉴于此,再次尝试采用Geneious Prime软件对每种基因型的序列进行单独地比对分析,并充分考虑每种基因型序列在全部序列中占有的权重和比例,首先以丰度高达55.5% (3 561/6 419)的GⅡ.4型序列为主进行新引物的设计,然后,采用序列比对的方式评估新引物与其余22种基因型的保守序列的匹配情况,最终设计出含有大量简并碱基的新引物,并将其命名为NG2OF/NG2OR, 序列如表 1所示。
2.2 基因型覆盖度为全面分析引物所覆盖的病毒基因型,我们选取近年来在世界范围内广泛传播并且丰度较高的7种代表性GⅡ型诺如病毒阳性粪便样品(GⅡ.3, GⅡ.4, GⅡ.6, GⅡ.12, GⅡ.6[P7], GⅡ.2[P16]和GⅡ.17), 分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)进行巢式RT-PCR扩增和电泳分析。如图 1所示,除GⅡ.6[P7]型诺如病毒阳性粪便样品外,旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)均成功地扩增出其余6种GⅡ型诺如病毒阳性粪便样品,表明新旧引物在基因型覆盖度上基本一致。通过浓度测定发现,GⅡ.6[P7]型诺如病毒阳性粪便样品因降解导致其病毒浓度降低,在采用新引物(NG2OF/NG2OR)时胶图上呈现非常微弱但单一的条带,而采用旧引物(G2SKF/G2SKR)时胶图上并没有条带出现,表明新引物(NG2OF/NG2OR)在灵敏度方面具有一定的优势。
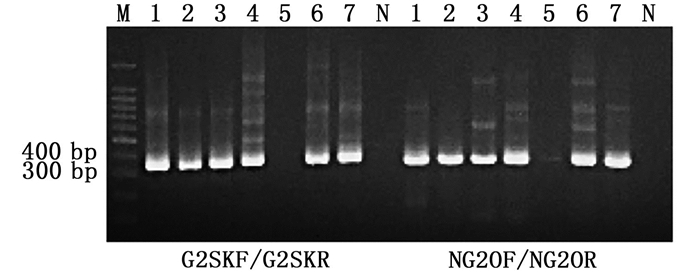
|
M. 100 bp DNA marker; 1~7. GⅡ.3, GⅡ.4, GⅡ.6, GⅡ.12, GⅡ.6[P7], GⅡ.2[P16]和GⅡ.17型诺如病毒阳性粪便样品;N. 阴性对照;G2SKF/G2SKR. 344 bp; NG2OF/NG2OR. 337 bp。 M. 100 bp DNA marker; 1-7. GⅡ.3, GⅡ.4, GⅡ.6, GⅡ.12, GⅡ.6[P7], GⅡ.2[P16] and GⅡ.17 norovirus contaminated stool samples; N. negative control; G2SKF/G2SKR. 344 bp; NG2OF/NG2OR. 337 bp. 图 1 基因型覆盖率的评估 Fig. 1 Evaluation of genotype coverage |
采用由实验室前期得到的有非特异性扩增现象的牡蛎样品和GⅠ型诺如病毒阳性的牡蛎样品,分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)进行巢式RT-PCR扩增和电泳分析。如图 2所示,所有3只牡蛎样品在采用旧引物(G2SKF/G2SKR)检测时均出现了不同大小的非特异性条带,经割胶回收后测序验证为牡蛎本身或其肠道微生物群的序列,检测结果全为GⅡ型诺如病毒阴性。1号样品(背景对照)在采用新引物(NG2OF/NG2OR)时成功扩增出亮且单一的目标条带,并经后续测序验证得出GⅡ.12型诺如病毒阳性,体现了新引物(NG2OF/NG2OR)在灵敏度方面具有一定的优势。2号样品(背景对照)在采用新引物(NG2OF/NG2OR)时胶图上没有出现信号,结果为阴性。3号样品(背景对照和阴性对照)在采用新引物(NG2OF/NG2OR)时胶图上背景干净、没有信号,结果为阴性。结果显示新引物(NG2OF/NG2OR)在检测牡蛎样品时特异性更好。特异性实验结果表明,无论是背景对照(牡蛎)还是阴性对照(GⅠ型诺如病毒阳性牡蛎), 都没有出现扩增现象,有效地纠正了使用旧引物(G2SKF/G2SKR)时出现的非特异性扩增的问题。
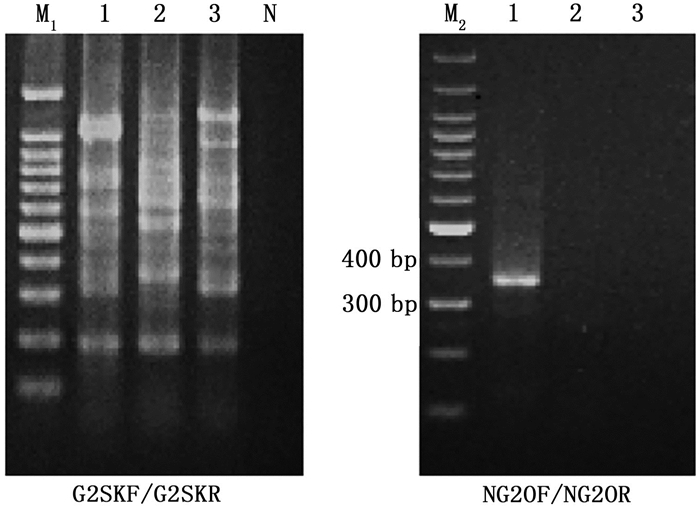
|
M1和M2.100 bp DNA marker; 1~2. 由实验室前期牡蛎中诺如病毒检测得到的有非特异性扩增的牡蛎样品;3. 由实验室前期牡蛎中诺如病毒检测得到的GⅠ型诺如病毒阳性牡蛎样品;N. 阴性对照(水); G2SKF/G2SKR. 344 bp; NG2OF/NG2OR. 337 bp。 M1, M2. 100 bp DNA marker; 1-2. Oyster samples with non-specific bands; 3. GⅠ norovirus contaminated oyster sample; N. negative control (ddH2O); G2SKF/G2SKR. 344 bp; NG2OF/NG2OR. 337 bp. 图 2 特异性的评估 Fig. 2 Evaluation of specificity |
为了评价检测方法的灵敏度(检测限), 采用3个随机选取的GⅡ型诺如病毒阳性并已知浓度的牡蛎消化腺RNA(编号1904047、1902263、1902264)和它们的10倍梯度稀释液作为模板,分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)进行巢式RT-PCR扩增和电泳分析。如表 2所示,在采用新引物(NG2OF/NG2OR)时最低可以稳定地检测到26.4个拷贝,成功检测到1次9.32个拷贝。采用旧引物(G2SKF/G2SKR)时最低稳定地检测到57.9个拷贝。从结果可以看出新引物(NG2OF/NG2OR)灵敏度更高。
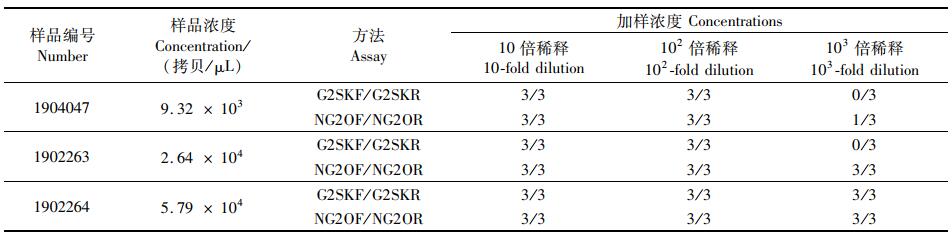
|
表 2 灵敏度的评估 Tab.2 Evaluation of sensitivity |
随机挑选新鲜采集的92只牡蛎样品,分别用旧引物(G2SKF/G2SKR)和新引物(NG2OF/NG2OR)进行巢式PCR扩增和电泳检测,结果如表 3所示。从表 4可以看出,使用旧引物(G2SKF/G2SKR)检测牡蛎中的诺如病毒时,阳性检出率为20.7%, 低于新引物(NG2OF/NG2OR, 28.3%)。其中,有7只牡蛎样品在使用旧引物(G2SKF/G2SKR)检测时呈阴性,而使用新引物(NG2OF/NG2OR)检测时呈阳性,后经测序并分型得到基因型分别为GⅡ.17(n = 2)、GⅡ.2(n = 2)、GⅡ.3(n = 3)。结果表明,采用旧引物(G2SKF/G2SKR)检测牡蛎样品会出现假阴性的情况,这种情况对GⅡ型基因型普遍存在,从而低估了牡蛎中的诺如病毒的污染情况和基因型的丰度,而采用新引物(NG2OF/NG2OR)可避免这样的情况。新引物(NG2OF/NG2OR)更适用于牡蛎中的诺如病毒检测。

|
表 3 样品信息 Tab.3 Information of samples |

|
表 4 牡蛎样品诺如病毒检测结果 Tab.4 Results of detection of norovirus in oyster samples |
目前诺如病毒分子检测手段中,荧光定量RT-PCR法技术成熟且灵敏度高,但在检测牡蛎样品时经常出现假阳性的情况[5, 15]。此外,荧光定量RT-PCR扩增产物过短,无法用于后续的基因型分析。而巢式RT-PCR方法较于常规RT-PCR方法更为灵敏,更适用于诺如病毒含量较低的牡蛎样品的检测。但是目前常用的巢式RT-PCR方法建立于20多年前,其间病毒的序列发生了显著的改变,新产生的序列数量是之前的上百倍。因此,建立一套新的具有更高特异性和灵敏度的巢式RT-PCR方法用于牡蛎样品中的诺如病毒检测是非常迫切的,为调查牡蛎消费的人中诺如病毒的感染情况和基因型研究提供重要支撑。
同时,为避免因基因型覆盖度低等原因造成的漏检问题,大量的序列分析对于引物设计是十分必要的[16-17]。本研究建立了含6 419条(截至2017年12月31日)GⅡ型诺如病毒序列的本地数据库,基于对所有序列的分析,进行了引物设计。从位置上看,新引物(NG2OF/NG2OR)与旧引物(G2SKF/G2SKR)只有几个碱基的差别(表 1), 却避免了引物3′端和5′端的错配,从而提高了扩增效率。这可能是新引物(NG2OF/NG2OR)灵敏度更高的原因。从简并碱基来看,引物NG2OR与引物G2SKR不同的是,基于对6 419条序列的分析,没有使用简并碱基H而是使用了V, 这样能覆盖更多的序列。2019年,诺如病毒基因群及基因型的分类进行了更新[1], GⅡ的基因型数量由23扩大为27。尽管基因型的数量增加了,由于数据库中用于引物设计的序列保持不变,因此这并不影响本研究所优化的GⅡ型诺如病毒巢式RT-PCR法。
本研究从基因型覆盖度、特异性、灵敏度和稳定性等4个方面对新引物(NG2OF/NG2OR)进行了评估。在基因型覆盖度方面,采用的是GⅡ型诺如病毒阳性粪便样品。与牡蛎样品不同,粪便样品中的诺如病毒含量高,通常应用常规RT-PCR检测。而采用巢式RT-PCR检测时,粪便中富余的病毒RNA可能导致PCR扩增效率过高,从而出现了非特异性扩增的现象。本研究采用了7种代表性的GⅡ型诺如病毒样本对基因型覆盖度进行了评估,这7种GⅡ基因型均为近年世界范围内的主要流行株,其序列丰度占NCBI在线数据库(截至2017年12月31日)中GⅡ型诺如病毒序列的89.1%, 代表了绝大多数GⅡ型诺如病毒。因此,使用这7种GⅡ基因型对基因型覆盖度进行评估的结果是具有代表性的。在特异性验证方面,采用了GⅠ型诺如病毒阳性的牡蛎样品作为阴性对照。在亲缘关系上,GⅠ型诺如病毒与GⅡ型诺如病毒关系最近。因此,使用GⅠ型诺如病毒阳性的牡蛎样品进行交叉实验的结果是非常具有代表性的。在灵敏度方面,新引物(NG2OF/NG2OR)的灵敏度是旧引物(G2SKF/G2SKR)的2倍,且结果稳定。采用实际样品对灵敏度进行评估的好处是可排除样品中存在的PCR抑制剂对结果的影响,比起采用质粒、标准品、人工阳性样品等,结果更可靠。在进行临床牡蛎样品诺如病毒筛查时发现,有7只牡蛎样品在使用旧引物(G2SKF/G2SKR)检测时呈阴性,而使用新引物(NG2OF/NG2OR)检测时呈阳性并经后续测序验证。这说明新引物(NG2OF/NG2OR)在检测低含量的牡蛎样品时更加具有优势。
综上所述,本研究所建立的GⅡ型诺如病毒巢式RT-PCR检测方法在检测牡蛎样品时特异性强,灵敏度高,应用性较好,为牡蛎中诺如病毒的检测提供了一种新的、可靠的技术手段。
| [1] |
CHHABRA P, DE GRAAF M, PARRA G I, et al. Updated classification of norovirus genogroups and genotypes[J]. Journal of General Virology, 2019, 100(10): 1393-1406. DOI:10.1099/jgv.0.001318 |
| [2] |
AO Y Y, WANG J J, LING H, et al. Norovirus GⅡ. P16/GⅡ. 2-associated gastroenteritis, China, 2016[J]. Emerging Infectious Diseases, 2017, 23(7): 1172-1175. DOI:10.3201/eid2307.170034 |
| [3] |
QIN S W, CHAN T C, CAI J, et al. Genotypic and epidemiological trends of acute gastroenteritis associated with Noroviruses in China from 2006 to 2016[J]. International Journal of Environmental Research and Public Health, 2017, 14(11): 1341. DOI:10.3390/ijerph14111341 |
| [4] |
CAMPOS C J A, LEES D N. Environmental transmission of human noroviruses in shellfish waters[J]. Applied and Environmental Microbiology, 2014, 80(12): 3552-3561. DOI:10.1128/AEM.04188-13 |
| [5] |
KAGEYAMA T, KOJIMA S, SHINOHARA M, et al. Broadly reactive and highly sensitive assay for Norwalk-like viruses based on real-time quantitative reverse transcription-PCR[J]. Journal of Clinical Microbiology, 2003, 41(4): 1548-1557. DOI:10.1128/JCM.41.4.1548-1557.2003 |
| [6] |
IMAMURA S, KANEZASHI H, GOSHIMA T, et al. Next-generation sequencing analysis of the diversity of human Noroviruses in Japanese oysters[J]. Foodborne Pathogens and Disease, 2017, 14(8): 465-471. DOI:10.1089/fpd.2017.2289 |
| [7] |
YU Y X, CAI H, HU L H, et al. Molecular epidemiology of oyster-related human Noroviruses and their global genetic diversity and temporal-geographical distribution from 1983 to 2014[J]. Applied and Environmental Microbiology, 2015, 81(21): 7615-7624. DOI:10.1128/AEM.01729-15 |
| [8] |
刘爱玲, 刘爱胜, 罗小芳, 等. 肠道轮状病毒巢式PCR检测方法的建立及初步应用[J]. 临床输血与检验, 2019, 21(6): 604-608. LIU A L, LIU A S, LUO X F, et al. Establishment and preliminary application of intestinal rotavirus(RV)nested PCR detection method[J]. Journal of Clinical Transfusion and Laboratory Medicine, 2019, 21(6): 604-608. DOI:10.3969/j.issn.1671-2587.2019.06.013 |
| [9] |
柴下, 陆宏达, 刘俊杰, 等. 武汉单极虫巢式聚合酶链反应检测方法的建立与优化[J]. 上海海洋大学学报, 2014, 23(4): 556-563. CHAI X, LU H D, LIU J J, et al. Establishment of nested PCR assay for detection of Thelohanellus wuhanensis in allo-gynogenetic crucian carp[J]. Journal of Shanghai Ocean University, 2014, 23(4): 556-563. |
| [10] |
KOJIMA S, KAGEYAMA T, FUKUSHI S, et al. Genogroup-specific PCR primers for detection of Norwalk-like viruses[J]. Journal of Virological Methods, 2002, 100(1/2): 107-114. |
| [11] |
NISHIDA T, KIMURA H, SAITOH M, et al. Detection, quantitation, and phylogenetic analysis of noroviruses in Japanese oysters[J]. Applied and Environmental Microbiology, 2003, 69(10): 5782-5786. DOI:10.1128/AEM.69.10.5782-5786.2003 |
| [12] |
GUO P, YU Y X, PAN Y J, et al. Design and evaluation of nested PCR primers for specific detection of genogroup I noroviruses in oysters[J]. Molecular and Cellular Probes, 2018, 40: 40-43. DOI:10.1016/j.mcp.2018.06.002 |
| [13] |
李志凯, 苏国成, 苏文金, 等. 诺如病毒检测方法研究进展[J]. 微生物学通报, 2014, 41(7): 1417-1424. LI Z K, SU G C, SU W J, et al. Progress of detection methods for norovirus[J]. Microbiology China, 2014, 41(7): 1417-1424. |
| [14] |
MOORE M D, JAYKUS L A. Development of a Recombinase polymerase amplification assay for detection of epidemic human Noroviruses[J]. Scientific Reports, 2017, 7(1): 40244. DOI:10.1038/srep40244 |
| [15] |
PU J, KAZAMA S, MIURA T, et al. Pyrosequencing analysis of norovirus genogroup II distribution in sewage and oysters: first detection of GⅡ. 17 Kawasaki 2014 in oysters[J]. Food and Environmental Virology, 2016, 8(4): 310-312. DOI:10.1007/s12560-016-9261-5 |
| [16] |
ANDO T, NOEL J S, FANKHAUSER R L. Genetic classification of "Norwalk-like viruses"[J]. The Journal of Infectious Diseases, 2000, 181(s2): 336-348. DOI:10.1086/315589 |
| [17] |
VINJÉ J, HAMIDJAJA R A, SOBSEY M D. Development and application of a capsid VP1 (region D) based reverse transcription PCR assay for genotyping of genogroup I and II noroviruses[J]. Journal of Virological Methods, 2004, 116(2): 109-117. DOI:10.1016/j.jviromet.2003.11.001 |
2. Laboratory of Quality and Safety Risk Assessment for Aquatic Products on Storage and Preservation, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China
 2021,
Vol. 30
2021,
Vol. 30


