2. 上海海洋大学 教育部水产遗传资源开发利用重点实验室, 上海 201306;
3. 上海海洋大学 水生动物遗传育种中心上海市协同创新中心, 上海 201306
三角帆蚌(Hyriopsis cumingii)是我国特有且重要的大型淡水珍珠蚌,具有很高的经济价值。我国从20世纪80年代初期开始研究淡水珍珠蚌的细胞培养技术[1],到目前为止,其细胞体外分裂迟缓仍是贝类细胞培养的一大难题。虽然石安静[2]在珍珠蚌外套膜细胞培养基中加入了胎牛血清及水解乳蛋白,以供细胞养分;李倩等[3]采用物理化学诱导法诱导三角帆蚌外套膜细胞体外增殖分裂,但至今未能解决该难题,无法建立贝类成熟的细胞系,制约了对三角帆蚌育珠育种、病害防治和基因功能等方面的进一步研究。
细胞周期(cell cycle)是保证细胞进行生命活动的基本过程,细胞通过细胞周期时相的变迁进入增殖、分化、衰老和死亡等生理过程[4-5]。细胞周期蛋白B(cyclin B)是卵细胞成熟促进因子(MPF)的主要成分,在细胞周期运转中主要参与调控M期,即细胞分裂期。目前,在水生生物中对cyclin B基因的研究多与性腺发育相关,FANG等[6]克隆出中华绒螯蟹(Eriocheir sinensis)cyclin B基因全长,并研究了其在卵黄发生期的表达模式;VISUDTIPHOLE等[7]在斑节对虾(Penaeus monodon)卵巢中克隆出cyclin B基因,并检测到该基因在性成熟个体的表达量高于未成熟个体等,但cyclin B作为细胞周期周转中重要的正向调控因子,主要诱导细胞分裂。XU等[8]鉴定了秀丽隐杆线虫(Caenorhabditis elegans)4种cyclin B基因,并发现cyclin B-3是染色体分离不可缺少的基因,但在贝类中对cyclin B基因与细胞分裂间直接关系的研究还未开展。
本研究对三角帆蚌cyclin B基因进行克隆,并对其进行了生物信息分析,通过RT-qPCR技术,检测了三角帆蚌不同性别及组织中cyclin B基因的表达水平,构建了三角帆蚌cyclin B基因的组织表达谱,利用RNA干扰(RNAi)技术对cyclin B基因功能进行了初步分析,为后续三角帆蚌细胞分裂机制、细胞体外培养及建系提供了分子基础。
1 材料与方法 1.1 实验材料三角帆蚌采购于上海滨海生产基地,选取喷水有力、大小均一、体质健康的1龄蚌,饲养于实验室玻璃缸内,以豆浆投喂并24 h不断冲氧。
三角帆蚌暂养于实验室2周,随机挑选4只雌蚌和4只雄蚌解剖后,取其闭壳肌、斧足、外套膜、心脏、血液、内脏团、鳃和性腺组织,经DEPC水洗净并迅速放置于液氮中,存于-80 ℃冰箱中备用。
1.2 实验方法 1.2.1 总RNA提取和检测根据Trizol® Reaget(Invitrogen,美国)的操作说明将上述采集的样品进行RNA提取。利用NANODROP 2000C(Thermo Fisher Scientific, 美国)测定总RNA OD260/280(1.8~2.0范围的RNA纯度较好)及浓度,再根据1%琼脂糖凝胶电泳检测RNA质量,所有RNA样品均符合实验要求后,置于-80 ℃保存备用。反转录过程按照PrimeScriptTM RT reagent Kit with gDNA Eraser (TaRaKa,日本)的操作步骤进行。
1.2.2 cyclin B基因的克隆及分析根据转录组数据库中cyclin B基因的部分序列,运用Primer Premier 5.0设计引物(表 1),由生工生物工程(上海)公司合成。用三角帆蚌各组织混合池cDNA为模板进行PCR扩增。PCR的扩增条件:94 ℃预变性3 min、94 ℃变性30 s、50 ℃退火30 s、72 ℃延伸40 s,共40个循环。扩增产物经1%的琼脂糖凝胶回收纯化与PMD19-T(TaKaRa, 日本)连接转化入感受态大肠杆菌DH5α(实验室保存)中,经菌落PCR验证后将阳性克隆送于生工生物工程(上海)公司测序。根据克隆出来的三角帆蚌cyclin B片段,设计3′RACE的特异性引物(表 1)。依照3′-Full RACE kit (TaKaRa, 日本)试剂盒说明,进行RACE PCR扩增。RACE PCR产物经1%琼脂糖凝胶电泳分离后,与PCR回收纯化目的片段,进行连接、转化、克隆和测序。
|
表 1 实验所用引物 Tab.1 Primers used in the experiment |
用DANMAN软件将所得到的三角帆蚌cyclin B片段去除序列间重叠部分以及RACE试剂盒添加的引物部分,拼接得到克隆片段。应用ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/orfig.cgi)对cDNA进行开放阅读框(ORF)分析,并进行比对、分析其氨基酸序列。应用DNAMAN软件将三角帆蚌cyclin B的碱基及氨基酸序列分别与GenBank数据库中其他物种的cyclin B的碱基和氨基酸序列进行比对,检测它们之间的序列相似性。通过ExPASy系统(https://web.expasy.org/protparam/)对三角帆蚌cyclin B的分子量和理论等电点进行预测。应用String(http://string-db.org)对能与其发生相互作用的靶蛋白进行预测。
1.2.3 RNA干扰(RNAi)实验siRNA的制备:根据小干扰RNA(siRNA)设计原则,针对克隆得到的cyclin B基因设计目的基因干扰链(siRNA-cyclin B)的正义链(sense)和反义链(antisense),加以碱基修饰后,交由上海吉玛制药技术有限公司合成;NC链是通用阴性对照链,购自吉玛公司。目的干扰链和NC链的序列信息见表 2。

|
表 2 RNA干扰实验所用引物 Tab.2 Primers used in the RNAi |
实验设置:从三角帆蚌暂养缸中随机挑选9只已暂养2周以上的健康1龄蚌随机平均分成3组,即cyclin B干扰组(注射cyclin B干扰链)、NC组(注射NC链)和正常组(不注射)。用1 mL无酶注射器将干扰链(20 μmol/L)和NC链(20 μmol/L)在0 h、24 h时注射进三角帆蚌性腺内,并在首次注射48 h后取三角帆蚌的性腺、鳃和外套膜组织与细胞,分别备用于荧光定量PCR和细胞周期测定。
1.2.4 荧光定量PCR根据克隆得到的cyclin B基因结构域保守区,运用Primer Premier 5.0软件设计特异性好的PCR引物(表 1),并交由生工生物工程(上海)公司合成。cyclin B基因的定量分析在CFX96 TouchTM Real-Time PCR Detection System (Bio-Rad,美国)平台进行。首先制备目的基因和内参基因(EF1A)的标准曲线。然后按照以下条件进行相对定量分析。反应体系(20 μL):1.0 μL cDNA,上下游引物各0.2 μL,10.0 μL 2 × iQTM SYBR Green Supermix(Bio-Rad, 美国)和8.6 μL ddH2O;反应条件:95 ℃ 2 min;95 ℃ 15 s,60 ℃ 30 s,采集荧光40次,然后进行溶解曲线的扩增。该实验中,生物学重复n=3,技术重复2次,目的基因和内参基因的扩增效率(E)均为95%~100%,R2均大于0.9。
cyclin B基因相对表达量数据运用2-ΔΔCT法进行计算。数值采用平均值±标准误差(Mean±SE)表示。不同样品间的相对表达差异分析利用SPSS 19.0软件中的One-Way方差分析(ANOVA), 使用Dunnett’s multiple comparisons进行组间多重比较,P < 0.05代表差异显著。定量结果用Origin 2017软件进行作图分析。
1.2.5 细胞周期测定将上述RNAi后的性腺、鳃及外套膜细胞固定于70%乙醇中,在-20 ℃冰箱中固定3 h以上。取1 mL已固定好的细胞悬液于1.5 mL无菌离心管中,室温300 g离心5 min,去上清;加入500 μL PBS缓冲液重新使细胞悬浮,室温300 g离心5 min,去上清;将细胞重新悬浮于500 μL PI/RNase染液(BD,美国),37 ℃避光孵育15~20 min后,用Accuri C6 PLUS流式细胞仪(BD,美国)检测细胞的细胞周期时相,用novo expression软件分析结果。
2 结果与分析 2.1 三角帆蚌cyclin B基因的克隆及序列分析通过克隆得到三角帆蚌cyclin B基因的cDNA长度为1 024 bp(图 1), 包括ORF 768 bp,3′-UTR 197 bp,编码255个氨基酸残基。核苷酸序列的3′-UTR中发现典型的多聚赖氨酸信号“AATAAA”和末尾的Poly A重复序列。对cyclin B蛋白保守结构域(图 2)分析,发现有一段含90个氨基酸残基的保守序列,即周期蛋白框(cyclin-box),在其框内存在两个周期蛋白依赖性蛋白激酶(CDK)结合位点。ExPASy分析三角帆蚌cyclin B基因的分子量为29.1 ku,理论等电点为8.84。

|
黑色下划线表示起始密码子和终止密码子;黑色双下划线表示poly A;黑色框内“AATAAA”为多聚腺苷酸信号;灰色阴影表示转录组库中cyclin B基因序列 Black underlines indicated start codon and stop codon; Black double underlines indicated poly A; The "AATAAA" in black frame is the polyadenosine plus tail signal; Gray shading indicates the cyclin B gene sequence in the transcriptome 图 1 三角帆蚌cyclin B基因的cDNA序列及预测的氨基酸序列 Fig. 1 cDNA sequence and predicted amino acid sequence of cyclin B in Hyriopsis cumingii |

|
灰色三角形为cyclin B与CDK结合的氨基酸残基;大括号表示cyclin B与CDK的结合位点;黑色方框内为cyclin B的周期蛋白框 The gray triangle is the amino acid residue of cyclin B binding to CDK; The braces indicate the binding site of cyclin B and CDK; The black box is the cyclicn-box of cyclin B 图 2 三角帆蚌cyclin B保守结构域 Fig. 2 Conserved domain of cyclin B in Hyriopsis cumingii |
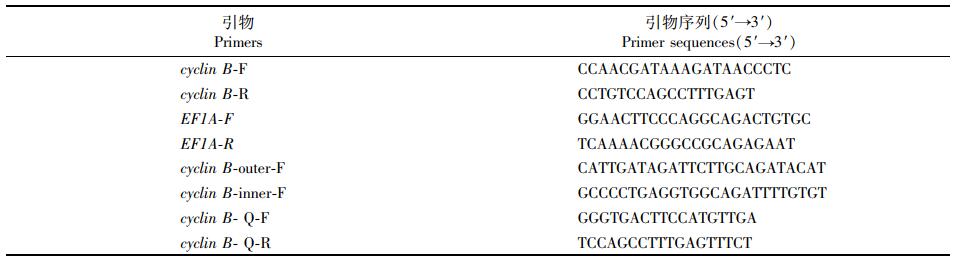
三角帆蚌与其他物种cyclin B基因序列相似性比对结果(图 3a)表明,其与牡蛎聚在一支,其次与扇贝、贻贝具有较高的序列相似性,cyclin B基因在贝类中具有保守性。通过多物种的cyclin B基因氨基酸序列分析显示(图 3b),三角帆蚌与斑马贻贝(Dreissena polymorphia)有73%的同源性,且均与牡蛎和扇贝有60%以上的同源性。通过String网站预测三角帆蚌cyclin B靶蛋白的结果表明,cyclin B蛋白与CDK1和CDK2有靶向关系。
|
Hyriopsis cumingii:三角帆蚌;Crassostrea virginic:美洲牡蛎[XM022479068.1];Crassostrea giga:太平洋牡蛎[XM020067773.1];Danio rerio:斑马鱼[NM131513.1];Mizuhopecten yessoensis:虾夷扇贝[XM021487977.1];Dreissena polymorph:斑马贻贝[AF086634.1];Xenopus laevis:非洲爪蟾[NM001089899.1];Penaeus monodo:斑节对虾[EU707332.1];Acanthaster planci:长棘海星[XM022232970.1] 图 3 三角帆蚌与其他物种cyclin B序列相似性比对 Fig. 3 cyclin B sequence similarity alignment between Hyriopsis cumingii and other species |
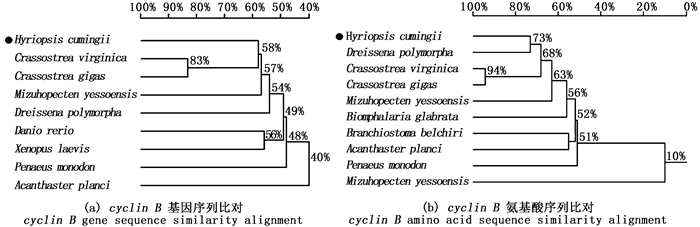
由图 4可知:三角帆蚌cyclin B基因mRNA在不同性别的闭壳肌、斧足、外套膜、心脏、血液、内脏团、鳃和性腺中均有表达。三角帆蚌cyclin B基因在其性腺中高表达,且在雌性个体中的表达量显著高于雄性个体(P < 0.05),cyclin B基因在闭壳肌、斧足、外套膜、心脏、血液、内脏团和鳃组织中的表达量无明显的雌雄差异,且各组织间也无显著性表达差异。
|
不同字母代表显著性差异(P < 0.05) Different letters represent significant differences(P < 0.05) 图 4 cyclin B基因在不同性别三角帆蚌不同组织中的表达 Fig. 4 Expression levels of cyclin B of different tissues in male and female Hyriopsis cumingii |
在RNAi后,cyclin B干扰链注射三角帆蚌48 h后,定量结果(图 5)表明:cyclin B基因在性腺和鳃中的表达量在正常组和NC组无显著差异(P>0.05),但在性腺的干扰组中cyclin B基因的表达水平显著下降(P < 0.05),为正常水平的39%,在鳃的干扰组中cyclin B基因的水平显著下降(P < 0.05),为正常水平的26%,而在干扰后的外套膜组织中,cyclin B基因的表达量与正常组无显著性差异(P>0.05)。

|
*代表显著性差异(P < 0.05) *Represents significant differences(P < 0.05) 图 5 RNA干扰后三角帆蚌性腺、鳃、外套膜中cyclin B的表达 Fig. 5 Expression levels of cyclin B gene of gonad, gill and mantle in Hyriopsis cumingii after RNA interfered |
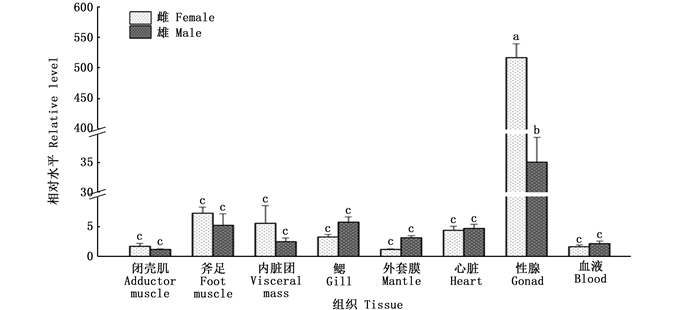
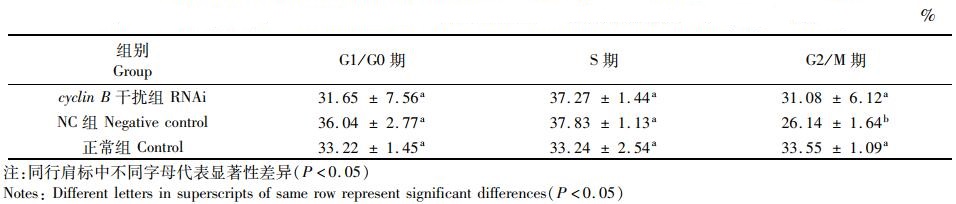
根据细胞分裂过程中DNA变化情况,可人为地将细胞周期划分为第一个间隔期(G1期)、DNA合成期(S期)、第二个间隔期(G2期)和分裂期(M期)4个阶段,有些细胞会暂时脱离细胞周期而停止分裂,被称为G0期细胞。对于性腺细胞(表 3)而言,在cyclin B干扰组、NC组及正常组中均呈现S期细胞的比例显著高于G1/G0期和G2/M期细胞的比例(P < 0.05),针对G2/M期细胞占比而言,cyclin B干扰组显著低于正常组(P < 0.05);对于鳃细胞(表 4)而言,NC组各时相细胞占比无显著性差异(P>0.05), cyclin B干扰组及正常组中均呈现S期细胞的比例显著高于G1/G0期和G2/M期细胞的比例(P < 0.05),针对G2/M期细胞占比而言,在cyclin B干扰组中其比例显著低于G1/G0期(P < 0.05),而在正常组中其比例显著高于G1/G0期(P < 0.05);对于外套膜细胞(表 5),cyclin B干扰组和正常组细胞时相分布趋势具有一致性,G1/G0、S及G2/M期细胞占比无显著性差异(P>0.05),针对G2/M期细胞占比而言, cyclin B干扰组及正常组显著性水平一致,均显著高于NC组(P < 0.05)。
|
表 3 RNA干扰后三角帆蚌性腺细胞的细胞周期时相分布 Tab.3 Cell cycle phase distribution of gonad cells after RNA interfered in Hyriopsis cumingii |

|
表 4 RNA干扰后三角帆蚌鳃细胞的细胞周期时相分布 Tab.4 Cell cycle phase distribution of gill cells after RNA interfered in Hyriopsis cumingii |
|
表 5 RNA干扰后三角帆蚌外套膜细胞的细胞周期时相分布 Tab.5 Cell cycle phase distribution of mantle cells after RNA interfered in Hyriopsis cumingii |
本研究首次通过克隆得到三角帆蚌cyclin B基因的cDNA长度为1 024 bp, 包括768 bp的ORF,197 bp的3′-UTR,编码255个氨基酸残基。克隆结果显示cyclin B基因的3′-UTR区有两个多聚赖氨酸信号,多聚赖氨酸尾主要控制mRNA的稳定性和调节mRNA的翻译效率,根据三角帆蚌cyclin B基因的克隆结果显示,其3′-UTR区有两个多聚赖氨酸加尾信号,这与吴萍[9]在日本沼虾卵巢中克隆得到cyclin B基因的3′-UTR区的结构相似,表明这是cyclin B作为M期周期蛋白在细胞周期中相对稳定的原因。自1983年首次发现细胞周期蛋白后,许多科学家对细胞周期蛋白开展了研究,目前从高等动物中克隆分离的cyclin就超过18种,在人体中也发现了25种周期蛋白,对水生生物中细胞周期蛋白的克隆研究也日渐成熟,目前已从大黄鱼(Larimichthys crocea)[10]、拟穴青蟹(Scylla paramamosain)[11]、长牡蛎(Crassostrea gigas)[12]等中克隆得到cyclin B基因。不同cyclin之间具有共同的分子结构,具体表现在其氨基酸序列中有一段含100~150个氨基酸残基的保守序列,即周期蛋白框(cyclin-box)[13],不同的周期蛋白框可以识别不同的CDK,形成cyclin-CDK复合体,从而调控细胞周期运转。在本研究中推导的三角帆蚌cyclin B的氨基酸序列中找到了周期蛋白框,并且发现其中含有2个与CDK结合的作用位点,通过String数据库的分析,预测所结合的CDK为CDK1和CDK2。1个标准的细胞周期含有2个时间间隔(G1期和G2期)、DNA合成期(S期)及分裂期(M期),cyclin B-CDK1的复合物在G2/M期执行其功能,使细胞由G2期向M期转化[14],诱导细胞分裂。
cyclin B是细胞周期周转中重要的调控因子,其积累与表达在细胞分裂过程中呈周期性变化,cyclin B一般在S期开始表达和积累,但到M期才表现出调节功能,主要在G2/M交界期诱导细胞分裂,所以被称为M期周期蛋白[15-16]。cyclin B在mRNA及蛋白水平上均存在细胞周期调控机制,MARKIEWICZ等[17]通过电离辐射诱导大鼠胚胎细胞,发现诱导细胞G2期延迟与cyclin B基因mRNA水平降低有关。目前在水生生物中对cyclin B基因的研究多与性腺发育相关。本研究通过构建的不同性别三角帆蚌组织表达谱发现,cyclin B基因在性腺中的表达量显著高于其他各组织,并且均在雌性个体中的表达量显著高于雄性个体,而在外套膜中的表达量均远远低于性腺且无显著的雌雄表达差异。LOHKA等[18]首次从非洲爪蟾(Xenopus laevis)成熟的卵细胞中分离出了能诱导卵母细胞成熟的成熟促进因子(MPF),之后被证明在细胞周期调控中起到重要作用,cyclin B是MPF的主要成分,在配子发生过程中起到重要作用[19],有研究[20-22]通过原位杂交和免疫印迹发现,cyclin B在早期精子细胞核周围和正在生长的卵泡中表达。性腺作为细胞分裂旺盛的部位,cyclin B基因在性腺中的高表达与他们在生殖细胞减数分裂中重要的调控作用具有一致性。外套膜是三角帆蚌珍珠形成的重要组织,本研究结果表明与体细胞有丝分裂相关的cyclin B基因在外套膜中低表达,说明三角帆蚌外套膜不是细胞分裂旺盛的部位[23],这可能是其细胞在体外培养分裂速度迟缓的原因。本实验cyclin B基因在三角帆蚌中各组织的表达差异情况与王桐[12]在长牡蛎中构建cyclin B基因组织表达谱的结果一致,并且其研究中发现cyclin B基因的表达水平随着长牡蛎性腺发育程度的提高而提高,本实验的样品均采集于性发育成熟的三角帆蚌,表明这也是cyclin B基因在三角帆蚌性腺中高表达的原因之一。
RNA干扰(RNAi)技术是目的基因序列同源的dsRNA被其特异性核酸酶裂解成siRNA后,由siRNA作为介导子引起特异性地降解相同序列的mRNA,从而阻断相应基因的转录后沉默机制[24]。近些年来,在水生动物中RNAi技术逐渐发展成熟,在贝类基因功能的研究中也被广泛应用,FANG等[25]应用RNAi技术发现了敲降牡蛎外套膜PFN23基因,结果导致贝壳珍珠层结构发生紊乱;刘晓丽[26]研究表明马氏珠母贝(Pinctada martensii)外套膜几丁质结合蛋白进行RNAi,导致其珍珠层晶体排列疏松且不规则等,由此证明贝类中存在着RNAi的机制。
本研究分别对三角帆蚌性腺、鳃及外套膜中cyclin B基因进行沉默,荧光定量结果表明:注射cyclin B干扰片段的三角帆蚌性腺及鳃中的cyclin B基因沉默,而外套膜中的cyclin B基因表达水平并未下降,可见RNAi对三角帆蚌不同组织的沉默效果不同。三角帆蚌外套膜是珍珠形成的重要部位,可分为前端缘膜、中央膜和后端缘膜,后缘膜被认为是人工有核珍珠培育的最适插核部位[27],该部位的细胞表现出较好的自我修复能力。对三角帆蚌某些基因与贝类矿化的研究中,外套膜的3个部位因其表现出不同的生物学功能,常被分别做为研究对象[28],王芹等[29]在研究三角帆蚌CA3基因对贝类矿化作用的影响时,运用RNAi技术分别对三角帆蚌外套膜前、后端缘膜及中央膜注射了干扰片段,发现该干扰片段在外套膜不同部位表现出不同的干扰效果。本研究为探讨外套膜细胞体外分裂能力与cyclin B基因之间的关系,样品取自细胞自我修复能力较好的后缘膜,表明本实验设计的cyclin B的siRNA片段对外套膜后缘膜无干扰效应,而对前端缘膜和中央膜的干扰效果需要进一步研究。三角帆蚌注射cyclin B干扰片段后其性腺、鳃和外套膜细胞的细胞时相变化结果显示,干扰后性腺和鳃细胞中G2/M期细胞占比显著底于正常组,而外套膜细胞G2/M期的细胞占比在干扰前后无显著性变化,该细胞周期变化情况与定量结果一致,表明在三角帆蚌中cyclin B基因参与调控G2/M期,其mRNA水平下降,使cyclin B积累降低到不能激活CDK活性的程度,从而使G2期延长,处于分裂状态的细胞数量减少。
本研究为进一步研究三角帆蚌生物学功能提供分子基础,为三角帆蚌细胞体外培养分裂机制及建立细胞系提供理论支持,也为优化育珠工艺、推动我国珍珠产业蓬勃发展奠定了科研基础。
| [1] |
石安静. 河蚌外套膜的组织培养[J]. 水产学报, 1983, 7(2): 153-157. SHI A J. The tissue culture of the mantle of freshwater clams[J]. Journal of Fisheries of China, 1983, 7(2): 153-157. |
| [2] |
石安静. 人工育珠河蚌钩介幼虫的原代细胞培养[J]. 四川大学学报, 1983(4): 88-91. SHI A J. Primary cell culture of freshwater shdll glochidium[J]. Journal of Sichuan University, 1983(4): 88-91. |
| [3] |
李倩, 施志仪, 李文娟, 等. 三角帆蚌外套膜细胞体外培养优化及体内植入培养对细胞生长的影响[J]. 中国水产科学, 2014, 21(2): 225-234. LI Q, SHI Z Y, LI W J, et al. Impact of in vitro optimization and in vivo implantation culture on the growth of Hyriopsis cumingii mantle cells[J]. Journal of Fishery Sciences of China, 2014, 21(2): 225-234. |
| [4] |
翟中和, 王喜忠, 丁明孝. 细胞生物学[M]. 北京: 高等教育出版社, 2000. ZHAI Z H, WANG X Z, DING M X. Cell biology[M]. Beijing: Higher Education Press, 2000. |
| [5] |
HARTWELL L H, KASTAN M B. Cell cycle control and cancer[J]. Science, 1994, 266(5192): 1821-1828. DOI:10.1126/science.7997877 |
| [6] |
FANG J J, QIU G F. Molecular cloning of cyclin B transcript with an unusually long 3' untranslation region and its expression analysis during oogenesis in the Chinese mitten crab, Eriocheir sinensis[J]. Molecular Biology Reports, 2009, 36(6): 1521-1529. DOI:10.1007/s11033-008-9346-9 |
| [7] |
VISUDTIPHOLE V, KLINBUNGA S, KIRTIKARA K. Molecular characterization and expression profiles of cyclin A and cyclin B during ovarian development of the giant tiger shrimp Penaeus monodon[J]. Comparative Biochemistry and Physiology Part A:Molecular & Integrative Physiology, 2009, 152(4): 535-543. |
| [8] |
XU H, HEUVEL S V D, BENNETT K L, et al. The cyclin B and B3 genes have distinct functions during cell division in C. elegans[J]. East Coast Worm Meeting, 2000. |
| [9] |
吴萍.日本沼虾卵巢表达序列标签分析及生殖相关基因的克隆与表达[D].上海: 华东师范大学, 2009: 4. WU P. Analysis of Ovary ESTs and cDNA cloning and expression profile of reproduction-related genes from Macrobrachium nipponense[D].Shanghai: Esast China Normal University, 2009: 4. |
| [10] |
蔡明夷, 周鹏, 韩坤煌, 等. 大黄鱼cyclin B1和cdc2 cDNA序列特征及组织表达分析[J]. 厦门大学学报(自然科学版), 2014, 53(1): 132-141. CAI M Y, ZHOU P, HAN K H, et al. Characterization and tissue expression profiles of cyclin B1 and cdc2 in Large Yellow Croaker (Larimichthys crocea)[J]. Journal of Xiamen University (Natural Science), 2014, 53(1): 132-141. |
| [11] |
虞晋晋.三种虾蟹Cyclin B的定位和分子克隆[D].厦门: 厦门大学, 2008: 10. YU J J. Localization and clone of Cyclin B from three kinds of crab and shrimps[D]. Xiamen: Xiamen University, 2008: 10. |
| [12] |
王桐.长牡蛎(Crassostrea gigas)cdk1和cyclin B3基因的克隆及其在性腺发育中作用的初步研究[D].北京: 中国科学院研究生院(海洋研究所), 2011: 4. WANG T. Cloning of cdk1 and cyclin B3 genes from Crassostrea gigas and its preliminary role in gonadal development[D]. Beijing: Graduate School of the Chinese Academy of Sciences (Ocean Institute), 2011: 4. |
| [13] |
NOBLE M E M, ENDICOTT J A, BROWN N R, et al. The cyclin box fold:protein recognition in cell-cycle and transcription control[J]. Trends in Biochemical Sciences, 1997, 22(12): 482-487. DOI:10.1016/S0968-0004(97)01144-4 |
| [14] |
RICHARDSON H, LEW D J, HENZE M, et al. Cyclin-B homologs in Saccharomyces cerevisiae function in S phase and in G2[J]. Genes & Development, 1992, 6(11): 2021-2034. |
| [15] |
LE BRETON M, BELLÉR, CORMIER P, et al. M-phase regulation of the recruitment of mRNAs onto polysomes using the Cdk1/cyclin B inhibitor aminopurvalanol[J]. Biochemical and Biophysical Research Communications, 2003, 306(4): 880-886. DOI:10.1016/S0006-291X(03)01083-0 |
| [16] |
BORGNE A, OSTVOLD A C, FLAMENT S, et al. Intra-M phase-promoting factor phosphorylation of cyclin B at the prophase/metaphase transition[J]. The Journal of Biological Chemistry, 1999, 274(17): 11977-11986. DOI:10.1074/jbc.274.17.11977 |
| [17] |
MARKIEWICZ D A, MCKENNA W G, FLICK M B, et al. The effects of radiation on the expression of a newly cloned and characterized rat cyclin B mRNA[J]. International Journal of Radiation Oncology, Biology, Physics, 1994, 28(1): 135-144. DOI:10.1016/0360-3016(94)90151-1 |
| [18] |
LOHKA M J, HAYES M K, MALLER J L. Purification of maturation-promoting factor, an intracellular regulator of early mitotic events[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(9): 3009-3013. DOI:10.1073/pnas.85.9.3009 |
| [19] |
WACHIRACHAIKARN A, RUNGSIN W, SRISAPOOME P, et al. Molecular characterization and expression analysis of Cyclin B and Cell division cycle 2 in gonads of diploid and triploid bighead catfish, Clarias macrocephalus Günther, 1864[J]. Agriculture and Natural Resources, 2017, 51(2): 117-130. DOI:10.1016/j.anres.2016.05.004 |
| [20] |
刁红录, 徐立滨, 杨增明. 细胞周期蛋白和哺乳动物生殖[J]. 细胞生物学杂志, 2002, 24(5): 286-289. DIAO H L, XU L B, YANG Z M. Cyclin and mammalian reproduction[J]. Chinese Journal of Cell Biology, 2002, 24(5): 286-289. |
| [21] |
ABRIEU A, DOREE M, FISHER D. The interplay between cyclin-B-Cdc2 kinase (MPF) and MAP kinase during maturation of oocytes[J]. Journal of Cell Science, 2001, 114(2): 257-267. |
| [22] |
GAUTIER J, MALLER J L. Cyclin B in Xenopus oocytes:implications for the mechanism of pre-MPF activation[J]. The EMBO Journal, 1991, 10(1): 177-182. DOI:10.1002/j.1460-2075.1991.tb07934.x |
| [23] |
谢先中, 施志仪, 王永树. 三角帆蚌内脏团大颗粒有核珍珠培育技术的研究[J]. 水产学杂志, 2015, 28(1): 45-48. XIE X Z, SHI Z Y, WANG Y S. Culture of large nucleated pearl in visceral mass of freshwater pearl mussel Hyriopsis cumingii (Lea)[J]. Chinese Journal of Fisheries, 2015, 28(1): 45-48. DOI:10.3969/j.issn.1005-3832.2015.01.009 |
| [24] |
TIJSTERMAN M, PLASTERK R H A. Dicers at RISC:the mechanism of RNAi[J]. Cell, 2004, 117(1): 1-3. |
| [25] |
FANG D, PAN C, LIN H J, et al. Novel basic protein, PfN23, functions as key macromolecule during nacre formation[J]. The Journal of Biological Chemistry, 2012, 287(19): 15776-15785. DOI:10.1074/jbc.M112.341594 |
| [26] |
刘晓丽.马氏珠母贝外套膜不同区域差异矿化基因的克隆及功能研究[D].湛江: 广东海洋大学, 2015: 12. LIU X L. Cloning and functional studies of new genes related to mineralization in different regions of mantle from Pinctada martensii[D]. Zhanjiang: Guangdong Ocean University, 2015: 12. |
| [27] |
李庆乐. 关于提高细胞片活力和三角帆蚌外套膜不同部位培育有核珍珠的对比试验[J]. 广西农业科学, 1990(3): 46-49. LI Q L. Comparative on improving the viability of cell sheets and the cultivation of nuclear pearls in different parts of the mantle of Hyriopsis cumingii[J]. Guangxi Agricultural Sciences, 1990(3): 46-49. |
| [28] |
董绍建.三角帆蚌贝壳基质蛋白基因silkmapin的生物矿化功能研究[D].上海: 上海海洋大学, 2015: 6. DONG S J. Studies on biomineralization of shell matrix protein-silkmapin of Hyriopsis cumingii[D]. Shanghai: Shanghai Ocean University, 2015: 6. |
| [29] |
王芹, 汪桂玲, 陈亚, 等. RNA干扰沉默三角帆蚌HcCA3基因对贝类矿化作用的影响[J]. 基因组学与应用生物学, 2018, 37(2): 741-747. WANG Q, WANG G L, CHEN Y, et al. Effect of silencing HcCA3 Gene in Hyriopsis cumingii by RNA interference on mineralization of shellfish[J]. Genomics and Applied Biology, 2018, 37(2): 741-747. |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. Shanghai Collaborative Innovation for Aquatic Animal Genetics and Breeding, Shanghai Ocean University, Shanghai 201306, China
 2020,
Vol. 29
2020,
Vol. 29


