2. 中国水产科学研究院淡水渔业研究中心 农业农村部淡水渔业与种质资源利用重点实验室, 江苏 无锡 214081
罗非鱼是联合国粮组织向全世界推广淡水养殖的主要对象,也是中国优先发展的淡水养殖优势品种[1]。2018年全球养殖罗非鱼产量超过630万t[2]。虽然近年来罗非鱼的产量一直在增加,但其产业发展也面临着诸多挑战和问题。其中,野生品种体色暗淡并不太受消费者喜欢,选择生长快、肉质好的罗非鱼新品种,提高肉质是解决罗非鱼产业发展瓶颈的有效措施之一。红罗非鱼作为一种优良的杂交品种,存在多个品种或品系。目前,红罗非鱼是由突变的红橙色莫桑比克罗非鱼(Oreochromis mossambicus)和其他罗非鱼杂交而来,经多代选育而成的优良品种[3]。它具有生长快、适口性好、易于繁殖等生产优点,因其肉色轻、蛋白质含量高,深受消费者青睐[4]。随着我国市场需求的不断增长,从国外引进罗非鱼种群也越来越多。然而,引进后的养殖场经营者存在不重视遗传监测和缺乏科学管理等问题,在长期的繁育过程中存在基础群体减少、近交衰退等问题,导致国内罗非鱼群体出现种质退化现象,并表现出生长速度下降、体色混杂、性早熟等现象[5]。过去几十年,基于各种遗传标记手段,对中国的罗非鱼群体开展了较多的遗传分析。如:颜标等[6]利用10个微卫星标记对尼罗罗非鱼(O. niloticus)、萨罗罗非鱼(Sarotherodon melanotheron)及其正反杂交F1的遗传结构进行了研究;陈增祥等[7]基于线粒体D-loop序列对吉富罗非鱼(GIFI strain O.niloticus)遗传变异进行分析;杨洁等[8]利用线粒体DNA控制区部分序列对中国8个尼罗罗非鱼养殖群体(埃及、吉拉达、美国、鹭业、吉诺玛、宝路、广东、新吉富)的遗传多样性和相互间遗传关系进行了分析。然而,考虑到罗非鱼繁殖周期短,有些品种在中国已经历多代繁养,对目前遗传多样性水平缺乏认识;再加上近年来也有新的红罗非鱼种质资源引入,对其遗传背景分析开展较少。因此对国内罗非鱼群体的遗传变异研究,可为国内罗非鱼种群资源保护和进一步选育利用提供一定的理论基础。
研究罗非鱼遗传多样性的方法主要有形态学差异和分子标记等。对比形态学的观察和描述,DNA分子标记能更准确、快速地鉴别群体[9-10]。COI基因作为DNA分子标记中物种鉴定的有效工具,具有结构简单、进化速度适中、母系遗传等特点,且大多数鱼类的COI基因能被通用引物所扩增,可用于分析群体的遗传多样性和推断其系统发育关系[11-12]。到目前为止,COI基因已经广泛应用于锦鲤(Cyprinus carpio koi)[13]、鲷属(Sparus)[14]等鱼类群体研究。关于中国罗非鱼群体遗传分析的研究也已有报道[15],但并不充分。本研究利用mtDNA COI对中国养殖的7个罗非鱼群体进行遗传变异分析,为罗非鱼遗传资源的保护、利用和开发提供理论依据。
1 材料与方法 1.1 样本的来源和采集共采集7个群体的罗非鱼标本336尾,其中包括红罗非鱼3个品系[中国台湾红罗非鱼(TW)、马来西亚红罗非鱼(MY)、以色列红罗非鱼(IL)]和其他4个罗非鱼群体[尼罗罗非鱼(NL)、莫桑比克罗非鱼(MS)、奥利亚罗非鱼(O. aureus, AR)、吉富罗非鱼(GIFT)]。马来西亚红罗非鱼是2009年淡水渔业研究中心从马来西亚引进的,一直在宜兴实验基地繁殖养殖;中国台湾红罗非和以色列红罗非是2013年从福建引进的,在当地已经进行了多年的养殖驯化,并保留较明显的区域品种特征;莫桑比克罗非鱼是从中国水产科学研究院珠江所实验基地采集的;其他罗非鱼均来自宜兴实验基地,为保种或选育群体。每个群体各采样48尾,将采集的尾鳍保存在装有无水乙醇的冻存管中,直到DNA提取。
1.2 方法 1.2.1 DNA的提取用消毒的剪刀剪取罗非鱼个体尾鳍下叶一小块放入灭过菌的2 mL离心管中,采用苯酚/氯仿法提取DNA,用1%琼脂糖凝胶电泳检测其完整性,经NanoDrop分光光度仪检测其纯度及浓度。每个样本提取的DNA用灭菌的ddH2O稀释到最终浓度20 ng/μL,4 ℃保存备用。
1.2.2 COI基因片段的扩增和测序在NCBI上找到红罗非鱼的mtDNA全序列(序列号:NC_014060),将1 596 bp的COI序列分为2段,使用Primer premier 6.0软件进行引物设计(表 1),并送生工生物工程(上海)股份有限公司合成。PCR反应混合体系总共50 μL,包括5 μL 10×PCR Buffer、3 μL MgCl2(0.25 mmol/L)、4 μL dNTPs(2.5 mmol/L)、1 μL Taq polymerase(2.5 U/μL)、1 μL上、下游引物(10 μmol/L)、2 μL DNA模板(20 ng/μL)、33 μL无酶去离子水。PCR扩增反应在Eppendorf Mastercycler Pro 384 PCR热循环仪(Eppendorf,Germany)上进行。反应条件如下:94 ℃预变性2 min,94 ℃变性40 s,55 ℃退火55 s,72 ℃延伸1 min,35个循环,最后在72 ℃延伸10 min。将PCR产物用1%琼脂糖凝胶电泳检测,紫外灯下观察电泳结果,将确定的PCR扩增产物送上海美吉生物有限公司进行双向测序。

|
表 1 COI序列引物表 Tab.1 Primers of COI sequence |
所有的序列使用BioEdit version 7.0.9软件[16]进行编辑分析。为了保证准确性,所有的DNA序列都进行正反2个方向测序,手工拼接COI基因2段重叠区域,去除因测序误差造成的错误碱基。剪接完成后,用DnaSP 5.0软件[17]计算出多态性位点(S)、单倍型(h)、单体型多样性(Hd)、核苷酸多样性(π)、平均核苷酸差异(k)、Tajima’s D值和其他遗传多样性指数。使用MEGA 5软件[18-19]计算群体间的K2P遗传距离,并利用UPGMA法绘制群体间遗传聚类树。采用Arlequin 3.5软件[20]计算核苷酸组成、群体间遗传分化指数(F-statistics,Fst)和分子方差分析(AMOVA)。
2 结果 2.1 7个群体罗非鱼COI序列的变异位点与单倍型分布对采集的样品进行测序,共扩增出罗非鱼COI序列336条(1 596 bp)。经过同源比对和测序,剔除了误差较大的序列,最终获得了324条同源序列。核苷酸组成显示,A、T、C、G碱基的平均含量分别为28.6%、28.7%、25.2%、17.5%,且A+T(57.3%)的含量明显高于G+C(42.7%),与其他鱼类的COI基因序列研究结果一致。
在324条同源序列中共检测到98个单倍型(GenBank收录号:MH515186-MH515294)。其中,群体间的共享单倍型和特有单倍型分别如表 2和表 3所示,在7个罗非鱼群体中检测到不同数量的单倍型(5~29)。其中,2个共享单倍型由MS和TW群体(Hap_53、Hap_56)组成,4个共享单倍型由MY、TW、IL群体(Hap_21、Hap_27、Hap_28、Hap_37)组成,1个共享单倍型由NL和GIFT群体组成(Hap_10),其余单倍型为各群体所特有。在98个单倍型中存在6个优势单倍型(Hap_10、Hap_21、Hap_27、Hap_42、Hap_53和Hap_56),分别占个体数的34.70%、35.70%、22.40%、42.80%、24.40%和20.40%。
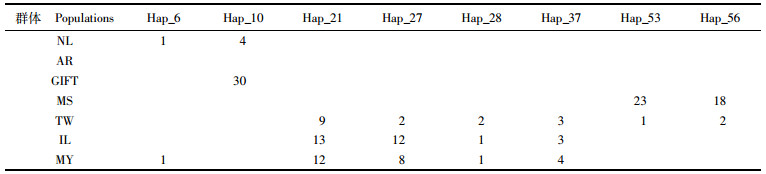
|
表 2 7个罗非鱼群体中CO Ⅰ基因共享单倍型分布情况 Tab.2 Distribution of the shared haplotypes in 7 tilapia populations |

|
表 3 7个罗非鱼群体中CO Ⅰ基因特有单倍型分布情况 Tab.3 Distribution of the unique haplotypes in 7 tilapia populations |
7个罗非鱼群体整体单倍型多样性为0.944(表 4)。各群体的单倍型多样性介于0.234~0.952,核苷酸多样性介于0.000~0.036,基于COI序列的7个罗非鱼群体的遗传多样性比较,AR罗非鱼遗传多样性水平最低(Hd=0.234,π=0.000),TW罗非鱼具有最多的单倍型(H=29),NL具有最大的平均核苷酸差异(k=56.84)。中性检验显示,AR、GIFT、MS罗非鱼的Tajima’s D值均为负值,其余群体均为正值,其中NL群体达到显著水平(P < 0.05),GIFT、MY群体达到极显著水平(P < 0.01)。
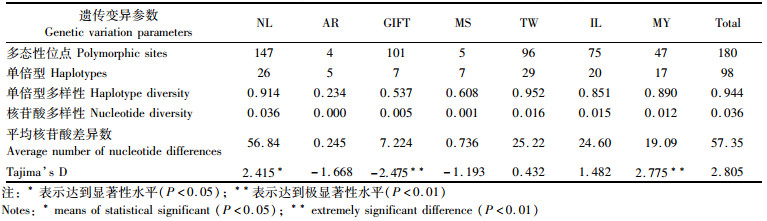
|
表 4 罗非鱼7个群体COI基因的遗传变异参数 Tab.4 Genetic variation parameters of COI gene of seven tilapia populations |
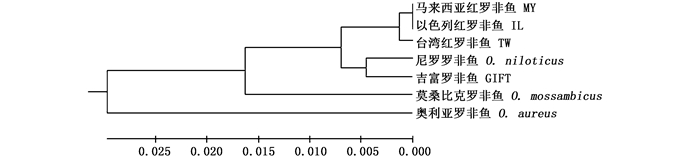
利用K2P模型计算群体间遗传距离的范围为0.000~0.071(表 5,下对角线)。其中,AR和MS的遗传距离最远(D = 0.071),IL和MY的遗传距离最近(D = 0.000)。基于K2P遗传距离构建UPGMA聚类关系(图 1)显示:7个罗非鱼群体聚为2个分支,第一个分支首先由MY,IL和TW这3种红罗非鱼群体聚类,然后与NL和GIFT群体聚类,最后和MS群体聚类;第2个分支为AR一个群体。
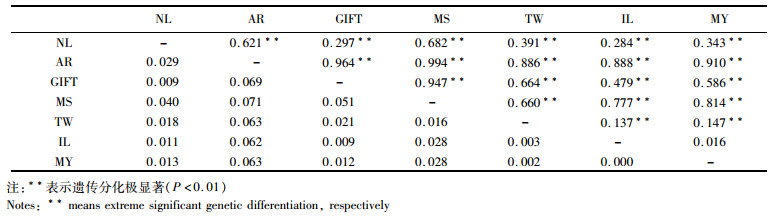
|
表 5 罗非鱼群体间的K2P遗传距离(左下角)和遗传分化指数Fst(右上角) Tab.5 Pairwise K2P genetic distances (below diagonal) and fixation indexes (Fst, above diagonal) among tilapia populations using COI |
7个罗非鱼群体的分子方差分析(AMOVA)显示,在不分组的情况下,群体间的方差组分占70.780%,遗传分化指数为0.708,达到了极显著的水平(P < 0.01,见表 6)。将罗非鱼群体分成2组:红罗非鱼和野生或者选育群体,群体遗传分化指数为0.717,也达到极显著的水平(P < 0.01)。这一结果和未分组的结果显示相同,进一步说明7个罗非鱼群体间存在明显的遗传分化。群体间遗传分化指数F-statistics统计值(Fst)分析显示(表 5,右上角),除了MY和IL群体,其他群体的遗传分化均到达了极显著水平(P < 0.01)。
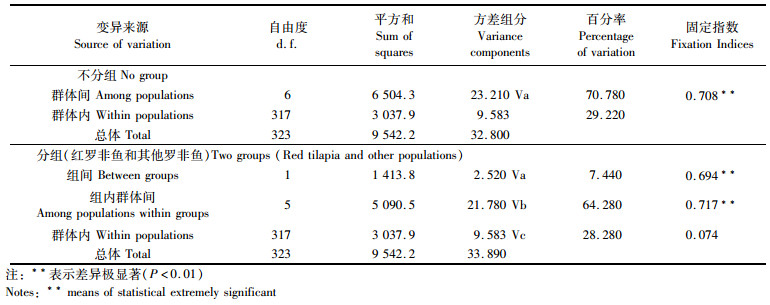
|
表 6 7个罗非鱼群体COI序列变异的分子方差分析(AMOVA) Tab.6 Analysis of molecular variances of COI gene of seven tilapia populations |
该研究对罗非鱼群体的COI基因序列的分析结果显示4种核苷酸的不均匀分布,属动物线粒体基因组的共同特征[21]。在7个罗非鱼群体中共检测到180个多态性位点和98个单倍型,说明COI基因在该群体样本中具有较高多态性,适用于系统发育进化和遗传多样性等分析。研究发现,序列的多态位点和单倍型高于ORDONEZ等[22]在罗非鱼群体中的研究结果,推测这与本研究所用基因序列更长、涉及样本量更大和所用群体更多等有关,当然也说明该研究所述7个罗非鱼群体具有较高的整体遗传多样性水平。
|
图 1 基于COI基因7个罗非鱼群体的UPGMA树 Fig. 1 UPGMA tree based on COI gene of seven tilapia populations |
该研究发现,GIFT和MS群体具有较高单倍型多样性(Hd>0.5)和较低核苷酸多样性(π < 0.005),在其他报道,如斑重唇鱼(Diptychus maculatus)[23]、草鱼(Ctenopharyngodon idella)[24]和真鲷(Pagrosomus major)[25]中也有类似结果。推测这种情况可能是经历瓶颈效应后群体数量的再次快速增长和突变积累,且单倍型多样性比核苷酸多样性易于在短时间内积累[26]。NL和红罗非鱼群体(TW、IL、MY)具有较高的遗传多样性,与杨慧等[27]利用SSR的分析结果相符。这可能是在生产或选育过程中维持了较大的繁育群体,导致其遗传多样性保持较高水平。研究中检测到AR群体遗传多样性最低(Hd < 0.5,π < 0.005),这一结果与夏德全等[28]和王进科等[29]分别利用RAPD和SSR检测结果相似。分析其主要原因是AR群体引入中国后,在生产或选育过程中应用基础群体较小,并经历多代选育留种后,使其种群遗传多样性下降。该研究表明,NL和3种红罗非鱼群体具有一定的进一步选育潜力,而AR群体的遗传多样性相对较低,但维持了更高的种质纯度,可以考虑更新引进优良种质,或者基于当前种质材料开展杂交优势利用。
对7个罗非鱼群体的遗传距离分析显示AR群体和MS群体之间的遗传距离最大为0.071。此外,IL与MY(D=0.000)之间的遗传距离最小,说明其母体遗传来源可能是一致的。对于红罗非鱼,TW、IL和MY之间的遗传距离远低于不同种群的分类基础(D=0.050)[30],说明红罗非鱼群体之间的遗传分化较弱,这在亲缘关系较近的群体中较为典型。NJ树显示罗非鱼的群体结构被分为2支,其中1个分枝是除AR群体外的其他6个群体,表明这6个群体可能是从一个共同的原始祖先进化而来。
对红罗非鱼群体与其他罗非鱼群体的分组和不分组分析显示,所述群体存在显著的遗传变异和分化,主要变异来源于群体间,且研究所用群体均为选育或生产应用中的繁殖群体,可能受到不同程度的选择压力影响。在分组情况下,种群间群体内发生了显著的遗传分化,表明经过几代养殖后,红罗非鱼群体与其他罗非鱼群体之间缺乏基因交流,导致了明显的遗传分化。其中,IL和MY群体间的遗传分化程度不显著(P>0.05),推测它们可能存在母系的同源性。其他种群间显著的遗传分化水平(Fst)表明亲本种群的起源差异及其在生长应用(或选育)中发生遗传漂变的可能。吉富罗非鱼是利用4个非洲引进群体和4个亚洲养殖群体,通过人工选育而成的新品种[31]。对于GIFT与NL群体表现出的遗传分化差异,分析是由于长期的地理分离和人工选择压力造成的。红罗非鱼的起源问题,一直以来不同学者各有不同的看法。大部分学者认为红罗非鱼是由莫桑比克罗非鱼橙色突变体和尼罗罗非鱼、奥利亚罗非鱼或者霍诺鲁姆罗非鱼(O. hornorum)杂交而来的[3],但其父、母本还未确定。目前,我国主要养殖的红罗非鱼群体有TW、IL、MY,研究结果表明,NL与红罗非鱼的遗传分化相对较小,亲缘关系较近,这与聚类分析的结果相符,且COI是母系遗传基因,推测其母系来源可能是NL。这与李莉好等[32]对3种罗非鱼(尼罗罗非鱼、奥利亚罗非鱼、红罗非鱼)遗传多样性比较和李学军等[33]对6种罗非鱼多样性比较的分析结果一致,而与郭奕惠等[15]用COI基因分析中国主要养殖罗非鱼亲缘关系的结果不同,推测出现这种情况的原因可能与本文所用COI基因序列片段更长、涉及样本量更大或样本来源不同有关,进而也说明红罗非来源的复杂性。AMOVA结果显示,红罗非鱼群体属于杂交且人工选育群体,与推测的原始亲本来源群体分化出一定程度的遗传变异。
| [1] |
GUPTA M V, ACOSTA B O. A review of global tilapia farming practices[J]. Aquaculture Asia, 2004, 9(1): 7-12. |
| [2] |
Food and Agriculture Organization of the United Nations (FAO). The State of World Fisheries and Aquaculture 2018-Meeting the sustainable development goals[M]. Rome Italy Fao, 2018: 16-24.
|
| [3] |
李学军, 李思发, 冯金海, 等. 以色列红罗非鱼耐盐性的初步研究[J]. 上海水产大学学报, 2003, 12(3): 205-208. LI X J, LI S F, FENG J H, et al. Preliminary study on salinity tolerance of Israel red tilapia[J]. Journal of Shanghai Fisheries University, 2003, 12(3): 205-208. |
| [4] |
李思发, 蔡完其. 我国尼罗罗非鱼和奥利亚罗非鱼养殖群体的遗传渐渗[J]. 水产学报, 1995, 19(2): 105-111. LI S F, CAI W Q. Introgression in hatchery stocks of Tilapia Nilotica and Tilapia Aurea in China[J]. Journal of Fisheries of China, 1995, 19(2): 105-111. |
| [5] |
RANDALL J E. Introductions of marine fishes to the Hawaiian Islands[J]. Bulletin of Marine Science, 1987, 41(2): 490-502. |
| [6] |
颜标, 李思发, 蔡完其. 尼罗罗非鱼与萨罗罗非鱼及其正反杂交后代的微卫星分析[J]. 水产学报, 2007, 31(3): 411-415. YAN B, LI S F, CAI W Q. Microsatellite analysis of Oreochromis niloticus, Sarotherodon melanotheron and their reciprocal hybrids[J]. Journal of Fisheries of China, 2007, 31(3): 411-415. |
| [7] |
陈增祥, 刘丽, 袁吉贵, 等. 基于D-loop序列的吉富罗非鱼遗传变异分析[J]. 海南热带海洋学院学报, 2018, 25(2): 80-83. CHEN Z X, LI L, YUAN J G, et al. Genetic variation of GIFT strains of Oreochromis niloticus based on D-loop sequence[J]. Journal of Hainan Tropical Ocean University, 2018, 25(2): 80-83. |
| [8] |
杨洁, 何安元, 何学军, 等. 尼罗罗非鱼8个养殖群体线粒体控制区遗传多样性和遗传关系分析[J]. 中国水产科学, 2014, 21(4): 693-699. YANG J, HE A Y, HE X J, et al. Genetic diversity and relationships of mitochondrial DNA control region from the eight domesticated populations of Nile tilapia[J]. Journal of Fishery Sciences of China, 2014, 21(4): 693-699. |
| [9] |
NEI M. Molecular Evolutionary Genetics[M]. New York: Columbia University Press, 1987: 159-164.
|
| [10] |
MA H Y, MA C Y, MA L B. Molecular identification of genus Scylla (Decapoda:Portunidae) based on DNA barcoding and polymerase chain reaction[J]. Biochemical Systematics and Ecology, 2012, 41: 41-47. DOI:10.1016/j.bse.2011.12.016 |
| [11] |
彭士明, 施兆鸿, 侯俊利. 基于线粒体D-loop区与COI基因序列比较分析养殖与野生银鲳群体遗传多样性[J]. 水产学报, 2010, 34(1): 19-25. PENG S M, SHI Z H, HOU J L. Comparative analysis on the genetic diversity of cultured and wild silver pomfret populations based on mtDNA-loop and COI gene[J]. Journal of Fisheries of China, 2010, 34(1): 19-25. |
| [12] |
陈月华, 何培民, 杨金权. 基于DNA条形码的如东海域浒苔附着鱼卵的物种鉴定[J]. 上海海洋大学学报, 2018, 27(1): 1-7. CHEN Y H, HE P M, YANG J Q. Molecular identification of fish eggs in Enteromorpha of Rudong sea area based on DNA barcode[J]. Journal of Shanghai Ocean University, 2018, 27(1): 1-7. |
| [13] |
李梦荣, 田雪, 庞小磊, 等. 基于线粒体COI和Cytb基因序列的6种锦鲤(Cyprinus carpio Koi)遗传多样性分析[J]. 淡水渔业, 2018, 48(3): 13-18. LI M R, TIAN X, PANG X L, et al. Genetic diversity of six species of Cyprinus carpio Koi based on mitochondrial COIand Cytb gene sequences[J]. Freshwater Fisheries, 2018, 48(3): 13-18. DOI:10.3969/j.issn.1000-6907.2018.03.003 |
| [14] |
张凤英, 马凌波, 施兆鸿, 等. 两种鲷属鱼类线粒体COI基因片段序列的比较[J]. 上海水产大学学报, 2006, 15(4): 403-408. ZHANG F Y, MA L B, SHI Z H, et al. Studies on mitochondrial cytochrome oxidase subunit I fragments of two genera of Sparus fish[J]. Journal of Shanghai Fisheries University, 2006, 15(4): 403-408. |
| [15] |
郭奕惠, 喻达辉, 黄桂菊, 等. 中国主要养殖罗非鱼亲缘关系的COI序列分析[J]. 华中农业大学学报, 2009, 28(1): 75-79. GUO Y H, YU D H, HUANG G J, et al. Phylogenetic relationship of Oreochromis tilapias cultured in Mainland China based on COIsequences[J]. Journal of Huazhong Agricultural University, 2009, 28(1): 75-79. DOI:10.3321/j.issn:1000-2421.2009.01.019 |
| [16] |
HALL T A. BioEdit:a user-friendly biological sequence alignment editor and analysis program for windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1998, 41: 95-98. |
| [17] |
LIBRADO P, ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [18] |
KIMURA M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16(2): 111-120. |
| [19] |
TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [20] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [21] |
BOWEN B W, BASS A L, ROCHA L A, et al. Phylogeography of the trumpetfishes (Aulostomus):ring species complex on a global scale[J]. Evolution, 2001, 55(5): 1029-1039. DOI:10.1554/0014-3820(2001)055[1029:POTTAR]2.0.CO;2 |
| [22] |
ORDOÑEZ J F F, VENTOLERO M F H, SANTOS M D. Maternal mismatches in farmed tilapia strains (Oreochromis spp.) in the Philippines as revealed by mitochondrial COI gene[J]. Mitochondrial DNA Part A, 2017, 28(4): 526-535. DOI:10.3109/24701394.2016.1149824 |
| [23] |
MENG W, YANG T Y, HAI S, et al. Extensive genetic divergence among Diptychus maculatus populations in Northwest China[J]. Chinese Journal of Oceanology and Limnology, 2015, 33(3): 577-584. DOI:10.1007/s00343-015-4137-3 |
| [24] |
傅建军, 王荣泉, 沈玉帮, 等. 我国草鱼野生群体D-loop序列遗传变异分析[J]. 水生生物学报, 2015, 39(2): 349-357. FU J J, WANG R Q, SHEN Y B, et al. Genetic variation analysis based on D-loop sequences of wild populations of grass carp (Ctenopharyngodon idella) in China[J]. Acta Hydrobiologica Sinica, 2015, 39(2): 349-357. |
| [25] |
乐小亮, 章群, 赵爽, 等. 中国近海真鲷遗传变异的线粒体控制区序列分析[J]. 广东农业科学, 2010, 37(2): 136-139. YUE X L, ZHANG Q, ZHAO S, et al. Genetic variation of red seabream (Pagrus major) in coastal waters of China inferred from mitochondrial DNA control region sequence analysis[J]. Guangdong Agricultural Sciences, 2010, 37(2): 136-139. DOI:10.3969/j.issn.1004-874X.2010.02.046 |
| [26] |
BOWEN B W, BASS A L, ROCHA L A, et al. Phylogeography of the trumpetfishes (Aulostomus):ring species complex on a global scale[J]. Evolution, 2001, 55(5): 1029-1039. DOI:10.1554/0014-3820(2001)055[1029:POTTAR]2.0.CO;2 |
| [27] |
杨慧, 朱文彬, 董在杰, 等. 3个红罗非鱼群体的形态差异分析[J]. 上海海洋大学学报, 2015, 24(5): 678-684. YANG H, ZHU W B, DONG Z J, et al. Morphological variation analysis of three populations of red tilapia[J]. Journal of Shanghai Ocean University, 2015, 24(5): 678-684. |
| [28] |
夏德全, 曹萤, 吴婷婷, 等. 用RAPD分析对罗非鱼遗传变异的研究及其对杂种优势的应用[J]. 水产学报, 1999, 23(1): 27-32. XIA D Q, CAO Y, WU T T, et al. A study on genetic variation of Tilapias Fish with RAPD analysis and its application to Heterosis[J]. Journal of Fisheries of China, 1999, 23(1): 27-32. |
| [29] |
王进科, 夏德全, 吴婷婷. 中国奠基群奥利亚罗非鱼遗传多态性的DNA指纹图谱[J]. 南京农业大学学报, 2000, 23(3): 61-63. WANG J K, XIA D Q, WU T T. Studies on genetic polymorphism of O. aureus of founder in China with DNA fingerprinting[J]. Journal of Nanjing Agricultural University, 2000, 23(3): 61-63. |
| [30] |
SHAKLEE J B, TAMARU C S, WAPLES R S. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J]. Pacific Science, 1982, 36(2): 141-157. |
| [31] |
EKNATH A E, TAYAMEN M M, PALADA-DE VERA M S, et al. Genetic improvement of farmed tilapias:the growth performance of eight strains of Oreochromis niloticus tested in different farm environments[J]. Aquaculture, 1993, 111(1/4): 171-188. |
| [32] |
李莉好, 喻达辉, 黄桂菊, 等. 尼罗罗非鱼Oreochromis niloticus、奥利亚罗非鱼O. aureus和红罗非鱼O. sp.群体遗传多样性的比较[J]. 热带海洋学报, 2012, 31(2): 102-109. LI L H, YU D H, HUANG G J, et al. Comparison of genetic diversity among stocks of Oreochromis niloticus, O. aureus and red tilapia based on microsatellite DNA[J]. Journal of Tropical Oceanography, 2012, 31(2): 102-109. DOI:10.3969/j.issn.1009-5470.2012.02.014 |
| [33] |
李学军, 李爱景, 李思发, 等. 萨罗罗非鱼(Sarotherodon melanotheron)与其他5种罗非鱼遗传多样性比较研究[J]. 海洋与湖沼, 2010, 41(1): 85-90. LI X J, LI A J, LI S F, et al. Comparison on genetic diversity between Sarotherodon melanotheron and other five tilapias species[J]. Oceanologia et Limnologia Sinica, 2010, 41(1): 85-90. |
2. Freshwater Fisheries Research Center of Chinese Academy of Fishery Sciences, Key Laboratory of Freshwater Fisheries and Germplasm Resources Utilization, Ministry of Agriculture and Rural Affairs, Wuxi 214081, Jiangsu, China
 2019,
Vol. 28
2019,
Vol. 28


