2. 上海深渊科学工程技术研究中心, 上海 201306;
3. 大洋渔业资源可持续开发省部共建教育部重点实验室, 上海 201306;
4. 国家远洋渔业工程技术研究中心, 上海 201306;
5. 上海彩虹鱼海洋科技股份有限公司, 上海 201306
海沟是五大洋海底中比相邻海底深2 000 m以上的狭长凹陷。水深超过6 000 m的长形洼地都可以叫做海沟。目前的研究发现,最深的海沟是马里亚纳海沟,水深在11 000 m左右[1]。海洋最深处的区域称为深渊区[2],深渊区主要由海沟组成,占地球表面的0.24%。深渊海沟包括6 000~11 000 m的深度,为海洋总深度的41%[3-4]。深渊区由较浅的区域分隔开来,由于其深度人类难以到达,因此深渊区域仍然是地球上最不了解的区域之一。
目前人类对深渊区域生物的研究相对匮乏,3 500 m以下的海域基本尚未勘探[5]。有研究[6-7]表明,深渊海沟占了海洋底栖生物栖息地的2%。海沟中形成了离散的超深环境生物群体,破坏了深渊海底的连续性。在这种极端的环境中,依然生存着很多生物,主要有多毛类、腹足类、异足类和角足类[8-10]。它们适应了低温、静水压力高、食物资源匮乏、缺少光照的生存环境,从而具有独特的生物多样性和独特的生态系统[11]。BELIAEV等[12]指出,在深渊区域发现的近700种生物中有56%是深渊环境特有的生物。
生活在深渊海沟中的生物,其在水平分布上也存在着很大的差异。在相邻的两个深渊海沟之间,也仅有大约5%的种群群落是相同的[13]。因此,有研究认为深渊海沟中的生物群落在水平分布以及垂直分布尺度上均表现出其特有的区域专属性[14]。
在生物圈中,无论是生态平衡还是化学平衡,微生物均起到了不可估量的作用[15]。研究肠道微生物多样性,有助于人们了解肠道菌群的微生态,对研究肠道疾病、其生存环境的微生物菌群结构研究也具有很大的帮助。如:利用肠杆菌基因间重复共有序列指纹图谱分析方法可以寻找健康肠道共有菌群的分子标记,从而对肠道疾病做出准确的判断[16]。利用16S rRNA PCR-RFLP方法可以有效地实现海产品微生物的物种鉴定[17]。
随着分子生物学技术的发展,人们发现了一种研究肠道微生物多样性的新方法,即利用16S rRNA技术,可有效克服传统方法的局限性[18-20]。如:利用16S rRNA-RFLP技术对酸马奶中的乳杆菌进行分类和鉴定[21],以及对新疆小叶白蜡丛枝病植原体的鉴定[22]。利用16S rRNA的测序方法来分析微生物多样性的研究也很广泛[23]。利用16S rRNA技术对肠道菌种进行精确定量,已成为研究肠道微生物多样性的主要方法[24]。
为了解深渊海沟宏生物的肠道微生物组成,基于细菌16S rRNA-RFLP的方法,本研究对不同深渊海沟端足类生物的肠道微生物组成与群落结构进行研究。本研究分析获得的深渊海沟端足类钩虾的肠道微生物多样性组成与群落结构特征,将有助于探知深渊物种的肠道菌群微生态,为后续研究深渊端足类生物的肠道微生物理论和应用研究提供基础资料。
1 材料与方法 1.1 样品采集2016年12月至2017年2月,搭乘“张謇”号科学考察船,利用我国自主研制的全海深着陆器,在马里亚纳海沟(Mariana Trench, MT)、玛索海沟(Marceau Trench, MCT)、新不列颠海沟东部(East New Britain, ENBT)、新不列颠海沟中部(Central New Britain, CNBT)等4个采样点进行深海采样。全海深着陆器拥有两个自主研制的诱捕笼,以鲐鱼为饵料、荧光棒为诱捕光源,从“张謇”号科考母船布放,在海底诱捕10余小时后回收回科考母船,收集采集到的钩虾样品,立即放入-80 ℃冰箱,待后续研究。
1.2 试剂基因组DNA提取试剂盒(Fast DNA Spin Kit for Feces,MP公司);PCR rTaq酶以及T4连接酶(大连宝生物工程公司,TaKaRa);Msp Ⅰ限制性内切酶(TaKaRa);氨苄青霉素(上海生工);Ecoli DH5α感受态细胞(天根生化科技有限公司);pMD 18-T载体(上海生工);胶回收试剂盒(OMEGA);琼脂糖(上海生工)、EB溶液(上海生工)等。
1.3 基因组总DNA的抽提以及16S rRNA基因的扩增利用动物粪便基因组DNA抽提试剂盒(MP Biomedicals公司), 提取4个采样点钩虾的肠道基因组DNA,具体操作步骤参照试剂盒说明书进行。提取出的DNA保存于-20 ℃冰箱中备用。分别以提取出的钩虾肠道微生物基因组总DNA为模板,利用细菌通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1 492R(5′-TACGGTTACCTTGTTACGACTT -3′)进行PCR扩增[25],得到16S rRNA基因片段。
PCR扩增体系(50.0 μL)包括:2.5 μL 10×高保真聚合酶(TaKaRa公司),2.0 μL基因组总DNA模板,27F和1 492引物(10 μmol)各1.0 μL,用经灭菌处理后的去离子水补充至50.0 μL。PCR进行扩增的条件为:95 ℃预变性5 min;94 ℃变性45 s;56 ℃退火45 s;72 ℃,2 min,35个循环;72℃最终延伸10 min。4 ℃保存,所得产物使用1%的琼脂糖凝胶进行电泳分离。分离条带用DNA纯化试剂盒(Ackgen公司)纯化回收。
将回收纯化的PCR产物连接到载体pMD18-T(TaKaRa公司)中。在42 ℃条件下,将连接产物转化到Ecoli DH5α感受态细胞(天根公司)中,在LB/Ampr平板上过夜培养。第2天,从LB/Ampr平板上挑取阳性克隆放入LB培养基中进行6 h摇菌培养。利用M13引物鉴定阳性克隆,每个采样点挑选63个阳性克隆。
1.4 细菌16S rRNA-RFLP分析以阳性克隆菌液为模板,利用pMD18-T载体(TaRaKa公司)、通用引物M13重新扩增插入的16S rRNA基因片段,PCR产物即为连接到载体上的16S rRNA片段,大小约为1 000 bp。将PCR产物用Msp Ⅰ核酸限制性内切酶进行单酶切反应,酶切反应体系按照说明书进行。得到的酶切产物用2%琼脂糖凝胶进行电泳分离,分离后进行限制性片段长度多态性(restriction fragment length polymorphism, RFLP)分析,得出不同的酶切图谱,每种谱型初步判定为一种微生物的类型。
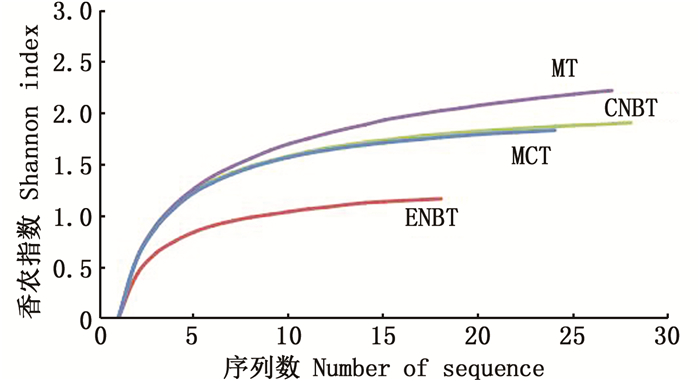
1.5 生物信息学分析测序结束后进行样本间的差异比较,通过Mothur生物学软件,用相似度95%的操作分类单元(operational taxonomic unit, OTU)进行肠道微生物菌群分析(Shannon指数,Simpson指数,Evenness指数)和丰度(Chao指数,ACE指数)分析(表 1),并利用QIIME软件制成稀释曲线(图 1)。

|
表 1 样品的α多样性 Tab.1 Alpha diversity of samples |
|
图 1 Alpha指数稀疏曲线 Fig. 1 Alpha index shannon rarefraction plot |
16S rRNA基因片段的种类鉴定是通过核酸胶图来做的。挑选代表性克隆子进行测序,将测序结果在NCBI (National Center for Biotechnology Information)数据库中与最相似的序列进行比对。将所比对出的细菌进行分类统计,并进行主要成分分析(principal component analysis, PCA)。通过分析不同采样点钩虾肠道微生物的OTU组成可以反映出样品间的差异与距离,从而观察不同采样点、不同样品间微生物群落多样性的分类学差异和优势菌群。
2 结果 2.1 肠道微生物16S rRNA基因文库的构建和PCR-RFLP分析针对4个采样点采集获得的钩虾样本,首先进行形态学鉴定,同时辅以mtDNA 16S rRNA和COⅠ基因进行物种鉴定。4个采样点一共获得3个钩虾物种:马里亚纳海沟的Hirondellea gigas、玛索海沟的Scopelocheirus schellenbergi以及新不列颠海沟东部和中部的Alicella gigantean。
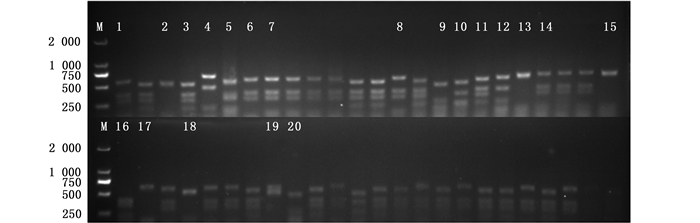
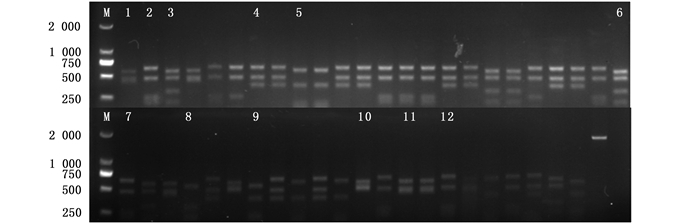
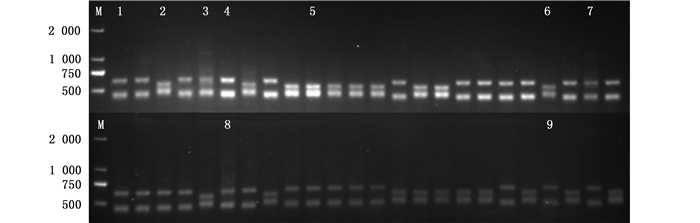
针对每个采样点,分别取30尾钩虾个体进行基因组DNA的提取。以4个采样点钩虾的基因组DNA为模板,利用细菌16S rRNA基因通用引物进行PCR扩增,得到大小约为1 000 bp的PCR扩增片段。将纯化后的PCR产物进行TA克隆,并利用M13引物鉴定阳性克隆,每个采样点得到63个阳性克隆。利用限制性内切酶Msp Ⅰ将上述阳性克隆M13片段进行酶切,每个实验样品保持相同的酶切反应次数及酶切反应条件,结果见图 2~5。马里亚纳海沟的Hirondellea gigas钩虾肠道微生物有20个酶切图谱(图 2);玛索海沟采样点的Scopelocheirus schellenbergi钩虾肠道微生物含有12种酶切图谱(图 3);新不列颠海沟东部、新不列颠海沟中部Alicella gigantean钩虾肠道微生物分别含有9种、11种酶切图谱(图 4和5)。每种谱型初步定为一种微生物类型。
|
M. DNA Marker; 阿拉伯数字代表不同的酶切图谱型 M. DNA Marker; The Arabic numbers indicate different restriction patterns 图 2 马里亚纳海沟优势RFLP型的Msp Ⅰ酶切图谱 Fig. 2 MspⅠ restriction map of predominant RFLP patterns in Mariana Trench |
|
M. DNA Marker; 阿拉伯数字代表不同的酶切图谱型 M. DNA Marker; The Arabic numbers indicate different restriction patterns 图 3 玛索海沟优势RFLP型的Msp Ⅰ酶切图谱 Fig. 3 MspⅠ restriction map of predominant RFLP patterns in Marceau Trench |

|
M. DNA Marker; 阿拉伯数字代表不同的酶切图谱型 M. DNA Marker; The Arabic numbers indicate different restriction patterns 图 4 新不列颠海沟东部优势RFLP型的Msp Ⅰ酶切图谱 Fig. 4 MspⅠ restriction map of predominant RFLP patterns in East of the New British Trench |
|
M. DNA Marker; 阿拉伯数字代表不同的酶切图谱型 M. DNA Marker; The Arabic number indicate different restriction patterns 图 5 新不列颠海沟中部优势RFLP型的Msp Ⅰ酶切图谱 Fig. 5 MspⅠ restriction map of predominant RFLP patterns in Central of the New British Trench |
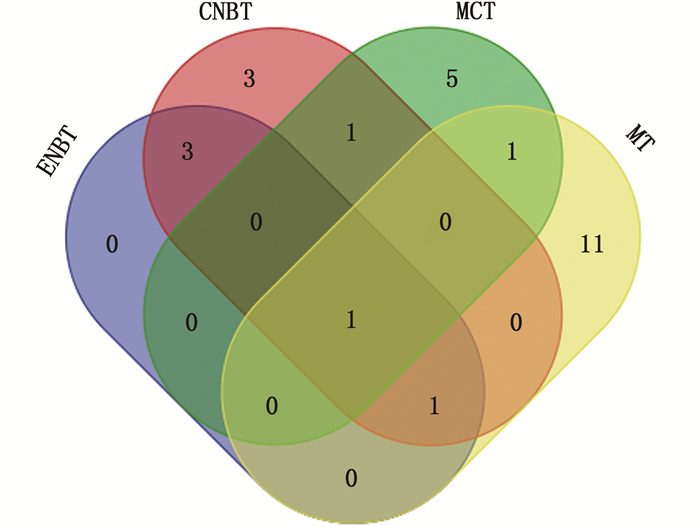
将所有样本序列按照序列间的距离进行聚类,根据序列之间的相似性将序列分成不同的操作分类单元(OTU)。Coverage是各样品文库的覆盖率,其数值越高则样本中序列没有被测出的概率越低。通过Mothur软件分析,以95%相似性水平为标准划分OUT(图 6),总共得到36个有效OUT。CNBT、ENBT、MCT、MT地区的钩虾肠道微生物分别测得9个、5个、8个和14个OUT。4个采样点共有的OTU个数为1个,MT、MCT、CNBT和ENBT特有的OTU分别为11个、5个、3个和0个。OTU在CNBT、ENBT、MCT和MT采样点的覆盖率分别为88.9%、85.7%、87.5%和63.0%(表 1),表明挑选的单克隆子数量已能够反映样品文库中优势细菌类群的数量关系。
|
图 6 四个采样点钩虾OTU样本分布韦恩图 Fig. 6 Amphipod samples Operational Taxonomic Unit |
通过样品的α多样性分析可以反映微生物群落的丰度和多样性。Shannon和Simpson均是用来估算样品中微生物多样性的指数之一:Shannon指数越大,说明群落多样性越高;Simpson指数越大,说明群落多样性越低。通过ACE指数和Chao1指数可以估计样品的群落丰度,其数值越大,说明群落结构多样性越大。由表 1可知,4个采样点样品的群落多样性和丰度上存在着很大的差异(表 1)。马里亚纳海沟采样点的钩虾肠道微生物群落多样性和丰度水平均最高,其他3个采样点的多样性和丰度水平相对较低(表 1)。
Alpha指数稀疏曲线可以用来比较测序数据量在不同样本中的物种丰富度,反映样本测序数量是否合理。曲线趋向平坦,说明测序数据量合理,更多的数据量只会产生少量新的OTU,反之则说明继续测序还可能产生较多新的OTU。由图 6可以看出,曲线趋于平坦,说明测序量合理。
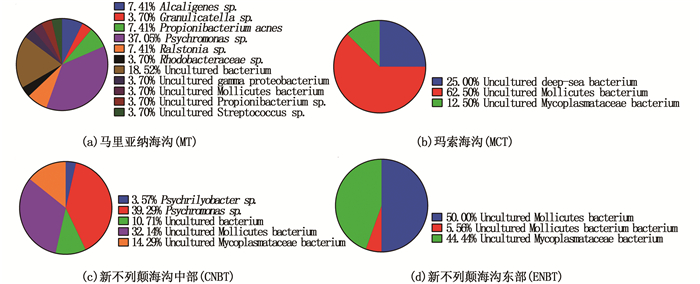
2.3 肠道微生物种类分析选取代表不同酶切类型的克隆子进行测序,并在NCBI上进行序列比对。分析不同采样点不同微生物所占比例(图 7)。结果表明:马里亚纳海沟Hirondellea gigas钩虾肠道微生物中含量最丰富的细菌是嗜冷单胞菌属(Psychromonas sp.)(37.05%),其次为Uncultured bacterium(10.52%),见图 7a。在玛索海沟Scopelocheirus schellenbergi钩虾肠道中,Uncultured Mollicutes bacterium这一微生物含量最为丰富,高达62.50%,其次为Uncultured deep-sea bacterium,含量占到了25.00%(图 7b)。采样于新不列颠海沟中部Alicella gigantean钩虾肠道微生物中Psychromonas sp.含量最高(39.29%),其次为Uncultured Mollicutes bacterium(32.14%),见图 7c。采样于新不列颠海沟东部海域的Alicella gigantean钩虾,其肠道微生物中Uncultured Mollicutes bacterium含量最高(50.00%),其次为Uncultured Mycoplasmataceae bacterium (44.44%),见图 7d。
|
图 7 不同采样点微生物所占比例 Fig. 7 The intestinal microbial community in different sampling sites |
相比于其他3个采样点,马里亚纳海沟中钩虾肠道微生物的种类最丰富(图 7a)。马里亚纳海沟采样点肠道微生物中嗜冷单胞菌属为优势菌群。嗜冷单胞菌是对原料乳危害最大的嗜冷菌之一,在低温条件下容易成为优势菌群[26],表明在深渊低温条件下,嗜冷单胞菌属在深渊冷适应中起重要作用。
3 讨论深渊海沟特殊的环境条件导致深渊海沟生物的独特性。深渊微生物具有较强的环境适应性。本研究通过16S rRNA-RFLP方法鉴定了来自4个海沟采样点3种深渊钩虾的微生物组成、多样性信息以及肠道微生物群落结构特征。
国内外学者先后对水生动物肠道细菌进行了可培养技术的分离和鉴定。宛立等[27]从养殖场的南美白对虾肠道中分离出106株革兰阴性菌、5株革兰阳性菌。这些分离菌株分别属于发光杆菌属(Photobacterium)等13个属(科)。李可等[28]利用RFLP方法对实验室养殖条件下的南美白对虾肠道细菌进行分析,发现2种不同的细菌类群:变形细菌门(Proteobacteria)和厚壁细菌门(Firmicutes), 分别为希瓦氏菌属(Shewanella)、泛菌属(Pantoea)、Aranicola属、假单胞菌属(Pseudomonas)和弧菌属(Vibrio)。王祥红等[29]从野生中国对虾肠道中分离出弧菌属、发光杆菌属等8个菌属。以上研究中发现的菌属存在于浅海水生动物的肠道中。本研究并没有发现这些菌属的存在。由此推测,深渊海沟钩虾肠道微生物和浅海虾的肠道微生物种类差异甚大。推测这可能与深渊钩虾生长在高压、低温、缺氧的极端环境下缺乏营养物质有关。
虾肠道微生物的组成不仅受宿主的影响,还与其生存的环境有关[30]。研究[31-32]表明:海洋生物在幼体胃肠道尚未发育完全的阶段就开始从生活的水体中摄取食物;水体中的微生物随着水流进入体内,进而形成复杂的微生物群落。可以推测,深渊钩虾肠道中的微生物与其生活环境水体中的微生物密切相关。马里亚纳海沟为两个板块辐辏俯冲带,是太平洋板块俯冲到菲律宾板块之下形成的[33]。本研究对4个不同采样点钩虾肠道微生物的分析发现,马里亚纳海沟和玛索海沟、新不列颠海沟东部、新不列颠海沟中部分别有2、2、2个相同的OUT(图 5),这说明马里亚纳海沟和其他3个海沟的钩虾肠道微生物细菌群落结构相似度较低,微生物种群结构存在着较大差异。这可能与马里亚纳海沟独特的形成方式、其深度较深(10 840 m)以及钩虾生长环境差异较大有关。新不列颠海沟东部和中部钩虾Alicella gigantean肠道微生物具有较高的相似性,微生物种群结构相似,原因可能是同属于新不列颠海沟,钩虾生长的环境差异不大,且是同一个物种,因此其肠道微生物类群相似。
嗜冷单胞菌Psychromonas sp.是马里亚纳海沟地区钩虾肠道微生物中含量最丰富的细菌,不可培养细菌Uncultured bacterium也占有很大的比重(图 7)。这说明在马里亚纳海沟中嗜冷菌和不可培养的细菌为优势菌种。值得一提的是,Uncultured Mollicutes bacterium这一柔膜细菌在玛索海沟占了62.50%,在新不列颠海沟东部含量为50.00%,在新不列颠海沟中部含量为32.14%,在马里亚纳海沟仅占3.7%(图 7)。这提示该微生物类群不易在马里亚纳海沟生存。马里亚纳海沟是4个采样点中最深的地区,可以简单地推测深度可能是影响Uncultured Mollicutes bacterium这一柔膜细菌生长的原因。而Uncultured deep-sea bacterium这一类群仅在玛索海沟钩虾肠道中发现(图 7),推测可能与钩虾宿主本身有关。
嗜冷微生物被认为能够生活在永久寒冷的环境中,例如极地、深海和海洋中的冷洋流区域。本研究也发现了大量嗜冷微生物。研究[34]表明,弧菌属和假单胞菌属是甲壳类动物肠道中最常见的菌属,而本研究未发现这两种常见菌属。不过,其他因素会影响到水产动物肠道微生物的组成,如:采样地点、季节、动物发育阶段[35-37]。目前的技术虽不能实现将这些深渊钩虾肠道内的菌种分离,但是,本研究结果初步揭示了深渊海沟中端足目钩虾的肠道微生物组成,为后续进一步研究深海微生物提供了一定的理论基础和依据。
| [1] |
刘方兰, 杨胜雄, 邓希光, 等. 马里亚纳海沟"挑战者深渊"最深点水深探测[J]. 海洋测绘, 2013, 33(5): 49-52. LIU F L, YANG S X, DENG X G, et al. Depth detection in the challenger deep of mariana trench[J]. Hydrographic Surveying and Charting, 2013, 33(5): 49-52. DOI:10.3969/j.issn.1671-3044.2013.05.013 |
| [2] |
WOLFF T. The hadal community, an introduction[J]. Deep Sea Research, 1959-1960, 6: 95-124.
|
| [3] |
RIEHL T, LINS L, BRANDT A. The effects of depth, distance, and the Mid-Atlantic Ridge on genetic differentiation of abyssal and hadal isopods (Macrostylidae)[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2018, 148: 74-90. DOI:10.1016/j.dsr2.2017.10.005 |
| [4] |
JAMIESON A. The hadal zone: life in the deepest oceans[M]. Cambridge: Cambridge University Press, 2015.
|
| [5] |
RIEHL T, KAISER S, BRANDT A. Vema-TRANSIT-An interdisciplinary study on the bathymetry of the Vema-Fracture Zone and Puerto Rico Trench as well as abyssal Atlantic biodiversity[J]. Deep Sea Research Part Ⅱ: Topical Studies in Oceanography, 2018, 148: 1-6. DOI:10.1016/j.dsr2.2018.01.007 |
| [6] |
JAMIESON A J, KILGALLEN N M, ROWDEN A A, et al. Bait-attending fauna of the Kermadec Trench, SW Pacific Ocean: Evidence for an ecotone across the abyssal-hadal transition zone[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2011, 58(1): 49-62. DOI:10.1016/j.dsr.2010.11.003 |
| [7] |
JAMIESON A. The hadal zone: life in the deepest oceans[M]. Cambridge: Cambridge University Press, 2015.
|
| [8] |
JUMARS P A, HESSLER R R. Hadal community structure: implications from the aleutian trench[J]. Journal of Marine Research, 1976, 34(4): 547-560. |
| [9] |
T IETJEN J H, DEMING J W, ROWE G T, et al. Meiobenthos of the hatteras abyssal plain and Puerto Rico trench: abundance, biomass and associations with bacteria and particulate fluxes[J]. Deep Sea Research Part A. Oceanographic Research Papers, 1989, 36(10): 1567-1577. DOI:10.1016/0198-0149(89)90058-7 |
| [10] |
DANOVARO R, GAMBI C, CROCE N D, et al. Meiofauna hotspot in the Atacama Trench, eastern South Pacific Ocean[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2002, 49(5): 843-857. DOI:10.1016/S0967-0637(01)00084-X |
| [11] |
NUNOURA T, TAKAKI Y, HIRAI M, et al. Hadal biosphere: insight into the microbial ecosystem in the deepest ocean on earth[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 112(11): E1230-E1236. |
| [12] |
BELIAEV G M, VINOGRADOV M Y. Deep sea ocean trenches and their fauna[R]. California: Scripps Institution of Oceanography, 1989.
|
| [13] |
VINOGRADOVA N G. Zoogeography of the abyssal and Hadal Zones[J]. Advances in Marine Biology, 1997, 32: 325-387. DOI:10.1016/S0065-2881(08)60019-X |
| [14] |
陈俊, 张奇峰, 李俊, 等. 深渊着陆器技术研究及马里亚纳海沟科考应用[J]. 海洋技术学报, 2017, 36(1): 63-69. CHEN J, ZHANG Q F, LI J, et al. Research on the application of the Hadal Lander technology in the Mariana trench[J]. Journal of Ocean Technology, 2017, 36(1): 63-69. |
| [15] |
许文涛, 郭星, 罗云波, 等. 微生物菌群多样性分析方法的研究进展[J]. 食品科学, 2009, 30(7): 258-265. XU W T, GUO X, LUO Y B, et al. Research progress on analysis methods of diversity of microbial Flora[J]. Food Science, 2009, 30(7): 258-265. DOI:10.3321/j.issn:1002-6630.2009.07.060 |
| [16] |
魏桂芳.肠道微生物群落结构的分子分析[D].上海: 上海交通大学, 2005. WEI G F. Molecular approach based analysis of microbial community structure in gastronintestinal tracts[D]. Shanghai: Shanghai Jiao Tong University, 2005. |
| [17] |
文菁, 胡超群, 张吕平, 等. 16种商品海参16S rRNA的PCR-RFLP鉴定方法[J]. 中国水产科学, 2011, 18(2): 451-457. WEN J, HU C Q, ZHANG L P, et al. PCR-RFLP identification of 16 commercial sea cucumber species on the basis of 16S rRNA gene[J]. Journal of Fishery Sciences of China, 2011, 18(2): 451-457. |
| [18] |
杨青青, 南小宁, 王云果, 等. 柏大蚜和松大蚜体内细菌的16S rRNA-RFLP分析[J]. 西北林学院学报, 2015, 30(6): 168-173. YANG Q Q, NAN X N, WANG Y G, et al. Composition of the Bacteria Associated with Cinara tujafilina and C. pinitabulaeformis Based on 16S rRNA-RFLP Analysis[J]. Journal of Northwest Forestry University, 2015, 30(6): 168-173. DOI:10.3969/j.issn.1001-7461.2015.06.31 |
| [19] |
The Human Microbiome Project Consortium. Structure, function and diversity of the healthy human microbiome[J]. Nature, 2012, 486(7402): 207-214. DOI:10.1038/nature11234 |
| [20] |
RANJAN R, RANI A, METWALLY A, et al. Analysis of the microbiome: advantages of whole genome shotgun versus 16S amplicon sequencing[J]. Biochemical and Biophysical Research Communications, 2016, 469(4): 967-977. DOI:10.1016/j.bbrc.2015.12.083 |
| [21] |
高娃, 于洁, 乌兰, 等. 蒙古国传统发酵乳中部分乳杆菌16S rRNA-RFLP的分类[J]. 中国乳品工业, 2010, 38(1): 4-7. GAO W, YU J, WU L, et al. Classify research of Lactobacillus from traditional fermented milk in Mongolia by 16S rRNA-RFLP methods[J]. China Dairy Industry, 2010, 38(1): 4-7. DOI:10.3969/j.issn.1001-2230.2010.01.001 |
| [22] |
李成亮, 都业娟, 石宝萍, 等. 新疆小叶白蜡丛枝病植原体的鉴定及16S rRNA基因序列分析[J]. 微生物学通报, 2014, 41(4): 601-606. LI C L, DU Y J, SHI B P, et al. The identification and sequence analysis of 16S rRNA gene of Fraxinus sogdiana Bunge witches' broom phytoplasma[J]. Microbiology China, 2014, 41(4): 601-606. |
| [23] |
CELIKKOL-AYDIN S, GAYLARDE C C, LEE T, et al. 16S rRNA gene profiling of planktonic and biofilm microbial populations in the Gulf of Guinea using Illumina NGS[J]. Marine Environmental Research, 2016, 122: 105-112. DOI:10.1016/j.marenvres.2016.10.001 |
| [24] |
李东萍, 郭明璋, 许文涛. 16S rRNA测序技术在肠道微生物中的应用研究进展[J]. 生物技术通报, 2015, 31(2): 71-77. LI D P, GUO M Z, XU W T. Advances and applications on methodology of 16S rRNA sequencing in gut microbiota analysis[J]. Biotechnology Bulletin, 2015, 31(2): 71-77. |
| [25] |
HE H, CHEN Y Y, ZHANG Y L, et al. Bacteria associated with gut lumen of Camponotus japonicus Mayr[J]. Environmental Entomology, 2011, 40(6): 1405-1409. DOI:10.1603/EN11157 |
| [26] |
徐煜, 任婧. 原料乳中嗜冷假单胞菌危害及控制研究进展[J]. 食品工业科技, 2015, 36(13): 380-384. XU Y, REN J. Research progress in hazards and control of psychrotrophilic Pseudomonas in raw milk[J]. Science and Technology of Food Industry, 2015, 36(13): 380-384. |
| [27] |
宛立, 王吉桥, 高峰, 等. 南美白对虾肠道细菌菌群分析[J]. 水产科学, 2006, 25(1): 13-15. WAN L, WANG J Q, GAO F, et al. Bacterial flora in intestines of white leg shrimp (Penaeus vannamei Booen)[J]. Fisheries Science, 2006, 25(1): 13-15. |
| [28] |
李可, 郑天凌, 田蕴, 等. 南美白对虾肠道微生物群落的分子分析[J]. 微生物学报, 2007, 47(4): 649-653. LI K, ZHENG T L, TIAN Y, et al. Bacterial community structure in intestine of the white shrimp, Litopenaeus vannamei[J]. Acta Microbiologica Sinica, 2007, 47(4): 649-653. DOI:10.3321/j.issn:0001-6209.2007.04.016 |
| [29] |
WANG X H, LIU H R, ZHNG X H, et al. Microbial flora in the digestive tract of adult penaeid shrimp (Penaeus chinensis)[J]. Journal of Ocean University of Qingdao, 2000, 30(3): 493-498. |
| [30] |
王芳, 杨季芳, 陈吉刚, 等. 海水养殖蟹体内与外部环境中菌群结构的PCR-DGGE比较——以三疣梭子蟹和锯缘青蟹为例[J]. 海洋学研究, 2010, 28(4): 59-64. WANG F, YANG J F, CHEN J G, et al. Comparison of the bacterial community structure in the crab seawater cultured and the outside environment by PCR-DGGE fingerprint technique: Portunus trituberculatus and Scylla serrata[J]. Journal of Marine Sciences, 2010, 28(4): 59-64. DOI:10.3969/j.issn.1001-909X.2010.04.008 |
| [31] |
TIMMERMANS L P M. Early development and differentiation in fish[J]. Sarsia, 1987, 72(3/4): 331-339. |
| [32] |
VADSTEIN O. The use of immunostimulation in marine larviculture: possibilities and challenges[J]. Aquaculture, 1997, 155(1/4): 401-417. |
| [33] |
STERN R J. Subduction zones[J]. Reviews of Geophysics, 2002, 40(4): 1012. DOI:10.1029/2001RG000108 |
| [34] |
MORIARTY D J W. Interactions of microorganisms and aquatic animals, particularly the nutritional role of the gut flora[M]//Lésel R. Microbiology in Poecilotherms. Amsterdam: Elsevier, 1990: 217-222.
|
| [35] |
SUGITA H, TSUNOHARA M, OHKOSHI T, et al. The establishment of an intestinal microflora in developing goldfish (Carassius auratus) of culture ponds[J]. Microbial Ecology, 1988, 15(3): 333-344. DOI:10.1007/BF02012646 |
| [36] |
SUGITA H, OSHIMA K, TAMURA M, et al. Bacterial flora in the gastrointestine of freshwater fishes in the river[J]. Nippon Suisan Gakkaishi, 1983, 49(49): 1387-1395. |
| [37] |
YOSHIMIZU M, KIMURA T. Study on the intestinal Microflora of Salmonids[J]. Fish Pathology, 1976, 10(2): 243-259. DOI:10.3147/jsfp.10.243 |
2. Hadal Science and Technology Research Center, Shanghai 201306, China;
3. Key Laboratory of Sustainable Exploitation of Ocean Fisheries Resources, Ministry of Education, Shanghai 201306, China;
4. National Distant-water Fisheries Engineering Research Center, Shanghai 201306, China;
5. Shanghai Rainbowfish Ocean Technology Co., Ltd., Shanghai 201306, China
 2020,
Vol. 29
2020,
Vol. 29


