2. 上海海洋大学 海洋科学学院, 上海 201306;
3. 农业农村部大洋渔业开发重点实验室, 上海 201306;
4. 国家远洋渔业工程技术研究中心, 上海 201306;
5. 大洋渔业资源可持续开发教育部重点实验室, 上海 201306
营养位置(trophic position,TP)是食物网科学的核心概念之一,TP的准确估算可量化生物间的营养关系,是了解生态系统结构和功能的基础。传统的TP研究方法有胃含物分析法(stomach content analysis,SCA)和“整体”稳定同位素分析法(bulk stable isotope analysis,BSIA)。前者是基于对生物胃肠中近期摄食的未消化食物的观察结果,可能因低估易消化的食物组分而造成TP估算误差[1]。BSIA是生态学家在SCA基础上引入的最常见的TP分析方法,但该方法在估算TP时需要测定氮稳定同位素基线值(δ15Nbase)[2]。而基线生物或物质的选取需要考虑其分布范围和所能反映的时间尺度等多方面因素,这在大洋生态系统研究中尤为困难。
茎柔鱼(Dosidicus gigas)是一种高度洄游的大洋性头足类,在栖息环境发生变化或洄游到一个新栖息地时,其机体的δ15N值会受到食物来源和δ15Nbase变化的共同影响[3-4]。在缺少基线生物或物质时,利用BSIA较难区分食性变化和洄游行为对机体δ15N值的影响,难以准确估算其TP[2]。氨基酸特定化合物氮稳定同位素技术(amino acid compound specific stable nitrogen isotope analysis,AA-CSIA)是近年来兴起的新方法[5]。研究发现:谷氨酸(Glu)等氨基酸在机体内会发生转氨(transamination)和脱氨(deamination)作用,其δ15N值在TP间具有稳定的富集现象(>5 ‰),被归类为“营养”氨基酸(trophic amino acid,Tro-AA);苯丙氨酸(Phe)等氨基酸的δ15N值在TP间基本不发生变化,能够反映初级生产者的δ15N值,被称为“源”氨基酸(source amino acid,Src-AA)[6]。据此,通过分析茎柔鱼机体氨基酸的δ15N值,可更本质地揭示其机体δ15N值变化的原因,并估算其TP[7]。
本研究以采集自东太平洋赤道海域、秘鲁和智利外海的茎柔鱼为研究对象,对其肌肉氨基酸δ15N值进行测定和比较,分析各海域茎柔鱼氨基酸δ15N值的潜在差异,探讨造成δ15N值空间差异的主要氨基酸种类和可能原因,利用氨基酸δ15N值测定结果估算茎柔鱼TP,并对比不同TP模型的计算结果。
1 材料与方法 1.1 实验材料茎柔鱼样品来自东太平洋3个海域,分别是东太平洋赤道海域(EEP)、秘鲁(PER)和智利外海(CHI),样品经冷冻保存(-20 ℃)运回实验室。样品在实验室解冻后进行基础生物学参数测量。选取个体大小相近的24尾茎柔鱼进行氨基酸氮稳定同位素分析,每个海域各8尾(表 1)。取茎柔鱼胴体漏斗锁软骨处的肌肉4 cm2,去除表皮,并用去离子水(Milli-Q-Water)清洗,置于冷冻干燥机(Christ Alpha 1-4,Martin Christ)内-55 ℃干燥24 h,取出后用混合型球磨仪(Mixer mill MM440,Retsch)研磨呈均匀的细粉末状。

|
表 1 茎柔鱼采样点和基础生物学参数比较 Tab.1 Sampling locations and biological parameters of Dosidicus gigas |
称取干燥的肌肉粉末2 mg加入配有聚四氟乙烯瓶盖的玻璃瓶,而后缓慢加入0.5 ml的HCl(6 mol/L),充入N2后封口,在150 ℃恒温干燥箱中水解70 min。水解结束后,样品根据YARNES等[8]的方法进行衍生化,即氨基酸N-乙酰基异丙酯衍生化法(N-Acetyl Methyl Ester Derivatization)。使用异亮氨酸(L-Norleucine)作为内标。
氨基酸氮稳定同位素分析是采用气相色谱仪(Trace Ultra GC,Thermo Scientific)与质谱仪联用(Thermo Delta V Plus,Thermo Scientific)。毛细管柱型号为Agilent DB-1301(60 m×0.25 mm×1 μm)。进样口温度为250 ℃。升温程序:初始温度为125 ℃,以15 ℃/min升温至140 ℃,然后以8 ℃/min升温至255 ℃并保持35 min。每个样品测定2次以计算标准偏差。所有样品氨基酸δ15N值的平均标准偏差为0.37 ‰。氨基酸氮稳定同位素分析在美国加州大学戴维斯分校下属的稳定同位素分析实验室进行。
1.3 营养位置根据现有研究成果,本研究选取了2种常见的计算模型估算茎柔鱼TP。
第1种模型形式是基于CHIKARAISHI等[7]提出的计算公式,仅根据Glu和Phe的δ15N值计算茎柔鱼TP:
 (1)
(1)
式中:T1为第1种模型估算的茎柔鱼TP;δ15NGlu和δ15NPhe分别是Glu和Phe的δ15N值;βGlu-Phe是初级生产者Glu和Phe的δ15N值的差值,对于水生生态系统,取值3.4 ‰;EGlu-Phe是Glu和Phe在营养级(trophic level)之间富集度的差值,取值7.6 ‰[7]。
第2种模型是BRADLEY等[9]提出的基于3种Tro-AA(Ala、Leu和Glu)和3种Src-AA(Phe、Lys和Gly)的δ15N值计算TP,计算公式如下:
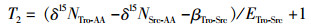 (2)
(2)
式中:T2为第2种模型估算的茎柔鱼TP;δ15NTro-AA和δ15NSrc-AA分别是上述Tro-AA和Src-AA的δ15N值的均值;βTro-Src是Tro-AA和Src-AA在海洋自养生物中δ15N值的均值差,参考有关文献[9],取值3.6‰;ETro-Src是Tro-AA和Src-AA在营养级间富集度的差值,取值5.7‰[9-10]。
1.4 数据统计应用Kruskal-Wallis非参数检验或单因素方差分析(one-way ANOVA)对组间数据进行差异显著性分析。由于不同物种机体内氨基酸氮分馏机制存在差异,有学者提出在MCCLELLAND等[6]与CHIKARAISHI等[7]对Tro-AA和Src-AA的分类基础上,针对特定的物种,需要首先排除部分具有特殊氮分馏机制的氨基酸[5, 11-12]。据此,利用主成分分析(principal component analysis,PCA)对测定出的氨基酸进行筛选[13],特征向量相似的氨基酸将用于后续分析。利用逐步判别分析(stepwise discriminant analysis,SDA)检验造成茎柔鱼氨基酸氮稳定同位素空间差异的主要氨基酸种类。统计分析使用SPSS 25.0软件进行。
2 结果与分析 2.1 茎柔鱼肌肉氨基酸氮稳定同位素比值利用上述样品处理和分析方法,茎柔鱼肌肉共测得12种氨基酸的δ15N值(表 2)。EEP茎柔鱼肌肉的氨基酸δ15N值范围为-21.17‰~26.55 ‰,PER个体的范围为-16.20‰~33.27 ‰,而CHI的样品为-26.53‰~39.73 ‰。苏氨酸(Thr)是茎柔鱼肌肉中δ15N值最低的氨基酸,而各海域δ15N值较高的氨基酸存在差异。来自EEP和PER的茎柔鱼Ala和Val的δ15N值较高,而CHI个体Leu和Val的δ15N值较高(表 2)。根据MCCLELLAND等[6]与CHIKARAISHI等[7]的研究结果,12种氨基酸可分为6种Src-AA(Met、Ser、Thr、Gly、Lys和Phe)和6种Tro-AA(Ala、Glu、Leu、Pro、Val和Asp)。

|
表 2 茎柔鱼肌肉氨基酸δ15N值(均值±标准偏差) Tab.2 The δ15N values of amino acids (mean ± standard deviation) of Dosidicus gigas muscle tissues |
对3个海域茎柔鱼肌肉的12种氨基酸的δ15N值进行主成分分析。结果发现,有2个主成分初始特征值大于1,方差贡献率依次为82.11%和14.48%,累计96.59%。从图 1可以看出,除Thr和Gly外,其余氨基酸(特征向量)的权重均接近于1(>0.9),说明这些氨基酸具有较高的相似度,因此这10种氨基酸将用于后续分析。

|
图 1 茎柔鱼肌肉氨基酸主成分载荷图 Fig. 1 Loading diagram for the principal component of amino acids of Dosidicus gigas muscle tissues |
以3个海域茎柔鱼样品为分组单元,10种氨基酸的δ15N值作为逐步判别分析的输入因子。SDA结果显示,第一个判别函数解释了总判别率81.3%的方差,有5种氨基酸的δ15N值可用于区分茎柔鱼地理来源,即Pro、Glu、Asp、Ser和Lys,并且判别正确率达100 %(图 2)。

|
图 2 来自3个海域的茎柔鱼逐步判别分析散点图 Fig. 2 Stepwise discriminant analysis plots of Dosidicus gigas from three regions |
2种计算模型的计算结果显示,各海域茎柔鱼的TP存在显著差异(表 3)。CHI个体的营养位置显著高于来自EEP和PER的个体,而后两者无显著差异(P> 0.05,表 3)。并且,模型2(4.38 ± 0.35)计算出的茎柔鱼TP显著高于模型1(3.42 ± 0.32)的计算结果(P<0.01)。

|
表 3 来自3个海域的茎柔鱼营养位置 Tab.3 The trophic position of Dosidicus gigas from three regions |
大洋性头足类具有高度洄游特性,其机体的δ15N值会受到不同栖息地食物来源差异和δ15Nbase值变化的共同影响[3-4],单纯利用BSIA较难对其食性变化和洄游行为进行准确判断。AA-CSIA的发展将稳定同位素技术扩展到了分子水平[5]。MCCLELLAND等[6]对海洋浮游生物氨基酸δ15N值的研究发现,伴随着TP的升高,生物体的δ15N值变化是其体内氨基酸δ15N值变化的加权平均结果。不同氨基酸在合成和代谢过程中存在不同的氮分馏机制。随TP升高,谷氨酸(Glu)等Tro-AA的15N存在稳定的富集现象,而苯丙氨酸(Phe)等Src-AA的δ15N值基本不发生变化,能够表征初级生产者的δ15N值[5, 7, 13]。因此,通过分析生物机体氨基酸的δ15N值,可以区分食性和δ15Nbase值的变化,从而更有效地开展大洋性头足类的生态学研究。
3.1 氨基酸氮稳定同位素特征本研究对东太平洋3个海域茎柔鱼肌肉中12种氨基酸的δ15N值分析发现,苏氨酸(Thr)在不同海域均具有最低的δ15N值。该结果与HETHERINGTON等[14]对哥斯达黎加外海茎柔鱼肌肉氨基酸δ15N值分析结果一致。Thr的这种现象也出现在其他海洋生物的氨基酸氮稳定同位素研究中,例如头足纲(Cephalopoda)的鸢乌贼(Sthenoteuthis oualaniensis)[14],哺乳纲(Mammalia)的绿海龟(Chelonia mydas)[15]和抹香鲸(Physeter macrocephalus)[16],硬骨鱼纲(Osteichthyes)的黄鳍金枪鱼(Thunnus albacares)和大眼金枪鱼(Thunnus obesus)[14]。研究发现,Thr具有特殊的氮分馏机制[5],其δ15N值与TP呈负相关,并且捕食者Thr的15N富集度会受到食物δ15NThr值的潜在影响[17],因此,有研究[5]认为Thr可能不适合作为Src-AA。本研究PCA结果也反映出Thr的异质性,但Thr的归类还需根据其在生物机体中的氮分馏机制做进一步论证。除Thr以外,Gly也具有特殊的氮稳定同位素特征,这可能与其受微生物降解有关[5]
对比各海域茎柔鱼肌肉氨基酸δ15N值发现,相同氨基酸的δ15N值存在空间异质性(表 2)。根据SDA结果,氨基酸δ15N值的空间差异主要是来自3种Tro-AA(Pro、Glu和Asp)和2种Src-AA(Ser和Lys)。这些氨基酸表明茎柔鱼肌肉的δ15N值同时与各海域食物来源和δ15Nbase值差异有关。茎柔鱼食性的空间差异已得到广泛验证,PARDO-GANDARILLAS等[18]对智利外海(CHI)采集的茎柔鱼胃含物分析发现,超过80%比例为鱼类,而秘鲁海域(PER)个体的胃含物主要包括头足类和灯笼鱼(Vinciguerria lucetia)[19]。虽然对EEP茎柔鱼食性分析的研究还未见报道,但Tro-AA的空间差异反映出该海域个体食物组成的特异性。这种现象在茎柔鱼肌肉的脂肪酸组成分析中也有发现[20]。相对于初级生产力较低的离岸海域(EEP),近岸海域一般具有较高的δ15Nbase值,例如营养盐含量较高的秘鲁寒流和上升补偿流海域(PER和CHI)[21]。δ15Nbase值的空间差异亦会通过捕食关系传递到茎柔鱼机体中,使得Src-AA的δ15N值出现空间异质性。此外,SDA结果表明,利用Pro、Glu、Asp、Ser和Lys这5种氨基酸的δ15N值可准确追溯茎柔鱼地理来源,说明AA-CSIA在大洋性头足类地理溯源中具有潜在作用。
3.2 茎柔鱼营养位置Src-AA的δ15N值可以反映δ15Nbase值,因此通过测定某些或特定Tro-AA和Src-AA的δ15N值,结合数学函数可估算消费者的TP[7, 13]。OHKOUCHI等[22]测定了1种旋壳乌贼(Spirula spirula)和3种乌贼(Sepia officinalis,S. latimanus和S. esculenta)肌肉和钙质内壳的δ15NPhe和δ15NGlu值,利用模型1估算各物种TP,结果表明旋壳乌贼TP为2.5~2.8,低于乌贼的TP(3.4~3.6)。CHOY等[10]选取了23种北太平洋中上层鱼类肌肉组织,基于模型2估算出TP,进而分析各鱼种营养关系和生态功能。选取多个Tro-AA和Src-AA估算TP可以降低因δ15NGlu和δ15NPhe测量误差造成的潜在影响。
本研究利用这2种常见的基于AA-CSIA的计算模型估算茎柔鱼TP。对比发现,模型1(T1=3.4)和2(T2=4.4)的计算结果存在显著差异,且模型2的结果与其他学者对茎柔鱼的SCA或BSIA的结果更为接近。利用SCA,FIELD等[23]发现加利福利亚海域和东太平洋热带海域的茎柔鱼TP分别为4.4和4.1。TAM等[24]和ESPINOZA等[25]对秘鲁海域茎柔鱼的BSIA结果表明,其TP范围为3.9~4.2。模型1和2都已广泛用于海洋生物营养级估算,但多数研究在计算时仅利用1种模型。本研究结果表明,在分析数据时,需要考虑不同计算方法可能导致的结果差异。有学者[9]认为模型1的计算仅利用Glu和Phe的δ15N值,其估算出的TP易受测量误差的影响,而基于6种氨基酸的模型2在一定程度上可以降低这种潜在影响。尽管如此,两种计算模型都显示出CHI个体具有最高的TP,这与前期对3个海域茎柔鱼肌肉BSIA的结果一致[26]。从两种计算模型可以看出,TP的估算与富集度有关。氨基酸15N的富集度会受食物蛋白质含量和质量的影响[17, 27]。本研究中CHI个体所处纬度最高,海水温度相对最低,该海域个体摄食的冷水性鱼类较多,而栖息于热带海域的EEP和PER个体可能主要摄食头足类和暖水性鱼类[17-18]。各海域茎柔鱼的食性差异会引起其摄入的蛋白质含量存在差别,进而影响富集度。这可能是造成CHI个体TP最高,而EEP和PER个体TP相近的原因。因此,在应用模型估算消费者TP时,针对研究对象选取富集度将进一步提高TP估算的准确性。
| [1] |
RUIZ-COOLEY R I, MARKAIDA U, GENDRON D, et al. Stable isotopes in jumbo squid (Dosidicus gigas) beaks to estimate its trophic position:comparison between stomach contents and stable isotopes[J]. Journal of the Marine Biological Association of the United Kingdom, 2006, 86(2): 437-445. DOI:10.1017/S0025315406013324 |
| [2] |
贡艺, 陈玲, 李云凯. 海洋生态系统稳定同位素基线的选取[J]. 应用生态学报, 2017, 28(7): 2399-2404. GONG Y, CHEN L, LI Y K. Selection of isotopic baselines in marine ecosystems[J]. Chinese Journal of Applied Ecology, 2017, 28(7): 2399-2404. |
| [3] |
LI Y K, GONG Y, ZHANG Y Y, et al. Inter-annual variability in trophic patterns of jumbo squid (Dosidicus gigas) off the exclusive economic zone of Peru, implications from stable isotope values in gladius[J]. Fisheries Research, 2017, 187: 22-30. DOI:10.1016/j.fishres.2016.11.005 |
| [4] |
RUIZ-COOLEY R I, VILLA E C, GOULD W R. Ontogenetic variation of δ13C and δ15N recorded in the gladius of the jumbo squid Dosidicus gigas:geographic differences[J]. Marine Ecology Progress Series, 2010, 399: 187-198. DOI:10.3354/meps08383 |
| [5] |
MCMAHON K W, MCCARTHY M D. Embracing variability in amino acid δ15N fractionation:mechanisms, implications, and applications for trophic ecology[J]. Ecosphere, 2016, 7(12): e01511. DOI:10.1002/ecs2.1511 |
| [6] |
MCCLELLAND J W, MONTOYA J P. Trophic relationships and the nitrogen isotopic composition of amino acids in plankton[J]. Ecology, 2002, 83(8): 2173-2180. DOI:10.1890/0012-9658(2002)083[2173:TRATNI]2.0.CO;2 |
| [7] |
CHIKARAISHI Y, OGAWA N O, KASHIYAMA Y, et al. Determination of aquatic food-web structure based on compound-specific nitrogen isotopic composition of amino acids[J]. Limnology and Oceanography:Methods, 2009, 7(11): 740-750. DOI:10.4319/lom.2009.7.740 |
| [8] |
YARNES C T, HERSZAGE J. The relative influence of derivatization and normalization procedures on the compound-specific stable isotope analysis of nitrogen in amino acids[J]. Rapid Communications in Mass Spectrometry, 2017, 31(8): 693-704. DOI:10.1002/rcm.7832 |
| [9] |
BRADLEY C J, WALLSGROVE N J, CHOY C A, et al. Trophic position estimates of marine teleosts using amino acid compound specific isotopic analysis[J]. Limnology and Oceanography:Methods, 2015, 13(9): 476-493. DOI:10.1002/lom3.10041 |
| [10] |
CHOY C A, POPP B N, HANNIDES C C S, et al. Trophic structure and food resources of epipelagic and mesopelagic fishes in the north Pacific Subtropical Gyre ecosystem inferred from nitrogen isotopic compositions[J]. Limnology and Oceanography, 2015, 60(4): 1156-1171. DOI:10.1002/lno.10085 |
| [11] |
RUIZ-COOLEY R I, GERRODETTE T, FIEDLER P C, et al. Temporal variation in pelagic food chain length in response to environmental change[J]. Science Advances, 2017, 3(10): e1701140. DOI:10.1126/sciadv.1701140 |
| [12] |
NIELSEN J M, POPP B N, WINDER M. Meta-analysis of amino acid stable nitrogen isotope ratios for estimating trophic position in marine organisms[J]. Oecologia, 2015, 178(3): 631-642. DOI:10.1007/s00442-015-3305-7 |
| [13] |
CHIKARAISHI Y, STEFFAN S A, OGAWA N O, et al. High-resolution food webs based on nitrogen isotopic composition of amino acids[J]. Ecology and Evolution, 2014, 4(12): 2423-2449. DOI:10.1002/ece3.2014.4.issue-12 |
| [14] |
HETHERINGTON E D, OLSON R J, DRAZEN J C, et al. Spatial food-web structure in the eastern tropical Pacific Ocean based on compound-specific nitrogen isotope analysis of amino acids[J]. Limnology and Oceanography, 2017, 62(2): 541-560. DOI:10.1002/lno.10443 |
| [15] |
VANDER ZANDEN H B, ARTHUR K E, BOLTEN A B, et al. Trophic ecology of a green turtle breeding population[J]. Marine Ecology Progress Series, 2013, 476: 237-249. DOI:10.3354/meps10185 |
| [16] |
ZUPCIC-MOORE J R, RUIZ-COOLEY R I, PALIZA O, et al. Using stable isotopes to investigate foraging variation and habitat use of sperm whales from northern Peru[J]. Marine Ecology Progress Series, 2017, 579: 201-212. DOI:10.3354/meps12281 |
| [17] |
MCMAHON K W, THORROLD S R, ELSDON T S, et al. Trophic discrimination of nitrogen stable isotopes in amino acids varies with diet quality in a marine fish[J]. Limnology and Oceanography, 2015, 60(3): 1076-1087. DOI:10.1002/lno.10081 |
| [18] |
PARDO-GANDARILLAS M C, LOHRMANN K B, GEORGE-NASCIMENTO M, et al. Diet and parasites of the jumbo squid Dosidicus gigas in the Humboldt Current System[J]. Molluscan Research, 2014, 34(1): 10-19. DOI:10.1080/13235818.2013.860870 |
| [19] |
ALEGRE A, MÉNARD F, TAFUR R, et al. Comprehensive model of jumbo squid Dosidicus gigas trophic ecology in the northern Humboldt Current System[J]. PLoS One, 2014, 9(1): e85919. DOI:10.1371/journal.pone.0085919 |
| [20] |
贡艺, 李云凯, 陈玲, 等. 东太平洋不同海区茎柔鱼肌肉脂肪酸组成分析与比较[J]. 渔业科学进展, 2018, 39(6): 147-154. GONG Y, LI Y K, CHEN L, et al. A comparative analysis of fatty acid profiles in muscle of Dosidicus gigas from different harvest locations in the Eastern Pacific Ocean[J]. Progress in Fishery Sciences, 2018, 39(6): 147-154. |
| [21] |
ANDERSON C I, RODHOUSE P G. Life cycles, oceanography and variability:ommastrephid squid in variable oceanographic environments[J]. Fisheries Research, 2001, 54(1): 133-143. DOI:10.1016/S0165-7836(01)00378-2 |
| [22] |
OHKOUCHI N, TSUDA R, CHIKARAISHI Y, et al. A preliminary estimate of the trophic position of the deep-water ram's horn squid Spirula spirula based on the nitrogen isotopic composition of amino acids[J]. Marine Biology, 2013, 160(4): 773-779. DOI:10.1007/s00227-012-2132-1 |
| [23] |
FIELD J C, BALTZ K, PHILLIPS A J, et al. Range expansion and trophic interactions of the jumbo squid, Dosidicus gigas, in the California Current[J]. California Cooperative Oceanic Fisheries Investigations Report, 2007, 48: 131-146. |
| [24] |
TAM J, TAYLOR M H, BLASKOVIC V, et al. Trophic modeling of the northern Humboldt Current Ecosystem, part Ⅰ:comparing trophic linkages under La Niña and El Niño conditions[J]. Progress in Oceanography, 2008, 79(2/4): 352-365. |
| [25] |
ESPINOZA P, LORRAIN A, MÉNARD F, et al. Trophic structure in the northern Humboldt Current system:new perspectives from stable isotope analysis[J]. Marine Biology, 2017, 164(4): 86. DOI:10.1007/s00227-017-3119-8 |
| [26] |
GONG Y, LI Y K, CHEN X J, et al. Potential use of stable isotope and fatty acid analyses for traceability of geographic origins of jumbo squid (Dosidicus gigas)[J]. Rapid Communications in Mass Spectrometry, 2018, 32(7): 583-589. DOI:10.1002/rcm.8071 |
| [27] |
NUCHE-PASCUAL M T, LZAO J P, RUIZ-COOLEY R I, et al. Amino acid-specific δ15N trophic enrichment factors in fish fed with formulated diets varying in protein quantity and quality[J]. Ecology and Evolution, 2018, 8(18): 9192-9217. DOI:10.1002/ece3.2018.8.issue-18 |
2. College of Marine Sciences, Shanghai Ocean University, Shanghai 201306, China;
3. Key Laboratory of Oceanic Fisheries Exploration, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China;
4. National Engineering Research Center for Oceanic Fisheries, Shanghai 201306, China;
5. Key Laboratory of Sustainable Exploitation of Oceanic Fisheries Resources, Ministry of Education, Shanghai 201306, China
 2019,
Vol. 28
2019,
Vol. 28


