2. 上海海洋大学 水产动物遗传育种上海市协同创新中心, 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306
刀鲚(Coilia nasus)是一种重要的溯河产卵鱼类,是1846年根据一尾采集于日本的标本命名的[1],隶属鲱形目(Clupeiformes)、鳀科(Engraulidae)、鲚属(Coilia)[2-3]。刀鲚生活在西北太平洋沿海和河口地区,成熟后每年迁移至淡水区进行产卵。在长江流域,它们通常迁移至一些小支流和淡水湖泊进行产卵,例如洞庭湖、鄱阳湖和太湖等水域。此外,在长江流域中还存在2种相近的鲚属鱼类,即湖鲚(Coilia. nasus taihuensis)和短颌鲚(Coilia. brachygnathus)。1980年,袁传宓等[4]研究发现湖泊捕捞到的定居型“刀鲚”与长江主干流中捕捞的洄游型刀鲚存在形态差异。一些形态特征,例如椎体平均数、臀鳍鳍条的平均数,吻长与眼径及肝长之比等,被作为区分巢湖和太湖淡水定居型刀鲚和长江洄游型刀鲚的依据,并以此定名一个新亚种湖鲚[4]。湖鲚是刀鲚定居于淡水的一种生态型,生活在长江流域下游的一些淡水湖泊中,可以在淡水中完成它们的整个生命周期,如太湖和巢湖[4]。1908年,KREYENBERG等[5]描述了一尾从洞庭湖采集的标本,由于它的上颌较短,所以被命名为一个新种,短颌鲚。短颌鲚也是一种淡水定居型刀鲚,但是它们主要分布在与长江流域相通的更上游的淡水湖泊,例如鄱阳湖和洞庭湖[6]。对于短颌鲚和刀鲚、湖鲚和刀鲚之间是否互为有效种或亚种,在之前一直缺乏统一的认知。
刀鲚作为长江的名贵鱼类,以肉味鲜美而著称,曾为长江下游最重要的经济鱼类之一[7]。然而由于水体环境污染、洄游通道受阻以及捕捞过度等原因, 刀鲚的资源产量急剧下滑[8],已经不能形成渔汛,受供求关系的影响,价格一路飙升,为获取巨大利益,渔民们非法捕捞,对长江的生物资源造成严重影响。据统计,在2012年,由于长江刀鲚资源稀少,市场价达到了20 000元/kg。在巨大的利润推动下,许多商人利用短颌鲚和湖鲚冒充长江刀鲚[9]。短颌鲚、湖鲚和刀鲚之间如何区分,一直是很多学者研究的热点。
早期分类学研究为刀鲚的不同生态型提供了形态学判别特征,依据形态学特征鉴定物种方便易行又简单直观, 是研究鲚属鱼类分类问题最常使用的方法[10]。但是通过形态学方法进行物种鉴定往往得到矛盾的结果,可能是由于用来区分刀鲚和湖鲚、刀鲚和短颌鲚的形态测量特征不稳定造成的。例如,程起群等[11]早期使用框架分析方法比较了刀鲚和湖鲚形态差异,结果表明,两个群体之间的形态差异只属于种内不同地理种群之间的差距,并没有达到亚种水平的差异。向文殿等[12]通过对南四湖、洪泽湖和太湖的湖鲚进行形态差异分析表明,太湖湖鲚的群体形态差异最大,已有一定程度的形态分化。此外,研究发现短颌鲚的上颌没有到达鳃盖的边缘被用作区别于刀鲚的形态特征[13],但是其他研究中在太湖的湖鲚和从长江主支流中捕捞到的刀鲚中也发现有上颌较短的个体[13-14]。因此,形态学的方法并不能作为一种有效鉴别刀鲚的方法。
随后的分子研究表明,刀鲚、短颌鲚和湖鲚的遗传分化较低,在它们之间没有显著的遗传结构差异[15-16]。YANG等[16]利用扩增片段长度多态性(AFLP)技术,对鲚属鱼类进行遗传亲缘关系分析,表明短颌鲚、湖鲚和刀鲚应为同物异名的同一物种。杨金权等[17]使用线粒体控制区全序列证明了鄱阳湖短颌鲚、湖鲚与刀鲚是同一个物种,短颌鲚和湖鲚为刀鲚的淡水生态型,并非有效物种。张燕萍等[18]基于线粒体控制区序列,分析刀鲚和短颌鲚的遗传多样性,结果表明刀鲚和短颌鲚尚未达到种或亚种水平的分化。但是大多数分子研究是基于单个的线粒体基因,统计鉴定力低,不能够提供强有力的证据,所以不能有效地区分短颌鲚和刀鲚、湖鲚和刀鲚。
在刀鲚资源调查和物种保护的工作中,常常需要对刀鲚的生态型(物种)进行区分[19]。目前比较有效的方法是使用耳石微量元素检测,因为生活过不同环境的刀鲚耳石中微量元素的比率不一样,所以可以根据耳石中的微量元素的比率推断刀鲚的生态型。有基于耳石微量元素的研究[20]表明长江安徽和县江段刀鲚资源组成复杂,同时存在短颌鲚和刀鲚,即洄游群体中可能存在短颌鲚;而短颌鲚中也存在洄游型的刀鲚个体[21]。测量耳石的微量元素来探究每尾鱼的生活史,确定湖泊中的鱼是否为洄游型还是定居型的方法,虽然大大提高了准确性,但是检测方法较为繁琐且成本昂贵[22]。
由于长江流域刀鲚的成鱼数量急剧减少,所以对于产卵区的保护显得尤为重要[8]。JIANG等[1]认为,产卵地的保护可以确保刀鲚资源的可持续性,但耳石微化学的方法对鱼体的大小有一定的要求,不能够用于确定仔鱼和受精卵是否为洄游型刀鲚的后代。因此,急需一种快速高效的方法来区分短颌鲚和刀鲚、湖鲚和刀鲚,从而更有针对性地保护资源[23]。
本研究团队在前研究中,应用跨物种靶基因富集方法[24]收集了刀鲚、湖鲚和短颌鲚的基因组水平的数据,证明了洞庭湖和鄱阳湖的短颌鲚与刀鲚互为有效种,太湖和巢湖的湖鲚与洄游型的刀鲚差异较小,并且找到了在短颌鲚和刀鲚、湖鲚和刀鲚之间存在区别的SNP位点。本研究测试了以上SNP位点,用来开发区分短颌鲚和刀鲚、湖鲚和刀鲚的分子标记,希望为刀鲚的物种鉴定和保护提供一种有效的方法[25]。
1 材料与方法 1.1 实验材料与DNA提取共采集了20尾短颌鲚样本,分别来自2个湖泊鄱阳湖(PY,n=10,来自3个采样点)和洞庭湖(DT,n=10,来自3个采样点)。20尾湖鲚样本,分别来自2个湖泊太湖(TH,n=10,来自3个采样点)和巢湖(CH,n=10,来自2个采样点)。12尾洄游型刀鲚样本,分别来自长江的主河道和重要支流(CJ,n=12),如靖江、环崇明岛的长江分流和上海沿海的芦潮港(表 1和图 1)。

|
表 1 刀鲚、湖鲚和短颌鲚的样本采集信息 Tab.1 nformation of sample collection of C. nasus, C. nasus taihuensis and C. brachygnathus |

|
图 1 采样点分布图 Fig. 1 Collection Locations |
大多数的定居型群体样本都为10月份之后采集的。10月份之后洄游型的刀鲚已经离开淡水湖泊,增加了我们在湖泊中采到定居型样本的概率。我们也通过测定样本矢耳石的Sr/Ca来进一步验证生态型[1]。从每个湖泊中分别挑选多个采集点的样本测量矢耳石微量元素Sr/Ca(每个湖泊4~5尾),另选1尾洄游型的样本作为参照比较,发现从湖泊中采集的鱼矢耳石的中心、边缘和中间位置的Sr/Ca很稳定,几乎没有数量级的波动,认定它们是淡水定居型。与此相反,洄游的刀鲚矢耳石3个位置的Sr /Ca在中间和边缘处存在明显的差异。因此,进一步验证了湖泊采集的样本确实是淡水定居型。
取样本的胸鳍条和肌肉浸泡于95%的乙醇储存在4 ℃冰箱中。使用组织DNA试剂盒(Omega Bio-tek, Norcross, GA, USA)提取所有样本的DNA,使用琼脂糖凝胶电泳检测DNA提取的结果,然后置于-20 ℃的冰箱中以备后续实验使用。
将测序验证后的序列用MEGA7软件分析基因序列的变异位点(SNP位点),对比测序后的数据峰图,找到峰值图中对应MEGA7序列里观察到的变异位点,基因型统计时,二倍体纯合位点记为两个相同的碱基,显示为单峰图;而杂合位点则记为两个不同的碱基,显示为套峰,以此计算等位基因频率。基于测序结果,分别统计了短颌鲚与刀鲚、湖鲚与刀鲚之间的差异分子标记在各群体样本中的基因频率信息。
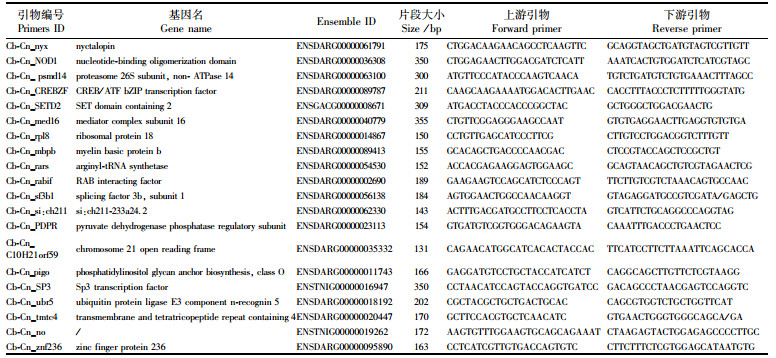
1.2 引物设计、PCR扩增和测序本实验使用的标记为之前靶基因富集研究中发现的SNP分子标记。在定居型的短颌鲚和洄游型的刀鲚之间共发现了120个分子标记位点,其中有68个Fst分化指数为1的位点。从68个标记位点中随机挑选20个用于引物设计(表 2)。在湖鲚和刀鲚之间发现了21个分子标记位点,按照Fst分化指数的由高到低和P显著性挑选了10个用于设计引物(表 3)。PCR反应体系为25 μL,包括2×Taq Master Mix 12.5 μL(南京诺唯赞生物科技有限公司:P111-01/02/03)、DNA样品1 μL、上下游引物各0.5 μL,最后使用ddH2O补充到25 μL。反应程序:94 ℃预变性5 min,30~35个循环, 94 ℃变性30 s、退火温度为每对上下游引物的平均数45 s、72 ℃延伸60 s,循环结束,72 ℃延伸7 min。每个样品PCR产物使用1.5%的琼脂糖凝胶电泳检测扩增情况,将扩增出的PCR目标产物送至上海生物工程公司单向测序。
|
表 2 用于区分短颌鲚与刀鲚的20个SNP分子标记的引物 Tab.2 Primers of 20 SNP markers distinguishing C. brachygnathus and C. nasus |

|
表 3 区分湖鲚和刀鲚的10个SNP分子标记的引物 Tab.3 Primers of 10 SNP markers distinguishing C. nasus taihuensis and C. nasus |
使用CondonCodeAligner 6.0.2软件(http://www.codoncode.com/aligner/download.htm)对测序结果进行初步的处理,并进行人工校正。根据软件显示的峰值图,将含有变异位点序列的前或后端的杂峰序列删除,以确保数据的准确性。最后,将所有生工测序的数据与设计引物所用的数据合并,用MEGA7软件(https://www.megasoftware.net/)进行比对,验证所得数据变异位点是否与最初设计引物的数据变异位点相一致。
本实验随机测序了10尾预先了解来源信息的湖鲚和刀鲚的样本,基于以上统计获得各SNP位点在湖鲚和刀鲚中的等位基因频率数据,判断该10个样本归类信息,进一步验证这些SNP位点用于判别刀鲚或湖鲚的准确性。
具体分析方法:针对个体成功获得扩增的共有分子标记,当只使用一个分子标记时,此分子标记含有一个SNP位点,由于并不知道要判断的序列样本是湖鲚还是刀鲚,所以根据已知此SNP标记在湖鲚和刀鲚群体中的基因频率,分别计算此样本为湖鲚或刀鲚的可能性,假设测定的结果为AA,那么它是湖鲚和刀鲚的可能性分别为湖鲚和刀鲚A的基因频率乘以A的基因频率,若湖鲚的乘积高则支持此样本为湖鲚。若1个分子标记含有2个SNP位点,那么分别独立计算,即相当于2个SNP标记,将2次获得的数值分别依次相乘获得综合判定数值,依据数值大小认定所属群体的可能性。判定正确则记录为正确1次,最后计算验证准确率。计算公式如下:
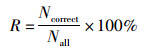 (1)
(1)
式中:R为计算的验证准确率及判断正确次数占总次数的比值;Ncorrect为判断正确的次数;Nall为判断总次数。随后,依次增加位点数并计算验证准确率。对10尾刀鲚和湖鲚的样本分别进行判断。
2 结果 2.1 短颌鲚和刀鲚间特异性SNP位点分析结果使用MEGA7进行比对,验证了所得数据变异位点与最初设计引物的数据变异位点相一致。用于测试区分短颌鲚和刀鲚的20对引物平均在13尾短颌鲚和11尾刀鲚样本中成功扩增。用于测试区分湖鲚和刀鲚的10对引物只有8对引物成功扩增,平均获得17尾湖鲚和11尾刀鲚的数据,另外2对引物扩增出的序列经2次测序都为杂峰。
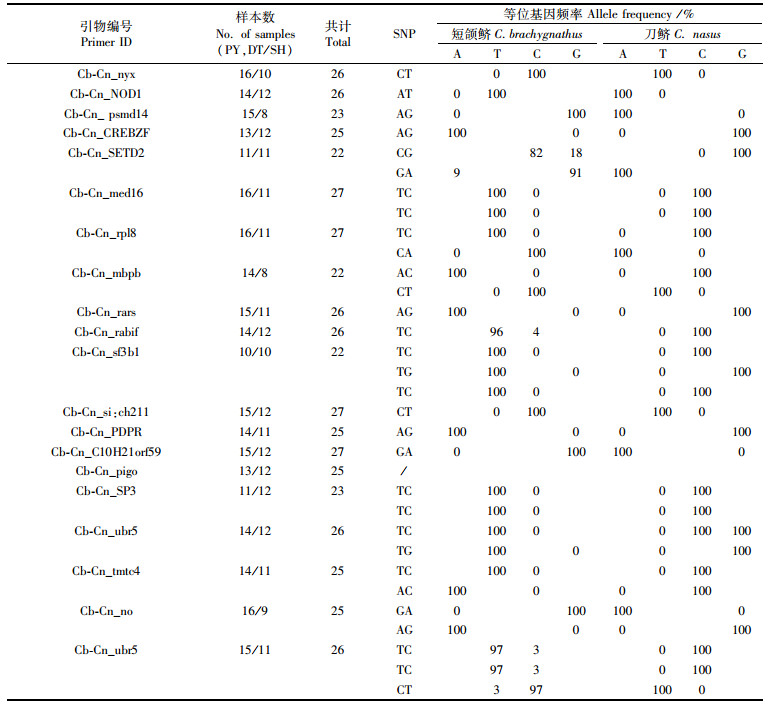
用于区分短颌鲚和刀鲚的20个分子标记中有1个没有观察到SNP位点,其他19个标记序列在进行人工校正和比对后发现,19个SNP分子标记可以将短颌鲚和刀鲚完全区分开,其中16个分子标记在短颌鲚和刀鲚中都是100%纯合的。部分基因存在2~3个SNP位点(表 4)。
|
表 4 20个SNP分子标记的基因频率 Tab.4 Gene frequency of 20 SNP markers |
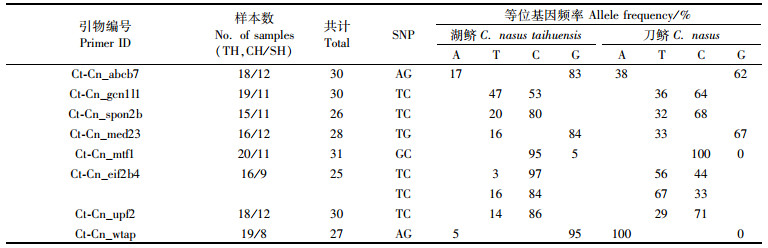
用区分湖鲚和刀鲚的10对引物成功扩增出了8个SNP分子标记,其中引物编号为Ct-Cn_eif2b4的分子标记含有2个SNP位点。湖鲚的等位基因频率范围为3%~95%,刀鲚的基因频率范围为0~100%(表 5)。其中引物编号Ct-Cn_wtap的分子标记在湖鲚和刀鲚之间的基因频率相差最大,分别为5%和100%、95%和0,最有可能将湖鲚和刀鲚区分开。
|
表 5 8个SNP分子标记的基因频率 Tab.5 Gene frequency of 8 SNP markers |
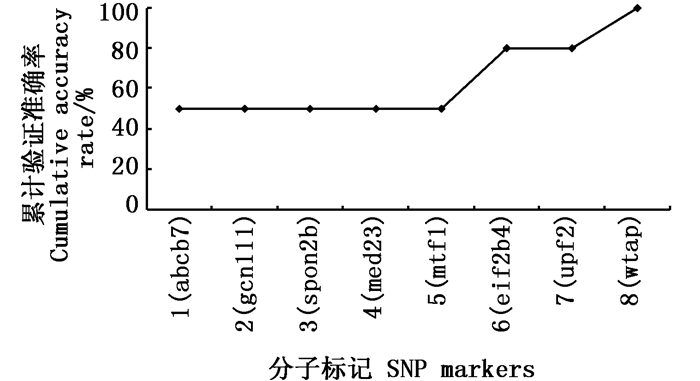
为了检验用于区分湖鲚与刀鲚的8个SNP标记的实际应用效果,随机验证10尾湖鲚或刀鲚的测试样本,使用表 5的信息鉴定湖鲚与刀鲚物种,与实际的情况进行对比。例如,当使用Ct-Cn_abcb7这个分子标记(表 5)时,随机测取的1尾鱼样本的基因型,假设测定结果为AA,那么它是湖鲚和刀鲚的可能性分别为0.028 9(0.17×0.17)和0.144 4(0.38×0.38),因为0.144 4 > 0.028 9,所以,判定该尾鱼为刀鲚,如判定正确则记录为正确1次,最后计算验证准确率。之后分别使用总数为1、2、3、…、8个分子标记,检验10尾混合湖鲚与刀鲚样本的准确率(图 2)。结果发现,验证准确率可以达到100%(图 2)。当使用前5个分子标记Ct-Cn_abcb7、Ct-Cn_gcn1l1、Ct-Cn_spon2b、Ct-Cn_med23和Ct-Cn_mtf1时,准确率并没有上升;当加入分子标记Ct-Cn_eif2b4时,明显发现判断准确率提高至80%,证明分子标记Ct-Cn_eif2b4区分性更高,当加入Ct-Cn_wtap时,混合样本的判断准确率高达100%。见图 2和表 6。
|
图 2 分子标记和累计验证准确率 Fig. 2 SNP markers and cumulative accuracy rate |
|
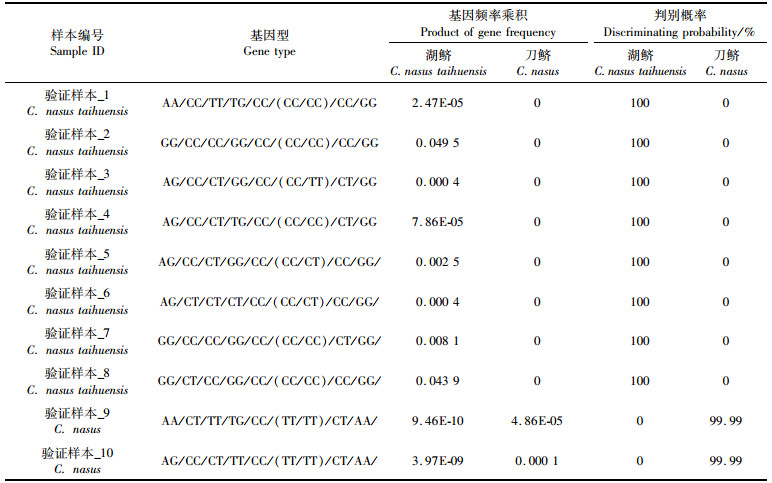
表 6 基于8个SNP分子标记测取的10个样本的判别概率 Tab.6 Discriminating probability of 10 samples using 8 SNP markers |
表 6列出了10尾刀鲚和湖鲚待验证样本的基因频率乘积及判别概率(将湖鲚和刀鲚的基因频率乘积相加为总和,计算湖鲚和刀鲚各自所占总和的比率,作为判别概率)。当使用8个SNP分子标记时,每个样本的基因频率乘积出现了明显的差别。若为湖鲚样本,刀鲚样本的基因频率乘积都为0;若为刀鲚样本,湖鲚样本的基因频率乘积远远小于刀鲚。结果表明湖鲚和刀鲚样本的判别概率都可以达到或接近100%(表 6)。
3 讨论目前,形态学的方法并不能用来有效鉴别刀鲚、湖鲚和短颌鲚[13-14],而常用的分子标记或者显示刀鲚、短颌鲚和湖鲚没有显著的遗传分化[15-16],或者是基于单个的线粒体基因,统计鉴定力低,不能有效区分短颌鲚、湖鲚和刀鲚[17]。测量耳石的微量元素来探究每尾鱼的生活史,确定湖泊中的鱼是洄游型还是定居型,虽然大大提高了准确性,但是检测方法较为繁琐且成本昂贵[22]。
之前研究采用跨物种靶基因富集的方法[24],使用的2 869个基因位点已经证实了短颌鲚和刀鲚之间互为有效种,已达到了种的分化层次,并且我们找到了120个可能用于区分短颌鲚和刀鲚的SNP分子标记位点。湖鲚与刀鲚之间没有达到亚种分化的水平,湖鲚只是刀鲚的淡水生态型,这有可能是由于湖鲚和刀鲚的分化时间较短,这与之前的诸多研究结果[26-30]相一致。但是,在不同的生态型之间,我们仍然发现了21个可能用于区分湖鲚和刀鲚的标记位点,它们可能是湖鲚对淡水生活环境做出的快速适应性改变的变异位点。
本研究中,区分短颌鲚和刀鲚的20对分子标记中19对可以直接用于实验鉴定,鉴定准确率为100%。区分湖鲚和刀鲚的8对分子标记,经实验验证发现,当加入分子标记Ct-Cn_eif2b4和Ct-Cn_wtap分析时,验证准确率得到提升,可以达到100%。根据我们的测试样本结果,不建议使用Ct-Cn_abcb7、Ct-Cn_gcn1l1、Ct-Cn_spon2b、Ct-Cn_med23和Ct-Cn_mtf1分子标记,因为这几个分子标记的基因频率没有较大的偏差倾向。例如,引物编号为Ct-Cn_abcb7的分子标记,在湖鲚和刀鲚中,A的基因频率分别为17%和38%,所占的比例都偏小,G的基因频率分别为83%和62%,所占的比例都偏高,所以即使共同使用以上几个位点也不能够提高验证准确率。我们最后使用的10尾验证样本表明,若只使用Ct-Cn_eif2b4和Ct-Cn_wtap这2个基因频率相差大的位点共同判断,得到的判别准确率可以达到100%。因此,我们推荐使用引物编号为Ct-Cn_eif2b4和Ct-Cn_wtap的分子标记。
基于开发的2组SNP分子标记位点,可以快速高效地区分湖鲚和刀鲚、短颌鲚和刀鲚。从规范市场上来讲,应用这些位点可以检测到用湖鲚冒充刀鲚的行为。从资源利用和保护上来说,鄱阳湖是刀鲚的一个重要的产卵场[31],通过标记位点的检测,可以区分刀鲚和短颌鲚的产卵场,更有利准确地调查刀鲚资源,有针对性地保护洄游型刀鲚幼苗和产卵群体。此外,短颌鲚生活在长江中上游的一些淡水湖泊中,这些湖泊位于经济高度发达地区,受人类活动的影响较严重,而短颌鲚与刀鲚互为有效种,因此建议短颌鲚的保护问题也应该受到更多的关注,参照研究结果,更好地指导鲚属鱼类资源的进一步恢复[32]。
| [1] |
JIANG T, YANG J, LV M J, et al. Discovery of a spawning area for anadromous Coilia nasus Temminck et Schlegel, 1846 in Poyang Lake, China[J]. Journal of Applied Ichthyology, 2017, 33(2): 189-192. DOI:10.1111/jai.13293 |
| [2] |
LIU D, LI Y, LI Y Y, TANG W Q, et al. Population structure of Coilia nasus in the Yangtze River revealed by insertion of short interspersed elements[J]. Biochemical Systematics and Ecology, 2014, 54: 103-112. DOI:10.1016/j.bse.2013.12.022 |
| [3] |
YANG Q L, GAO T X, MIAO Z Q. Differentiation between populations of Japanese grenadier anchovy (Coilia nasus) in Northwestern Pacific based on ISSR markers:implications for biogeography[J]. Biochemical Systematics and Ecology, 2011, 39(4/6): 286-296. |
| [4] |
袁传宓, 秦安黔, 刘仁华, 等. 关于长江中下游及东南沿海各省的鲚属鱼类种下分类的探讨[J]. 南京大学学报(自然科学版), 1980(3): 67-82. YUAN C M, QING A Q, LIU R H, et al. On the classification of the anchovies, Coilia from the lower Yangtze River and the southeast coast of China[J]. Journal of Nanjing University (Natural Sciences), 1980(3): 67-82. |
| [5] |
KREYENBERG W, PAPPENHEIM P. Ein Beitrag zur Kenntnis der Fische der Jangtze und seiner Zuflüsse[J]. Sber Ges Naturf Freunde Berl, 1908, 95-109. |
| [6] |
吴斌, 方春林, 傅培峰, 等. 鄱阳湖通江水道短颌鲚生长特性初探[J]. 水生态学杂志, 2015, 36(3): 51-55. WU B, FANG C L, FU P F, et al. Growth characteristics of Coilia brachygnathus in the Poyang Lake-Yangtze River waterway[J]. Journal of Hydroecology, 2015, 36(3): 51-55. |
| [7] |
董文霞, 唐文乔, 王磊. 长江刀鲚繁殖群体的生长特性[J]. 上海海洋大学学报, 2014, 23(5): 669-674. DONG W X, TANG W Q, WANG L. Growth characteristics of reproductive population of Coilia nasus in the Yangtze River[J]. Journal of Shanghai Ocean University, 2014, 23(5): 669-674. |
| [8] |
顾海龙, 冯亚明, 游华斌, 等. 长江刀鲚资源调查与人工养殖研究进展[J]. 江苏农业科学, 2016, 44(3): 265-267. GU H L, FENG Y M, YOU H B, et al. Research progress of resource investigation and artificial breeding of Coilia nasus of the Yangtze River[J]. Jiangsu Agricultural Sciences, 2016, 44(3): 265-267. |
| [9] |
王庆萍, 甘江英. 人类活动对鄱阳湖刀鲚的影响及其保护性开发对策[J]. 渔业致富指南, 2017(7): 18-21. WANG Q P, GAN J Y. Effects of human activities on Coilia nasus in Poyang Lake and protective development strategies[J]. Fishery Guide to be Rich, 2017(7): 18-21. |
| [10] |
ERGUDEN D, TURAN C. Examination of genetic and morphologic structure of seabass (Dicentrarchus labrax L., 1758) populations in Turkish coastal waters[J]. Turkish Journal of Veterinary & Animal Sciences, 2005, 29(3): 727-733. |
| [11] |
程起群, 李思发. 刀鲚和湖鲚种群的形态判别[J]. 海洋科学, 2004, 28(11): 39-44. CHENG Q Q, LI S F. Morphological discrimination between two populations of Coilia ectenes[J]. Marine Sciences, 2004, 28(11): 39-44. DOI:10.3969/j.issn.1000-3096.2004.11.009 |
| [12] |
向文殿, 谢佳燕, 林佳. 不同湖泊湖鲚种群形态差异的研究[J]. 湖北农业科学, 2011, 50(21): 4445-4447. XIANG W D, XIE J Y, LIN J. Morphological variations of Coilia ectenes in different lakes[J]. Hubei Agricultural Sciences, 2011, 50(21): 4445-4447. DOI:10.3969/j.issn.0439-8114.2011.21.040 |
| [13] |
倪勇, 伍汉霖. 江苏鱼类志[M]. 北京: 中国农业出版社, 2006. NI Y, WU H L. Fishes of Jiangsu Province[M]. Beijing: China Agriculture Press, 2006. |
| [14] |
TANG W Q, HU X L, YANG J Q. Species validities of Coilia brachygnathus and C. nasus taihuensis based on sequence variations of complete mtDNA control region[J]. Biodiversity Science, 2007, 15(3): 224-231. DOI:10.1360/biodiv.060263 |
| [15] |
王倩, 程方圆, 李晨虹. 鲚属单核苷酸多态性位点(SNPs)标记开发及在物种界定中的应用初探[J]. 上海海洋大学学报, 2017, 26(1): 8-16. WANG Q, CHENG F Y, LI C H. Developing SNP markers for Coilia and its application in species delimitation[J]. Journal of Shanghai Ocean University, 2017, 26(1): 8-16. |
| [16] |
YANG Q L, HAN Z Q, SUN D R, et al. Genetics and phylogeny of genus Coilia in China based on AFLP markers[J]. Chinese Journal of Oceanology and Limnology, 2010, 28(4): 795-801. DOI:10.1007/s00343-010-9093-3 |
| [17] |
杨金权, 胡雪莲, 唐文乔. 长江及其南部邻近水域刀鲚的种群遗传结构及种群历史[J]. 上海水产大学学报, 2008, 17(5): 513-519. YANG J Q, HU X L, TANG W Q. Genetic structure and population history of Coilia nasus in Yangtze River and its south adjacent water[J]. Journal of Shanghai Fisheries University, 2008, 17(5): 513-519. |
| [18] |
张燕萍, 贺刚, 王生, 等. 基于线粒体DNA D-loop序列分析刀鲚与短颌鲚的遗传多样性[J]. 江苏农业科学, 2017, 45(20): 59-64. ZHANG Y P, HE G, WANG S, et al. Genetic diversity analysis of Coilia nasus and Coilia brachynathus based on mitochondrial DNA D-loop sequence[J]. Jiangsu Agricultural Sciences, 2017, 45(20): 59-64. |
| [19] |
姜涛, 刘洪波, 李孟孟, 等. 溯河洄游长江刀鲚(Coilia nasus)摄食虾类的调查[J]. 湖泊科学, 2018, 30(2): 458-463. JIANG T, LIU H B, LI M M, et al. Investigation on shrimp feeding of Coilia nasus during its anadromous migration along the Yangtze River[J]. Journal of Lake Sciences, 2018, 30(2): 458-463. |
| [20] |
李孟孟, 姜涛, KHUMBANYIWAD D, 等. 基于耳石微化学的长江安徽和县江段刀鲚生境履历重建[J]. 水生生物学报, 2017, 41(5): 1054-1061. LI M M, JIANG T, KHUMBANYIWA D D, et al. Reconstructing habitat history of Coilia nasus from the Hexian section of the Yangtze river in Anhui province by otolith microchemistry[J]. Acta Hydrobiologica Sinica, 2017, 41(5): 1054-1061. |
| [21] |
HATCHER P E, WILKINSON M J, ALBANI M C, et al. Conserving marginal populations of the food plant (Impatiens noli-tangere) of an endangered moth (Eustroma reticulatum) in a changing climate[J]. Biological Conservation, 2004, 116(3): 305-317. DOI:10.1016/S0006-3207(03)00200-3 |
| [22] |
马春艳, 刘敏, 马凌波, 等. 长江口刀鲚遗传多样性的随机扩增多态DNA(RAPD)分析[J]. 海洋水产研究, 2004, 25(5): 19-24. MA C Y, LIU M, MA L B, et al. Genetic diversity in Coilia ectenes by RAPD analysis[J]. Marine Fisheries Research, 2004, 25(5): 19-24. |
| [23] |
王美垚, 管建洪. 刀鲚遗传多样性研究进展[J]. 安徽农业科学, 2015, 43(10): 149-151. WANG M Y, GUAN H J. Research progress on genetic diversity of Coilia nasus[J]. Journal of Anhui Agricultural Sciences, 2015, 43(10): 149-151. DOI:10.3969/j.issn.0517-6611.2015.10.054 |
| [24] |
LI C H, HOFREITER M, STRAUBE N, et al. Capturing protein-coding genes across highly divergent species[J]. BioTechniques, 2013, 54(6): 321-326. |
| [25] |
程起群, 温俊娥, 王云龙, 等. 刀鲚与湖鲚线粒体细胞色素b基因片段多态性及遗传关系[J]. 湖泊科学, 2006, 18(4): 425-430. CHENG Q Q, WEN J E, WANG Y L, et al. Genetic diversity and genetic differentiation between Coilia ectenes and Coilia ectenes taihuensis inferred from cytochrome b gene segment sequence of mitochondrial DNA[J]. Journal of Lake Sciences, 2006, 18(4): 425-430. DOI:10.3321/j.issn:1003-5427.2006.04.016 |
| [26] |
ZHOU X D, YANG J Q, TANG W Q. Species validities analyses of Chinese Coili a fishes based on mtDNA COI barcoding[J]. Acta Zootaxonomica Sinica, 2010, 35(4): 819-826. |
| [27] |
JORDAN D S, SEALE A. List of fishes collected in 1882-83 by Pierre Louis Jouy at Shanghai and Hong Kong, China[J]. Proceedings of the United States National Museum, 1905, 29(1433): 517-29. DOI:10.5479/si.00963801.29-1433.517 |
| [28] |
葛家春, 曹廷, 陈婵娟, 等. 利用扩增片断长度多态性技术分析长江刀鲚的遗传多样性[J]. 南京大学学报(自然科学版), 2008, 44(3): 332-338. GE J G, CAO Y, CHEN C J, et al. Analysis of genetic diversity based on amplified fragment length polymorphism fingerprint of Coilia nasus from Yangtze River[J]. Journal of Nanjing University (Natural Sciences), 2008, 44(3): 332-338. DOI:10.3321/j.issn:0469-5097.2008.03.013 |
| [29] |
李红东.不同年间长江刀鲚形态差异及其遗传多样性研究[D].扬州: 扬州大学, 2011: 1-43. LI H D. Morphological variations and genetic diversity of Coilia ectenes in different years[D]. Yangzhou: Yangzhou University, 2011: 1-43. |
| [30] |
CHENG Q Q, ZHANG Q Y, MA C Y, et al. Genetic structure and differentiation of four lake populations of Coilia ectenes (Clupeiformes:Engraulidae) based on mtDNA control region sequences[J]. Biochemical Systematics and Ecology, 2011, 39(4/5): 544-552. |
| [31] |
姜涛, 周昕期, 刘洪波, 等. 鄱阳湖刀鲚耳石的两种微化学特征[J]. 水产学报, 2013, 37(2): 239-244. JIANG T, ZHOU X Q, LIU H B, et al. Two microchemistry patterns in otoliths of Coilia nasus from Poyang Lake, China[J]. Journal of Fisheries of China, 2013, 37(2): 239-244. |
| [32] |
田思泉, 田芝清, 高春霞, 等. 长江口刀鲚汛期特征及其资源状况的年际变化分析[J]. 上海海洋大学学报, 2014, 23(2): 245-250. TIAN S Q, TIAN Z Q, GAO C X, et al. Analyzing of annual changes for the stock status of Coilia nasus in fishing season in Yangtze River estuary[J]. Journal of Shanghai Ocean University, 2014, 23(2): 245-250. |
2. Shanghai Collaborative Innovation Center for Aquatic Animal Genetics and Breeding, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China
 2019,
Vol. 28
2019,
Vol. 28


