2. 中国水产科学研究院黄海水产研究所 青岛海洋科学与技术国家实验室海洋渔业科学与食物产出过程功能实验室, 山东 青岛 266071
凡纳滨对虾(Litopenaeus vannamei)自1987年引进中国,已成为我国重要的养殖品种。苗种质量的优劣,直接影响对虾养殖的生长速度与成活率,关系到整个对虾养殖过程的成功与否[1]。在对虾苗种繁育过程中,常发生鳗弧菌(Vibrio anguillarum)、溶藻弧菌(Vibrio alginolyticus)和副溶血弧菌(Vibrio parahaemolyticus)等引起的弧菌病,以及毛霉亮发菌(Leucothrix mucor)等引发的丝状细菌病[2]。鉴此,抗生素和化学试剂等常用于对虾育苗过程中的疾病控制。然而,这些药物容易导致对虾幼体变态时间长、糠虾幼体前成活率低、种苗大小参差不齐、抗病力差、养殖过程中生长慢等问题[3]。另外,这些药物的长期使用还能够增加细菌耐药性、破坏育苗水体正常的微生态平衡[4]。益生菌已经广泛用于对虾的生态养殖,通过调节和改善对虾养殖生态环境,控制和减少高密度对虾养殖过程中病害的发生[5]。目前,益生菌尤其是芽孢杆菌(Bacillus)在对虾育苗过程的应用研究也取得了一定进展。在凡纳滨对虾苗种培育水体中添加枯草芽孢杆菌(B. subtilis),幼体发育速度明显加快,耐胁迫能力也显著增强[6]。NIMRAT等在凡纳滨对虾早期发育时添加复合芽孢杆菌,研究结果表明,复合芽孢杆菌能够促进仔虾生长,并显著降低育苗水体中氨氮和亚硝氮的含量[7]。另外,芽孢杆菌和光合细菌(photosynthetic bacteria)、芽孢杆菌和溶藻弧菌配伍应用于凡纳滨对虾育苗的研究表明,复合益生菌的添加降低了水体中H2S、NH4+-N和NO2--N等对幼体有毒性的物质的含量[1],提高了幼体的成活率[8]。因此,鉴于芽孢杆菌与其他益生菌的复合制剂在改善对虾育苗水环境,促进幼体发育,提高幼体的成活率等方面的价值,筛选具有不同生态学功能的非芽孢杆菌的益生菌,并开展芽孢杆菌与之配伍的复合益生菌在对虾苗种培育中研究,对对虾苗种健康培育模式的建立具有重要意义。
本研究以无节Ⅴ期的凡纳滨对虾幼体为研究对象,通过在水体中添加配伍的、具有不同生态学功能的益生菌组合,分析复合益生菌幼体体内微生物组的影响,探究复合益生菌在凡纳滨对虾零换水苗种培育中的应用潜力,藉此为复合益生菌在凡纳滨对虾零换水苗种中的的应用提供理论依据。
1 材料与方法 1.1 益生菌菌株及培养条件育苗实验采用的4株益生菌分别为假交替单胞菌(Pseudoalteromonas sp.)CDM8、短小芽孢杆菌(B.pumilus)LV012和芽孢杆菌(Bacillus sp.)BC109以及肠球菌(Enterococcus sp.)BC702,均由本实验室筛选、鉴定和保存。其中,CDM8具有降低对虾肠道中弧菌含量的功能[9],LV012能够提高对虾抵抗WSSV的感染[10],BC109具有硝化作用,BC702能够产酸。细菌接种于2216E固体培养基进行活化,于28 ℃恒温培养过夜。挑取活化后的细菌单菌落接种于2216E液体培养基28 ℃振荡(150 r/min)培养过夜后制备种子液,然后,按照10%接种量接种新鲜2216E液体培养基进行扩大培养。
1.2 拮抗实验采用纸片法测定4株益生菌之间的拮抗效果。将过夜培养的细菌(1×106 CFU)涂布2216E固体培养基,然后在平板上均匀放置4个干燥的无菌滤纸片(直径6 mm)。其中,3个滤纸片上滴加7.5μl待测菌(1×106 CFU),另外1个滤纸片上滴加7.5 μL无菌2216E液体培养基。每个细菌设置3个平行。将2216E固体平板于28 ℃恒温培养24 h。
1.3 复合益生菌制备及使用将4株益生菌的过夜培养液离心收集菌体(5 000 g,10 min),用无菌海水重悬,再经5 000 g离心10 min,弃去上清,重复洗涤菌体两次。最后,使用无菌海水重悬,使用分光光度计(上海精仪,上海)在580 nm下调整菌悬液浓度(3×106 CFU/mL)。
4株益生菌按照潜在的生态学功能配伍形成2组,即PBN和PBE。PBN组由CDM8、LV012和BC109配伍组成,PBE组由CDM8、LV012和BC702配伍组成。PBN和PBE的使用周期为3天,均采用向水体添加活菌的方式,使各益生菌成分在水体的终浓度达到106 CFU/mL。另外,PBN或PBE中的CDM8早于其他益生菌成分24 h添加。
1.4 育苗条件零交换水育苗实验于2017年6月至7月在河北鑫海生物技术有限公司的养殖车间进行。将凡纳滨对虾无节Ⅴ期幼体(N5)随机分到18个装有75 L海水的PVC桶,初始密度约为20万尾/m3(即:200尾/L)。将桶随机分为3组,编号分别为PBE、PBN和K,每组包含6个桶,其中3个桶用于微生物分析,另3个桶用于对虾幼体变态率和存活率的统计。PBN组和PBE组的育苗过程分别使用复合益生菌PBN和PBE,而K组作为对照组,育苗过程不使用益生菌。育苗期间水体持续通气,水温维持30~33 ℃。当幼体发育到蚤状幼体Ⅰ期时,开始投喂福瑞派微胶囊育苗饲料(英伟,比利时)和八卦虾片(博尚,中国台湾),蚤状Ⅰ期至糠虾Ⅱ期加投螺旋藻,每2小时投喂1次(0.03 g/桶次),每天投喂12次。当幼体发育到仔虾第5天(PL5)时,育苗实验结束。整个育苗过程全程不换水。
1.5 幼体变态率和存活率的检测在蚤状Ⅲ期(ZⅢ)、糠虾Ⅲ期(MⅢ)和仔虾第五天(PL5)时进行幼体抽样,检测幼体变态率;在育苗结束(PL5)时检测仔虾的存活率。
1.6 育苗水体和幼体体内可培养细菌总数和假定弧菌总数的检测在ZⅢ、MⅢ和PL5期进行育苗水和对虾幼体的取样,每个处理每次取水1 mL和幼体10尾。用无菌海水冲洗幼体体表 3次,将幼体放入无菌1.5 mL离心管中,添加适量无菌海水进行研磨,补加无菌海水至1 mL。将育苗水样和幼体匀浆液进行10倍梯度稀释,选取合适稀释度的稀释液涂布100 μL至2216E和TCBS固体培养基。平板于28 ℃恒温培养24 h后统计菌落数,将2216E固体培养基上的菌落数作为可培养细菌总数(total culturable bacterial count,TBC),将TCBS固体培养基上的菌落数作为假定弧菌总数(total presumptive Vibrio bacteria,TPVB)。
1.7 不同发育阶段幼体体内免疫相关基因的检测在ZⅢ、MⅢ和PL5期采集对虾幼体(~0.1 g)后立即用加入RNA储存液(天根,北京),于-20 ℃暂存,转运时储存于干冰中。
按照Trizol(Invitrogen,美国)说明书提取对虾幼体样品的总RNA。用Nanodrop 2000 c(ThermoScientific,美国)和1%琼脂糖凝胶电泳分别检测RNA样品的含量和质量。利用PrimeScriptTM RT reagent Kit With gDNAEraser(Perfect Real Time)反转录试剂盒(TaKaRa,大连)将总RNA反转录为cDNA。将总RNA和cDNA于-80 ℃保存。
采用实时荧光定量PCR(qPCR)检测不同时期对虾幼体样本中热休克蛋白(heat shock protein,Hsp 70)、β-1, 3-葡聚糖结合蛋白-脂蛋白(βGBP)和Crustin编码基因的表达情况。将凡纳滨对虾β-actin编码基因作为内参基因。4种基因的检测引物信息见表 1,引物由生工生物工程(上海)股份有限公司合成。
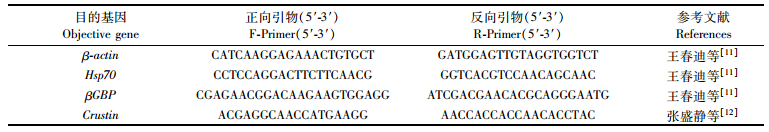
|
表 1 实时荧光定量PCR所用的引物信息 Tab.1 Information of primers used in real-time PCR |
qPCR反应体系(25 μL))包括SYBR Premix Ex Taq TM(2×)(Roche,瑞士)12.5 μL,cDNA模板1 μL,正、反向引物各0.5 μL(10 μmol/L)),DEPC处理水10.5 μL。PCR扩增条件:94 ℃,5 min;94 ℃ 30 s;58 ℃ 30;,72 ℃ 30 s;40个循环;扩增完成后进行熔解曲线分析。每个样品设置3个重复。采用2-ΔΔCt法[13]对实验结果进行相对定量分析。
1.8 仔虾体内细菌群落的高通量测序和数据分析在育苗结束时,各实验组取PL5仔虾用于体内微生物组的高通量测序。每个实验组设置3个平行,每个平行使用仔虾约0.5 g。仔虾经无菌海水冲洗体表后,放入无菌的1.5 mL离心管,用液氮速冻后于-80 ℃储存。将采集的9份仔虾样品用CTAB法[14]提取总DNA,作为模板DNA。采用细菌16S rRNA基因V4区的通用引物515F和806R扩增各样品的模板DNA,PCR反应体系和扩增条件按Peiffer等的方法进行[15]。将PCR产物的目的片段切胶回收和定量检测,合格后构建扩增子文库。利用Illumina MiSeq测序平台对文库进行双向测序,由诺禾致源生物信息科技有限公司完成。参考朱鹏飞[16]的方法对测序原始数据进行处理。由UPARSE程序将高质量的序列在97%相似性水平上聚类成OTUs (operational taxonomic units),并通过SILVA数据库确定各OTU的分类地位。每个样品随机挑选43839条序列(所有样品中的最低序列数)进行分析。在Qiime中计算Chao1和Shannon指数,用t-test检验仔虾体内细菌群落多样性和丰富度之间的差异,P < 0.05时表示差异显著。
1.9 统计分析运用SPSS 16.0软件进行数据单因素方差分析(ANOVA),以Duncan’s多重比较进行不同处理间的显著性分析,P < 0.05作为检验标准。
2 结果 2.1 4株益生菌间的拮抗4株益生菌之间拮抗实验的结果表明:CDM8能够抑制LV012、BC109和BC702的生长(图 1A-C),而后三者之间不存在拮抗效果(图 1D-G)
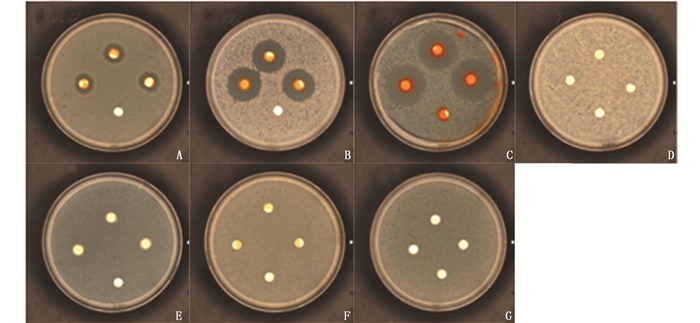
|
A. CDM8与LV012拮抗实验(其中,前者编号为滤纸片上添加的待测菌株,后者编号为固体培养基涂布的菌株;B-G同此);B. CDM8与BC109拮抗实验;C. CDM8与BC702拮抗实验;D. LV012与BC109拮抗实验;E. LV012与BC702拮抗实验;F. BC109与LV012拮抗实验;G. BC702与LV012拮抗实验 A. Antagonism among CDM8 and LV012.The former was the bacterial strain added to the filter paper, and the latter was the bacterial strain plated on the agar medium. It was the same treatment in figures B-G; B. Antagonism among CDM8 and BC109; C. Antagonism among CDM8 and BC702; D. Antagonism among LV012 and BC109; E. Antagonism among LV012 and BC702; F. Antagonism among BC109 and LV012; G. Antagonism among BC702 and LV012 图 1 4株益生菌之间的拮抗作用 Fig. 1 Antagonism among four probiotic bacteria |
当幼体发育到ZⅢ时,K组、PBE组和PBN组变态率分别为69.19%±3.15%、85.33%±7.81%和83.41%±3.15%,PBE组和PBN组中对虾幼体的变态率显著高于对照组(K组)(P < 0.05),而PBE组和PBN组中对虾幼体的变态率差异不显著(P>0.05)。当幼体发育到MⅢ和PL5时,3个实验组中对虾幼体的变态率无显著性差异(P>0.05)(图 2)。
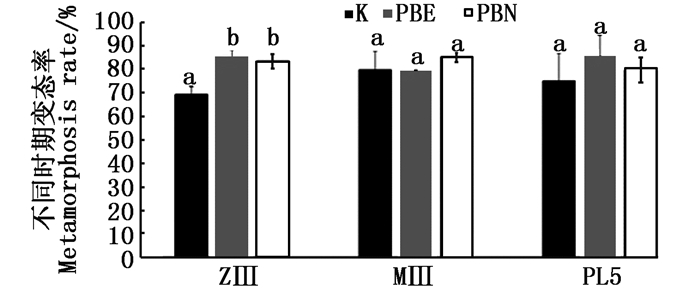
|
柱子上方不同的字母代表差异显著(P < 0.05) Different letters above bars indicate significant differences (P < 0.05) 图 2 不同发育时期对虾幼体的变态率 Fig. 2 Metamorphosis rates of larvae at different larval phases |
育苗结束时,仔虾在K组、PBE组和PBN组中的存活率分别为40.89%±3.80%、55.70%±1.43%、56.30%±2.57%,PBE组和PBN组的存活率显著高于K组(P < 0.05) (图 3)。相较对照组而言,仔虾在添加复合益生菌的实验组中的存活率增加了约15%。

|
柱子上方不同的字母代表差异显著(P < 0.05) Different letters above bars indicate significant differences (P < 0.05) 图 3 凡纳滨对虾仔虾的存活率 Fig. 3 The survival rate of postlarvae |
在MⅢ和PL5,PBE和PBN组中幼体体内TBC显著高于对照组(P < 0.05)(图 4A),而TPVB显著低于对照组(P < 0.05)(图 4B)。在ZⅢ、MⅢ和PL5,PBE和PBN组的对虾幼体体内TPVB/TBC的比例也显著低于对照组(P < 0.05)(图 4C)。
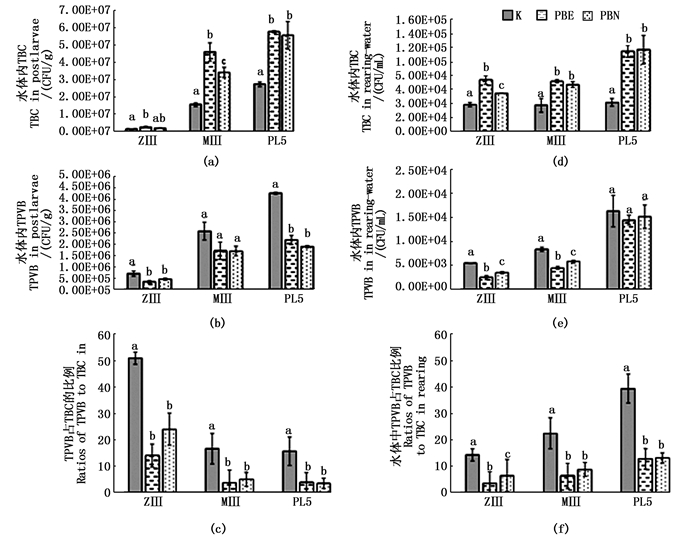
|
A-C分别为不同时期对虾幼体体内TBC、TPVB和TPVB/TBC的比例;D-F分别为不同时期水体中TBC、TPVB和TPVB/TBC的比例 A-C, TBC, TPVB and ratios of TPVB to TBC in larvae, respectively; D-F, TBC, TPVB and ratios of TPVB to TBC in rearing-water, respectively 图 4 不同时期对虾幼体体内和水体中TBC、TPVB和TPVB/TBC的比例 Fig. 4 At different larval phases, the TBC, TPVB and ratios of TPVB to TBC in larvae and rearing-water |
在ZⅢ、MⅢ或PL5期,育苗水体中TBC和TPVB的变化情况与对虾幼体体内的相似。在ZⅢ、MⅢ和PL5期,PBE和PBN组育苗水体的TBC相比对照组显著升高(P < 0.05)(图 4D)。在ZⅢ和MⅢ,PBE、PBN组育苗水体中TPVB相比对照组显著降低(P < 0.05),但在PL5期,PBE组、PBN组和对照组间无显著性差异(图 4E)。另外,在对虾幼体的不同时期,PBE和PBN组水体中TPVB/TBC的比例显著低于比对照组(P < 0.05),但是,PBE组和PBN组的差异较小(图 4F)。
2.4 免疫基因的相对表达量在ZⅢ期,Hsp70、βGBP和Crustin的编码基因在PBE组的表达量显著低于对照组(P < 0.05);Hsp70基因在PBN组和对照组的表达量差异显著(P < 0.05),而βGBP和Crustin基因在PBN组和对照组的表达量差异不显著(P>0.05)。在MⅢ期,3个免疫相关基因在3个实验组中的表达量无显著性差异(P>0.05)。在PL5期,Crustin基因在PBE组的表达量最高,并且显著高于在PBN组和对照组的表达量(P < 0.05);Hsp70和βGBP基因在PBE、PBN和对照组对虾幼体体内的表达量无显著性差异(P>0.05, 图 5)。

|
柱子上方不同的字母代表差异显著(P < 0.05) Different letters above bars indicate significant differences (P < 0.05) 图 5 对虾幼体不同时期Hsp70(a)、βGBP(b)、Crustin(c)基因的相对表达量 Fig. 5 Relative expression levels of Hsp70(a), βGBP(b), Crustin(c)at different larval phases |
通过高通量测序分析了复合益生菌对仔虾PL5体内微生物群落结构和组成的影响, 结果表明:PBN组中仔虾体内细菌群落的多样性明显低于PBE组和对照组,但差异未达到显著水平(表 2);在细菌群落的组成上,PBE组和对照组中仔虾体内细菌群落的优势物种比PBN组的更加多样(图 6)。

|
表 2 仔虾体内细菌群落的α-多样性 Tab.2 Alpha diversity of bacterial communities in postlarvae |

|
(a)相对丰度最高的10个细菌门;(b)相对丰度最高的10个细菌科 (a)ten most abundant bacterial phyla; (b) ten most abundant bacterial families 图 6 仔虾体内微生物群的组成 Fig. 6 Compositions of microbial communities in postlarvae |
在门水平上,变形菌门(Proteobacteria)是仔虾体内微生物群落中最具优势的菌群,它在PBE、PBN和对照组中的相对丰度分别是62.8%±11.6%、91.1%±6.5%和81.9%±8.1%。另外,拟杆菌门(Bacteroidetes)和厚壁菌门(Firmicutes)是PBE组和对照组中的优势菌群(相对丰度≥5%)。其中,拟杆菌门在PBE组和对照组中的相对丰度分别是8.53%±6.2%和5.1%±4.2%, 厚壁菌门在PBE组中的相对丰度是9.2%±9.2%(图 6a)。
在科水平上,红杆菌科(Rhodobacteraceae)在仔虾体内微生物群落的相对丰度最高,在PBE、PBN和对照组中的相对丰度分别是40.69%±14.6%、86.0%±9.8%和55.3%±5.3%。此外,假单胞菌科(Pseudomonadaceae)(5.5%±5.2%)也是PBE组的优势菌群, 盐单胞菌科(Halomonadaceae)(7.6%±12.2%)和假单胞菌科(6.73%±6.3%)是对照组中的优势菌群(图 6b)。
3 讨论本研究的育苗实验采用零换水育苗模式。零换水育苗可以降低换水对对虾幼体的伤害,但是,也会造成水体化学耗氧量(COD)、氨氮和亚硝酸氮逐渐升高以及硝酸盐的积累。COD升高易造成水体中溶解氧急剧下降,并促进弧菌大量繁殖;高浓度的氨氮和亚硝酸氮对对虾有毒害作用,使对虾处于亚健康状态,易导致对虾发病[17];虽然硝酸盐对对虾没有毒性,但过高的硝酸盐能够抑制对虾生长[18]。在本研究的零换水育苗水体中施用根据不同生态学功能配伍的复合益生菌,以维持育苗水体微生态的稳定。本研究的结果表明,复合益生菌能够显著提高ZⅢ幼体的变态率及PL5的成活率,这应该与复合益生菌对零换水育苗水环境的调控和改善作用密不可分。另外,潘康成等[19]和于鹏等[20]的研究结果表明,在水体中添加芽孢杆菌的微生态制剂或复合益生菌制剂能够显著提高对虾幼体的成活率,缩短幼体变态时间,促进幼体生长。
对虾育苗生产中,残饵及对虾幼体的分泌物、排泄物和残骸等的积累易导致水体中条件致病菌尤其是弧菌大量滋生,造成蚤状幼体后的幼体大量死亡[21]。在本研究中,配伍的复合益生菌显著提高了育苗水体和对虾幼体中TBC的含量,并降低了TPVB相对含量。因此,复合益生菌对幼体变态率和成活率的提高与零换水育苗水体和幼体体内相对较低的弧菌含量有着密切的联系。ZOKAEIFAR等在水体中应用枯草芽孢杆菌(108 CFU/mL)能有效降低哈维氏弧菌引起的对虾死亡率[22]。胡毅等[23]在饲料中添加芽孢杆菌等益生菌饲喂凡纳滨对虾显著降低了肠道和粪便中的弧菌含量。在凡纳滨对虾育苗和养殖生产中,应用益生菌可有效降低或预防弧菌病的发生。零换水概念应用于对虾苗种繁育可最大限度的利用养殖水体,为未被利用的饲料中营养物质的循环再利用提供空间和时间,能够减少养殖池与外环境的水交换,降低流行性病原的传播风险,减少养殖废水对近岸海洋环境的污染[24]。
凡纳滨对虾作为一种无脊椎动物,通过非特异性免疫来抵御病原微生物的入侵。热休克蛋白[25]、β-1, 3-葡聚糖结合蛋白(βGBP)[26]和Crustin[27]是凡纳滨对虾非特异性免疫中重要的免疫相关蛋白。HSP是所有原核细胞和真核细胞遭受高温或其它应激产生的一组非常保守的蛋白分子家族,HSP70家族是最保守和最主要的一类[25]。βGBP是甲壳动物体内一种重要的免疫应答蛋白,它通过结合真菌细胞壁的葡聚糖,进一步引发免疫级联反应[26]。Crustin属于AMPS家族,具有抗菌作用,还可以通过调节信号转导及细胞因子的产生和释放参与免疫调节,在无脊椎动物的免疫过程中发挥着重要作用[27]。芽孢杆菌产生的多种酶类和多糖在一定程度上可促进对虾的生长和提高非特异性免疫功能[28]。在本研究添加复合益生菌的实验组中,ZⅢ期幼体的Hsp70和βGBP基因的表达量显著低于对照组的,这可能与育苗水体和幼体体内相对较低的弧菌含量密切相关;在育苗后期,水体和幼体体内弧菌的数量相比育苗前期明显增加,两种免疫基因的表达量也相应上调。因此,在零换水育苗过程中,凡纳滨对虾幼体的Hsp70和βGBP基因的表达情况与体内和水体中弧菌的相对含量密切相关。
变形菌门和厚壁菌门是健康幼虾肠道微生物群的优势菌群[29],在本研究中,它们同样是PL5期仔虾体内的核心微生物群。郁维娜等[30]的研究结果表明,γ-变形菌纲和厚壁菌门等细菌在患病对虾肠道中含量相对较高。在门水平上通过比较微生物群的相对丰度的高低来评估对虾健康状况是不准确的,因此,XIONG等[31]发现在科水平上通过比较微生物群的相对丰度进而评估对虾的健康状况具有较高的准确性。在本研究中,红杆菌科是仔虾体内最主要的核心菌群。凡纳滨对虾养成期间疾病暴发与浮游细菌群落结构的骤变密切相关,13个细菌科(包括假单胞菌科和弧菌科等)可作为预测病害出现与否的指示菌群[31]。本研究的各实验组在整个育苗实验周期内未发生弧菌病。弧菌科在本研究各实验组的PL5仔虾体内均为非优势菌群,表明仔虾仍处于健康状态。值得注意的是,虽然在育苗实验中持续添加复合益生菌,但是,益生菌并未在仔虾微生物组中成为核心微生物群。这与已有的一些应用芽孢杆菌进行对虾养殖的研究[32-33]结果相似。
综上所述,将不同生态学功能的益生菌配伍成的益生菌组合应用于凡纳滨对虾的零换水育苗具有现实可行性。复合益生菌能够提高育苗水体和凡纳滨对虾幼体体内可培养细菌的总菌数,降低弧菌的相对含量,从而,提高幼体的变态率和成活率;但是,复合益生菌并未造成仔虾体内核心微生物群多样性的显著变化。这些研究结果为凡纳滨对虾的零换水育苗提供了技术支持。
| [1] |
叶乐, 杨莺莺, 吴开畅, 等. 益生菌在凡纳滨对虾育苗中的应用[J]. 南方水产, 2006, 2(4): 13-18. YE L, YANG Y Y, WU K C, et al. Effect of probiotics on breeding of Litopeuaeus vannamei[J]. South China Fisheries Science, 2006, 2(4): 13-18. DOI:10.3969/j.issn.2095-0780.2006.04.003 |
| [2] |
宫春光, 于清海, 陈福杰. 南美白对虾育苗中弧菌病和丝状细菌病的防治对策[J]. 科学养鱼, 2008(9): 50-51. GONG C G, YU Q H, CHEN F J. Prevention and treatment of vibrio and filamentous bacterial disease in Litopenaeus vannamei[J]. Scientific Fish Farming, 2008(9): 50-51. |
| [3] |
刘建勇. 复合微生物制剂在凡纳滨对虾育苗中的应用[J]. 海洋科学, 2005, 29(4): 36-40. LIU J Y. Effect of microecological compound agent on breeding of Litopenaeus vannamei Boon[J]. Marine Sciences, 2005, 29(4): 36-40. DOI:10.3969/j.issn.1000-3096.2005.04.009 |
| [4] |
姚晓东. 抗生素在水产养殖中应用存在的问题及对策[J]. 农业与技术, 2016, 36(24): 103. YAO X D. The problems and countermeasures of antibiotics in aquaculture[J]. Agriculture & Technology, 2016, 36(24): 103. |
| [5] |
CHIU C H, CHENG C H, GUA W R, et al. Dietary administration of the probiotic, Saccharomyces cerevisiae P13, enhanced the growth, innate immune responses, and disease resistance of the grouper, Epinephelus coioides[J]. Fish & Shellfish Immunology, 2010, 26(5): 691-698. |
| [6] |
LIU K F, CHIU C H, SHIU Y L, et al. Effects of the probiotic, Bacillus subtilis E20, on the survival, development, stress tolerance, and immune status of white shrimp, Litopenaeus vannamei larvae[J]. Fish & Shellfish Immunology, 2010, 28(5/6): 837-844. |
| [7] |
NIMRAT S, SUKSAWAT S, BOONTHAI T, et al. Potential Bacillus probiotics enhance bacterial numbers, water quality and growth during early development of white shrimp (Litopenaeus vannamei)[J]. Veterinary Microbiology, 2012, 159(3/4): 443-450. |
| [8] |
吴翔宇, 于明超, 单洪伟, 等. 两株益生菌在凡纳滨对虾育苗生产中的应用[J]. 科学养鱼, 2014(2): 54-55. WU X Y, YU M C, SHAN H W, et al. The application of two probiotics in the production of prawn seedlings in Litopenaeus vannamei[J]. Scientific Fish Farming, 2014(2): 54-55. |
| [9] |
王春迪.凡纳滨对虾防病益生菌的筛选和效果评价[D].上海: 上海海洋大学, 2016. WANG C D. Screening and evalution of potential probiotics against disease in Litopenaeus vannamei[D]. Shanghai: Shanghai Ocean University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10264-1016912456.htm |
| [10] |
李玉宏.养殖对虾肠道微生物区系的特征与功能[D].上海: 上海海洋大学, 2013. LI Y H. The characteristic and function research on the intestinal microflora of shrimp[D]. Shanghai: Shanghai Ocean University, 2013. http://d.wanfangdata.com.cn/Thesis/D372740 |
| [11] |
王春迪, 宋晓玲, 张晓静, 等. 养殖水体中添加蜡样芽孢杆菌PC465对凡纳滨对虾抗病力的影响[J]. 中国水产科学, 2016, 23(1): 146-155. WANG C D, SONG X L, ZHANG X J, et al. Effects of adding Bacillus cereus PC465 to rearing water on disease resistance of Litopenaeus vannamei[J]. Journal of Fishery Sciences of China, 2016, 23(1): 146-155. |
| [12] |
张盛静, 宋晓玲, 赵小金, 等. 饲料中添加益生菌对凡纳滨对虾抗感染和5种免疫基因表达的影响[J]. 水产学报, 2015, 39(6): 899-907. ZHANG S J, SONG X L, ZHAO X J, et al. Effects of adding probiotics to the feed on anti-infection and five kinds of immune gene expression of Litopenaeus vannamei[J]. Journal of Fisheries of China, 2015, 39(6): 899-907. |
| [13] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ct method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [14] |
隋昶生, 王利华. 崂山奶山羊瘤胃微生物总DNA提取方法的优化[J]. 中国农学通报, 2009, 25(5): 18-21. SUI C S, WANG L H. Extraction method optimizing of total DNA in rumen microorganism of Laoshan dairy[J]. Chinese Agricultural Science Bulletin, 2009, 25(5): 18-21. |
| [15] |
PEIFFER J A, SPOR A, KOREN O, et al. Diversity and heritability of the maize rhizosphere microbiome under field conditions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(16): 6548-6553. DOI:10.1073/pnas.1302837110 |
| [16] |
朱鹏飞.大菱鲆养殖环境细菌多样性分析及水产病原菌基因芯片检测体系的构建[D].青岛: 国家海洋局第一海洋研究所, 2015. ZHU P F. Analysis of bacterial communities from turbot (Scophthalmus maximus) aquaculture system and construction of DNA microarray detection system for aquaculture pathogenic bacteria[D]. Qingdao: The First Institute of Oceanography, 2015. http://cdmd.cnki.com.cn/Article/CDMD-85301-1015372085.htm |
| [17] |
邱德全, 周鲜娇, 邱明生. 氨氮胁迫下凡纳滨对虾抗病力和副溶血弧菌噬菌体防病效果研究[J]. 水生生物学报, 2008, 32(4): 455-461. QIU D Q, ZHOU X J, QIU M S. Study on anti-disease ability of Litopenaeus vannamei and the biological control of Vibrio parahaemolyticus bacteriophage under stresses of ammonia nitrogen[J]. Acta Hydrobiologica Sinica, 2008, 32(4): 455-461. DOI:10.3321/j.issn:1000-3207.2008.04.002 |
| [18] |
徐纪萍, 谈小兰, 钱辉仁. 凡纳滨对虾高密度零换水高产养殖技术[J]. 水产科技情报, 2012, 39(1): 26-29. XU J P, TAN X L, QIAN H R. The high-density zero-exchange and high-water aquaculture technology for shrimp[J]. Fisheries Science & Technology Information, 2012, 39(1): 26-29. DOI:10.3969/j.issn.1001-1994.2012.01.007 |
| [19] |
潘康成, 杨金龙, 王振华, 等. 枯草芽孢杆菌制剂在南美白对虾育苗上的应用[J]. 饲料研究, 2004(12): 33-34. PAN K C, YANG J L, WANG Z H, et al. Application of Bacillus subtilis in the breeding of Litopenaeus vannamei[J]. Feed Research, 2004(12): 33-34. DOI:10.3969/j.issn.1002-2813.2004.12.011 |
| [20] |
于鹏, 王腾, 于明超, 等. 三株潜在益生菌在凡纳滨对虾苗种培育过程中的应用[J]. 河北渔业, 2018(1): 12-16, 45. YU P, WANG T, YU MC, et al. Application of three potential probiotics in the cultivation of Litopenaeus vannamei larva[J]. Hebei Fisheries, 2018(1): 12-16, 45. DOI:10.3969/j.issn.1004-6755.2018.01.003 |
| [21] |
周丽, 宫庆礼. 海水鱼虾蟹贝病害防治技术[M]. 青岛: 青岛海洋大学出版社, 1998: 83-86. ZHOU L, GONG Q L. Prevention and treatment of Marine fish, shrimp, crab and shellfish[M]. Qingdao: Qingdao Ocean University Press, 1998: 83-86. |
| [22] |
ZOKAEIFAR H, BABAEI N, SAAD C R, et al. Administration of Bacillus subtilis strains in the rearing water enhances the water quality, growth performance, immune response, and resistance against Vibrio harveyi infection in juvenile white shrimp, Litopenaeus vannamei[J]. Fish & Shellfish Immunology, 2014, 36(1): 68-74. |
| [23] |
胡毅, 谭北平, 麦康森, 等. 饲料中益生菌对凡纳滨对虾生长、肠道菌群及部分免疫指标的影响[J]. 中国水产科学, 2008, 15(2): 244-251. HU Y, TAN B P, MAI K S, et al. Effects of dietary probiotic on growth, immunity and intestinal bacteria of juvenile Litopenaeus vannamei[J]. Journal of Fishery Sciences of China, 2008, 15(2): 244-251. DOI:10.3321/j.issn:1005-8737.2008.02.008 |
| [24] |
王志杰.两株益生菌在凡纳滨对虾生物絮团养殖中的应用[D].青岛: 中国海洋大学, 2014. WANG Z J. The application of two strains probiotics in Litopenaeus vannamei biological floccules breeding[D]. Qingdao: Ocean University of China, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10423-1014203904.htm |
| [25] |
ZHOU J, WANG L, XIN Y, et al. Effect of temperature on antioxidant enzyme gene expression and stress protein response in white shrimp, Litopenaeus vannamei[J]. Journal of Thermal Biology, 2010, 35(6): 284-289. DOI:10.1016/j.jtherbio.2010.06.004 |
| [26] |
李晓华.中国明对虾双功能分子β-1, 3-葡聚糖结合蛋白-脂蛋白的研究[D].南昌: 南昌大学, 2008. LI X H. Study on the β-1, 3-glucan-binding protein-Lipoprotein in shrimp Fenneropenaeus chinensis[D]. Nanchang: Nanchang University, 2008. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=Y1539920 |
| [27] |
张艳艳, 刘小林, 黄海洪, 等. 凡纳滨对虾Crustin-like基因的克隆及在副溶血弧菌感染条件下的表达分析[J]. 西北农林科技大学学报(自然科学版), 2012, 40(12): 41-48. ZHANG Y Y, LIU X L, HUANG H H, et al. Molecular cloning of Crustin-like gene in the white shrimp (Litopenaeus vannamei) and its mRNA expression with Vibrio parahaemolyticus challenge[J]. Journal of Northwest A & F University (Natural Science Edition), 2012, 40(12): 41-48. |
| [28] |
BOOPATHY R, KERN C, CORBIN A. Use of Bacillus consortium in waste digestion and pathogen control in shrimp aquaculture[J]. International Biodeterioration & Biodegradation, 2015, 102: 159-164. |
| [29] |
杨坤杰, 王欣, 熊金波, 等. 健康和患病凡纳滨对虾幼虾消化道菌群结构的比较[J]. 水产学报, 2016, 40(11): 1765-1773. YANG K J, WANG X, XIONG J B, et al. Comparison of the bacterial community structures between healthy and diseased juvenile shrimp (Litopenaeus vannamei) digestive tract[J]. Journal of Fisheries of China, 2016, 40(11): 1765-1773. |
| [30] |
郁维娜, 戴文芳, 陶震, 等. 健康与患病凡纳滨对虾肠道菌群结构及功能差异研究[J]. 水产学报, 2018, 42(3): 399-409. YU W N, DAI W F, TAO Z, et al. Characterizing the compositional and functional structures of intestinal microflora between healthy and diseased Litopenaeus vannamei[J]. Journal of Fisheries of China, 2018, 42(3): 399-409. |
| [31] |
XIONG J B, ZHU J L, ZHANG D M. The application of bacterial indicator phylotypes to predict shrimp health status[J]. Applied Microbiology and Biotechnology, 2014, 98(19): 8291-8299. DOI:10.1007/s00253-014-5941-y |
| [32] |
李卓佳, 林亮, 杨莺莺, 等. 芽孢杆菌制剂对虾池环境微生物群落的影响[J]. 农业环境科学学报, 2007, 26(3): 1183-1189. LI Z J, LIN L, YANG Y Y, et al. Influence of Bacillus on the microbial communities in shrimp ponds[J]. Journal of Agro-Environment Science, 2007, 26(3): 1183-1189. DOI:10.3321/j.issn:1672-2043.2007.03.075 |
| [33] |
林亮, 李卓佳, 郭志勋, 等. 施用芽孢杆菌对虾池底泥细菌群落的影响[J]. 生态学杂志, 2005, 24(1): 26-29. LIN L, LI Z J, GUO Z X, et al. Influence of Bacillus on the bacterial communities in the sediment of shrimp ponds[J]. Chinese Journal of Ecology, 2005, 24(1): 26-29. DOI:10.3321/j.issn:1000-4890.2005.01.006 |
2. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences; Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Sciences and Technology, Qingdao 266071, Shandong, China
 2019,
Vol. 28
2019,
Vol. 28


